结核分枝杆菌甲硫氨酰tRNA合成酶与底物及其类似物的结合比较
2021-05-06任伟宏陈明心
王 炜,任伟宏,陈明心,蒋 露,张 岱,陈 磊
氨酰tRNA合成酶(aminoacyl-tRNA synthetases, aaRS)是一类细胞内高度保守蛋白酶家族,参与到生命遗传信息翻译过程中,是确保细胞遗传信息正确翻译的酶。不同物种来源的MetRS具有结构上的多样性,适合特异性抑制剂的筛选,已被证明是理想的抗生素作用靶点[1]。在近几年的aaRS抑制剂研究中,发现的一些氨酰tRNA合成酶抑制剂,具有高选择性和强抑制能力。AN3016是一种亮氨酰tRNA合成酶抑制剂,动物实验显示,其抑制结核分枝杆菌的能力强于异烟肼[2]。
结核分枝杆菌MetRS(MycobacteriumtuberculosisMethionyl-tRNA synthetase, MtMetRS)的研究始于上个世纪,相关文献报道其氨酰化反应后就没有新的研究报道。MtMetRS研究进展缓慢的原因与其重组蛋白制备困难有关[3]。由于MetRS抑制剂存在专一性强、抗菌谱覆盖面窄的问题。为了筛选结核分枝杆菌MetRS的抑制剂,首先需要大量获得MtMetRS。我们通过构建不同重组质粒,进行重组蛋白表达筛选,最终解决了MtMetRS重组蛋白制备问题,为抑制剂的筛选提供基础条件。此外,我们使用Thermal Shift Assay(TSA)验证了重组MtMetRS与底物及其类似物的结合能力,为抑制剂的选择提供方向。
1 材料与方法
1.1材料 pET32a、pQE60、pGEX-6P-1、pTrcHisB质粒、M15大肠杆菌、BL21大肠杆菌本实验室保存。高保真DNA聚合酶、限制性核酸内切酶、T4 DNA连接酶、DNA marker购自Takara公司。质粒提取、凝胶回收试剂盒购自Axygen公司。引物及基因合成、测序在生工生物工程(上海)股份有限公司完成。青霉素、IPTG购自索莱宝公司。Milipure超滤管购自默克公司。HisPur Ni-NTA树脂、蛋白质Marker购自Thermo Scientific。ATP、Met、ADO、AMP-PNP、Sypro Orange购自Merck公司。
1.2 方 法
1.2.1MetRS序列分析 氨基酸序列决定蛋白质结构。为了分析人MetRS和结核分枝杆菌MetRS的差异,选用GenBank登录号为AAH15011.1、AAH09115.1、WP_003911321.1氨基酸序列为分析对象。氨基酸序列以FASTA格式整理为一个文件。使用MEGA软件中的MUSCLE[4]Neighbor-Joining算法对MetRS氨基酸序列进行多序列比对分析。以比对好的文件和人胞质MetRS晶体结构(PDB ID:5GL7)为材料,使用ESPript网络服务制作基于结构的多序列比对图,并指示出参与底物结合的氨基酸残基。
1.2.2重组基因的表达筛选 根据GenBank登录号CP003248.2核酸序列,由公司合成MtMetRS基因序列并插入到pET32a的NcoI、XhoI限制性核酸内切酶识别序列之间。通过PCR引物设计,在MtMetRS基因的两端加入相应的限制性核酸内切酶识别序列,通过限制性核酸内切酶酶切及T4连接酶链接,MtMetRS基因连接到pQE60、pGEX-6P-1、pTrcHisB载体上。使用菌落PCR鉴定阳性克隆后送公司测序确认MtMetRS基因开放读码框是否正确。pQE60-MtMetRS重组质粒的启动子为T5启动子,使用M15宿主菌进行诱导表达。pET32a-MtMetRS的T7启动子、pGEX-6P-1-MtMetRS的tac启动子、pTrcHisB-MtMetRS的trc启动子可以在BL21宿主菌中转录表达。pQE60-MtMetRS/M15、pET32a-MtMetRS/BL21、pGEX-6P-1-MtMetRS/BL21、pTrcHisB-MtMetRS/BL21,在18 ℃、250 r/min、1 mM IPTG条件下过夜诱导重组MtMetRS表达。诱导后4 ℃离心收集细菌,菌泥重悬到500 mmol/L NaCl、50 mmol/L Tris pH8.0裂解缓冲液中。菌液超声裂解后,15 000 g离心45 min去除未破碎的细菌及不溶性颗粒,可溶性部分用Ni-NTA树脂结合。首先用20 mL裂解缓冲液洗涤树脂,然后用15 mL含25 mmol/L咪唑的相同缓冲液洗涤。最后,用10 mL含300 mmol/L咪唑的缓冲液洗脱树脂结合的蛋白。分别取裂解液和Ni-NTA树脂洗脱液加入4×蛋白上样缓冲液,90 ℃加热10 min后用SDS-PAGE分析。
1.2.3重组MtMetRS的制备 通过表达筛选,确定使用pQE60-MtMetRS质粒制备重组MtMetRS。使用上面相同的条件诱导表达2 L菌液,经超声破碎、高速离心、Ni-NTA树脂结合与洗脱后,使用截留分子量为50 kDa的Milipure超滤管浓缩洗脱液至1.5 mL。浓缩液注入kta层析仪中,使用100 mmol/L NaCl、50 mmol/L Tris pH8.0缓冲液进行色谱分离。收集洗脱峰样品,使用Milipure超滤管浓缩。浓缩蛋白经Nanodrop调整浓度为10 mg/mL。50 μL分装后经液氮速冻后-80 ℃保存。
1.2.4MtMetRS与配体的结合分析 使用100 mmol/L NaCl、50 mmol/L Tris pH8.0缓冲液稀释Sypro Orange到10×。重组MtMetRS蛋白稀释到20 μmol/L浓度后与5 mmol/L浓度的ATP、Met、AMP-PNP、ADO、Met+ATP、Met+ADO、Met+AMP-PNP按照1∶1体积比混合,在冰上孵育15 min,然后与10× Sypro Orange按照1∶1体积比混合后冰上孵育15 min。在Bio-Rad CFX实时定量PCR仪上设置程序:温度范围20~80 ℃,从20 ℃起始,温度每升高0.5 ℃,抚育1 min,使用Cal Gold540通道检测Sypro Orange荧光信号。
2 结 果
2.1多序列比对结果 MetRS的氨基酸多序列比对结果如图1所示,红色五星指示出参与结合中间产物MetAde的氨基酸残基。MetRS虽然催化相同的化学反应,但这些氨基酸残基在人MetRS和结核分枝杆菌MetRS中并不完全一致。这个差异提示不同MetRS结合相同化合物的能力可能存在差异。而这种差异是设计筛选特异性、低毒性MetRS抑制剂的前提。
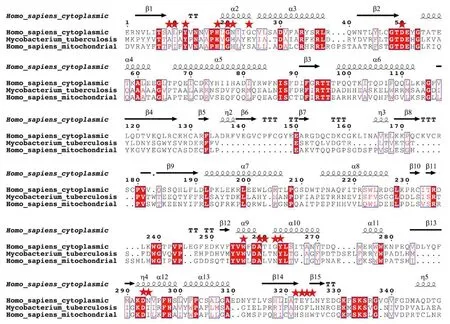
图1 MetRS的多序列比对Fig.1 Multiple sequence alignment of MetRS
2.2重组基因载体的构建 针对pQE60、pGEX-6P-1、pTrcHisB载体多克隆位点的限制性核酸内切酶识别序列设计MtMetRS基因引物。以pET32a-MtMetRS质粒为模板扩增MtMetRS基因。扩增结果如图2所示,在1 500 bp marker旁有一条带,与MtMetRS基因大小近似。PCR产物经琼脂糖凝胶纯化后,经限制性核酸内切酶消化后连接入相应载体中。重组质粒经测序验证读码框完全正确。
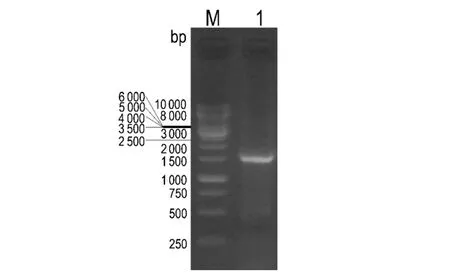
M: The product of MtMetRS gene amplification;1: GeneRuler 1 kb Marker图2 MtMetRS基因 PCR扩增结果Fig.2 Result of MtMetRS gene amplification
2.3重组载体的表达筛选 为获得可溶性重组蛋白,在重组质粒表达筛选时我们只验证细菌裂解液上清是否可以纯化到重组蛋白。重组质粒转化宿主菌后诱导表达500 mL菌液,经超声裂解后使用Ni-NTA树脂纯化。分别取细菌裂解液和Ni-NTA树脂洗脱液,使用SDS-PAGE分析。电泳结果如图3所示,第3、5泳道分别是pQE60-MtMetRS、pTrcHisB-MtMetRS质粒表达纯化的重组蛋白,与55 kDa蛋白marker位于同一水平。1、7泳道分别是pET32a-MtMetRS、pGEX-6P-1-MtMetRS质粒表达纯化的重组蛋白。2、4、6、8泳道分别为pET32a-MtMetRS/BL21、pQE60-MtMetRS/M15、pTrcHisB-MtMetRS/BL21、pGEX-6P-1-MtMetRS/BL21诱导表达后的裂解液。由于pTrcHisB-MtMetRS质粒表达的重组蛋白氨基端含有一个短的标签。为避免重组蛋白氨基端多余多肽对蛋白质活性的影响,我们最终选择pQE60-MtMetRS质粒作为重组蛋白MtMetRS的生产质粒。
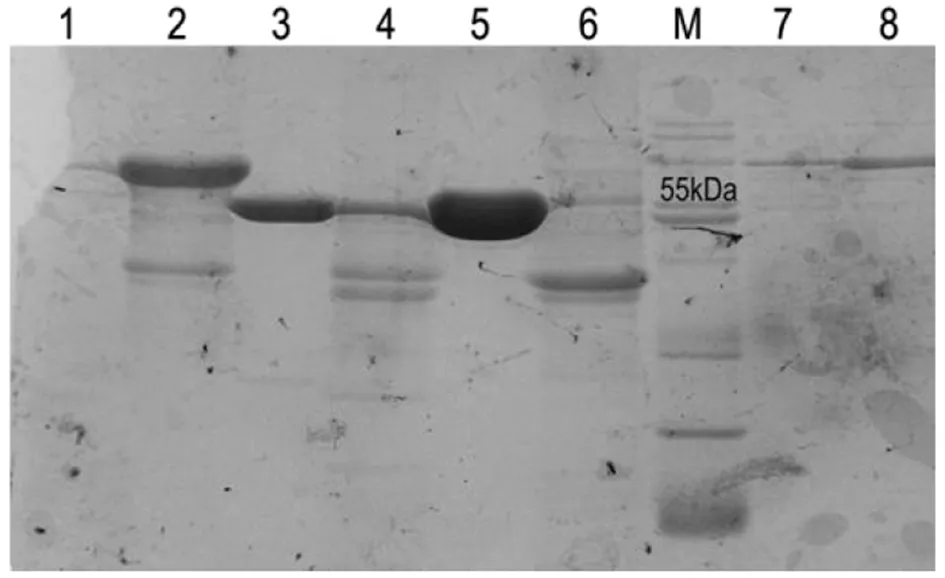
M:Protein marker;1:Recombinant MtMetRS expressed by pET32a-MtMetRS/BL21;2:Lysis solution of pET32a-MtMetRS/BL21 induced by IPTG;3:Recombinant MtMetRS expressed by pQE60-MtMetRS/M15;4:Lysis solution of pQE60-MtMetRS/M15 induced by IPTG;5: Recombinant MtMetRS expressed by pTrcHisB-MtMetRS;6: Lysis solution of pQE60-MtMetRS/M15 induced by IPTG;7: Recombinant MtMetRS expressed by pGEX-6P-1-MtMetRS/BL21;8: Lysis solution of pGEX-6P-1-MtMetRS/BL21 induced by IPTG图3 重组蛋白的表达筛选Fig.3 Expression screening of recombinant protein
2.4MtMetRS重组蛋白的制备 pQE60-MtMetRS/M15接种到2 L LB培养中,18 ℃ 0.5 mmol/L IPTG过夜诱导表达。重组MtMetRS的分子排阻层析结果如图4所示。MtMetRS的洗脱峰成对称图形,提示重组蛋白的结构均一。根据标准品的分子量及洗脱峰值计算获得的方程式为Y=7.900 35-0.186 09X。重组MtMetRS的洗脱峰体积为16.86 mL,依据上述方程计算出分子排阻层析测量的重组蛋白分子量为57.93 kDa,与MtMetRS的理论分子量59.12 kDa近似。使用Nanodrop的测定值除以重组蛋白的消光系数1.371,调整蛋白质的浓度为10 mg/mL,50 μL液氮速冻后-80 ℃保存。
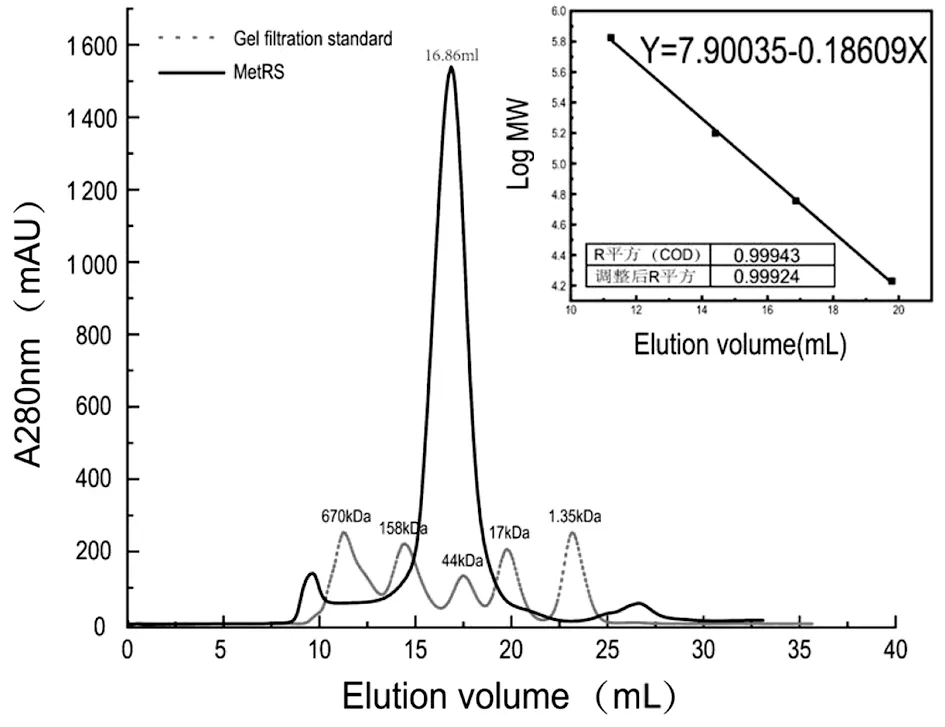
图4 重组MtMetRS及标准品的gel filtration洗脱峰Fig.4 Gel filtration elution peaks of recombinant MtMetRS and standard

图5 重组MtMetRS的TSA分析Fig.5 TSA to assess ligand:MtMetRS binding
为了进一步分析TSA的结果,我们在图6中展示了利什曼原虫及耻垢分枝杆菌MetRS与底物结合的晶体结构。从图6A中可以看到Met结合区是一个疏水口袋,其通过疏水区域的大小和疏水特性识别和结合Met[3]。Met的这种结合方式对提高MetRS的热稳定性贡献有限。而MetRS与ADO和Met结合时,不能引起催化口袋的封闭,催化口袋处于开放状态,如图6A所示。只有MetRS与Met和ATP结合后,催化形成的MetAde和PPi可以引起催化loop的构象变化,封闭催化口袋,如图6B所示。形成封闭的催化口袋可能是MtMetRS热稳定性提高的基础。
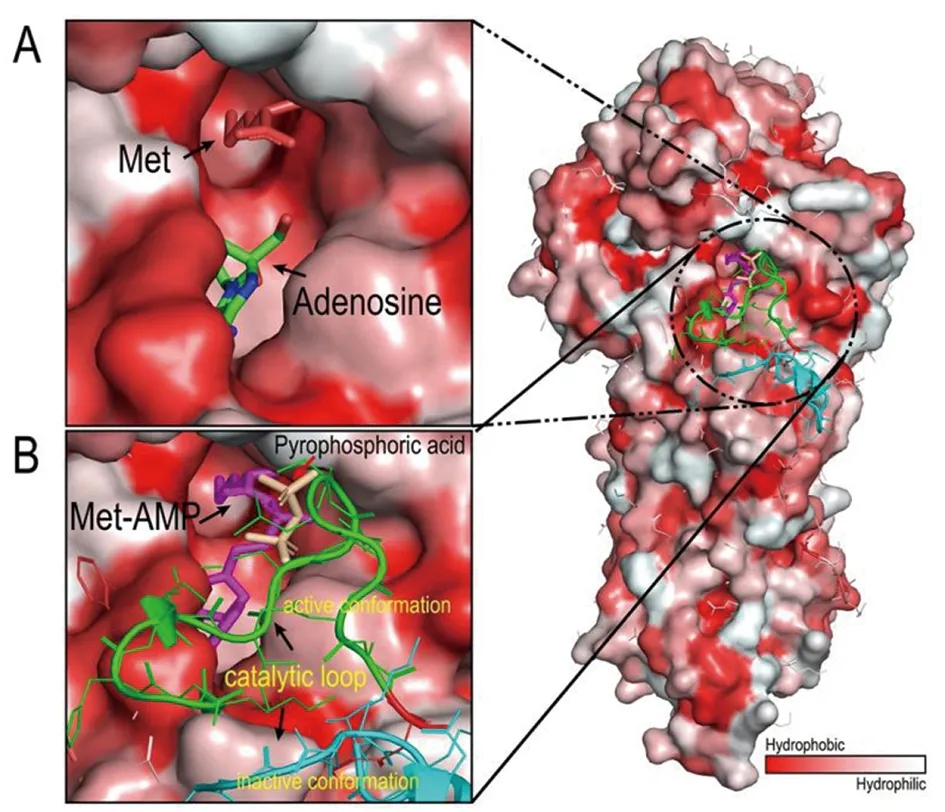
A: MetRS combined with Met and ADO(PDB:2X1L);B: MetRS combined with MetAde and PPi(PDB:3KFL)图6 MetRS与配体结合的分子结构Fig.6 Molecular structure of MetRS and ligand binding
3 讨 论
人们已经从天然来源或通过筛选合成文库方法鉴定到了许多aaRS抑制剂[5-7]。甲硫氨酰tRNA合成酶与其他aaRS最大的区别是不同物种来源的MetRS显示了结构的多样性[8]。人们早就认识到MetRS作为药物靶标的重要性,并不断在MetRS抑制剂的研究中取得进展[9-16]。上个世纪90年代人们已经意识到MtMetRS在抗结核分支杆菌抑制剂研究中的重要性,初步报道了MtMetRS的部分生化参数[17]。但由于MtMetRS重组制备方法没有突破,此后再没有MtMetRS的研究报道。Ingvarsson等对MtMetRS晶体学的研究也是由于难以大量获得重组MtMetRS而改为使用耻垢分枝杆菌MetRS为研究对像。
影响重组基因在原核细胞中表达的因素有密码子频率、蛋白质表达速度、目的蛋白的溶解性、mRNA结构等。在之前的MetRS研究报道中均使用原核表达系统获得重组蛋白,因此我们认为密码子频率和蛋白的溶解性不是阻碍获得重组MtMetRS的主要原因。pTrcHisB-MtMetRS质粒在插入基因的5′端存在一个小的顺反子结构,可以改善质粒转录的mRNA 5′端结构,有利于mRNA与核糖体的结合,但是在表达筛选结果中没有看出其明显增加重组蛋白的表达。使用弱启动子降低蛋白质表达速度可能是获得重组MtMetRS的主要原因。
MetRS分子具有底物诱导的构象变化。在未结合底物时,催化口袋呈现开放状态,而结合底物后催化口袋封闭。以往针对MetRS抑制剂的设计和筛选以开放状态为对象,选择化合物配体去结合催化口袋。我们的TSA分析结果和利什曼原虫与MetAde结合模型显示其催化口袋的封闭状态对配体结合最强。这提示结合MetRS并促使其催化口袋封闭的化合物是很好的抑制剂。以往以MetRS催化口袋的开放状态为目标设计、筛选抑制剂可能不是最优的方法。
总而言之,我们建立了重组MtMetRS的制备方法,为研究和筛选结核分枝杆菌抑制剂提供了基础。同时,TSA结果更进一步为抗结核分枝杆菌抗生素的研究指明方向。
利益冲突:无
