基于SSR标记的雪茄烟种质资源指纹图谱库的构建及遗传多样性分析
2021-04-27王琰琰刘国祥张华述骆铮珍陈志华戴培刚张兴伟杨爱国
王琰琰 王 俊 刘国祥 钟 秋 张华述 骆铮珍 陈志华 戴培刚 佟 英 李 媛 蒋 勋 张兴伟,* 杨爱国,*
基于SSR标记的雪茄烟种质资源指纹图谱库的构建及遗传多样性分析
王琰琰1,**王 俊2,**刘国祥1钟 秋2张华述2骆铮珍2陈志华3戴培刚1佟 英1李 媛1蒋 勋1张兴伟1,*杨爱国1,*
1中国农业科学院烟草研究所, 山东青岛 266101;2四川省烟草公司德阳市公司, 四川德阳 618400;3四川省烟草科学研究所, 四川成都 610000
为从分子水平研究我国雪茄烟种质资源的遗传多样性差异并建立雪茄烟品种的DNA指纹图谱数据库, 本研究利用43对多态性好的SSR引物对220份雪茄烟种质进行遗传多样性分析, 筛选出14对核心引物对雪茄烟种质进行指纹图谱的构建。结果表明, 43对SSR引物在220份雪茄烟种质材料中共扩增出243个等位基因, 平均每个标记5.65个, 变幅为2~13, 每个位点的多态性信息量(polymorphism information content, PIC)变化为0.2078~0.9087, 平均为0.6360。有效等位基因数(number of effective alleles,e)范围为1.3081~11.7876, 平均有效等位基因数为3.9077; 观测杂合度(observed heterozygosity,o)变化范围为0.0828~0.7639, 平均为0.3191; 预期杂合度(expected heterozygosity,e)的变化范围为0.2361~0.9172, 平均为0.6809; 种群平均Shannon遗传多样性指数(Shannon genetic diversity index,)为1.3756, 遗传距离在0.0233~0.9286之间, 平均遗传距离0.6816。聚类分析表明, 在遗传距离为0.74处, 可将供试雪茄烟资源分为3个类群。Structure群体遗传结构分析和主成分分析将所有的供试材料划分为2个类群。根据引物的分析和表型鉴定结果, 确定良种、辅善和满耳朵, 山东大叶和牡丹江05-1, Florida 513和CA0701为异名同种, 一个品种保留一份种质, 剩余216份不同种质。从43对SSR引物中筛选出14对可区分所有供试材料的SSR引物作为核心引物构建了216个雪茄烟品种的指纹图谱。我国雪茄烟种质资源具有较高水平的遗传多样性, 本研究构建的雪茄烟种质资源SSR指纹图谱库及遗传分析的结果在分子水平上为筛选、鉴定优质雪茄烟种质资源、挖掘重要基因以及拓宽雪茄烟遗传育种基础等工作提供科学依据。
雪茄烟; 种质资源; SSR标记; 遗传多样性; 指纹图谱
烟草(L.)为双子叶植物纲(Dicotyledoneae)管花目(Tubiflorae)茄科(Solanaceae)烟属()一年生或有限多年生草本植物[1], 种植范围十分广泛, 是我国重要的经济作物之一, 也是最早应用于分子生物学和基因工程研究的模式植物之一[2]。我国烟草种质资源十分丰富。目前已有76个烟属植物种被发现, 其中可直接利用的栽培种有普通烟草(L.)和黄花烟草(L.)[3-5]。雪茄烟是普通烟草的一种, 我国雪茄烟种质资源数量比较匮乏, 其仅占烟草种质资源的3%[6], 且主要为国外引进种质。由于我国雪茄烟种质资源引种渠道多元, 保存较为分散, 种质保存机构间相互引种且独立命名, 因此存在种质重复引进、品种同种异名、同名异种, 以及种质间亲缘关系不清等问题, 给烟草种质资源编目保存、育种亲本选择及优良品种推广应用带来不便。因此, 应用分子标记技术分析不同雪茄烟品种间的亲缘关系和遗传差异, 探索其遗传多样性的丰富程度, 并在此基础上构建雪茄烟种质资源指纹图谱库, 对于我国雪茄烟资源品种鉴定、品种选育及资源高效利用具有重要意义。
目前, 常用的分子标记有随机引物多态性标记(random amplified polymorphic DNA, RAPD)[7-9]、扩增片段长度多态性标记(amplified fragment length polymorphism, AFLP)[10-12]、简单重复序列(simple sequence repeats, SSR)、相关序列扩增多态性(sequence-related amplified polymorphism, SRAP)[13-14]、核糖体DNA和简单序列重复(inter-simple sequence repeat, ISSR)[15-17]、单核苷酸多态性(single nucleotide polymorphism, SNP)[18-19]等。在众多分子标记手段中, SSR分子标记由于具有灵敏度高、多态性好、稳定可靠等优点, 是分析遗传多样性及群体亲缘关系很好的标记方法。SSR标记技术已经被广泛用于作物品种鉴定[20-21]、多样性分析[22-24]、指纹图谱的构建[25-26]等研究中。近些年在烟草中也得到了较多应用, Bindler等[27]在2007年公布了第1张烟草SSR标记遗传连锁图谱, 并于2011年在原有的工作基础上又构建了包括2317个标记, 2363个位点的高密度遗传图谱[28], 为SSR分子标记的广泛应用奠定基础; Moon等[29]在2008年利用46对SSR引物分析了3种类型的54份烟草属材料的遗传多样性, 于2009年利用71对SSR引物对117个美国主栽烟草品种进行遗传多样性分析[30], 同年用70个SSR标记对来自美国烟草种质资源收集的702个材料进行了遗传多样性分析[31]; 马冰等[32]利用24对核心引物对20份烤烟种质进行了遗传多样性分析; 张雪廷等[33]利用30对SSR引物分析了38份晾晒烟种质资源的遗传关系; 童治军等[34]利用120个SSR标记分析了231份烤烟种质资源的遗传多样性及群体遗传结构; 刘国祥等[35]利用25对SSR标记对33份晒烟材料进行了遗传多样性分析并构建了DNA指纹图谱; 张铭真等[36]利用64对引物分析了81份烟草种质资源的遗传多样性; 陈芳等[37]利用48对SSR标记对80份烟草种质资源进行遗传多样性分析, 并绘制了SSR计算机数字指纹图谱; 目前为止国内外报道的有关烟草方面遗传多样性研究多为烤烟和晒晾烟, 而利用SSR标记对雪茄烟种质资源遗传关系进行分析并构建DNA指纹图谱, 在国内外尚未见报道。
本研究利用SSR分子标记对供试雪茄烟种质资源进行遗传多样性分析和指纹图谱的绘制, 以期确定烟草种质材料间的遗传背景差异及品种间亲缘关系, 为雪茄烟种质资源的鉴定、研究和利用提供依据, 并为后续充分发掘利用雪茄烟种质资源、选配合适的育种亲本及种质资源收集、鉴定、编目、保存等育种工作奠定基础。
1 材料与方法
1.1 试验材料
试验所采用的220份雪茄烟种质材料(附表1)由国家烟草种质资源中期库(中国农业科学院烟草研究所)、德阳市烟草公司提供。表1中的晒烟、晾烟、白肋烟及烤烟等类型材料均为选育或改良后的具有雪茄型香气的品种, 经多年试验已投入到雪茄烟选育及生产中, 因此这里统一称为雪茄烟。
1.2 基因组DNA的提取
供试材料于2019年种植在四川省什邡市师古镇苏家桥试验基地。取苗期幼嫩叶片于2 mL离心管中, 并快速放入干冰中冷冻, 带回实验室后置于-80℃的低温冰箱中保存。置于磨样机磨碎后利用SLS法提取DNA, 加ddH2O在4℃条件下溶解12 h。用核酸测定仪(Thermo Scientific NanoProp 2000)测定DNA浓度, 将DNA浓度统一稀释到50 ng μL-1,-20℃低温下保存。
1.3 SSR引物
试验所用烟草SSR标记引物是从Bindler等[28]公布的2317对SSR引物中经初步筛选得到的2000对扩增效果好的SSR引物, 由六合华大(北京)基因科技有限公司合成。
1.4 PCR扩增
扩增体系为10 μL, 包括1 μL的DNA (浓度50 ng μL-1)、2×RapidMaster Mix (含DNA聚合酶、dNTPs、MgCl2以及反应缓冲液) 5 μL、上游引物1 μL、下游引物1 μL, 剩余体系用ddH2O定量。总反应程序为95℃预变性5 min; 95℃变性15 s, 58℃(根据m值进行调整)退火15 s, 72℃延伸15 s, 循环35次; 72℃延伸8 min。PCR扩增产物在6%的非变性聚丙烯酰胺凝胶上电泳分离, 上样量为1.0 μL, 电压1200 V, 功率60 W下电泳1.5 h后银染显色, 照相保存。
1.5 数据分析
1.6 指纹图谱构建
根据利用尽量少的引物区分尽量多的品种的原则, 首先选取能够把供试材料完全区分开的部分引物作为核心引物构建供试材料指纹图谱代码, 然后参考王静毅等[44]和宋海斌等[45]的图谱代码构建方法进行构建。把每对核心SSR引物扩增出的条带, 按其分子量从小到大的顺序按1、2、3……进行编号, 记录各引物对材料扩增出的条带编号, 没有扩增出条带的用0表示。将核心引物按照顺序用A、B、C……进行编号, 每个品种的DNA经不同的引物扩增后将会形成由字母和阿拉伯数字组成的一系列带型编号, 便形成了该品种的SSR指纹图谱代码。利用在线软件草料二维码生成器(http://cli.im/)对每个品种进行编码, 将各个品种的品种名称、全国统一编号、类型、植物学分类、指纹图谱代码等信息一并录入, 生成供试雪茄种质的二维条码。
2 结果与分析
2.1 SSR引物的筛选
本研究利用6份烟草品种(Little Dutch、H211、Beinhart 1000-1、Hicks (Broad Leaf)、Havana 10、H382)对2000对候选引物进行筛选, 获得了43对多态性好、条带清晰的引物(表1)。
2.2 遗传多样性分析
43对SSR引物在220份供试材料中共检测出243个基因位点(表2), 每对引物可检测的等位基因数(a)为2~13, 平均为5.65个, 其中PT52689的最多, PT52176的最少。有效等位基因数(e)在1.3081~11.7876之间, 平均为3.9077, 有效等位基因所占比例为69.15%。引物的多态性信息含量(PIC)在0.2078~0.9087之间, 平均约为0.6360。其中有35对引物的PIC值大于0.5, 为高度多态性信息引物, 说明所选引物的多态性普遍较高。观测杂合度(o)的变化范围为0.0828~0.7639, 平均值为0.3191;群体平均Shannon’s遗传多样性指数()为1.3756, 其中PT52689的最高, 为2.5112, 而PT52176的最低, 为0.3983; 平均Nei’s多样性指数()为0.6793。期望杂合度(e)的平均值为 0.6809, 最大是0.9172 (PT52689), 最小为0.2361 (PT52176)。e的平均值大于0.5, 说明供试雪茄烟种质的遗传多样性较高[34]。
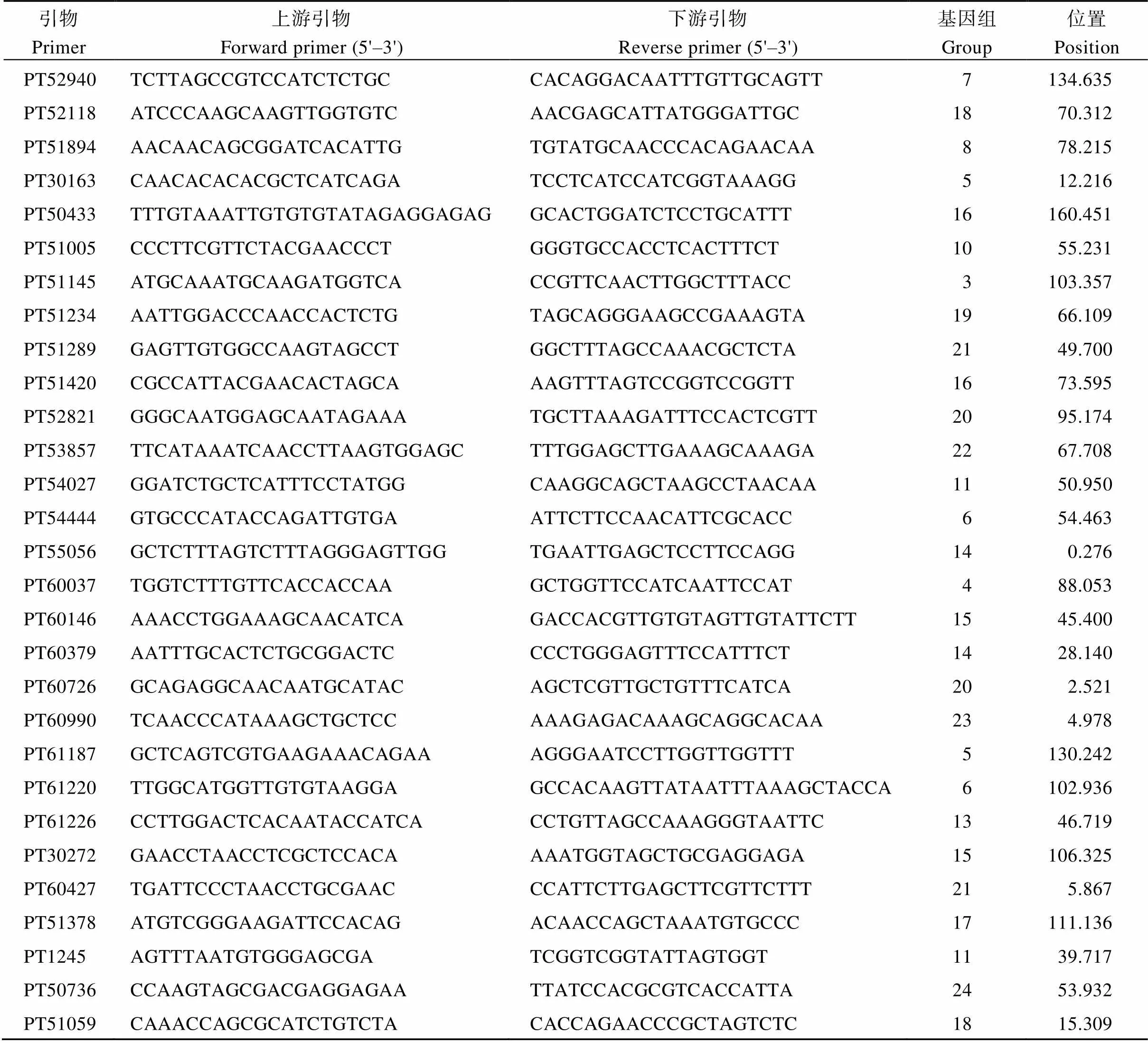
表1 SSR引物信息

(续表1)

表2 43对SSR在220份材料中的遗传多样性参数
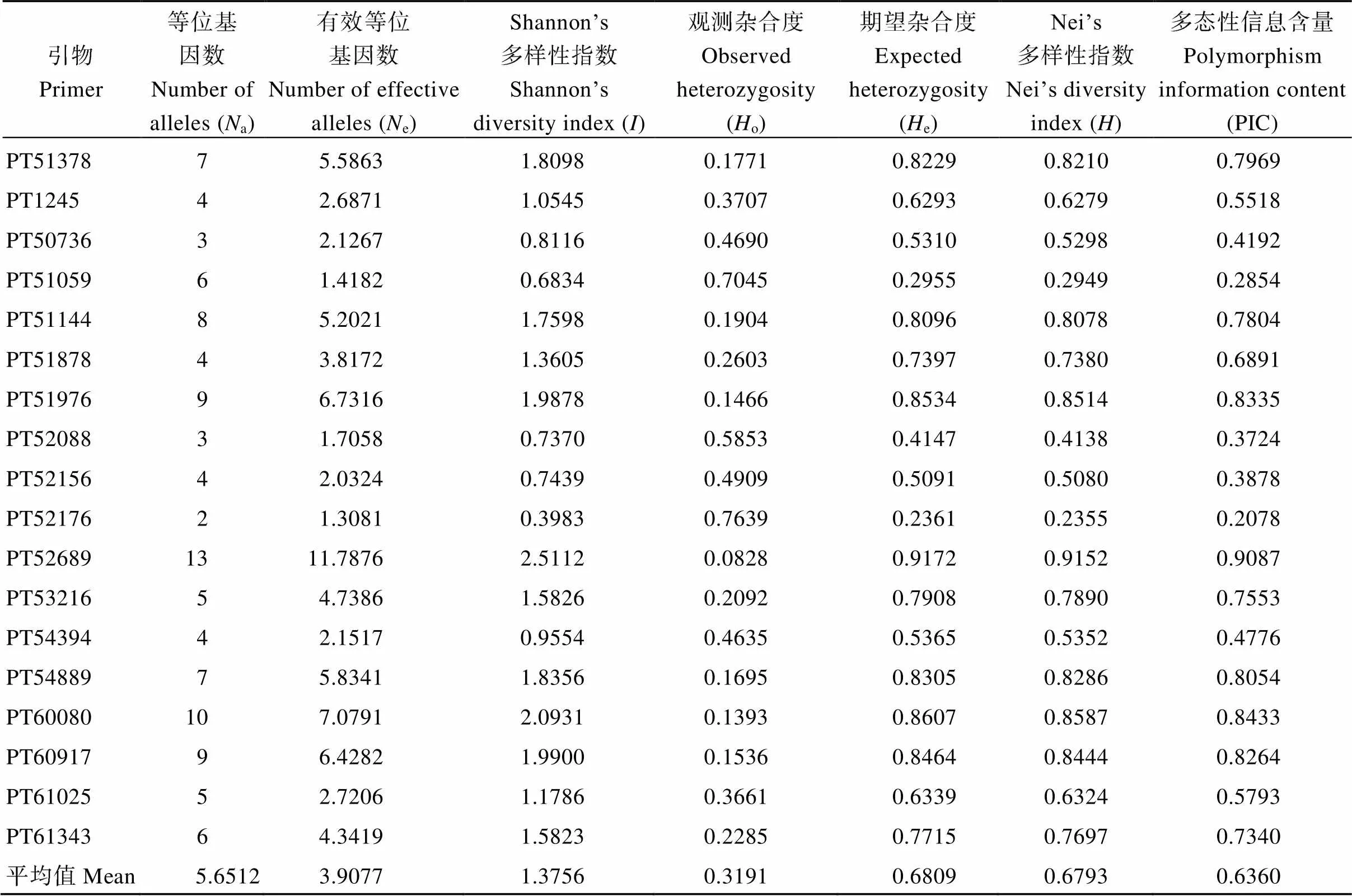
(续表2)
2.3 种质间遗传距离
通过SSR标记分析, 220份雪茄烟种质材料间的遗传距离(genetic distance, GD)在0.0233~0.9286之间(表略), 平均为0.6816。其中良种(No.211)、辅善(No.212)和满耳朵(No.213) 3个材料, 山东大叶(No.75)和牡丹江05-1 (No.76) 2个材料, Florida 513 (No.125)和CA0701 (No.141) 2个材料间的遗传距离均为0, 即品种间差异位点数为0, 判定为疑同品种。古巴2号-2 (No.11)和古巴2号-3 (No.12)、Connecticut Broad Leaf (No.40)和CA0708 (No.147)、Havana 211 (No.17)和CA0709 (No.148)、2040 (No.67)和CS0707 (No.160)、Florida 301 (No.36)和florida30 (No.166)、松阳(No.206)和连选(No.207)材料间的遗传距离最小, 为0.0233, 品种间差异位点数为1, 判定为相近品种。这些材料有的是从同一国家的相近地区引进的, 如古巴2号-2 (No.11)和古巴2号-3 (No.12)、松阳(No.206)和连选(No.207); 有的原本是同一个品种在不同地区长期种植过程中被命名了不同的名称, 如Florida 513 (No.125)和CA0701 (No.141)、Connecticut Broad Leaf (No.40)和CA0708 (No.147)、Havana 211 (No.17)和CA0709 (No.148)、2040 (No.67)和CS0707 (No.160)、Florida 301 (No.36)和florida30 (No.166)。需结合田间表型调查结果对疑同品种和相近品种进行进一步鉴定, 从而确定是否为同一种质。Mont Calme Brun (No.63)和什新-2 (No.72)、万毛1号(No.82)和松阳(No.206)、万毛1号(No.82)和连选(No.207)的遗传距离最大, 达到0.9286, 说明两者的亲缘关系较远。材料间遗传距离的变化区间在0.8821左右, 平均遗传距离为0.6816, 表明供试材料间变异范围较大, 亲缘关系较远, 遗传差异性较显著。
2.4 参试种质的聚类分析
基于Nei’s 遗传距离, 利用UPGMA方法对220份雪茄烟材料进行聚类分析, 其结果(图1)表明, 利用SSR标记可以将220份雪茄烟资源相互区分开, 在GD=0.74时可将材料分为3个类群(I为第1类群, II、III、IV为第2类群, V、VI为第3类群); 而在遗传距离GD=0.70处, 其中第2类群又可细分为II、III、IV 三个小类, 第3类群又被划分为V、VI两个小类, 按此可将220份雪茄烟种质资源分为6个类群。类群I由万毛1号(No.82)、龙井香叶子(No.84)和牡晒89-23-1 (No.182) 3个国内晒烟和国外引进的贝拉烟(No.181)一共4个地方性晒烟品种组成。聚类分析表明, 这4个品种在遗传距离较大时(GD=0.76), 就能明显与其他品种区分开来, 可能是由于这些品种为地方性品种, 种植面积小, 从而保留了较丰富的遗传变异; 类群II由E18 (No.37)和Trapesond 288 (No.58) 2个从不同国家引进的品种组成; 类群III由3个国外引进品种Zrenjanin (No.32)、Copus Pobrecene No.1241 (No. 39)、Begej (No.43)和1个国内选育品种世纪一号(No.52)组成; 类群IV共有40个品种, 其中土耳其雪茄(No.3)、Manila (No.4)、Comstock Spanish (No.41)、Havana IIc (No.14)、Connecticut Broad Leaf (No.40)等37个国外引进品种, 和江西晒烟(No.205)、城口晒烟(No.208)、青州一号(No.134) 3个国内地方品种组成。美国康州阔叶(No.136)、Florida 513 (No.51)、古巴2号(No.9)、德雪1号(No.129)、德雪2号(No.130)、德雪3号(No.131)、Cubra-Brazil (No.135)、Habana92 (No.132)、Beinhart 1000-1 (No.42)、beinhart 1000-1 (No.165)、什邡枇杷柳(No.91)、吉县大烟叶子(No.100)等48个品种聚于类群V。印尼雪茄包皮(No.54)、Zimmer Spanish (No.31)、Segedinska Ruca (No.1)、哈瓦娜(No.13)、小牛舌(No.47)等35个雪茄烟品种, 和牡晒81-21-2 (No.179)、利川乌烟(No.77)、桐乡晒烟(No.79)、沂水大弯筋(No.70)等87个晾晒烟品种, 共122个品种聚于类群VI。从聚类图中可以发现, 我国的品种和从各国引进的雪茄烟品种并没有很明显的分开, 每个类群中的品种不是单一的来自同产地, 各国的品种分散在每个类群中, 这说明聚类结果与雪茄烟种质的地域来源并没有显著的相关性。
类群I: 红色; 类群II: 蓝色; 类群III: 橙色; 类群IV: 湖蓝色; 类群V: 紫色; 类群VI: 绿色。
Group I: red; group II: blue; group III: orange; group IV: lake blue; group V: red; group VI: green.
2.5 主成分分析
根据SSR分子标记的结果用NTSYS软件根据位置近则亲缘关系近、位置远则亲缘关系远的原则[46], 以第1主成分和第2主成分为二维坐标图的横坐标和纵坐标, 构建了220份雪茄烟种质资源的二维主坐标分析图。由图2可知, 220份雪茄烟种质材料划分为2个类群, 其中类群I包含36份种质, 类群II共有184份雪茄烟种质, 每个类群都含有不同地理来源的雪茄烟种质资源, 从PCA图中可以清晰的看出, PCA主成分分析与UPGMA聚类分析得出相同的结果, 即雪茄烟种质资源并没有按地理来源进化划分, 地理来源相同的种质资源交错分布于2个类群中。
2.6 遗传结构分析
利用Structure软件对220份雪茄烟种质的群体遗传结构分析中发现, 在=1~15中, ln()值随假定亚群数值的增大而持续增大(图3-A), 则参照Evanno等[43]的方法, 由Δ值来确定220份雪茄烟资源的最优群体数值, 当=2时似然值最大(图3-B), 即所分析的雪茄烟种质从遗传结构上可划分为2个类群。参考苏一钧等[47]的研究认为在structure群体遗传结构分析中, 当某一材料的值≥0.6表示该材料的遗传背景相对比较单一, 而当值<0.6, 则说明该材料的遗传背景较为复杂。分析我国雪茄烟种质资源的值发现, 有38份资源(No.24、No.28、No.30、No.31、No.32、No.36、No.38、No.39、No.43、No.50、No.56、No.75、No.76、No.81、No.83、No.87、No.98、No.104、No.105、No.114、No.118、No.125、No.131、No.137、No.141、No.155、No.158、No.164、No.165、No.166、No.172、No.179、No.183、No.191、No.192、No.196、No.218、No.220)的值<0.6, 说明这38份雪茄烟资源的遗传组成较为复杂, 没有明确归属于哪个类群, 形成一个混合群体, 其余的182份雪茄烟种质资源的值≥0.6, 此部分资源的遗传结构较为单一, 可以划在某一个类群中。220份雪茄烟种质资源的群体遗传结构如图4, 其中第I类群包括92份材料, 第II类群含有90份雪茄烟种质资源。所有的供试材料没有按照地理来源进行分类, 分析的结果与UPGMA聚类分析和PCA主成分分析的结果基本一致。
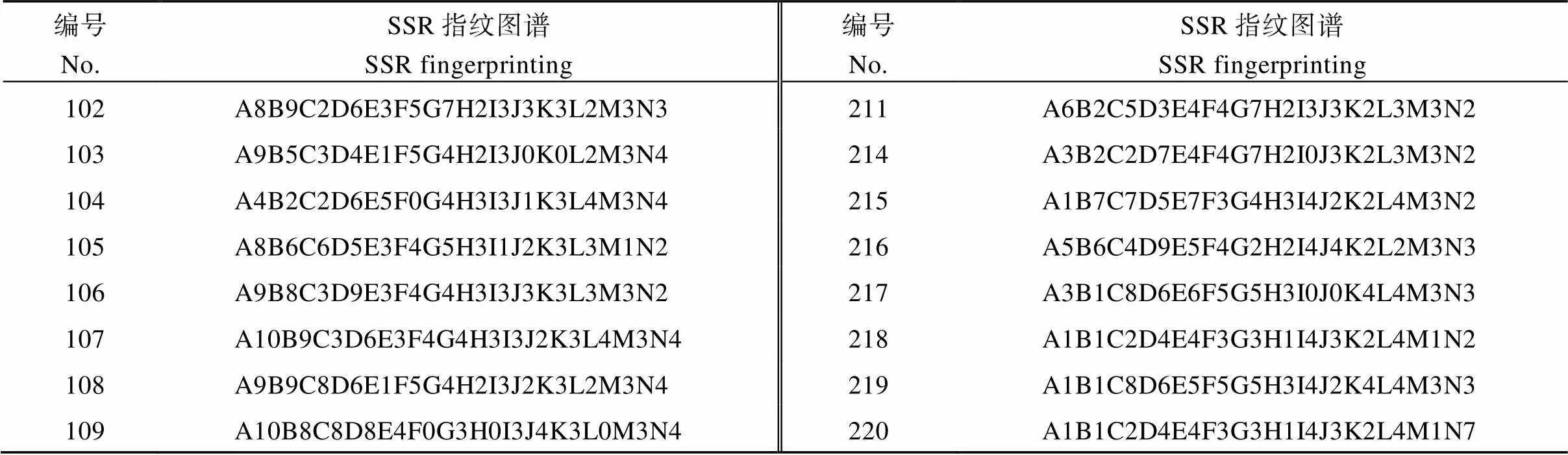
2.7 DNA指纹图谱的构建
本研究从43个SSR标记中选取14对SSR标记(PT52689、PT60080、PT51976、PT60917、PT61187、PT61226、PT54889、PT60726、PT51878、PT51145、PT52118、PT52821、PT60037、PT52940), 就可将供试雪茄烟烟种质完全区分开来。对于疑同品种和相近品种, 结合田间表型鉴定结果确定No.211、No.212和No.213三个材料, No.75和No.76两个材料, No.125和No.141两个材料为异名同种, 因此作为同一品种(No.211、No.212和No.213三个材料保留No.211; No.75和No.76两个材料保留No.75; No.125和No.141两个材料保留No.125)进行指纹图谱的构建。保留的216份雪茄烟种质资源指纹图谱代码构建方法依据之前设计的方案, 将14个引物依此用字母A~N表示, 每个引物生成的有差别的带型用该引物的字母和代表等位基因的数字编号表示, 为每份材料建立惟一能够区别于其他供试材料的指纹图谱代码(表3)。利用在线软件草料二维码生成器(http://cli.im/)对所有供试材料进行编码, 为每份材料构建了一份指纹图谱二维条码(图5), 其中包含了品种名称、全国统一编号、类型、植物学分类、指纹图谱代码等信息。如图1中A包含以下信息: 名称为Segedinska Ruca, 全国统一编号为00001238, 类型为雪茄烟, 植物学分类为双子叶植物纲(Dicotyledoneae)管花目(Tubiflorae)茄科(Solanaceae)烟属()普通烟草(L.), 指纹代码为A7B7C4D1E3F7G2H2I2J3K3L3M3N2。
A: ln()值随K值变化折线图; B: Δ值随K值变化折线图。
A: line chart of ln() with change of-values; B: line chart of Δwith change of-values.
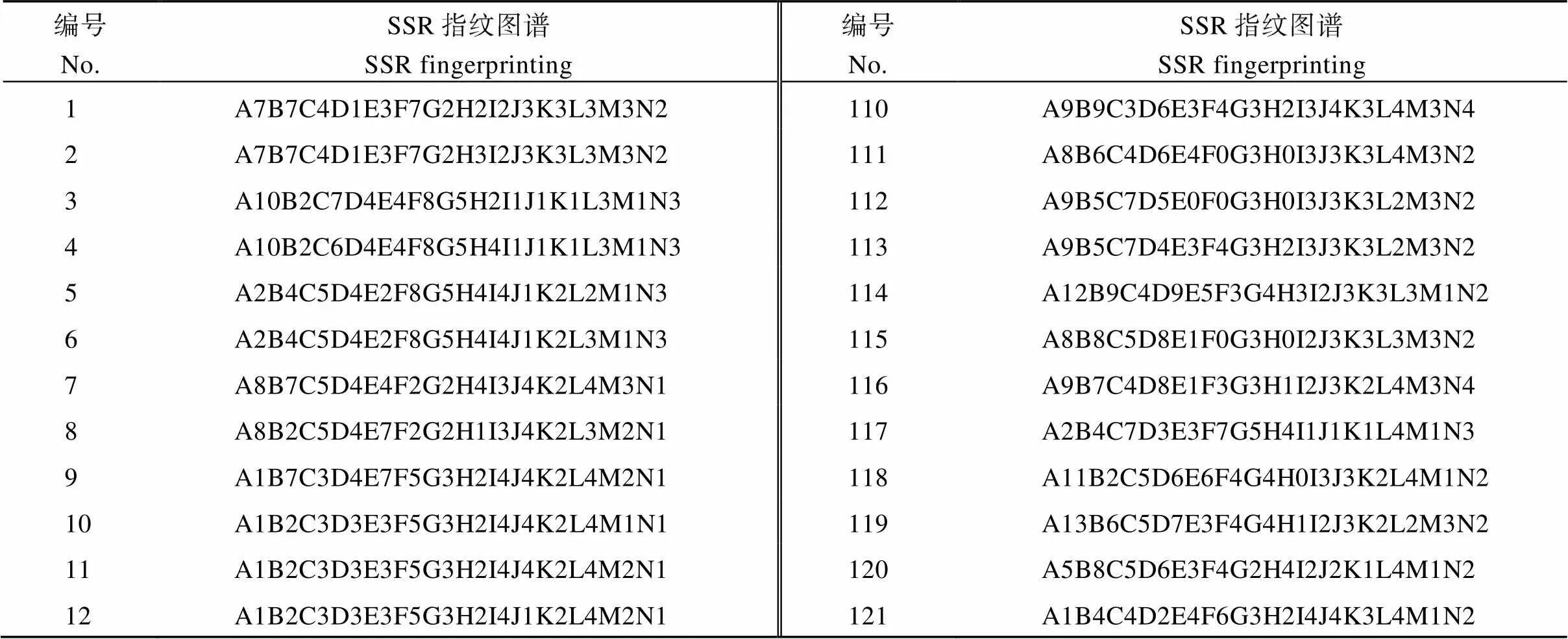
表3 216份雪茄烟种质资源的SSR指纹图谱代码

(续表3)
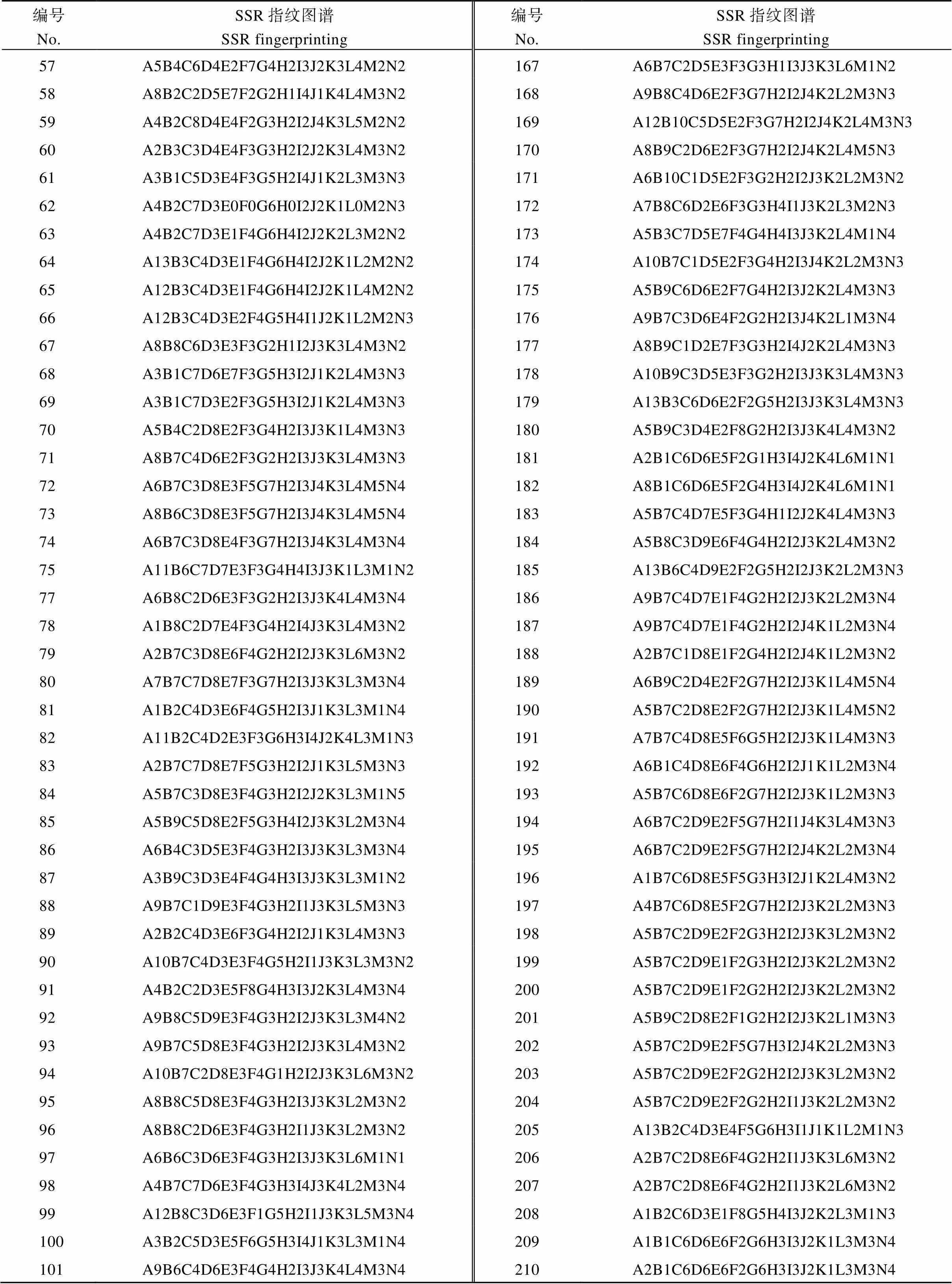
(续表3)
(续表3)
表4中编号对应表1同编号品种; A~N分别代表SSR引物PT52689、PT60080、PT51976、PT60917、PT61187、PT61226、PT54889、PT60726、PT51878、PT51145、PT52118、PT52821、PT60037、PT52940。
The number in Table 4 corresponds to the same number in Table 1; A–N represent primer of SSR PT52689, PT60080, PT51976, PT60917, PT61187, PT61226, PT54889, PT60726, PT51878, PT51145, PT52118, PT52821, PT60037, PT52940, respectively.
A~F分别为雪茄烟品种Segedinska Ruca、Scafati、土耳其雪茄、Manila、Connecticut Shade和Bad Geudertheimer Landsorte。
A–F represent the melon samples Segedinska Ruca, Scafati, Turkey Cigar, Manila, Connecticut Shade, and Bad Geudertheimer Landsorte, respectively.
3 讨论
3.1 雪茄烟种质资源遗传多样性分析
进行雪茄烟种质资源遗传多样性的研究对我国雪茄烟种质的鉴定、新基因的挖掘、新品种选育等育种工作至关重要。目前有不少烟草方面遗传多样性研究的报道, 但多为烤烟和晾晒烟, 而雪茄烟只进行过形态学上的鉴定[48-49], 受外界环境条件和栽培措施的影响较大, 极易产生误差导致结果不准确。作物品种鉴定的最可靠方法是品种的基因型鉴定, 利用分子标记技术从DNA水平上区分鉴别雪茄烟基因型间的遗传差异, 不受外界环境条件和栽培措施的影响, DNA分析方法是目前为止最有效的遗传分析方法[50]。操作简单、多态性好、耗费低的SSR标记已逐渐取代存在诸多缺点的通用型DNA标记RAPD、AFLP等。SSR标记已被广泛应用于烟草种质资源与品种遗传多样性分析[4,20-22,51-52], 因其成本较低也被视为烟草品种鉴定的理想工具[18-19,53]。
本研究利用SSR标记首次分析了雪茄烟种质资源遗传多样性, 因此, 没有与之相关的研究报道可供参考和比较。本研究利用43对SSR分子标记对收集到的220份雪茄烟种质资源的遗传多样性进行分析发现, 平均等位基因数为5.65,为0.6793, PIC为0.6360, 说明供试种质具有较高的遗传多样性, 引物总体上有较好的鉴别能力。供试材料间的遗传距离(GD)在0.0233~0.9286之间, 平均为0.6816, 说明雪茄烟种质资源的遗传距离较大, 遗传多样性比较丰富。主要原因可能为: 虽然我国最近十年才开始进行雪茄烟的大规模引种和栽培, 但是晾晒烟的种植历史悠久, 我国使用优质地方晾晒烟品种当做茄芯来使用, 当地也利用地方晾晒烟品种和引进的各种雪茄烟品种进行杂交, 以获得品种改良, 这在无形中拓宽了我国雪茄烟种质资源的遗传多样性。在220份雪茄烟种质资源中, 良种(No.211)、辅善(No.212)和满耳朵(No.213)、山东大叶(No.75)和牡丹江05-1 (No.76)、Florida 513 (No.125)和CA0701 (No.141)等种质被判定为疑同品种; 古巴2号-2 (No.11)和古巴2号-3 (No.12)、Connecticut Broad Leaf (No.40)和CA0708 (No.147)、Havana 211 (No.17)和CA0709 (No.148)、2040 (No.67)和CS0707 (No.160)、Florida 301 (No.36)和florida30 (No.166)、松阳(No.206)和连选(No.207)等种质被判定为相近品种, 具有非常高的遗传相似性。结合田间表型性状鉴定结果发现, 辅善(No.212)和满耳朵(No.213)、山东大叶(No.75)和牡丹江05-1 (No.76)、Florida 513 (No.125)和CA0701 (No.141)等种质为同一品种, 即异名同种。由此可见, SSR标记适用于雪茄烟品种鉴定和遗传多样性的研究中。SSR标记在雪茄烟遗传多样性分析、种质资源品种鉴定方面的应用, 为雪茄烟新品种的管理、杂交育种、产权保护提供科学依据。
本研究通过UPGMA聚类分析、PCA主成分分析和Structure软件的遗传结构分析3种分析方法对供试材料进行划分, 三者在分类结果上存在差异: UPGMA聚类分析在GD=0.74处将供试材料划分为3个大类, 而群体遗传结构和PCA主成分分析则将供试材料划分为2个亚群; UPGMA聚类分析和PCA主成分分析在类群内品种分布的结果上高度一致, 而群体遗传结构分析结果与这两者在类群内品种的分布上存在较大差异。造成差异的主要原因可能是各自的分类依据不同: 聚类分析和PCA主成分分析分别依据的是遗传距离(GD)和遗传相似系数(GS), 反映的都是品种间亲缘关系的远近, 这2种分析方法在类群内具体品种的分布结果上高度一致; 而群体遗传结构分类的依据服从Hardy-Weinberg平衡的亚群数目, 是建立在数学模型的基础上, 并计算各供试材料间相应的值。因此, Structure软件的遗传结构分析的结果避免了人为因素对群体划分的影响, 具有较高的准确性[34]。3种分析方法的结果均显示, 收集的这220份来自各地的雪茄烟资源交错分布于每个类群中, 遗传分类与地域因素没有显著相关性。主要因为我国是从引种鉴定、推广适宜各产区栽培条件的优良品种开始雪茄烟种植的, 并在此基础上发展新的雪茄烟品种。我国的雪茄烟资源大多是从国外引进, 有些雪茄烟资源有较好的香气, 如Havana系列品种; 有的是好的抗源, 如高抗黑胫病的Florida301, 高抗黑胫病、赤星病的Beinhart 1000-1等, 这些优质、抗病品种被广泛用作亲本与其他不同品种进行杂交选育出品质较好、抗病能力强的品种或是作为试验材料进行雪茄烟分子育种研究, 也进一步表明我国雪茄烟种质资源的遗传多样性丰富。本研究中的220份雪茄烟种质资源的聚类结果与地理来源无相关关系, 说明我国雪茄烟种质资源遗传背景比较复杂。
3.2 雪茄烟种质资源指纹图谱库的构建
DNA指纹图谱是在分子标记的基础上绘制而成的, 这种图谱多态性丰富, 具有高度的个体特异性和环境稳定性, 犹如人的指纹一样能够鉴别生物个体之间的差异, 因此被称为DNA指纹图谱(DNA fingerprinting)[54]。DNA指纹图谱具有快速、准确的特点, 是鉴别品种、品系的有力工具, 也非常适应于种质资源的鉴定工作[55]。SSR分子标记特异性强、谱带清晰、数据准确, 适合大量资源的指纹图谱构建工作[35], 利用SSR标记进行烟草种质资源指纹图谱的构建已有报道[35,37,56]。本研究利用筛选出的14对可以完全区分所有供试材料的SSR引物, 和根据供试材料分别在14对引物扩增出条带的分子量大小及条带类型构建了由字母和阿拉伯数字组成的216份雪茄烟种质资源DNA指纹图谱代码; 结合供试材料的相关信息和指纹图谱代码进行编码又构建了216份雪茄烟种质资源的DNA指纹图谱二维条码。本研究通过这2种方式对雪茄烟种质资源进行指纹图谱库的构建, 使每个品种都有唯一的一套DNA指纹, 可在分子水平上解决同种异名和同名异种等品种混乱现象, 若2份材料的指纹数据一致则被认为是同一品种。雪茄烟种质资源SSR指纹图谱库的建立使品种鉴定更加高效准确, 为以后雪茄烟种质资源的鉴定、保护及育种研究奠定理论基础。
4 结论
本研究利用43对SSR引物对220份雪茄烟种质资源进行遗传多样性分析表明, 这些材料亲缘关系较远, 遗传差异性显著, 遗传多样性丰富, 这对我国雪茄烟种质资源的利用及品种选育方面具有重要意义; 利用从43对SSR引物中筛选出的14对核心引物构建了216份雪茄烟种质资源的指纹图谱库, 为今后我国雪茄烟品种鉴定体系的研究提供技术支撑。
附表 请见网络版: 1) 本刊网站http://zwxb.chinacrops.org/; 2) 中国知网http://www.cnki.net/; 3) 万方数据http://c.wanfangdata.com.cn/Periodical-zuo wxb.aspx。
[1] 刘国顺. 烟草栽培学. 北京: 中国农业出版社, 2003. pp 21–22. Liu G S. Tobacco Cultivation. Beijing: China Agriculture Press, 2003. pp 21–22 (in Chinese).
[2] 童治军. 烟草微卫星标记的开发与应用. 浙江大学博士学位论文, 浙江杭州, 2012. Tong Z J. Development and Application of Microsatellite Markers in Flue-cured Tobacco (L). PhD Dissertation of Zhejiang University, Hangzhou, Zhejiang, China, 2012 (in Chinese with English abstract).
[3] 任民, 王志德, 牟建民,刘艳华, 张兴伟. 我国烟草种质资源的种类与分布概况. 中国烟草科学, 2009, 30(增刊1): 8–14.Ren M, Wang Z D, Mu J M, Liu Y H, Zhang X W. Overview of species and distribution of tobacco germplasm resources in China., 2009, 30(S1): 8–14 (in Chinese with English abstract).
[4] 杨柳, 汪斌, 童治军, 黄勇兵, 曹梅秀, 吴海乔, 巫升鑫, 陈顺辉, 兰涛. 25份普通烟草种质资源遗传多样性的SSR标记分析. 福建农林大学学报(自然科学版), 2013, 42(2): 171–175. Yang L, Wang B, Tong Z J, Huang Y B, Cao M X, Wu H Q, Wu S X, Chen S H, Lan T. Genetic diversity analysis of 25L. germplasm resources based on SSR marker.(Nat Sci Edn), 2013, 42: 171–175 (in Chinese with English abstract).
[5] 尹国英. 烟草种质资源遗传多样性和应用潜力分析以及核心SSR引物筛选. 西南大学硕士学位论文, 重庆, 2013. Yin G Y. Analysis of Genetic Diversity and Application Potential of Tobacco Germplasm Resources and Screening of Core SSR Primers. MS Thesis of Southwest University, Chongqing, China, 2013 (in Chinese with English abstract).
[6] 李爱军, 秦艳青, 代惠娟, 范静苑, 曾代龙, 王华敬, 潘运金. 国产雪茄烟叶科学发展刍议. 中国烟草学报, 2012, 18(1): 112–114. Li A J, Qin Y Q, Dai H J, Fan J Y, Zeng D L, Wang H J, Pan Y J. On scientific development of China’s cigar leaf., 2012, 18(1): 112–114 (in Chinese with English abstract).
[7] Piano L D, Abet M, Sorrentino C, Acanfora F, Cozzolino E, Muro A D. Genetic variability inandspecies as revealed by RAPD markers: 1. Development of the RAPD procedure., 2000, 19: 1–15.
[8] 常爱霞, 瞿永生, 贾兴华. 烟草RAPD反应体系优化及品种多态性标记研究. 中国烟草科学, 2004, 25(2): 9–13. Chang A X, Qu Y S, Jia X H. Optimalization of tobacco RAPD reaction system and studies on polymorphic marker of tobacco varieties., 2004, 25(2): 9–13 (in Chinese with English abstract).
[9] Arslan B, Okumus A. Genetic and geographic polymorphism of cultivated tobaccos () in Turkey., 2006, 42: 818–823.
[10] Dadras A R, Sabouri H, Nejad G M, Sabouri A, Shoai-Deylami M. Association analysis, genetic diversity and structure analysis of tobacco based on AFLP markers., 2014, 41: 3317–3329.
[11] 杨友才, 周清明, 尹晗琪, 朱列书. 烟草种质资源遗传多样性及亲缘关系的AFLP分析. 中国农业科学, 2006, 39: 2194–2199. Yang Y C, Zhou Q M, Yin H Q, Zhu L S. Studies on genetic diversity and relationship in tobacco germplasms by AFLP analysis., 2006, 39: 2194–2199 (in Chinese with English abstract).
[12] Ren N, Timko M P. AFLP analysis of genetic polymorphism and evolutionary relationships among cultivated and wildspecies., 2001, 44: 559–571.
[13] 龙腾, 刘雷, 黄玉碧. 四川部分晾晒烟种质遗传关系的SRAP分析. 作物学报, 2009, 35: 173–178. Teng L, Liu L, Huang Y B. Genetic analvsis of Sichuan sun-cured tobacco germplasm by SRAP., 2009, 35: 173–178.
[14] 张吉顺, 王仁刚, 杨春元, 吴春, 史跃伟, 王志红, 王轶, 任学良. 国内外烤烟品种农艺性状的遗传多样性及与SRAP标记的关联分析. 作物学报, 2012, 38: 1029–1041. Zhang J S, Wang R G, Yang C Y, Wu C, Shi Y W, Wang Z H, Wang Y, Ren X L. Genetic diversity of agronomic traits and association analysis with SRAP markers in flue-cured tobacco () varieties from China and abroad., 2012, 38: 1029–1041 (in Chinese with English abstract).
[15] Qiong N, Renxiang L, Wei L. Fingerprint map construction of core tobacco germplasm by ISSR markers., 2010, 23: 335–339.
[16] Edrisi M K, Samizadeh L H, Shoaei D M. Assessing the genetic diversity of tobacco (L.) varieties., 2012, 2: 125–132.
[17] 梁景霞, 祁建民, 方平平, 王涛, 陈顺辉, 周东新, 陶爱芬, 梁康迳, 吴为人. 烟草种质资源遗传多样性与亲缘关系的ISSR聚类分析. 中国农业科学, 2008, 41: 286–294. Liang J X, Qi J M, Fang P P, Wang T, Chen S H, Zhou D X, Tao A F, Liang K J, Wu W R. Genetic diversity and genetic relatives analysis of tobacco germplasm based on inter-simple sequence repeat (ISSR)., 2008, 41: 286–294 (in Chinese with English abstract).
[18] Salazar J A, Rubio M, Ruiz D, Ruiz D, Tartarini S, Gómez P M, Dondini L. SNP development for genetic diversity analysis in apricot., 2015, 11: 15.
[19] Wang W S, Mauleon R, Hu Z Q, Chebotarov D, Tai S S, Wu Z C, Li M, Zheng T Q, Fuentes R R, Zhang F, Mansueto L, Copetti D, Sanciangco M, Palis K C, Xu J L, Sun C, Fu B Y, Zhang H L, Gao Y M, Zhao X Q, Shen F, Cui X, Yu H, Li Z C, Chen M L, Detras J, Zhou Y L, Zhang X Y, Zhao Y, Kudrna D, Wang C C, Li R, Jia B, Lu J Y, He X C, Dong Z T, Xu J B, Li Y H, Wang M, Shi J X, Li J, Zhang D B, Lee S, Hu W S, Poliakov A, Dubchak I, Ulat V J, Borja F N, Mendoza J R, Ali J, Li J, Gao Q, Niu Y C, Yue Z, Naredo M E B, Talag J, Wang X Q, Li J J, Fang X D, Yin Y, Glaszmann J C, Zhang J W, Li J Y, Hamilton R S, Wing R A, Ruan J, Zhang G Y, Wei C C, Alexandrov N, McNally K L, Li Z K, Leung H. Genomic variation in 3,010 diverse accessions of Asian cultivated rice., 2018, 557: 43–49.
[20] 尚维, 赵申清玉, 党江波, 郭启高, 梁国鲁, 杨超, 张艳, 陈益银. 基于SSR分子标记的异源染色体植株的鉴定与筛选. 作物学报, 2018, 44: 1640–1649. Shang W, Zhao S Q Y, Dang J B, Guo Q G, Liang G L, Yang C, Zhang Y, Chen Y Y. Identification and screening ofheterologous chromosome plants based on SSR marker., 2018, 44: 1640–1649 (in Chinese with English abstract).
[21] 焦荻, 商纪鹏, 高素燕, 王钦, 郝建全, 何伟, 焦定量, 吕敬刚, 陈则明. 西瓜品种‘蜜多’种子纯度SSR标记鉴定. 中国瓜菜, 2019, 32(7): 19–22. Jiao D, Shang J P, Gao S Y, Wang Q, Hao J Q, He W, Jiao D L, Lyu J G, Chen Z M. Identification of ‘Miduo’ watermelon seed purity using SSR marker., 2019, 32(7): 19–22 (in Chinese with English abstract).
[22] 卢媛, 艾为大, 韩晴,王义发, 李宏杨, 瞿玉玑, 施标, 沈雪芳. 糯玉米自交系SSR标记遗传多样性及群体遗传结构分析. 作物学报, 2019, 45: 214–224. Lu Y, Ai W D, Han Q, Wang Y F, Li H Y, Qu Y J, Shi B, Shen X F. Genetic diversity and population structure analysis by SSR markers in waxy maize., 2019, 45: 214–224 (in Chinese with English abstract).
[23] 赵靓, 罗燕杰, 肖思文, 蔡亚南, 何立飞, 遆羽静, 李庆卫. 基于表型和SSR标记的梅花种质资源遗传多样性分析. 分子植物育种, 2019, 17: 4458–4469. Zhao L, Luo Y J, Xiao S W, Cai Y N, He L F, Di Y J, Li Q W. Genetic diversity analysis ongermplasm resources based on phenotypic traits and SSR markers., 2019, 17: 4458–4469 (in Chinese with English abstract).
[24] 张跃新, 胡伟, 郝艺铭, 闫宝松, 马凤, 么宏伟. 北方地区黑木耳部分主栽品种与野生菌株遗传多样性SSR分析. 中国食用菌, 2019, 38(5): 44–48. Zhang Y X, Hu W, Hao Y M, Yan B S, Ma F, Yao H W. Analysis on genetic diversity for the part of main cultivars and wild strains of auricularia auricula-judae from Northern China by SSR markers., 2019, 38(5): 44–48 (in Chinese with English abstract).
[25] 倪维晨, 李瑞霞, 陶启威, 张戟, 毕研飞, 钱春桃. 基于SSR标记的地方品种糯性小玉米自交系指纹图谱构建. 浙江农业科学, 2019, 60: 911–914. Ni W C, Li R X, Tao Q W, Zhang Q, Bi Y F, Qian C T. Construction of fingerprints of waxy maize inbred lines based on SSR markers., 2019, 60: 911–914 (in Chinese with English abstract).
[26] 马斌, 李军乔, 刘贺贺, 刘欣, 李风英, 富贵, 白世俊, 韦梅琴. 蕨麻品种SSR指纹图谱的构建及遗传相似性分析. 分子植物育种, 2019, 17: 4367–4377. Ma B, Li J Q, Liu H H, Liu X, Li F Y, Fu G, Bai S J, Wei M Q. Construction and analysis of genetic similarity of SSR fingerprints in Juema., 2019, 17: 4367–4377 (in Chinese with English abstract).
[27] Bindler G, Hoeven R V D, Gunduz I, Plieske J, Ganal M, Rossi L, Gadani F, Donini P. A microsatellite marker based linkage map of tobacco., 2007, 114: 341–349.
[28] Bindler G, Plieske J, Bakaher N, Gunduz I, Ivanov N, Hoeven R V D, Ganal M, Donini P. A high density genetic map of tobacco (L.) obtained from large scale microsatellite marker development., 2011, 123: 219–230.
[29] Lewis R S, Nicholson J S, Moon H S. Use of transferableL. microsatellite markers for investigating genetic diversity in the genus., 2008, 51: 547–559.
[30] Moon H S, Nicholson J S, Heineman A, Lion K, Hoeven R V D, Hayes A J, Lewis R S. Changes in genetic diversity of U.S. flue-cured tobacco germplasm over seven decades of cultivar development., 2009, 49: 498–508.
[31] Moon H S, Nifong J M, Nicholson J S, Heineman A, Lion K, Hoeven R V D, Hayes A J, Lewis R S. Microsatellite-based analysis of tobacco (L.) genetic resources., 2009, 49: 2149–2159.
[32] 马冰, 代帅帅, 程亚增, 蒋彩虹, 任民, 程立锐, 杨爱国. 烤烟种质资源SSR核心引物的筛选及验证. 中国烟草科学, 2016, 37(5): 1–5. Ma B, Dai S S, Cheng Y Z, Jiang C H, Ren M, Cheng L R, Yang A G. Screen and identification of SSR core primers for flue-cured tobacco germplasm., 2016, 37(5): 1–5 (in Chinese with English abstract).
[33] 张雪廷, 童治军, 焦芳婵, 肖炳光, 曾千春. 38份晾晒烟种质资源遗传关系的SSR分析. 植物遗传资源学报, 2013, 14: 653–658. Zhang X T, Tong Z J, Jiao F C, Xiao B G, Zeng Q C. Genetic relationship analysis of thirty-eight sun/air-cured tobacco germplasms based on simple sequence repeat (SSR) markers., 2013, 14(4): 653–658 (in Chinese with English abstract).
[34] 童治军, 陈学军, 方敦煌, 曾建敏, 吴兴富, 肖炳光. 231份烤烟种质资源SSR标记遗传多样性及其与农艺性状和化学成分的关联分析. 中国烟草学报, 2017, 23(5): 31–40. Tong Z J, Chen X J, Fang D H, Zeng J M, Wu X F, Xiao B G. SSR marker-based analyses on genetic diversity and relevant variations of agronomic traits and chemical composition of 231 flue-cured tobacco germplasm resources., 2017, 23(5): 31–40 (in Chinese with English abstract).
[35] 刘国祥, 邹昆晏, 任民, 佟英, 冯全福, 杨爱国, 戴培刚, 徐宜民, 张兴伟. 33份晒烟种质资源SSR标记的指纹图谱构建. 中国烟草学报, 2017, 23(5): 84–91. Liu G X, Zou K Y, Ren M, Tong Y, Feng Q F, Yang A G, Dai P G, Xu Y M, Zhang X W. Construction of SSR markers’ fingerprints of 33 sun-cured tobacco germplasm resources., 2017, 23(5): 84–91 (in Chinese with English abstract).
[36] 张铭真, 李晓辉, 王袁, 杨铁钊, 徐世晓. 81份烟草种质资源遗传多样性分析. 江西农业学报, 2017, 29(1): 62–68. Zhang M Z, Li X H, Wang Y, Yang T Z, Xu S X. Analysis of genetic diversity of 81 tobacco germplasm resources., 2017, 29(1): 62–68 (in Chinese with English abstract).
[37] 陈芳, 徐世晓, 李晓辉, 刘超, 周建飞, 王袁, 田培, 杨铁钊. 基于SSR标记的80份烟草种质指纹图谱的构建及遗传多样性分析. 作物杂志, 2019, (1): 28–37. Chen F, Xu S X, Li X H, Liu C, Zhou J F, Wang Y, Tian P, Yang T Z. Construction of molecular fingerprinting and analysis of genetic diversity for 80 tobacco () germplasms based on SSR markers., 2019, (1): 28–37 (in Chinese with English abstract).
[38] 樊文强, 盖红梅, 孙鑫, 杨爱国, 张忠锋, 任民. SSR数据格式转换软件DataFormater. 分子植物育种, 2016, 14: 265–270. Fan W Q, Ge H M, Sun X, Yang A G, Zhang Z F, Ren M. DataFormater, a software for SSR data formatting to develop population genetics analysis., 2016, 14: 265–270 (in Chinese with English abstract).
[39] Yen F, Boyle T. Population genetic analysis of co-dominant and dominant markers and quantitative traits., 1997, 129: 157.
[40] Sándor N, Péter P, István C, Mousapour G A, Géza H, János T. PICcalc: an online program to calculate polymorphic information content for molecular genetic studies., 2012, 50: 670–672.
[41] Sudhir K, Glen S, Koichiro T. MEGA7: molecular evolutionary genetics analysis Version 7.0 for bigger datasets., 2016, 33: 1870–1874.
[42] Pritchard J K, Stephens M, Donnelly P. Inference of population structure using multilocus genotype data., 2000, 155: 945–959.
[43] Evanno G, Regnaut S, Goudet J. Detecting the number of clusters of individuals using the software STRUCTURE: a simulation study., 2005, 14: 2611–2620.
[44] 王静毅, 陈业渊, 黄秉智, 于飞, 武耀廷. 部分香蕉品种SSR 指纹图谱的构建. 果树学报, 2009, 26: 733–738. Wang J Y, Chen Y Y, Huang B Z, Yu F, Wu Y T. Establishment of fingerprinting for bananas () by SSR marker., 2009, 26: 733–738 (in Chinese with English abstract).
[45] 宋海斌, 崔喜波, 马鸿艳, 朱子成, 栾非时. 基于SSR 标记的甜瓜品种(系)DNA 指纹图谱库的构建. 中国农业科学, 2012, 45: 2676–2689. Song H B, Cui X B, Ma H Y, Zhu Z C, Luan F S. Construction of DNA fingerprint database based on SSR marker for varieties (lines) ofL., 2012, 45: 2676–2689 (in Chinese with English abstract).
[46] 陈越, 陈玲, 李春花, 张敦宇, 付坚, 程在全. 中国南方地区水稻资源SSR指纹数据库的构建及遗传多样性分析. 分子植物育种, 2020, 18: 6502–6517. Chen Y, Chen L, Li C H, Zhang D Y, Fu J, Cheng Z Q. Construction of SSR fingerprint database and genetic diversity analysis of rice resources in Southern China., 2020, 18: 6502–6517 (in Chinese with English abstract).
[47] 苏一钧, 王娇, 戴习彬, 唐君, 赵冬兰, 张安, 周志林, 曹清河. 303份甘薯地方种SSR遗传多样性与群体结构分析. 植物遗传资源学报, 2018, 19: 243–251. Su Y J, Wang J, Dai X B, Tang J, Zhao D L, Zhang A, Zhou Z L, Cao Q H. Genetic diversity and population structure analysis of 303 sweet potato landraces using SSR markers., 2018, 19: 243–251 (in Chinese with English abstract).
[48] 许美玲, 贺晓辉, 宋玉川, 刘勇, 李梅云, 樊有银, 李永平. 72份雪茄烟种质资源的鉴定评价和聚类分析. 中国烟草学报, 2017, 23(5): 41–56. Xu M L, He X H, Song Y C, Liu Y, Li M Y, Fan Y Y, Li Y P. Identification, evaluation and cluster analysis of 72 cigar tobacco germplasm resources., 2017, 23(5): 41–56 (in Chinese with English abstract).
[49] 许美玲, 贺晓辉, 宋玉川, 李梅云, 方敦煌, 陈学军, 樊有银, 李永平. 76份雪茄烟资源鉴定评价. 中国烟草学报, 2018, 24(5):18–26. Xu M L, He X H, Song Y C, Li M Y, Fang D H, Chen X J, Fan Y Y, Li Y P. Identification and comprehensive evaluation of 76 germplasms of cigar tobacco., 2018, 24(5): 18–26 (in Chinese with English abstract).
[50] 陈杰, 杨静, 龙胜贤, 肖慈平, 杨昌义, 黄清忠, 王维. SSR分子标记在烟草研究中的应用进展. 生物技术通报, 2015, 31(3): 43–48. Chen J, Yang J, Long S X, Xiao C P, Yang C Y, Huang Q Z, Wang W. Advance of the application of SSR molecular markers in tobacco research., 2015, 31(3): 43–48 (in Chinese with English abstract).
[51] 赖瑞强, 李荣华, 夏岩石, 郭培国, 袁清华, 赵伟才. 烟草种质的SSR标记遗传多样性及青枯病抗性的关联分析. 中国烟草学报, 2018, 24(6): 71–81. Lai R Q, Li R H, Xia Y S, Guo P G, Yuan Q H, Zhao W C. SSR marker-based genetic diversity analysis of tobacco germplasm and association analysis with resistance to bacterial wilt., 2018, 24(6): 71–81 (in Chinese with English abstract).
[52] 丛鑫, 刘艳华, 戴培刚, 王志德, 张兴伟, 任民, 程立锐, 姚志敏. 抗PVY烟草种质的遗传多样性分析. 植物遗传资源学报, 2014, 15: 679–684. Cong X, Liu Y H, Dai P G, Wang Z D, Zhang X W, Ren M, Cheng L R, Yao Z M. Genetic diversity analysis of tobacco germplasm resistance to PVY., 2014, 15: 679–684 (in Chinese with English abstract).
[53] 孙九喆, 杨金初, 苏东嬴, 王二彬, 王君婷, 张顺峰. 基于SSR标记的初烤烟叶品种快速鉴别. 烟草科技, 2019, 52(3): 32–38.Sun J Z, Yang J C, Su D Y, Wang E B, Wang J T, Zhang S F. Rapid identification of cured tobacco leaf varieties based on SSR markers., 2019, 52(3): 32–38 (in Chinese with English abstract).
[54] Hilde N, Kurt W, Bjorn R. DNA fingerprinting in botany: past, present, future., 2014, 5: 1.
[55] 王忠华. DNA指纹图谱技术及其在作物品种资源中的应用. 分子植物育种, 2006, 4: 425–430. Wang Z H. DNA Fingerprinting technology and its application in crop germplasm resources., 2006, 4: 425–430 (in Chinese with English abstract).
[56] 徐军. 烟草核心种质SSR指纹图谱构建及遗传多样性分析. 中国农业科学院研究生院硕士学位论文, 山东青岛, 2011. Xu J. Construction of Fingerprinting and Analysis of Genetic Diversity with SSR Marker for Tobacco Core Germplasm. MS Thesis of Chinese Academy of Agricultural Sciences, Qingdao, Shandong, China, 2011 (in Chinese with English abstract).
Construction of SSR fingerprint database and genetic diversity analysis of cigar germplasm resources
WANG Yan-Yan1,**, WANG Jun2,**, LIU Guo-Xiang1, ZHONG Qiu2, ZHANG Hua-Shu2, LUO Zheng-Zhen2, CHEN Zhi-Hua3, DAI Pei-Gang1, TONG Ying1, LI Yuan1, JIANG Xun1, ZHANG Xing-Wei1,*, and YANG Ai-Guo1,*
1Tobacco Research Institute, Chinese Academy of Agricultural Sciences, Qingdao 266101, Shandong, China;2Deyang Tobacco Company of Sichuan Province, Deyang 618400, Sichuan, China;3Sichuan Institute of Tobacco Science, Chengdu 610000, Sichuan, China
In order to investigate the genetic diversity of Chinese cigar germplasm resources in molecular level and to establish the DNA fingerprint database of cigar varieties, 43 pairs of SSR primers with good polymorphic were used to analyze the genetic diversity of 220 cigar germplasms, and 14 pairs of core primers were screened from 43 pairs of SSR primers to construct the fingerprint database of 220 cigar germplasms in the study. Results showed that a total of 243 alleles were amplified by 43 pairs of SSR primers in 220 cigar germplasm materials, with an average of 5.65 per marker, ranging from 2 to 13. The polymorphic information content (PIC) of each locus varied from 0.2078 to 0.9087 with an average of 0.6360. The number of effective alleles (e) ranged from 1.3081 to 11.7876, and the average number of effective alleles was 3.9077. The observed heterozygosity (o) ranges from 0.0828 to 0.7639 with an average of 0.3191. The expected heterozygosity (e) ranged from 0.2361 to 0.9172 with an average of 0.6809. The average Shannon genetic diversity index () was 1.3756, the genetic distance was between 0.0233 and 0.9286, and the average genetic distance was 0.6816. Unweighted pair-group method with arithmetic means (UPGMA) cluster analysis showed that the tested cigar resources can be divided into three groups at the genetic distance of 0.74. All tested materials were divided into two groups by population genetic structure analysis and principal component analysis (PCA). Based on the primer analysis and phenotypic identification, it was determined that the ‘LZ’, ‘FS’ and ‘MED’, ‘SDDY’ and ‘MDJ 05-1’, ‘Florida 513’ and ‘CA0701’ were the same species with different names. One variety retained one germplasm, leaving 216 different germplasm. 14 pairs of SSR primers which could distinguish all the tested materials were screened to construct the fingerprint database of 216 cigar cultivars from 43 pairs of SSR primers as core primers. In conclusion, cigar germplasm resources in China had a high level of genetic diversity. In this study, the constructed SSR fingerprint database and genetic analysis of cigar germplasm resources provided the scientific basis for the screening and identification of high-quality cigar germplasms resources, the mining of important genes, and the broadening the basis of cigar genetic breeding.
cigar; germplasm resources; SSR marker; genetic diversity; fingerprint database
10.3724/SP.J.1006.2021.04183
本研究由中国烟草总公司四川省公司科技项目(SCYC201903), 中国农业科学院科技创新工程(ASTIP-TRIC01), 中央级公益性科研院所基本科研业务费专项(1610232019008), 农作物种质资源保护与利用专项(2020NWB038)和国家作物种质资源库(NCGRC-2020-18)资助。
This study was supported by the Science and Technology Project of China National Tobacco Corporation Sichuan Branch (SCYC201903), the Science and Technology Innovation Project of Chinese Academy of Agricultural Sciences (ASTIP-TRIC01), the Special Funds for Fundamental Scientific Research Operation of Central-level Public Welfare Scientific Research Institutes (1610232019008), the Special Project for Protection and Utilization of Crop Germplasm Resources (2020NWB038), and the National Crop Germplasm Bank (NCGRC-2020-18).
张兴伟, E-mail: zhangxingwei@caas.cn; 杨爱国, E-mail: yangaiguo@caas.cn
**同等贡献(Contributed equally to this work)
王琰琰, E-mail: wangyy101@163.com; 王俊, E-mail: 599187325@qq.com
2020-08-11;
2020-12-01;
2021-01-12.
URL: https://kns.cnki.net/kcms/detail/11.1809.S.20210111.1143.002.html
