小麦 BES1转录因子全基因组鉴定与分析
2021-04-26王书平张迎新尹军良马东方方正武
卢 晨,李 寒,王书平,2,张迎新,2,尹军良,2,马东方,2,方正武,2
(1.长江大学 农学院,主要粮食作物产业化湖北省协同创新中心,湖北荆州 434025;2.西北农林科技大学,旱区作物逆境生物学国家重点实验室,陕西杨陵 712100)
植物特异性转录因子(BRI1-EMS-Suppressor,BES1)是油菜素内酯(Brassinolide,BR)信号途径中的关键转录因子,对调节植物的生长发育和逆境胁迫具有非常重要的作用。植物经常遭受一系列生物和非生物胁迫,导致产量和质量下降。转录因子通过激活或抑制靶基因的转录,从而在植物生长发育和应激反应调控中发挥重要作用[1]。BES1是BRs信号通路的重要转录因子,通过激活下游基因的转录调控BRs靶基因的表达,最终调节植物的生长、发育和抗逆能力[2-4]。油菜素内酯(Brassinosteroids,BRs)是一类重要的植物内源激素,在提高作物的抗逆性以及调节植物衰老等生理功能方面发挥了重要作用,被国际上誉为第六激素[2]。已有研究结果表明,在BRs信号转导过程中,转录因子的磷酸化、去磷酸化和蛋白激酶的磷酸化是BRs调控植物生长发育以及提高作物抗逆性的内在生化机制[3]。
拟南芥的BR信号通路中,BES1和BZR1(Brassinazole Resistant 1)在细胞核内参与芥子油苷的生物合成[3]。在拟南芥主根维管束原组织中,BES1可以调节BR受体BRL3。Salazar等[5]研究发现,正向调节因子BES1在BRI1和BIN2的下游起作用,类固醇受体BRL3处理可以增加BES1基因表达水平和其核积累。相反,BES1蛋白表达受到BIN2激酶的负调控。基因突变的BES1蛋白在BES1-D中稳定且高水平积累[6]。BR信号通路中BES1的作用机理研究对于深入研究植物调控具有很重要的意义[3]。虽然目前对小麦(TriticumaestivumL.)中大多数转录因子的研究较多,但缺乏对BES1转录因子家族的系统研究。为全面解析小麦BES1转录因子家族信息,本研究拟通过构建系统发育树、蛋白质特征、基因结构、启动子、染色体分布、转录组及同源性分析,为利用小麦BES1基因奠定基础。
1 材料与方法
1.1 普通小麦、乌拉尔图小麦、野生二粒麦和粗山羊草BES1蛋白序列的检索与鉴定
乌拉尔图小麦T.urartu(v1.43),野生二粒麦T.dicoccoides(v1.0.43)和粗山羊草Ae.tauschii(v4.0.43)基因组数据从EnsemblPlants(http://plants.ensembl.org/index.html)数据库下载。普通小麦T.aestivum(IWGSC v1.1)来源于小麦研究联盟IWGSC(https://wheat-urgi.versailles.inra.fr/Seq-Repository/Assemblies)[7]。从HMMER 3.0下载pfam域PF05687(BES1_N)序列;搜集已鉴定的拟南芥中的8个BES1(AtBES1,TAIR10,http://www.arabidopsis.org/index.jsp)[8],玉米中的11个BES1(ZmBES1,maizeGDB,https://www.maizegdb.org/)[9]和水稻中的6个BES1(OsBES1,RGAP,http://rice.plantbiology.msu.edu/index.shtml)[10]。以上述获得的序列作为查询序列,设置阈值E<10-10,BLASTp(Protein-protein Basic Local Alignment Search Tool)搜索4个小麦蛋白质数据库,合并结果并删除冗余序列。利用Pfam(http://pfam.sanger.ac.uk/search)鉴定并删除不含有PF05687(BES1_N)结构域的序列,最终确定15个小麦BES1基因家族成员。
1.2 序列比对和系统进化树的构建
利用ClustalW2[11]将所有BES1基因的氨基酸序列对齐,然后利用MEGA 7.0最大似然法Maximum Likelihood(ML)生成系统发育树[12-13]。通过互动生命之树网站(IToL,version 3.2.317,http://itol.embl.de)绘制并美化系统发育树。
1.3 TaBES1的染色体定位
从基因组注释信息GFF3文件中提取包含各基因染色体起始位置信息的TaBES1的基因注释文件。然后通过MapInspect软件绘制物理图谱[14]。
1.4 预测TaBES1蛋白的特性
使用蛋白质分析工具ExPASy Server10(SIB Bioinformatics Resource Portal,https://prosite.expasy.org/PS50011)[15]预测TaBES1蛋白质长度,分子质量(MW),等电点(pI),稳定性和亲水性的均值(GRAVY)等特征。用SignalP 4.1 (http://www.cbs.dtu.dk/services/SignalP/)预测信号肽的长度,并通过网站WoLF PSORT(http://www.csbio.sjtu.edu.cn/bioinf/plant-multi/)和Plant-mPLoc(https://wolfpsort.hgc.jp/)进行亚细胞定位预测。
1.5 TaBES1基序(Motifs)、基因结构和启动子分析
使用MEME(v4.9.1,http://meme-suite.org/index.html)[16]和Smart Motif(http://smart.embl-heidelberg.de/)[17]搜索工具鉴定保守的TaBES1蛋白基序。以已知的AtBES1、OsBES1和ZmBES1蛋白序列为参考序列。TaBES1保守蛋白基序通过以下标准识别:(1)每条序列可以包含位置不重叠的多个基序;(2)最多20个不同的基序;(3)基序长度为6~50 aa。TBtools软件展示保守基序图。根据TaBES1基因组注释信息,使用GSDS 2.0(http://gsds.cbi.pku.edu.cn/index.php)[18-19]绘制基因(外显子-内含子)结构。为鉴定小麦BES1基因启动子序列中的顺式元件,从小麦数据库(IWGSC v1.1)中提取15个TaBES1基因上游序列1 500 bp,使用PlantCARE(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)标识启动子序列中的cis-elements。然后,用R软件包的“Pheatmap”展示预测结果。
1.6 TaBES1的多条件转录组分析
旱区作物逆境生物学国家重点实验室前期从NCBI的SRA数据库收集小麦多条件转录组分析的原始RNA-seq,通过sratoolkit子例程fastq-dump,将获得的SRA格式数据转换为fastq格式,通过Trimmomatic过滤低质量数据,通过Trinity(v2.0.6)进行从头组装,通过TransDecoder(v5.3.0)进行开放阅读框预测,最后通过FPKM计算转录本表达水平,所有程序均使用默认参数进行计算。基因表达水平通过片段按每百万碱基对外显子每千克碱基数(FPKM)模型进行归一化[20]。然后利用R软件包pheatmap根据FPKM值绘制热图。
1.7 4 种物种同源基因的鉴定
用互相BLASTp(阈值E<10-10,匹配度>80%)鉴定普通小麦,乌拉尔图小麦,野生二粒麦和粗山羊BES1的同源关系。利用R软件包“circlize”绘制同源关系图谱。直系同源的最原始定义为:两个不同物种中的两个基因,起源于这两个物种最后一个共同祖先的同一个基因。旁系同源被定义为:从一个基因组中复制衍生出来的基因[21]。
2 结果与分析
2.1 小麦基因组 BES1的鉴定及系统发育分析
由图1可知,通过25个(8个AtBES1、11个ZmBES1和6个OsBES1)已知BES1蛋白序列,最终确定了15个小麦BES1基因家族成员。pfam(PF05687BES1-N)和具有核心基序(E<10-5)的局部BLASTP进一步证实候选序列,去除没有BES1特异性蛋白活性区的序列。最后,利用MEGA 7.0最大似然(ML)建立系统发育树(图1)。结果表明,BES1可分为四大类(Group a、Group b、Group c、Group d),TaBES1在Group b中没有分布。
2.2 TaBES蛋白序列比对和特征分析
使用DNAMAN比对15个TaBES1蛋白序列(图2)。利用网站WebLogo(http://weblogo.berkeley.edu/logo.cgi)制作出TaBES1家族高保守区段BES1_N的比对结果(图3)。由表1可知TaBES1长度的平均值为312 aa,分布范围178~359 aa;分子质量平均值为33.08 ku;分布范围19.27~37.86 ku,等电点的平均值为8.66,分布范围8.13~9.4,为碱性蛋白质;不稳定指数平均值为61.62,分布范围50.78~ 69.99,属于不稳定蛋白质(指数大于40);脂肪族指数平均值为55.92,分布范围48.3~ 71.84;亲水性的平均值-0.59,分布范围-0.751~0.482,基本小于0,说明它们都是亲水性蛋白质。多网站预测亚细胞定位结果显示所有的TaBES1蛋白位于细胞 核内。
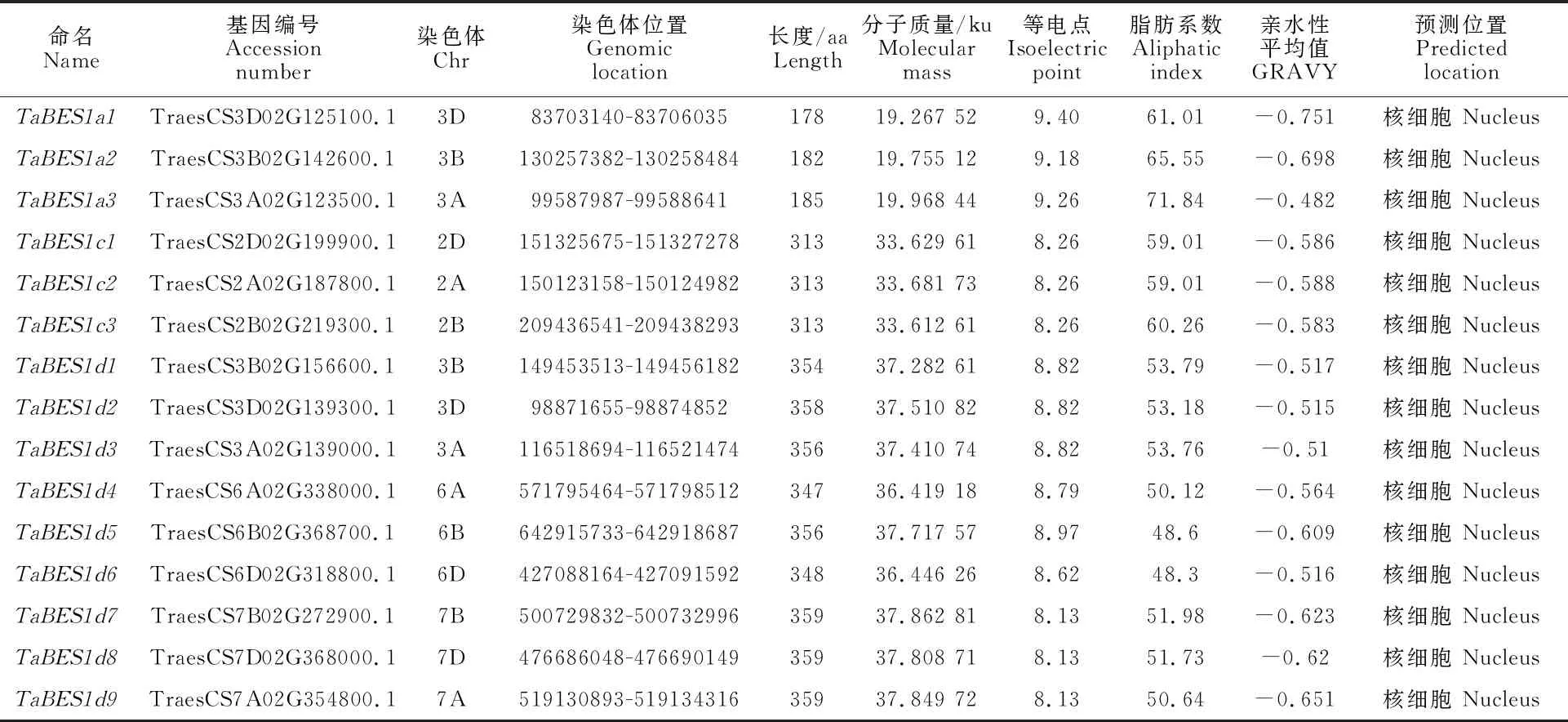
表1 小麦BES1家族的蛋白质特征及染色体位置Table 1 Protein characteristics and chromosomal location of wheat BES1 family
2.3 TaBES1 motif与基因结构分析、染色体定位和基因重复事件
外显子-内含子结构显示:几乎所有的TaBES1都含有一个内含子,大部分TaBES1都在序列两端具有UTR非编码区,仅TaBES1a3两端不具有UTR,TaBES1d5的5′端不具有UTR区。用MAST在线软件查询TaBES1蛋白序列中的20个保守motif,如图4所示,小麦BES1家族motif分析(b)和基因结构(c)高度的相似性说明TaBES1家族的高保守性。
由图5可知,15个TaBES1基因均匀地、相似度极高地分布在小麦A、B和D染色体上。TaBES1c2、TaBES1c3和TaBES1c1基因为三联体,分别分布在2A、2B和2D染色体的相似位置[21]。TaBES1a3、TaBES1a2和TaBES1a1为三联体,分布在3A、3B和3D染色体相似位置。TaBES1d3、TaBES1d2和TaBES1d1为三联体,TaBES1d4、TaBES1d5和TaBES1d6为三联体,TaBES1d9、TaBES1d7和TaBES1d8为三联体。
在小麦BES1基因家族中共找到13组基因重复,TaBES1a2与TaBES1a1、TaBES1a3与TaBES1a1、TaBES1a3与TaBES1a2、TaBES1c3与TaBES1c1、TaBES1c2与TaBES1c1、TaBES1c3与TaBES1c2、TaBES1d2与TaBES1d1、TaBES1d3与TaBES1d1、TaBES1d3与TaBES1d2、TaBES1d6与TaBES1d4、TaBES1d8与TaBES1d7、TaBES1d9与TaBES1d7、TaBES1d9与TaBES1d8(图5),这13组基因复制都是片段重复,在小麦BES1基因家族中未发现串联重复。
2.4 TaBES1的转录组学分析
从NCBI网站下载并整理的转录组数据分为三类,包括生长发育(图6)、非生物胁迫(图7)和生物胁迫(图8)。由图6可知,TaBES1a1、TaBES1a2、TaBES1a3、TaBES1d4、TaBES1d5和TaBES1d6基本不表达,所以其功能可能与生长发育无关。TaBES1d3、TaBES1d2和TaBES1d1基因在苗期、三叶期、分蘖期、孕穗期、抽穗期、开花期、结实期都有较高的表达量,推测其功能可能与生长发育有关。TaBES1c1、TaBES1c2、TaBES1c3、TaBES1d7、TaBES1d8和TaBES1d9在胚根苗期、叶鞘根顶端三叶期、根分蘖期、颖片孕穗期、抽穗期和根秧苗期高度表达,推测其功能可能与根和叶鞘的生长发育有关,具体功能有待进一步验证。
由图7可知,TaBES1d3、TaBES1d2和TaBES1d1基因在聚乙二醇6000、干旱、高温、寒冷胁迫下都有较好的表达,其功能可能与小麦抗逆有关。这一推论与已报道的其他物种BES1家族符合。TaBES1a1、TaBES1a2、TaBES1a3、TaBES1d4、TaBES1d5和TaBES1d6基本不表达,推测其功能可能与抗逆无关。TaBES1c1、TaBES1c2、TaBES1c3、TaBES1d7、TaBES1d8和TaBES1d9在中国春磷、饥饿处理下表达良好。
由图8可知,TaBES1a1、TaBES1a2、TaBES1a3、TaBES1d4、TaBES1d5和TaBES1d6基本不表达,推测其功能可能与抗病无关。TaBES1c1、TaBES1c2、TaBES1c3、TaBES1d1、TaBES1d2和TaBES1d3在条锈病、白粉病和禾谷镰刀菌的处理下与对照相比有明显的差异,其功能可能与抗病有关。条锈病病原菌CYR31处理小麦叶片 24 h~7 d后TaBES1d1和TaBES1d3高度表达,推测其功能可能与条锈相关。
2.5 TaBES1基因启动子顺式元件的研究
由图9可知,12个、11个和7个调控因子分别参与生物/非生物胁迫、生长发育和激素反应。分布最广的顺式元件是脱落酸反应顺式作用元件ABRE,核心启动子元件TATA box和CAAT box都是生长发育相关的元件。其次最常见的顺式元件是茉莉酸甲酯反应元件(CGTCA motif和TGACG motif),几乎分布在所有基因中 (93.3%),另外,光响应元件(g-box,85%)分布也较为广泛。
小麦BES1启动子分析发现,与生长发育有关相关的11个元件中,有3个与启动子启动相关的元件(TATA-box、TCA和CAAT-box),3个顺式作用调节相关的元件(A-box、box S和TCCC-motif),2个与生长素相关元件(W box和WRE3),2个元件与脱落酸相关元件(ABRE和AC-I),最后一个元件CCGTCC motif为分生组织表达相关的顺式作用调控元件。与生物/非生物胁迫相关的12个元件中,其中4个元件与光响应有关(G-Box、GC-motif、STRE和TCT-motif),3个参与低温反应的顺式元件(MBS、MRE和MYB),3个缺氧特异诱导的增强子元件(GT1-motif、I-box和LTR),2个厌氧诱导所必需的顺式作用调控元件(ARE和as-1)。与激素反应相关的7个元件中,其中5个都为茉莉酸甲酯反应元件(CGTCA-motif、chs、CTAG-motif、DRE core、TGACG-motif和TGA-element),1个与水杨酸反应相关元件Sp1。
小麦BES1启动子区域一共含有838 个顺式元件,471(56.2%)个与生长发育有关,201(24%)个与非生物/生物胁迫有关,166(19.8%)个与激素反应有关。
2.6 普通小麦、乌拉尔图小麦、野生二粒麦和粗山羊草中 TaBES1基因的同源性分析
经鉴定,普通小麦、乌拉尔图小麦、野生二粒麦和粗山羊草中分别包含15、13、39和13个TaBES1基因(图10-a)。BES1蛋白可分为3大类(Group a、Group c和Group d),普通小麦(3、3和9),乌拉尔图小麦(8、3和2),野生二粒麦(8、9和22),粗山羊草(6、2和5)。如图10-b所示,共鉴定出90对同源基因。与普通小麦相关的同源物共52对,其中有13对旁系同源物,普通小麦与乌拉尔图小麦,野生二粒麦和粗山羊草分别有9、18和12对直系同源物。与乌拉尔图小麦相关的同源物共18对,没有旁系同源物,乌拉尔图小麦与普通小麦,野生二粒麦和粗山羊草分别有9、6和3对直系同源物。与野生二粒麦相关的同源物共52对,有21对旁系同源物,野生二粒麦与普通小麦,乌拉尔图小麦和粗山羊草分别有18、6和8对直系同源物。与粗山羊草相关的同源物共12对,没有旁系同源物,粗山羊草与普通小麦,乌拉尔图小麦和野生二粒麦分别有12、3和8对直系同源物。
3 讨 论
目前,在149种植物种中发现BES1转录因子,其数目有2~42个不等,其已有30个转录因子被深入研究[22]。吴鹏等[23]在中国白菜中鉴定了15个BES1基因,发现BES1基因只存在于陆生植物中,且高等植物一般比低等植物的转录因子要多,这也表明BES1转录因子在植物的进化过程中可能起重要作用。本研究利用生物信息学的方法对小麦BES1转录因子家族进行全面的研究,共获得的15个TaBES1基因与搜集到的8个AtBES1、11个ZmBES1和6个OsBES1共同构建系统发育树,TaBES1成员分为Group a、Group c和Group d三组(图1)。
通过DNAMAN比对获得的15个TaBES1蛋白序列(图2),显示所有TaBES1蛋白都含有BES1_N结构域。外显子-内含子结构显示TaBES1a3两端不具有UTR,TaBES1d5的5′端不具有UTR区,TaBES1a3和TaBES1d5在后续的转录组分析中基本都不表达,据此推测缺失UTR可能导致TaBES1不表达。接下来的研究发现,小麦BES1基因家族中共找到13组基因重复和同源分析13对旁系同源物数量一致。
关于TaBES1基因的表达模式,不同组的基因表达水平有显著差异。在生长发育、生物/非生物胁迫条件下TaBES1a1、TaBES1a2、TaBES1a3、TaBES1d4、TaBES1d5和TaBES1d6,基本不表达,推测其功能可能与生长发育和非生物胁迫无关。TaBES1c1、TaBES1c2和TaBES1c3基因的表达水平在生物胁迫下表达较好,推测其功能可能与胁迫有关。TaBES1d3、TaBES1d2和TaBES1d1在生长发育、非生物胁迫和生物胁迫下都高度表达,推测其功能与生长发育、生物/非生物胁迫相关。
异源六倍体小麦是一种古老的多倍体植物,至少经历两轮全基因组复制(WGD)事件,这导致小麦基因组包含超过85%的重复基因[24]。重复基因的存在可以通过新功能化,亚功能化和非功能化为基因进化提供更多机会[25]。因此,有必要检测TaBES1的基因重复,进而初步分析出其进化关系。普通小麦相关的同源物性共52对,有13对旁系同源物(25%)与小麦LSD基因家族中13组基因重复数量一致。小麦与乌拉尔图小麦、野生二粒麦和粗山羊草的直系同源物分别有9对(13%)、18对(35%)和12对(32%),共39对(75%)。由此推测,根普通小麦BES1基因的进化关系如下,小部分(13对,25%)来源于自身的进化,大部分(39对 75%)来源于3种亚基因组 供体。
