生物信息学软件预测猪氨基肽酶N 功能与结构
2021-04-22曹金山魏战勇
贾 燕,曹金山,魏战勇
(1.内蒙古农业大学兽医学院,内蒙古 呼和浩特 010018;2.河南农业大学动物医学院,河南 郑州 450046)
氨基肽酶N(aminopeptidase N,APN)是一种大小为150 kDa 且高度糖基化的膜结合Ⅱ型锌金属跨膜蛋白酶,主要分布在肠道上皮绒毛细胞中。APN 生理功能为消化肽链, 使多肽在小肠中消化, 并与细胞运动及多种冠状病毒的黏附作用有关[1-2]。 猪氨基肽酶N(porcine aminopeptidase N,pAPN)由963 个氨基酸编码,在多种上皮细胞和组织中分布, 尤其在小肠刷状缘高效表达[3-4]。pAPN 在胰蛋白酶的作用下可分解为2 个亚基,分别是95 kDa 的N 端和50 kDa 的C 端[2]。 有研究表明pAPN 可能是多种猪肠道冠状病毒在感染宿主时的侵入受体。 猪传染性胃肠炎病毒(transmissible gastroenteritis virus,TGEV) 是最先被鉴定出以pAPN 作为关键性受体的猪肠道冠状病毒,并以Ser-506 至Ile-728 位氨基酸与pAPN 的第717~813 位氨基酸在感染过程中进行特异性结合[5-6]。 猪流行性腹泻病毒(porcine epidemic diarrhea virus,PEDV) 与TGEV 同属α-冠状病毒属,有学者证明pAPN 与PEDV S1 的C 末端可以相互作用, 也有较多研究显示pAPN 与PEDV 没有直接关系[7-9]。 猪德尔塔冠状病毒 (porcine delta coronavirus,PDCoV) 是引起猪肠道疾病的丁型冠状病毒,其S1 亚基结构域B 与pAPN 存在相互作用[10]。 猪肠道甲型冠状病毒(swine enteric alphacoronavirus,SeACoV)是近年发现的猪肠道冠状病毒, 已有研究报道猪肠道甲型冠状病毒不以常见的其他猪肠道冠状病毒的受体为功能性受体,但也没有明确指出使用哪种受体侵入宿主细胞[4]。APN 在不同物种间保守性较高, 不同冠状病毒利用APN 作为主要感染性受体或者辅助功能性受体,可能更便于跨种属传播。还有一些冠状病毒可能使用APN 作为受体,也可能以多种方式感染宿主,感染机制尚不清楚。该研究利用多种生物信息学软件对pAPN 的生物学功能及结构特征进行预测分析。 为深入研究pAPN 生物学过程及特异性功能域提供背景知识, 寻找pAPN 与相关病毒进行互作的结合位点。
1 材料和方法
1.1 基因序列及氨基酸序列
通过NCBI 数据库获取pAPN 核苷酸数据(ID:HQ824547);从Uniprot 数据库获得pAPN 蛋白氨基酸序列及protein accession:ADX53333.1。
1.2 生物信息学分析
利用pAPN 蛋白序列进行以下生物学分析,利 用ProtParam(https://web.expasy.org/protparam/)分析pAPN 蛋白质理化性质; 利用ExPASy-ProtScale (https://web.expasy.org/protscale/) 分 析pAPN 蛋白质亲疏水性; 利用SingalP 4.0(http://www.cbs.dtu.dk/services/SignalP-4.0/) 预 测pAPN蛋白质信号肽序列; 利用TMHMM Server v.2.0(http://www.cbs.dtu.dk/services/TMHMM/) 分 析pAPN 蛋白质跨膜区结构;通过NetNGlyc 4.0 Serve(http://www.cbs.dtu.dk/services/NetNGlyc/) 预 测pAPN 蛋白质N 型糖基化位点; 使用Predicting Antigenic Peptides (http://imed.med.ucm.es/Tools/antigenic.pl) 预测pAPN 蛋白质抗原表位; 利用SMART (http://smart.embl-heidelberg.de/) 预 测pAPN 蛋白质结构域; 利用Python molecule(Py-MOL)观察pAPN 蛋白质空间三维结构。
2 结果
2.1 pAPN 蛋白理化性质分析
将pAPN 氨基酸序列(ADX53333.1) 上传到ProtParam 在线软件分析框, 点击计算参数(compute parameters)。 pAPN 蛋白可编码963 个氨基酸, 分子量和等电点分别为108 885.20 Da 和5.09。 该蛋白质包含106 个带负电荷的残基和73个带正电荷的残基;pAPN 蛋白的不稳定系数为37.14, 脂肪族氨基酸指数为87.51, 平均疏水性为-0.236。 pAPN 蛋白理化性质分析结果见表1。
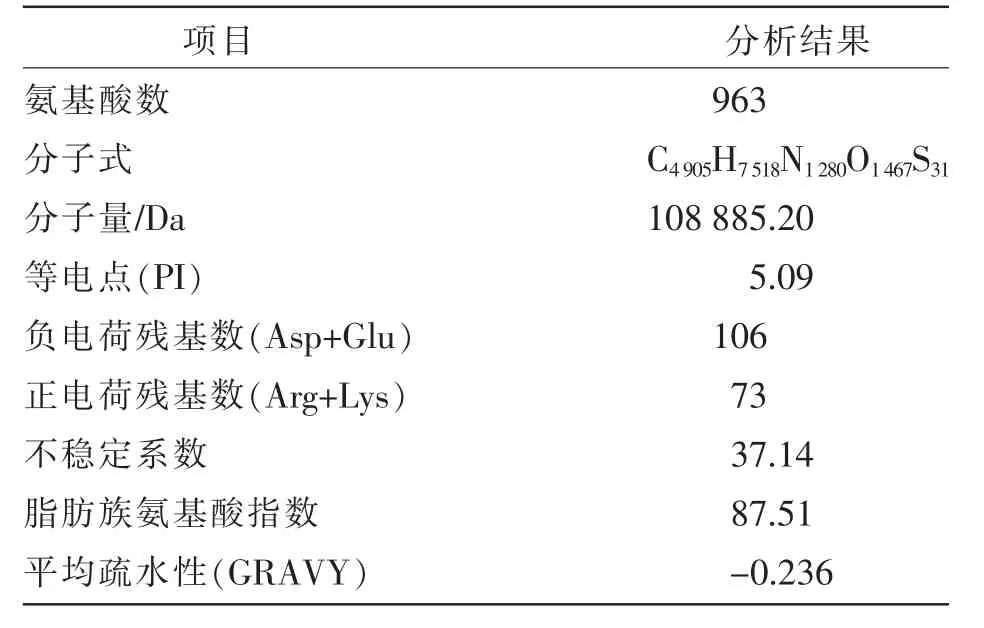
表1 pAPN 蛋白理化性质分析
2.2 pAPN 蛋白疏水性分析
通过ExPASy-ProtScale 软件对pAPN 氨基酸序列进行亲疏水性分析,分析结果依据正值代表疏水性,负值表示亲水性的规律呈现。 ProtScale 查询结果可用数值格式(numerical format)或者图表格式(image in GIF-format)分别展现每个氨基酸的亲疏水性具体数值。 如图1 所示, 利用image in GIF-for mat 说明pAPN 的第963 位氨基酸整体亲疏水性分布情况。 第17 位亮氨酸(Leu)数值最大为2.844,证明该位氨基酸疏水性最强,第39 位赖氨酸(Lys)数值最小为-2.967, 证明该位氨基酸亲水性最强。由此得知,pAPN 属于不稳定的可溶性蛋白质。
2.3 pAPN 蛋白信号肽分析
信号肽是由长度为5~30 个疏水性氨基酸组成,可引导新合成的蛋白质向分泌通路转移,通常位于氨基酸序列的N 端。 信号肽的作用是促进蛋白分泌到胞外,主要存在于分泌蛋白、跨膜蛋白和真核生物细胞器内。在进行重组蛋白表达前,需要对氨基酸序列进行信号肽预测。 运用SingalP 4.0在线软件预测pAPN 蛋白显示不含有信号肽序列,在后续载体构建过程中可以加上一段信号肽,以增加分泌蛋白的表达量,见图2。
2.4 pAPN 蛋白跨膜区分析
TMHMM 是基于马尔可夫模型并结合跨膜区疏水性、螺旋长度、电荷偏倚和膜蛋白拓扑学限制等性质预测跨膜螺旋的程序, 可对跨膜区及膜内外区进行整体预测。 TMHMM Server v.2.0 对pAPN氨基酸序列(ADX53333.1)预测结果如图3 所示,紫色线条表示第35~963 位氨基酸在胞外的概率,红色区域表示第12~34 位氨基酸之间可形成典型的跨膜螺旋区, 表明pAPN 蛋白主要分布于细胞膜,且可能作为病毒侵入细胞的膜受体。
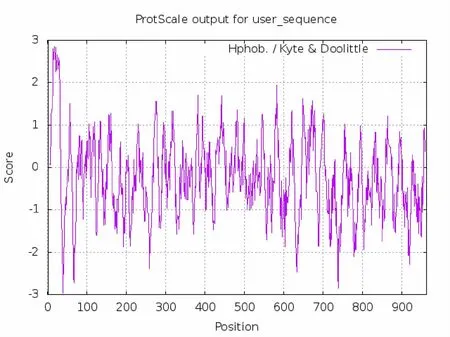
图1 pAPN 蛋白亲疏水性分析

图2 pAPN 蛋白信号肽预测结果
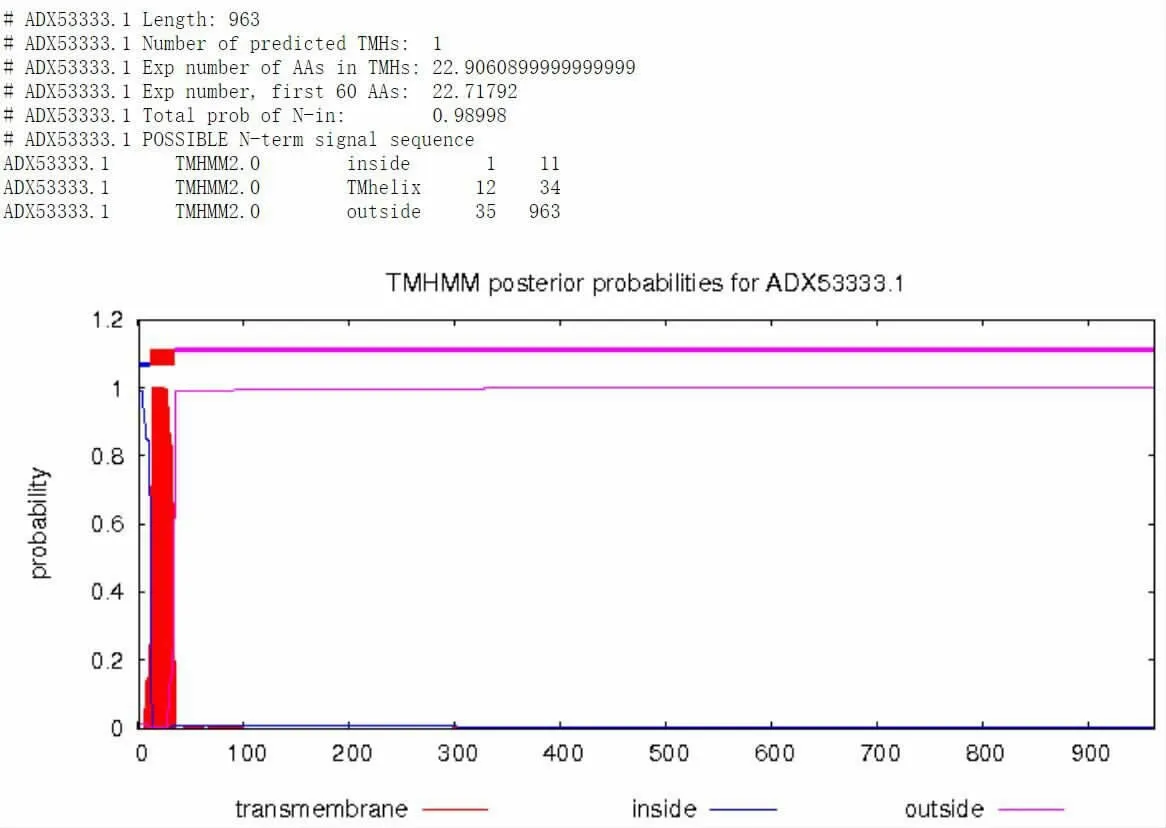
图3 pAPN 蛋白跨膜区结构分析
2.5 预测pAPN 蛋白糖基化位点
糖基化修饰可对蛋白质功能进行调节,包括酰胺链连接的糖链(N-糖基化) 和羟基连接的糖链(O-糖基化)。 利用NetNGlyc 4.0 Serve/NetOGlyc 4.0 Serve 在线软件对pAPN 进行N 型和O 型糖基化修饰位点预测。 预测结果显示,pAPN 含有11 个O-糖基化修饰位点,分别位于第49、50、52、54、55、56、60、64、447、451、452 位氨基酸。 同时pAPN 含有12 个N-糖基化修饰位点, 分别位于第229、237、258、286、314、328、506、556、569、622、646、736位氨基酸。 打分平均值在0.5 以上(见图4、图5)。在线数据库查询和在线软件预测结果基本一致。
2.6 预测pAPN 蛋白抗原表位
蛋白质的特异性取决于抗原决定簇, 即由抗原决定簇的种类、性质、数目和空间构型决定与什么抗体或者受体进行特异性结合, 从而完成病毒的生命活动。 利用predicting antigenic peptides 在线软件对pAPN 进行抗原决定簇预测。 结果显示,pAPN 拥有42 个抗原决定簇(见图6、图7),图7中红色部分即为抗原表位可能出现的区域。
2.7 预测pAPN 蛋白结构域
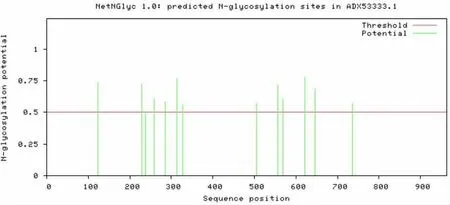
图4 pAPN 蛋白糖基化位点分析
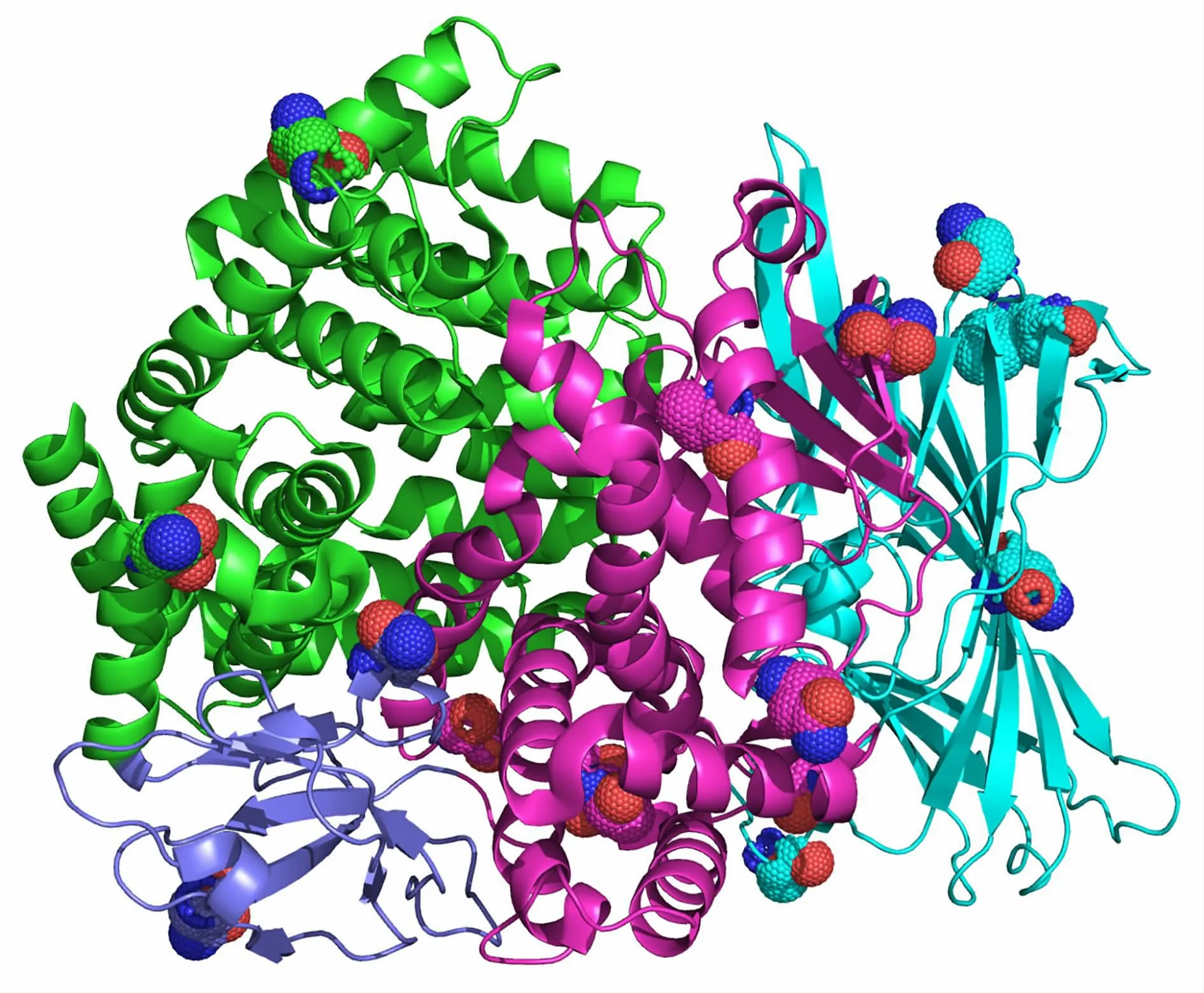
图5 pAPN 蛋白单体糖基化位点
通过简单模块构架搜索工具分析,发现pAPN含有2 个典型的功能结构域, 分别是第291~539位氨基酸高度保守的功能域peptidase_M1 和第616~943 位是高度保守的结构功能域内质网氨基肽酶 (endoplasmic reticulum aminopeptidase1,ERAP1)_C(见图8)。
2.8 pAPN 空间结构
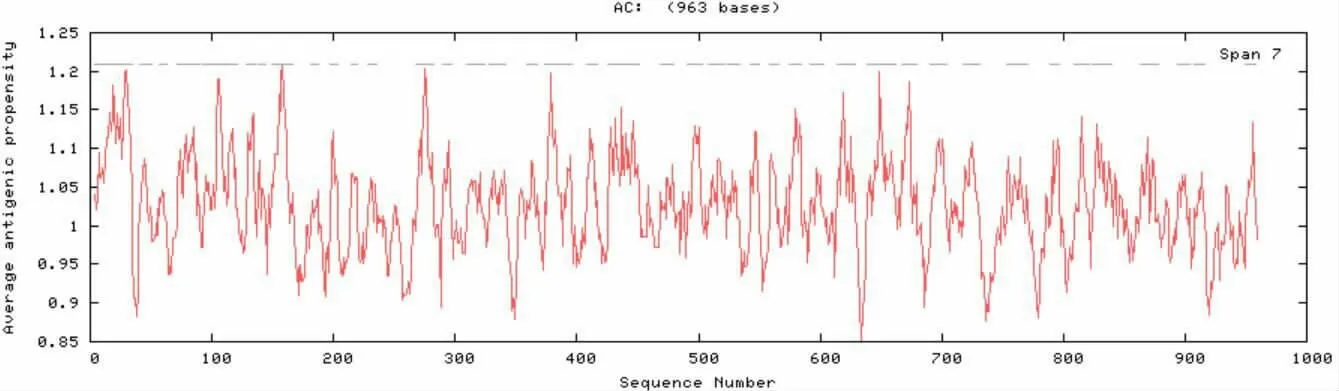
图6 pAPN 蛋白抗原表位分析

图7 pAPN 蛋白单体抗原表位
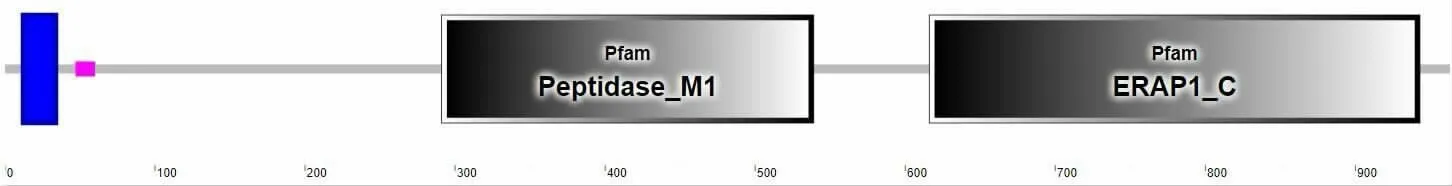
图8 pAPN 蛋白结构域分析

图9 pAPN 蛋白二聚体空间结构
pAPN 胞外区以二聚体形式发挥生物学功能,每个单体都可被分为4 个功能结构域, 分别命名为Ⅰ~Ⅳ。 并通过蛋白质数据库(protein data bank,PDB)下 载pAPN(PDB ID code 5LDS)[10-12],运 用PyMOL 显示pAPN 二聚体的空间结构 (见图9),并对pAPN (protein accession:ADX53333.1) 使用SWISS-MODEL 进行单体空间结构域预测, 预测结果显示与5LDS 相似度高达99.14%。 已有研究报道了pAPN 单体空间三维结构,结构域Ⅰ为35~281 位氨基酸,由15 链β 桶状折叠组成,用豆青色表示。 第282~543 位氨基酸为结构域Ⅱ,包含2 个亚结构域:N 末端亚结构域(残基282~369)由5 链β折叠连接1 个α 螺旋组成;C 末端亚结构域(残基370~543)包含7 个α 螺旋超螺旋,用粉紫色表示。结构域Ⅲ为第544~632 位氨基酸, 采用7 链β 夹心折叠构成,用青紫色表示。 结构域Ⅳ为第633~963 位氨基酸包含16 个α 螺旋超螺旋,用绿色表示[4,12]。 pAPN 在总体结构域排列和结构域折叠方面与其他M1 家族金属酶非常匹配。
3 讨论
目前, 冠状病毒对全球畜牧业的危害日益严重, 对于引起猪肠道疾病的猪肠道冠状病毒是如何利用侵入受体的研究是目前急需解决的基础科学问题。 研究结果中对不同猪肠道冠状病毒的感染性受体的差异, 使得学者们对pAPN 这个经常被用来作为冠状病毒进入宿主易感细胞的主要受体产生较多关注[13-14]。pAPN 又名CD13,是金属蛋白酶家族中一种分子量约为150 kDa 的Ⅱ型跨膜蛋白。 Ⅰ、Ⅱ和Ⅲ型跨膜蛋白均属于单程分子,而Ⅳ型为多程分子。 Ⅰ型通过终止转移锚序列锚定在脂质膜上。Ⅱ型和Ⅲ型以信号锚序列锚定,Ⅱ型以其C 端结构域靶向ER 管腔, 而Ⅲ型的N 端结构域以ER 管腔为靶。 Ⅳ型又分为Ⅳ-A、Ⅳ-B 两型, 分别以其N 端结构域靶向细胞溶质和靶向内腔。pAPN 分布于多种细胞表面尤其在肠道上皮细胞表达水平相对较高。由此推断,该蛋白生物学功能较广泛, 可借助其N 端的螺旋产生跨膜方式进入细胞膜。 在其膜结合形式中参与了细胞内的复杂功能,如肽裂解、免疫细胞趋化。 同时也与多种冠状病毒的黏附与侵袭有关[1-4]。
对冠状病毒功能性受体的研究一直是病毒学领域的热点。 作为冠状病毒常见受体的pAPN 经常被用来做细胞试验,研究病毒感染机制。研究最早的冠状病毒是TGEV, 利用抗细胞受体法证明了pAPN 是TGEV 的感染性受体[5]。与TGEV 同属甲型冠状病毒的PEDV 与宿主受体互作的具体位置以及关键受体的研究有很多, 如Li 等[15]用PEDV 感染pAPN 稳定转染于敲除掉pAPN 的猪睾丸细胞和犬肾上皮连续细胞系(madin-daby canine kidney cells)MDCK 后, 发现pAPN 的有无对PEDV 感染这些细胞系没有影响。 Shirato 等[16]和Cui 等[17]证明了可溶性pAPN 不能中和PEDV 对Vero 细胞的感染,其他手段得出同样的结论。利用PDCoV 棘突蛋白S1 与内源性表达pAPN、过表达pAPN 及敲除pAPN 的细胞进行结合后再感染试验,证明pPAN 可以介导PDCoV 感染宿主细胞[18]。同年Zhu 等[19]、Li 等[15]报道PDCoV S1 可以与pAPN 发生特异性结合的结论。 可见,继续深入研究猪腹泻相关病毒的致病机理为早日攻克猪肠道疾病有重要意义。 作为目前受关注度较高的冠状病毒功能性受体pAPN,对其蛋白结构的精确解析也可为研究其功能提供帮助。
通过生物信息学软件分析得知pAPN 属于不稳定的亲水性蛋白酶且无信号肽,但有文献报道该蛋白含有信号肽[20]。 在第35~963 位氨基酸形成胞外蛋白,第12~34 位氨基酸形成典型的跨膜螺旋区, 表明该蛋白为细胞膜蛋白。 pAPN 拥有23 个糖基化修饰位点、42 个抗原表位。 功能结构域预测显示pAPN 含有peptidase_M1 和ERAP1_C 两个典型的功能结构域,以上生物学分析与 刘 颖 等[21]对pAPN 预 测 结 果 基 本 一 致。 运 用PyMOL 可视化软件显示pAPN 胞外域的空间三维结构其中结构域Ⅰ为35~281 位氨基酸, 由15链β 桶状折叠组成,结构域Ⅱ为282~543 位氨基酸,包含N 末端和C 末端2 个亚结构域,分别以5 链β 折叠连接1 个α 螺旋和7 个α 螺旋超螺旋构成,第544~632 位氨基酸采用7 链β 夹心折叠构成结构域Ⅲ, 结构域Ⅳ为633~963 位氨基酸,包含16 个α 螺旋超螺旋[10-11]。 pAPN 在总体结构域排列和结构域折叠方面与M1 家族其他金属酶非常相似,空间三维结构精确解析可为下一步寻找该蛋白与猪肠道冠状病毒棘突蛋白结合的关键位点提供结构方面素材, 为下一步研究pAPN 功能结构域与猪肠道冠状病毒膜蛋白相互作用提供理论依据。
