山羊PNPLA3 基因克隆及序列分析
2021-04-21苟圣松林亚秋朱江江
苟圣松,林亚秋,王 永,朱江江,3*
(1.西南民族大学生命科学与技术学院,四川成都 610041;2.青藏高原动物遗传资源保护与利用四川省重点实验室,四川成都 610041;3.青藏高原动物遗传资源保护与利用教育部重点实验室,四川成都 610041)
肌内脂肪是影响山羊肉品质的重要因素,筛选并鉴定山羊脂质代谢调控的关键基因是目前山羊分子育种的重要手段。PNPLA3(Patatin Like Phospholipase Domain-Containing 3)是由位于人22 号染色体上的PNPLA3基因所编码的一种非分泌性多功能蛋白,由481 个氨基酸组成,最初被鉴定为一种高脂肪特异性转录物,同时具有甘油三酯脂肪酶和酰基甘油转酰基酶的活性,能够快速地参与营养调节[1],主要参与脂肪的积累和动员[2],被称为脂肪滋养蛋白。PNPLA3基因在人类各组织中均表达,在肝脏和脂肪组织表达丰度最高[3-4]。
已有研究表明,PNPLA3可调节肝脏中糖脂代谢[5]。脂质代谢中许多基因的转录控制是通过固醇调节元件结合蛋白(SREBP)途径进行调控的。PNPLA3是SREBP-1c的靶基因[6],SREBP-1c可通过肝X 受体/类视黄醇X 受体(LXR/RXR)途径的激活进而影响PNPLA3的转录调控。此外,有研究证实PNPLA3可在转录后水平上通过SREBP-1c途径的终产物脂肪酸进行营养调节[7]。全基因组关联研究发现PNPLA3中的1个等位基因(rs738409[G],编码I148M)突变与肝脏脂肪水平升高、肝炎以及肝纤维化密切相关,为非酒精性脂肪肝病(NAFLD)的主要易感因素[8-9]。然而目前PNPLA3在山羊脂质代谢中的研究较少。
简州大耳羊是用努比羊与四川省简阳市当地山羊杂交选育而形成的我国第2 个肉用山羊新品种,具有肉质优良、胆固醇含量低、营养丰富等特点[10-11]。本研究以简州大耳羊为研究对象,利用T-A 克隆技术获得山羊PNPLA3基因序列并进行生物信息学分析,为进一步阐明PNPLA3基因在调控山羊脂质代谢中的作用及分子机制提供基础资料。
1 材料与方法
1.1 实验材料
1.1.1 实验动物与组织采集 于四川省简阳市大哥大牧业有限公司随机挑选3 只1 周岁无遗传相关性的健康简州大耳羊公羊,空腹颈动脉放血致死,无菌采集各山羊背部皮下脂肪组织,用0.1% DEPC 水冲洗干净裹上锡箔纸后装入冻存管,迅速放入液氮内带回实验室,等比例混合后用于提取组织总RNA。
1.1.2 主要试剂 试剂主要包括Trizol 试剂、反转录试剂盒(Thermo 公司,美国)、2×LongTaqPCR Master Mix、琼脂糖、胶回收试剂盒(QIAGEN 公司,德国)、DNA Marker DL2000、loading buffer 6x、pMD-19T(擎科生物科技有限公司,成都)、大肠杆菌感受态细胞DH5α(天根生化科技有限公司,北京)。
1.2 实验方法
1.2.1 组 织RNA 提取、反转录及PCR 扩增采用Trizol 法提取组织总RNA,用核酸蛋白浓度检测仪检测其浓度(ng/µL)及OD 值(OD260/OD280在1.8~2.0符合要求)。按反转录试剂盒说明书将RNA 反转录成cDNA。引物由成都擎科生物科技有限公司合成。根据NCBI 上山羊PNPLA3基因的预测序列(登录号:XM_005709739.3),利用Primer Premier 5 软件设计PCR 克隆引物,PNPLA3-F:5'-CCAACCTGCCGCCGC TAT-3';PNPLA3-R:5'-CCAGCCGAGCCAACGATA C-3',预测PCR 产物片段大小为1 564 bp。PCR 反应体系:2×Long Taq PCR Master Mix 12.5 μL,模板cDNA 1 μL,上、下游引物(10 μmol/L)各1 μL,最后加ddH2O 至25 μL。扩增条件:98℃预变性4 min;98℃变性20 s,68℃退火20 s,72℃延伸90 s,18 个循环;98℃变性20 s,53℃退火20 s,72℃延伸90 s,17 个循环;72℃延伸5 min;PCR 产物用1%琼脂糖凝胶电泳检测正确后,按胶回收试剂盒说明书回收目的片段。
1.2.2 山羊PNPLA3基因克隆及测序 将目的基因与pMD-19T 载体连接,并转化至大肠杆菌感受态细胞DH5α中,37℃培养过夜,挑取阳性单克隆菌落于1.5 mL LB 液体培养基中以37℃、180 r/min 扩大培养3~6 h,经菌液PCR 鉴定正确后送到成都擎科生物科技有限公司测序。
1.2.3 山羊PNPLA3基因序列分析 利用NCBI 网站中的ORF Finder 对所得到的序列进行开放式阅读框预测,获取其氨基酸序列,DNAMAN 进行氨基酸序列同源性对比;用MEGA 5.0 软件构建进化树;利用SignalP 4.1 Server(http://www.cbs.dtu.dk/services/SignalP-4.1/)进行信号肽分析;运用ProtParam(https://web.expasy.org/protparam/)在线分析PNPLA3 蛋白质的理化性质;使用TMHMM2.0(http://www.cbs.dtu.dk/services/TMHMM/)预测蛋白跨膜结构域;应用NetPhos3.1 Server(http://www.cbs.dtu.dk/services/NetPhos/)在 线分析PNPLA3 蛋白磷酸化位点;STRING 交互式数据库(https://string-db.org/cgi/input.pl)进行蛋白相互作用分析;通过SOPMA(https://npsa-prabi.ibcp.fr/cgi-bin/secpred_sopma.pl)在线分析PNPLA3 蛋白的二级结构;SWISSMODEL(https://swissmodel.expasy.org/) 在 线分析PNPLA3 蛋白的三级空间构型。
2 结果
2.1 山羊PNPLA3基因克隆 以简州大耳羊皮下脂肪组织cDNA 为模板用于PCR 扩增。扩增产物经1% 的琼脂糖凝胶电泳检测后得到单一明亮条带,且大小与预期片段相符。经测序获得PNPLA3基因序列1 564 bp(图1),其中包括CDS 区1 344 bp,5'UTR 序列234 bp和3'UTR 序列49 bp,编码447 个氨基酸残基,上传至NCBI 获得登录号:MN643056。
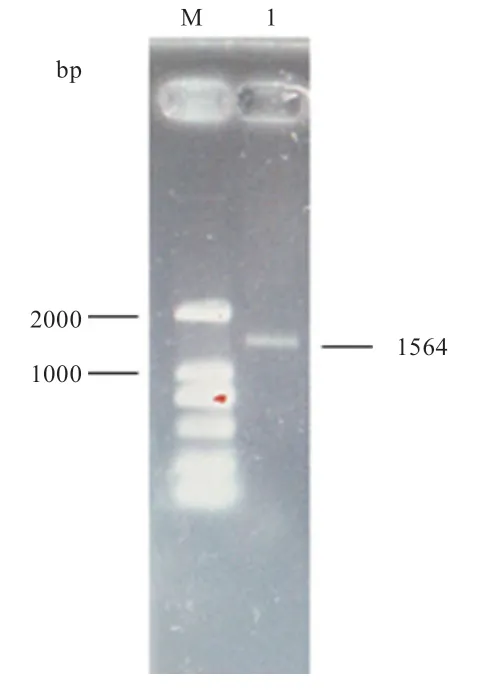
图1 PNPLA3 基因克隆
2.2 PNPLA3 氨基酸同源性分析 利用DNAMAN 将获得的山羊PNPLA3 氨基酸序列与其他物种进行比对发现,山羊PNPLA3 氨基酸序列与绵羊(XM_027968188.1)、牛(XM_024992822.1)、大鼠(NM_001282324.1)、小鼠(NM_054088.3)和人(NM_025225.3)的序列同源性分别为83.89%、81.21%、56.38%、56.15%、58.42%(图2)。构建的氨基酸序列系统进化树(图3)显示,PNPLA3蛋白在各物种进化上相对保守,尤其与绵羊亲缘关系最近,其次为牛、猪、人、小鼠、大鼠,而与原鸡的亲缘关系最远。

图2 PNPLA3 氨基酸序列同源性对比

图3 利用MEGA 5.0 构建山羊PNPLA3 氨基酸序列系统进化树
2.3PNPLA3基因生物信息学分析 ExPASy 在线工具对山羊PNPLA3基因编码的氨基酸序列进行理化性质分析发现,该蛋白分子式为C2230H3485N595O639S23,分子质量为49.592 ku,理论等电点为6.07,不稳定指数为49.40,亲水性总平均值为0.049,说明为不稳定疏水酸性蛋白(图4)。利用SignalP 4.1 Server 进行信号肽分析,结果显示无信号肽序列(图5)。利用SMART 和ScanProsite 软件预测蛋白结构域,发现该蛋白有2 个跨膜结构域,分别为Leu10-Ser30 和Phe43-Leu62。其中Leu10-Lys179 为PNPLA 家族同源结构域(图6)。蛋白质磷酸化是调节和控制蛋白质活力和功能的最基本的机制,利用NetPhos 3.1 软件对PNPLA3 蛋白进行磷酸化位点分析,结果显示该蛋白有18 个丝氨酸、3 个苏氨酸、3 个酪氨酸磷酸化位点(图7)。用STRING数据库检索到PNLIP、PNPLA2、LIPC、PNLIPRP3、LPL、AGK、MGLL 等蛋白可能与PNPLA3 蛋白存在相互作用关系(图8)。山羊PNPLA3 蛋白三级结构预测结果与二级结构预测一致,α螺旋、无规则卷曲、β折叠和β转角分别占比44.07%、39.6%、10.51%、5.82%(图9)。用PSORT Ⅱ进行亚细胞定位,发现该蛋白主要在线粒体(30.4%)、细胞质(30.4%)和高尔基体(13.0%)中发挥生物学作用。
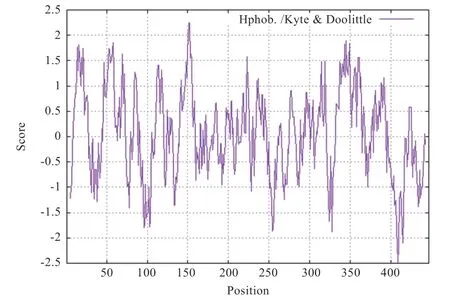
图4 山羊PNPLA3 蛋白质的亲疏水性的预测
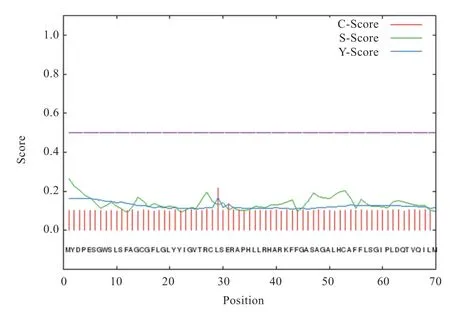
图5 山羊PNPLA3 蛋白的信号肽分析

图6 山羊PNPLA3 蛋白质的跨膜结构域分析

图7 山羊PNPLA3 蛋白的磷酸化位点预测
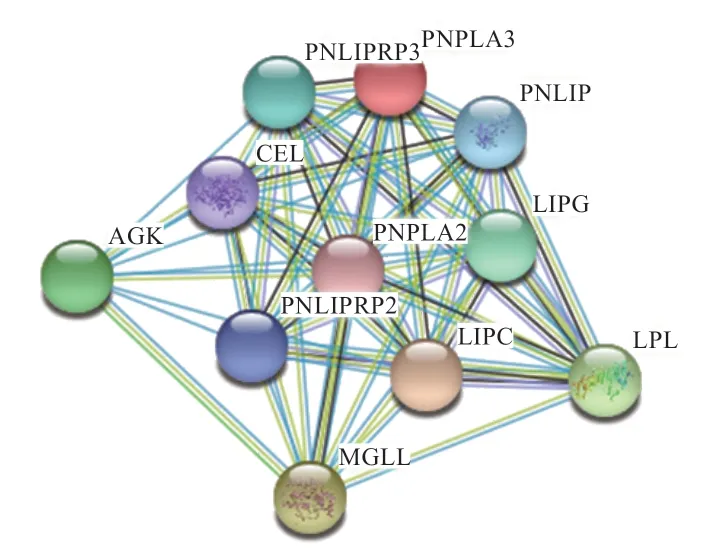
图8 绵羊PNPLA3 蛋白相互作用蛋白预测
3 讨 论

图9 PNPLA3 蛋白结构预测
本实验通过基因克隆技术获得山羊PNPLA3基因序列1 564 bq,其中CDS 区1 344 bp,编码447 个氨基酸,对比不同物种PNPLA3基因CDS 区序列得知,多数变异为碱基的转换或颠换,仅有少数位点发生碱基的插入或缺失。与NCBI 上山羊预测cDNA 序列相比较,山羊PNPLA3基因存在2 个突变位点,第1 149 位核苷酸由胸腺嘧啶突变成为胞嘧啶,第1 249 位发生插入突变,插入了鸟嘌呤碱基。此外,对比山羊和牛的PNPLA3 氨基酸序列发现,山羊PNPLA3 416 位插入了天冬氨酸,这是由于其PNPLA3 编码区序列1 249 bp 处插入了鸟嘌呤碱基,导致终止密码子TGA 后移21 bp,多编码7 个氨基酸。构建的系统进化树表明,山羊与绵羊的亲缘关系最近。
PNPLA3编码区序列分析显示,人和小鼠PNPLA3序列同源性为68%。人PNPLA3编码481 个氨基酸,小鼠PNPLA3编码384 个氨基酸。在小鼠中,PNPLA3在白色和棕色脂肪细胞中高度表达,在肝脏中适度表达[5]。然而,在人体中,PNPLA3在肝脏中的表达是脂肪组织的10 倍[12]。研究表明,人PNPLA3 I148M 基因突变与肝脏甘油三酯含量增加密切相关[12]。然而,含有PNPLA3 I148 M 突变基因的小鼠肝脏中甘油三酯含量与野生型小鼠无显著差异。PNPLA3基因敲除的小鼠并不表现肝脏甘油三酯积聚,因此不会发生脂肪变性,且过度表达人PNPLA3基因的小鼠具有与野生型小鼠相似的甘油三酯水平。有报道认为PNPLA3 在二级结构的基础上具有跨膜结构域,且与内质网和脂滴密切相关[13],这与本实验的分析结果有异曲同工之处。PNPLA3 蛋白三级结构预测结果显示,山羊、绵羊、牛、大鼠、小鼠的蛋白三级结构具有高度相似性,而人PNPLA3 蛋白三级结构与上述物种有着明显差异,这可能是导致PNPLA3 蛋白在人和小鼠中表现出功能差异的直接原因,目前暂未见山羊、绵羊、牛的相关报道。STRING 数据库检索到PNPLA3 蛋白与PNPLA2、LPL、PNLIP、LIPC 蛋白存在相互作用。有研究表明,PNPLA2 具有三酰甘油水解酶和酰基甘油转酰酶活性,LPL 在甘油三酯代谢中起重要调控作用,PNLIP 具有甘油三酯水解酶的活性,LIPC 也与脂肪代谢密切相关,且PNPLA2、LPL 已被证实与山羊肌肉生长发育,脂肪沉积能力相关[13-15]。因此推测PNPLA3可能与山羊脂质代谢调控密切相关。
目前,PNPLA3基因多态性与非酒精性脂肪肝病(NAFLD)的关联性研究是一个热点与难点。PNPLA3中的一个等位基因(rs738409[G],编码I148M)突变使得148 位氨基酸异亮氨酸转化为蛋氨酸,蛋氨酸将阻止底物结合到有催化活性的47 位氨基酸丝氨酸上,从而使其丧失脂肪酰基水解活性[16],进而导致肝脏脂肪变性和代谢紊乱。相关研究也表明PNPLA3-I148M能通过限制甘油三酯水解来促进甘油三酯的积累进而增加肝脏脂肪蓄积[17]。近年来,越来越多的证据表明PNPLA3-I148M 多态性在肝癌发病中具有独立的倾向性,肝细胞癌患者存在严重的肝纤维化[18]。同时,PNPLA3突变在肥胖人群中的作用被放大,使之对肝癌的易感性增高[19]。因此,PNPLA3rs738409 可用于更好地了解易感人群患肝病的风险,并做出早期的预防策略。已有研究表明,SREBP-1c和ChREBP2个转录因子参与调控PNPLA3基因的表达。ChREBP是葡萄糖代谢过程中的重要调控因子,葡萄糖摄入能上调人肝脏细胞PNPLA3mRNA 的表达,且ChREBP作为关键的转录调节因子参与介导了这种上调[20]。此外,PNPLA3已被报道为SREBP-1c的靶基因,SREBP-1c可通过结合到大鼠PNPLA3启动子区进而激活PNPLA3基因转录。敲除小鼠SREBP-1c基因,饱食后肝脏中甘油三酯和脂肪酸的合成减少,而敲低小鼠PNPLA3能部分抑制通过SREBP-1c 途径引起的甘油三酯的聚集[21-23]。目前暂未见PNPLA3在山羊肌肉生长发育中分子调控机制的相关报道,本实验为此提供了基础资料。
4 结 论
本研究克隆了山羊PNPLA3基因,获得其序列1 564 bp,其中CDS 区1 344 bp,编码447 个氨基酸残基,GenBank 登录号为MN 643056;山羊PNPLA3 氨基酸序列与绵羊、牛、大鼠、小鼠和人的氨基酸序列同源性分别达到83.89%、81.21%、56.38%、56.15%、58.42%;与绵羊亲缘关系最近,其次为牛、猪、人、小鼠、大鼠、原鸡,并预测与PNPLA2、LPL 等脂质代谢调控相关蛋白具有相互作用。
