陇东绒山羊KRTAP2-1基因鉴定及其对羊绒性状影响的研究
2021-04-19赵孟丽郝志云王建清沈继源宋宜泽王继卿
孙 凯 赵孟丽 郝志云 王建清 沈继源 宋宜泽 柯 娜 王继卿
(甘肃农业大学动物科学技术学院/甘肃省草食动物生物技术重点实验室,甘肃 兰州 730070)
山羊绒纤维由次级毛囊产生,具有细而柔软、轻而保暖、绒面柔滑丰满、弹性和吸湿性好、风格独特和穿着舒适等优点,其价格也远高于绵羊毛和马海毛,在业界被称为“软黄金”和“纤维钻石”[1-3]。产绒量和平均纤维直径是羊绒最重要的经济性状,直接决定了山羊绒的经济价值。角蛋白(keratins,Ks)和角蛋白关联蛋白(keratin-associated proteins,KAPs; 其编码基因标识为KRTAPs)是羊绒纤维的主要结构蛋白,约占其总重量的90%。前者形成角蛋白中间丝(keratin intermediate filaments,KIFs),后者交联形成KIFs 的基质。Ks 和KAPs 直接决定了羊绒纤维的理化特性[4]。KAPs 是一类复杂的蛋白质,最明显的特点是含有大量的半胱氨酸(cysteine,Cys)或甘氨酸(glycine,Gly)和酪氨酸(tyrosine,Tyr)[5]。根据氨基酸成分和含量,KAPs 可以分为3 类:高甘氨酸/酪氨酸KAPs(HGTKAPs,含有35 ~60 mol%的Gly 和Tyr)、高硫KAPs(HS-KAPs,含有≤30 mol%的Cys)和超高KAPs(UHS-KAPs,含有>30 mol%的Cys)[6]。根据序列相似性,可将KAPs 进一步分为不同的家族。目前,已经在哺乳动物中发现了来自27 个基因家族的100 多个KRTAPs基因[7]。然而,在山羊中只发现了来自11 个家族的14 个KRTAPs基因[1,8-10]。山羊基因组中还有大量的KRTAPs基因有待进一步鉴定。
已有研究表明,KRTAPs基因的核苷酸序列变异与山羊的产绒性状有很强的相关性。王杰等[11]在高原型藏山羊上发现了3 个基因型,其中BB基因型个体的羊绒纤维直径比AB基因型个体小0.9 μm,比AA基因型个体小0.37 μm(P<0.05)。Wang 等[1]发现存在KRTAP20-2 等位基因A的陇东绒山羊有较大的产绒量(P<0.001),存在等位基因B的个体有较小的产绒量和纤维自然长度(P<0.05),推测KRTAP20-2 基因可以作为陇东绒山羊产绒量和纤维长度的有效分子标记。Fang 等[12]研究表明,新疆绒山羊KRTAP13-1 中GT基因型个体的绒层厚度比GG基因型个体增加0.393 cm(P<0.05)。上述结果表明,研究山羊KRTAPs基因的核苷酸序列变异及其与产绒性状的相关性对山羊生产实践有重要意义。KRTAPs基因一般只含1 个不到1 000 bp 的外显子,无内含子。人类基因组中已鉴定出KRTAP2 基因家族,由KRTAP2-1、KRTAP2-2、KRTAP2-3、KRTAP2-4 和KRTAP2-5P(假基因)5 个成员组成[13],而鲜有关于山羊KRTAP2 基因家族的研究报道。本研究以陇东绒山羊为研究对象,利用比较基因组、生物信息学、聚合酶链反应-单链构象多态性( polymerase chain reaction-single stranded conformation polymorphism,PCR-SSCP)和测序等方法,在山羊基因组中鉴定KRTAP2-1 基因,研究该基因的单核苷酸多态性 ( single nucleotide polymorphisms,SNPs)和氨基酸序列特征,最后分析KRTAP2-1 基因的核苷酸序列变异对陇东绒山羊羊绒性状的影响,旨在为改良羊绒性状提供有效的分子遗传标记。
1 材料与方法
1.1 试验材料
在甘肃省庆阳市环县钰生绒山羊繁育有限责任公司,以8 只无血缘关系的陇东绒山羊作为父本,采用人工授精技术对陇东绒山羊母羊进行配种。在后代中随机选择249 只发育正常的陇东绒山羊,待其周岁时,现场测定产绒量和绒层高度。在背中线处采集羊绒样品,带回实验室测定羊绒平均纤维直径(mean fibre diameter,MFD)。对于上述249 只个体,每只羊颈静脉采血8 mL,加入酸性柠檬酸葡萄糖(citric acid dextrose,ACD)抗凝,-20℃冻存,采用苯酚-氯仿抽提法[14]提取基因组DNA。
1.2 山羊基因组序列分析
根据人类KRTAP2-1 基因的编码区序列(GenBank 登录号:NM001123387.1),利用GenBank 的BLAST 软件在山羊基因组GCF_001704415.1(www.ncbi.nlm.nih.gov/assembly/GCF_001704415.1)进行同源性搜索。在比对结果中,与人类KRTAP2-1 基因同源性最高的DNA 序列被假定为山羊KRTAP2-1 基因的序列。
1.3 KRTAP2-1 基因的引物设计和PCR 扩增
根据上述序列,设计引物,特异性扩增山羊KRTAP2-1 基因的假定序列,目的片段长度为565 bp。上游引物:5′-AACAAGGAATGGCATGAGTC-3′,下游引物:5′-G TTGCTTTATAGGAAAGTGGG-3′,由北京奥科鼎盛生物科技有限公司合成。PCR 反应总体系为20 μL,其中Taq 预混酶10 μL(上海华大基因科技有限公司),ddH2O 7.6 μL,0.25 μmol·L-1上下游引物各0.8 μL,约50 ng·μL-1的基因组DNA 模板0.8 μL。PCR扩增条件:94℃预变性5 min;94℃变性30 s,64℃退火30 s,72℃延伸30 s,共35 个循环;最后72℃延伸7 min,4℃保存PCR 产物。
1.4 山羊KRTAP2-1 基因的SSCP 分析
取1 μL PCR 扩增产物加入8 μL 变性上样缓冲液[98%去离子甲酰胺、10 mmol·L-1乙二胺四乙酸二钠(ethylene diamine tetraacetic acid,EDTA)、0.025%溴酚蓝和0.025%二甲苯氰],105℃变性10 min 后,立刻放入冰水混合物中,快速上样于14%的非变性聚丙烯酰胺凝胶(丙烯酰胺∶甲叉=37.5 ∶1)中,在0.5×Tris-硼酸缓冲液中,先于350 V、35℃条件下电泳40 min,最后在170 V、35℃条件下电泳23 h。电泳完成后,根据Byun 等[15]的方法对聚丙烯酰胺凝胶进行染色。
1.5 KRTAP2-1 等位基因的测序
通过SSCP 分析来判断个体基因型是纯合子还是杂合子。如个体基因型为纯合子,直接用PCR 扩增产物进行测序;如果个体基因型全部为杂合子(无纯合子),则采用Gong 等[16]的方法进行切胶测序。测序在西安奥科鼎盛生物科技有限公司完成。
1.6 数据统计分析
使 用 MEGA (V5. 2. 10,Lynnon BioSoft,Vaudreuil,加拿大)进行DNA 序列比对和系统发育树构建,构建系统发育树时用到的KAPs 序列有:JN012101.1 (gKAP1-4)、NM_001285774 (gKAP3-1)、 NM _ 001285767.1 (gKAP11 - 1)、 AY510115(gKAP13 - 1)、 JX426138 (gKAP13 - 3)、 X01610(sKAP1-1 和sKAP1-4)、HQ897973 (sKAP1-2)、X02925 (sKAP1-3)、P02443 (sKAP2-1)、P02441(sKAP2-3)、P02446 (sKAP3-1)、P02444 (sKAP3-2)、P02445 (sKAP3-3)、HQ595347 (sKAP11-1)、JN377429 (sKAP13-3)、KX817979 (sKAP15-1)、JX112014 (sKAP24-1)、KX644903 (sKAP26-1)、NM_030967.2 (hKAP1-1)、NM_030966.1 (hKAP1-3)、NM_001257305.1 (hKAP1-4)、NM_031957.1 (hKAP1-5)、NM_001123387.1 (hKAP2-1)、NM_033032.2(hKAP2-2)、NM_001165252.1 (hKAP2-3)、NM_033184.3 (hKAP2-4)、NM_031958.1 (hKAP3-1)、NM_031959.2 (hKAP3-2)、NM_033185.2 (hKAP3-3)、NM _ 175858.2 (hKAP11 - 1)、 NM _ 181599.2(hKAP13-1)、NM_181621.3 (hKAP13-2)、NM_181622.1 (hKAP13-3)、NM_181600.1 (hKAP13-4)、NM _ 181623.1 (hKAP15 - 1)、 NM _ 001146182.1(hKAP16-1)、NM_181624.1 (hKAP23-1)、NM_001085455.2 ( hKAP24 - 1)、 NM _ 001128598.1(hKAP25-1)、NM_203405.1 (hKAP26-1)和NM_001077711.1 (hKAP27-1)。
用GenBank 数据库BLAST 软件(http:/ /www.ncbi.nlm.nih.gov/)进行序列同源性比对,用Popgen(V32.0) 计算等位基因频率、 遗传杂合度(heterozygosity,He) 和有效等位基因数(effective number of alleles,Ne),用PIC 软件计算群体多态信息含量(polymorphism information content,PIC),用在线软件NetPhos 3.1 Server (http:/ /www. cbs. dtu. dk/services/NetPhos-3.1/)预测氨基酸序列的磷酸化位点。采用SPSS 20.0 软件的一般线性混合效应模型(general liner mixed-effect models,GLMMs)分析等位基因状态(存在或缺失)对陇东绒山羊羊绒性状的影响。为确定模型分析中的因素,进一步分析出生等级(单羔和双羔)、父本(配种公羊)和性别对249 只陇东绒山羊产绒性状的影响。结果表明,出生等级(单羔和双羔)对产绒性状无显著影响(P>0.05),父本(配种公羊)和性别对羊绒性状有极显著影响(P<0.01)。因此,在模型中,把基因型与父本作为固定效应,把性别作为随机效应。由于所有羊只年龄均相同,且在统一管理条件下饲养,因此在模型中未考虑年龄和环境因素。
2 结果与分析
2.1 山羊KRTAP2-1 基因鉴定
利用人类KRTAP2 - 1 基因的编码区序列(GenBank 登录号:NM001123387.1),用GenBank 中Blast 程序在山羊基因组中搜索,结果在山羊19 号染色体上发现一个399 bp(40927134 ~40927532)的开放阅读框,该序列与人类的KRTAP2-1 基因序列的相似性为97%。在该区域附近有4 个已被鉴定的山羊KRTAPs基因,分别是KRTAP1-3、KRTAP1-4、KRTAP1-1 和KRTAP9-2(图1)。
通过PCR-SSCP 分析发现,从249 只陇东绒山羊中检测到7 个等位基因(分别是A、B、C、D、E、F和G)和11 种基因型(AA、BB、CC、DE、AB、AC、AF、AG、BC、BF和BG)(图2)。7 个等位基因中,等位基因F的序列与山羊基因组完全相同,其他等位基因的核苷酸序列与山羊基因组的序列相似性均超过98%。
根据本研究发现的上述7 条A~G序列所编码的氨基酸序列,以及从人类、绵羊和山羊中鉴定出的HSKAPs 氨基酸序列,构建了系统发育树(图3)。结果表明,7 条氨基酸序列与人类和绵羊的KAP2-1 序列具有较高的同源性,说明这7 条序列是山羊KRTAP2-1的7 个等位基因。
2.2 山羊KRTAP2-1 基因的SNPs
测序结果表明(表1),陇东绒山羊KRTAP2-1 的7 个等位基因中存在12 个SNPs 位点和2 个插入/缺失位点,占序列位点总数的2.12%(表1)。12 个SNPs位点中,7 个SNPs(c.-121C>T、c.-108T>G、c.-103T>C、c.-80A>C、c.-74A>G、c.-38G>T 和c.-31G>A)位于5’UTR 区域,5 个SNPs(c.48C>T、c.67C>G、c.71A>G、c.99A>C 和c.117C>T)位于KRTAP2-1 基因的编码区域。c.67C>G 和c.71A>G 是非同义突变,分别引起了p.Arg23Gly 和p.Tyr24Cys 氨基酸的改变。与A、B、C、E、F和G等位基因序列相比,等位基因D发生了3-bp 的碱基缺失(c.-61_-63delCTC)。与A、C、D、E、F和G等位基因序列相比,等位基因B发生了3-bp 的碱基缺失(c.93_95delCCG),引起了第32 位精氨酸的移码缺失。
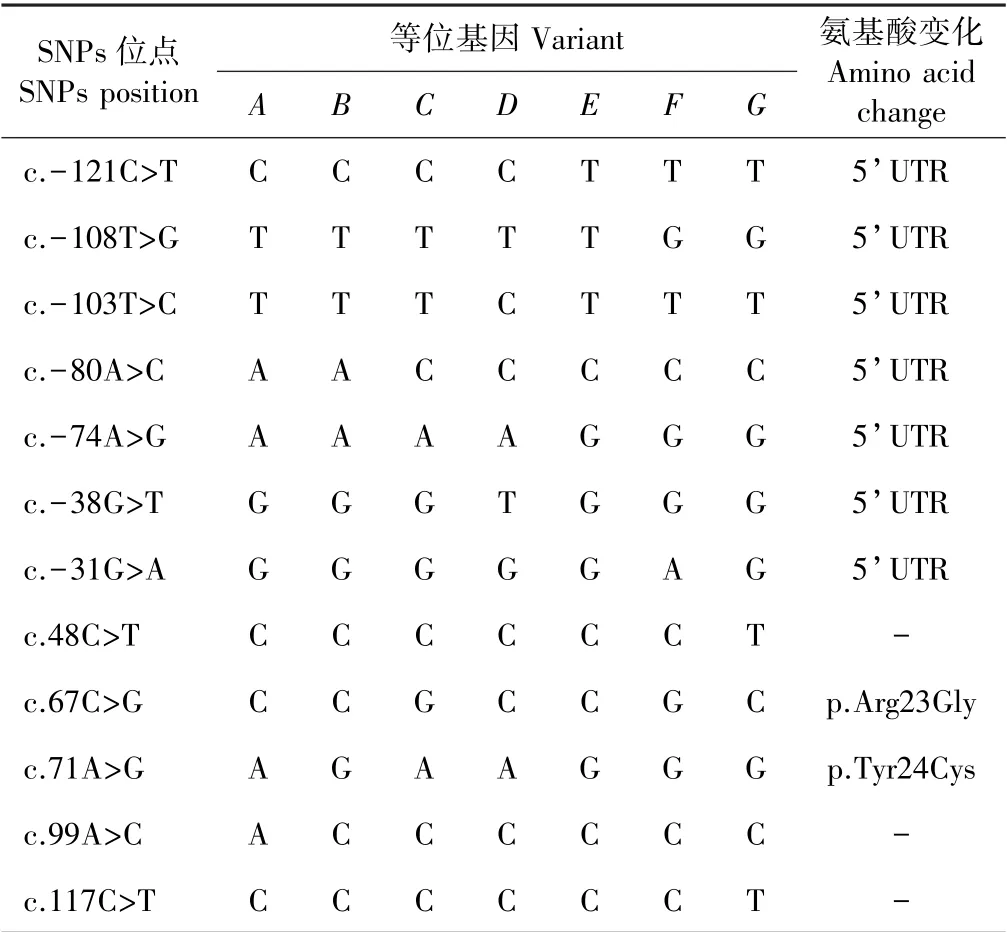
表1 陇东绒山羊KRTAP2-1 基因的SNPsTable 1 SNPs of KRTAP2-1 in Longdong cashmere goats
2.3 陇东绒山羊KAP2-1 多肽链的氨基酸组成
山羊KAP2-1 多肽链包含132 个氨基酸。其中半胱氨酸的比例最高,为23.48 mol%~24.24 mol%,其次为丝氨酸(9.85 mol%)、脯氨酸(13.64 mol%)和苏氨酸(11.36 mol%),天冬氨酸、组氨酸和赖氨酸在7条多肽链中未出现。在等位基因A、C、D、E、F和G翻译的多肽链中发现了15 个潜在的磷酸化位点,包含8个丝氨酸和7 个苏氨酸磷酸化位点。在等位基因B翻译的多肽链中发现了14 个潜在的磷酸化位点,包括7 个丝氨酸和7 个苏氨酸磷酸化位点(图4)。

图3 基于山羊、绵羊和人类的HS-KAPs 的氨基酸序列构建的系统进化树Fig.3 Phylogenetic tree of the amino acid sequences from high-sulphur (HS)-KAPs identified in sheep,humans and goats
2.4 陇东绒山羊KRTAP2-1 基因的遗传多态性分析

图4 山羊和人类KAP2-1 的氨基酸序列对比结果Fig.4 Alignment of KAP2-1 amino acid sequence from goat and human
在249 只陇东绒山羊中,各个等位基因的频率分别为:A:67.81%,B:12.75%,C:12.55%,D:1.01%,E:1.01%,F:4.66%,G:0.2%。共检测到11 种基因型,其中AA、AB、AC和AF为优势基因型,这4 种基因型的总频率为90.77%,其他7 种基因型(BB、CC、DE、AG、BC、BF和CF)的频率均小于5%。有效等位基因数(Ne)、遗传杂合度(He)和多态信息含量(PIC)3 个指标可以用来评价一个群体的遗传多态性。结果表明,陇东绒山羊的多态信息含量为0.48,属于中度多态,有效等位基因数为2.02,遗传杂合度为0.55。
2.5 KRTAP2-1 基因核苷酸序列变异与羊绒性状的关系
在发现的7 个等位基因中,由于等位基因D、E、F和G的频率都低于5%,因此在相关性分析中,只分析了等位基因A、B和C对陇东绒山羊羊绒性状的影响。结果表明,等位基因C的存在与较小的羊绒纤维直径相关(存在:13.4 μm;缺失:13.6 μm;P=0.010)(表2)。

表2 KRTAP2-1 基因核苷酸序列变异与陇东绒山羊羊绒性状的相关性Table 2 Association of caprine KRTAP2-1 variants with cashmere traits in Longdong cashmere goats
3 讨论
本研究在山羊基因组中鉴定了一个编码HS-KAP蛋白的KRTAP2-1 基因,同时分析了该基因的核苷酸序列变异对陇东绒山羊羊绒性状的影响。KRTAP2-1基因位于山羊19 号染色体上,与人类和绵羊的KRTAP2-1 序列具有较高的同源性。KRTAP2-1 的鉴定使得山羊基因组中已鉴定的KRTAPs基因总数从14个增加到15 个。应用比较基因组学和PCR-SSCP 相结合的方法是鉴定绵羊和山羊KRTAPs基因的常用方法,前人已用相同的方法在山羊基因组中鉴定出了KRTAP15- 1[10]、KRTAP20 - 1[8]、KRTAP20 - 2[1]和KRTAP24-1[9]基因,这也表明了本研究鉴定山羊KRTAP2-1 基因方法的可靠性和可行性。
虽然山羊和人类的KAP2-1 序列具有较高的相似性,但是两者的氨基酸序列间存在一定的差异。首先,两物种KAP2-1 序列的长度不同。人类KRTAP2-1 基因编码的多肽链共有128 个氨基酸[13],而山羊KAP2-1 多肽链有132 个氨基酸,它在C 末端比人类蛋白多了SPCC 4 个氨基酸残基。在KAP26-1 和KAP20-2蛋白上也发现了上述物种间的氨基酸序列差异[1,18]。其次,人类与山羊的KAP2-1 多肽链中含有不同比例的半胱氨酸。山羊KAP2-1 蛋白含有24.24 mol%的半胱氨酸,低于人类的27.34 mol%[13]。半胱氨酸对羊绒纤维合成至关重要,是合成羊绒纤维的第一限制性氨基酸,其形成的二硫键连接了KAPs 和KIFs[19]。半胱氨酸数量的改变,可能影响KAPs 与KIFs 或其他KAPs 的交联,从而导致羊绒纤维的结构和功能发生改变。最后,两物种的“CCXPX”五聚体重复序列数量也存在差异(图4)。在人的氨基酸序列中,存在7 个五聚体重复序列,而山羊的氨基酸序列中存在6 个。
值得注意的是,与其他等位基因相比,等位基因B发生了3-bp 的碱基缺失(c.93_95delCCG),导致了1个潜在磷酸化位点的消失。蛋白质磷酸化是一种广泛存在的翻译后修饰,参与和调控生物体内的许多生命活动[20-21]。虽然在KAPs 蛋白中鲜见磷酸化的相关报道,但它对Ks 的结构和功能有重要影响,进而对山羊的羊绒性状产生影响[22]。因此,鉴于在羊绒纤维形成过程中,KAPs 蛋白和Ks 的密切关系,本研究在KAP2-1 中发现的磷酸位点可能也有重要作用,需要进一步试验证实。
SNPs 是基因组上由单个核苷酸的变异所引起的DNA 序列多态性,是动物基因组中最常见的一种变异类型[23]。在绵羊基因组中每1 kb 碱基就有4.9 个SNPs[10],但山羊基因组中的SNPs 分布密度尚不清楚。本研究在山羊KRTAP2-1 基因524 bp 的目的片段内(除去两端的引物序列),检测到12 个SNPs,它的平均分布密度是23 SNPs/1 kb。由此发现,山羊KRTAP2-1 基因的SNPs 分布密度远高于绵羊基因组的平均密度,表明该基因具有非常丰富的多态性。山羊KRTAP2-1 编码区序列中共发现了5 个SNPs 位点,2 个SNPs 是非同义突变。其中,c.67C>G 导致了甘氨酸含量的改变(p.Arg23Gly)。甘氨酸是分子量最小的氨基酸,且缺少一个侧链,能破坏多肽链的螺旋结构,使KAPs 结构更加灵活,有助于KAPs 与Ks 之间更好地交联[24]。c.71A>G 引起了半胱氨酸含量的变化(p.Tyr24Cys),李少斌[24]研究表明半胱氨酸在羊绒纤维之间形成二硫键的过程中发挥重要作用,最终影响羊绒纤维的生长。等位基因B发生了3-bp 的碱基缺失(c.93_95delCCG),这种KRTAPs的长度变异在绵羊KRTAP6-3 基因中已有相关报道[19]。该3-bp 的碱基缺失引起第32 位精氨酸的缺失。精氨酸通常倾向于在蛋白质表面参与盐桥反应,它与带负电荷的天冬氨酸或谷氨酸配对,形成稳定的氢键,这对蛋白质的稳定性有重要作用。本研究中精氨酸含量的变化可能会影响KAP2-1 蛋白的稳定性。本研究还发现了3 个同义突变SNPs 和7 个位于5’UTR 区域的SNPs,虽然这些SNPs 不会引起氨基酸的改变,但仍会通过其他途径发挥生物学功能。Hurst[25]发现,同义突变SNPs 可能影响mRNA 的翻译率,进而改变蛋白质折叠的方式。非编码区的SNPs 能够影响mRNA 的加工速度,对维持染色体特定结构有重要作用,并在转录及转录后水平对基因表达起调控作用[26-27]。因此,这些SNPs 最终会影响KAP2-1 蛋白的表达和结构,进而影响羊绒纤维的结构和性状。
本研究发现陇东绒山羊KRTAP2-1 基因的PIC 值为0.48,表明该群体处于中度多态(0.25 <PIC <0.5),说明该群体中性状变异程度较大,这为羊绒性状选育提供了育种材料,也说明陇东绒山羊具有很大的育种空间。同时,陇东绒山羊群体的遗传杂合度为0.55,有效等位基因数为2.02,进一步证实了该群体有较大的遗传变异。相关性分析表明,等位基因C的存在与较小的羊绒纤维直径相关(表2)。因此,在生产实践中,可选留含有等位基因C的个体,以降低陇东绒山羊的羊绒纤维直径。序列分析发现,与另外2个优势等位基因A和B相比,等位基因C在非编码区有一个SNP (c.-80A>C),在编码区有一个非同义突变SNP(c.67C>G),导致精氨酸变为甘氨酸。因此,等位基因C有可能通过调节精氨酸和甘氨酸的含量,或通过影响mRNA 的转录和翻译,最终影响陇东绒山羊的羊绒纤维直径。目前在山羊基因组中已鉴定的14个KRTAPs基因中,KRTAP8 - 2、KRTAP15-1和KRTAP24-1 对羊绒纤维直径有影响[9-10,28],KRTAP1-1、KRTAP20-1、KRTAP20-2 对产绒量有影响[1,8,29],KRTAP13-1、KRTAP8-2 对绒层高度有影响[12,28]。本研究在KRTAP2-1 中发现的结果与KRTAP8-2、KRTAP15-1 和KRTAP24-1 基因一致。由于KRTAP2-1 基因与上述基因均不在同一个染色体上,故推测KRTAP2-1 对羊绒纤维直径有独立的影响,而不是与其他基因紧密连锁的结果。
4 结论
本研究在山羊19 号染色体上鉴定出了KRTAP2-1 基因,在249 只陇东山羊的该基因中检测出7 个等位基因,发现了12 个SNPs 和2 个3-bp 的碱基缺失。相关性分析表明,等位基因C对羊绒纤维直径有显著影响。本研究丰富了山羊基因组数据,可将该基因用作分子标记,以降低陇东绒山羊的羊绒纤维直径,从而提高羊绒纤维品质。
