小立碗藓PpLBD20基因敲除载体的构建及功能研究
2021-04-13闫慧清敖选真孙贵连
闫慧清,敖选真,刘 露,周 琴,孙贵连,黄 绒
小立碗藓基因敲除载体的构建及功能研究
闫慧清,敖选真,刘 露,周 琴,孙贵连,黄 绒
(贵州师范大学生命科学学院,贵阳 550025)
(Lateral organ boundaries domain)是植物中特有的基因家族。前期研究发现灰霉菌处理小立碗藓导致配子体中的上调表达,但的功能尚不明确。因此提取小立碗藓DNA,PCR扩增基因的上下游片段,依次插入PTN182载体,利用同源重组原理构建敲除表达载体,酶切和测序验证插入序列的正确性。通过工作浓度为20%的PGE 6000介导小立碗藓原生质体转化,筛选鉴定得到敲除后的突变体植株,观察到敲除后小立碗藓不形成茎叶体结构且配子体成丝状。结果为深入探究在小立碗藓的形态建成调控病原菌侵染的抗性作用奠定基础。
基因;载体构建;小立碗藓;配子体
基因家族是一类在植物中特有的基因家族,参与高等植物侧生器官的发育,对侧生器官边界的建成有一定作用,影响地上、地下器官的形成与发育,其家族的普遍功能可能是参与侧生器官近远轴极性的建立[1]。研究发现该基因家族还能参与植物体内激素积累和植物花青素等,与植物非生物胁迫以及病原菌抗性相关[2-3]。Shuai等利用从拟南芥()中发现一个植物特有的在侧生器官近轴端基部中表达的新基因,在拟南芥基因数据库找到的基因家族共有42个[4]。其编码的蛋白质产物中都包含着一段约有100个氨基酸残基且与基因相同的LOB保守结构域。
LBD蛋白由氨基端的LOB结构域和羧基端的可变C末端组成[5]。LOB结构域包含一个典型的由4个保守半胱氨酸残基组成的CX2CX6CX3C (C和X分别代表半胱氨酸和非保守的氨基酸残基)基序和一个保守的甘氨酸残基[6]。根据LOB结构域中亮氨酸拉链类似基序的存在与否,将基因归为两类:第一类是含有完整亮氨酸拉链基序的蛋白结构域 (class I);第二类是缺失亮氨酸拉链基序的蛋白结构域(class II),如图1所示。

图 1 LBD的两类不同结构
Figure 1 Schematic representation of two types of LBDs
前人研究表明,在拟南芥[1]、小麦[7]和玉米[8]等几类植物中,基因参与侧生分生组织的发育,调控植物发育[9]。此外,参与开花植物的非生物胁迫和生物胁迫反应。葡萄在盐胁迫、冷害、水分胁迫和ABA处理下上调表达[10]。香蕉果实中,在茉莉酸甲酯和冷害处理下可诱导的表达[11]。利用转录组和代谢组测定不同成熟时期葡萄果实被灰霉菌侵染的数据中,大部分的参与灰霉菌侵染的早期反应,尤其和都显著性地上调表达[12]。在灰霉菌处理后,葡萄上调表达,且与编码NBS-LRR和果胶酯酶的基因共表达,而后两者分别参与病原体的识别和细胞壁的修饰[12]。在由黄单胞菌引起的柑橘溃疡病中,发现CsLBD1能够调控植物的抗性反应。有研究表明,拟南芥被坏死型的灰霉菌侵染后,Class II AtLBD被诱导产生[13]。这些揭示了LBD在有花植物防卫机制中的作用。基因的启动子含有与非生物胁迫和茉莉酸甲酯反应相关的顺式元件。在拟南芥中,被发现在尖孢镰刀菌()对拟南芥根部的感染过程中起着负调控因子的作用,尖孢镰刀菌通过诱导的表达抑制茉莉酮酸酯信号通路(JA signal pathway)中抗性基因的表达,从而实现其对根部的侵染[14]。小立碗藓()是葫芦藓目葫芦藓科小立碗藓属的藓类植物,是高等植物的低等类群。小立碗藓拥有相对高的同源重组率,为10-3~10-4,即易与外源基因片段发生同源重组[15]。在植物系统进化树上,位于藻类,蕨类和种子植物都在苔藓植物之间。小立碗藓的基因组中有31个[16],其中(GeneBank: XM_024534713)所编码的蛋白质属于class I的LBD蛋白,前期RNA-seq实验及qRT-PCR分析结果表明,小立碗碗藓接种灰霉菌后,在侵染12 h后表达量显著性增加[16]。本实验利用同源重组原理构建敲除载体,获得基因敲除突变株,为今后研究在小立碗藓中的功能提供理论依据。
1 材料与方法
1.1 材料
植物材料小立碗藓(用于提取DNA,扩增目的片段)及PTN182载体(5 006 bp,用于构建基因敲除载体)均由首都师范大学何奕騉教授提供;DH5α感受态细胞由本实验室制备。DNA提取试剂盒、胶回收试剂盒、PremixTM、氨苄青霉素(Amp)、T4-DNA连接酶、1.5 K DNA Marker以及Ⅰ、RⅤ、Ⅰ和Ⅰ限制性内切酶等均购置于大连宝生物有限公司;Plasmid Mini Kit I试剂盒,购于OMEGA Biotek公司;卡那霉素购于美国Sigma公司;酵母提取物和胰蛋白胨购于英国OXOID公司;氯化钠、氢氧化钠、葡萄糖及琼脂等试剂购于国药集团化学试剂有限公司;引物合成委托武汉金开瑞生物工程有限公司完成;测序委托上海生工生物工程股份有限公司完成。

表1 PpLBD20上、下游片段扩增的引物序列
注:下划线部分为酶切位点的碱基序列。
1.2 载体的构建
1.2.1目的基因上、下游同源臂的获得 用的cDNA序列在NCBI上进行Blast查找对应的DNA序列(GenBank: XM_024534713),得到全长为5 400 bp的DNA序列,随机选取大小约1 000 bp的DNA序列的上、下游片段。利用Primer 5软件设计上、下游同源臂扩增出引物,结合PTN182载体的克隆位点选取酶切位点,计算GC含量(50%~55%)添加保护碱基。最终,以Ⅰ和RⅤ为上游臂酶切位点,以Ⅰ和Ⅰ为下游臂酶切位点。设计的引物序列如表1所示,扩增得到上游PCR产物大小为948 bp(),下游PCR产物大小为892 bp(),送武汉金开瑞生物工程公司合成上述引物。
1.2.2敲除载体的构建 取10 μL含PTN182质粒的大肠杆菌菌液加入50 mL含卡那霉素的LB液体培养基中恒温37℃、220 r·min-1振荡培养12~16 h。取5 mL含有菌液的培养液于圆底离心管,根据Plasmid Mini Kit I试剂盒说明书步骤提取PTN182质粒。用Ⅰ、RⅤ对和PTN182载体进行同步双酶切。在37℃恒温水浴锅中酶切25 min后,1%琼脂糖凝胶电泳验证,将满足上游臂片段和PTN182酶切片段大小的目的片段按照胶回收试剂盒说明书步骤进行胶回收。获得的目的片段与PTN182酶切片段在16℃恒温下连接12~16 h。将10 μL连接产物转化到大肠杆菌感受态细胞中,继而将转化菌液涂板到含卡那霉素的LB培养平板上,37℃恒温倒置过夜培养。挑取长出的单菌落至含卡那霉素的LB液体培养基中,37℃、200 r·min-1恒温振荡培养12~16 h。后续进行菌液PCR(引物为上游臂引物)、菌液质粒提取并酶切验证。将菌液送至上海生工生物工程股份有限公司进行测序。用Ⅰ、Ⅰ双酶切和-PTN182质粒,0.7%琼脂糖凝胶电泳验证酶切结果,对-pMD19-T酶切片段和-PTN182酶切片段(约5 000 bp)进行胶回收,恒温16℃连接目的片段与-PTN182酶切片段12~16 h,连接液转入大肠杆菌感受态细胞中,将转化菌液涂板于含卡那霉素的LB培养平板上培养,挑取单菌落扩大培养。对扩大培养的单菌落进行菌液PCR、质粒提取并酶切等验证后(酶切所用的酶为Ⅰ和Ⅰ),将菌液送至上海生工生物工程股份有限公司完成测序。
1.3 原生质体的制备
取培养大约5 d适量的小立碗藓原丝体,加入1%崩溃酶溶液中,避光反应30 min。150/300目的细胞筛进行过滤,收集过滤后的原生质体溶液,1 000 r·min-1,4 min,15℃离心,弃上清。收集等体积的8% D-甘露醇溶液重悬原生质体2次,用500 μL MMM溶液重悬。取20 μL原生质体溶液,用血细胞计数板计算原生质体浓度。
1.4 PEG介导外源基因PpLBD20转化和鉴定
将敲除载体菌液扩大培养,用Plasmid Mini Kit I试剂盒(OMEGA,D6943-02)提取质粒后,加入等体积的工作浓度为20%的PEG 6000溶液45 ℃热激5 min,20 ℃恢复15 min。用ABCG稀释转化本混合液。每次300 μL,都静置4 min,共5次。室温下避光恢复1 h。1 500 r·min-1,3 min离心,收集原生质体。用8 mL PRM/T培养基溶解。铺板,加到120 mm PRM/B培养基培养,在40 μmol·m-2·s-1持续光照下25 ℃培养。分别选用G418-30 μg·mL-1和G418-60 μg·mL-1的浓度进行抗性筛选,每次抗性筛选后,在不含抗性的培养基上恢复培养2周进行下一次筛选。分子水平上的筛选过程:设计3对引物(分别为P1、P2和P3)进行鉴定,引物序列如表2所示。

表2 PpLB20敲除后分子鉴定的引物序列
1.5 基因表达
用Primer Premier 5设计内参基因的引物。提取总RNA:称取150 mg的小立碗藓到研钵中,用液氮充分研磨2~3次至材料成白色粉状,加至1.5 mL RNAiso Plus的离心管中,静置5 min后,于4℃、12 000 r·min-1离心5 min;收集上清,加入200 μL氯仿静置5 min,于4℃、12 000 r·min-1离心5 min;再收集上清,加500 μL的异丙醇,静置15 min后,于4℃、12 000 r·min-1离心5 min;弃上清,加1 mL 75%的乙醇,于4℃、7 500 r·min-1离心5 min;去上清干燥,加20 μL DEPC水溶解沉淀即可。将获得的RNA利用反转录试剂盒(北京庄盟生物,Cat#ZR102)试剂盒进行反转录后,用SYBR®PremixTMⅡ试剂盒扩增,条件为:95℃,5 s;60℃,34 s,共40个循环。目的基因表达量以内参基因作为标准进行相对定量,相对表达量的计算用公式2-△△Ct法[17]。
2 结果与分析
2.1 PCR扩增获得PpLBD20目的基因的上、下游同源臂
利用同源重组原理将敲除载体的片段替换基因组中目的基因的片段。首先通过PCR扩增分别得到的上游同源臂片段和下游同源臂片段,将其命名为和,设计引物进行PCR扩增,得到的序列片段长度分别为948 bp和892 bp。经0.7%琼脂糖凝胶电泳验证PCR产物,结果(图2)显示,除空白对照外,其余3条扩增得到的条带均略低于1 000 bp,该结果符合扩增得到上游和下游目的片段的长度。
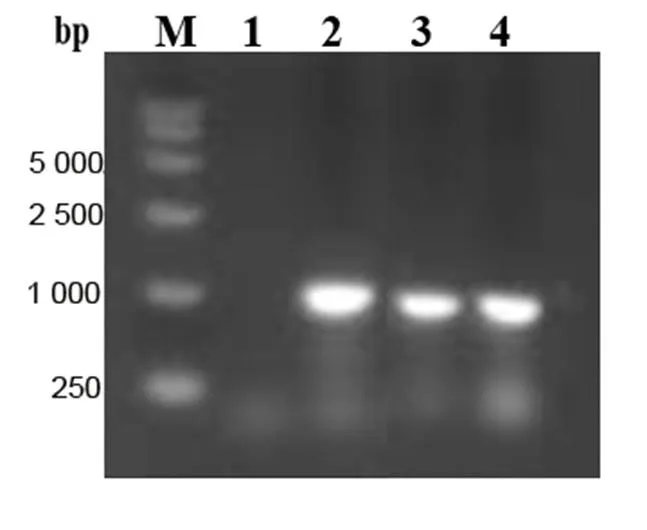
注:泳道1为空白对照,泳道2是上游的扩增片段,泳道3和4为下游扩增片段。
Figure 2 The amplified PCR products separated by agarose gel electrophoresis
2.2 PpLBD20敲除载体的构建
将酶切引物经PCR扩增连接到上游臂目的片段,同时用Ⅰ和R V酶切PTN182载体。胶回收扩增得到含有酶切片段的目的基因和PTN182酶切片段并过夜连接,进行菌液PCR(引物为上游臂引物)。结果(图3A)显示,PCR扩增得到的片段接近1 000 bp左右,与上游臂片段大小相似的片段。经Ⅰ和R V酶切(图3B),分别检测得到的片段大小约为5 000 bp和1 000 bp大小。该片段大小符合PTN182载体片段大小(5 005 bp)和上游臂片段的大小,后经测序验证得到-PTN182载体;再将酶切引物酶切经PCR扩增连接到下游臂目的片段,同时用Ⅰ和Ⅰ酶切-PTN182质粒;再经连接酶连接转化后进行检测,提取重组质粒。结果如图3C所示:泳道1、2和3均为构建后的重组载体,泳道4为Ⅰ和Ⅰ酶切后的产物,显示为两条带,片段显示接近于1 000 bp和5 000 bp,该片段大小符合下游臂()和- PTN182的大小。后经菌液PCR和测序结果表明-PTN182-载体连接成功,敲除载体构建完成。
2.3 敲除突变体小立碗藓的鉴定
通过同源重组的原理,利用PEG 6000介导的遗传转化方法将的敲除载体转化到小立碗藓原生质体中,转化的原生质体生长2周后状态良好。结果如图4(A-a)所示,经过2次不同浓度的G418抗性筛选,得到转化株图4(A-b)。设计3段引物(命名为P1、P2和P3)来验证敲除载体是否转入到小立碗藓原生质体中,其分布如图4(B)所示。3条引物分别为:P1为上游臂和下游臂外小立碗藓基因组的序列;P2的正向引物为上游臂的序列,反向引物为Ⅱ的序列;P3的正向引物为ⅡI的序列,反向引物为下游臂臂的序列。经过PCR扩增得到的结果(图4(C))显示:泳道1、2和3属于野生型,野生型经P1引物扩增得到的片段接近5 000 bp左右,与基因大小相符;经P2和P3扩增后没有检测到片段。泳道4、5和6属于敲除后植株,观察得到经P1扩增得到的区间略高于野生型;经P2和P3扩增后没有检测到约为4 000 bp的片段,表明Ⅱ已替换掉的部分序列。
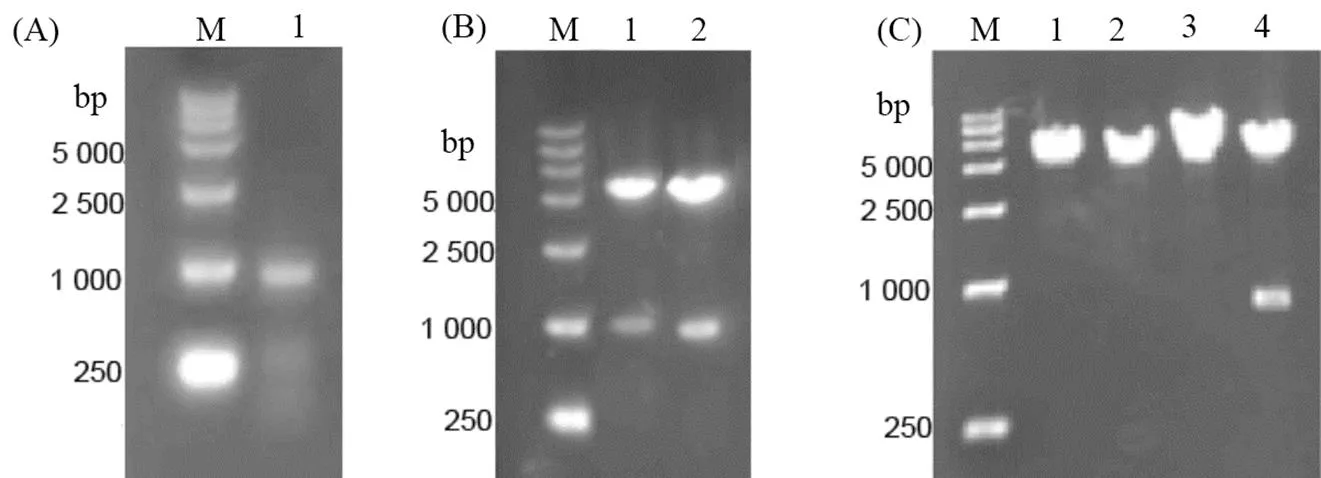
(A) PpLBD20L-PTN182菌液PCR验证;(B) PpLBD20L-PTN182酶切验证; (C) PpLBD20 L-PTN182-PpLBD20R载体质粒提取构建酶切验证。
Figure 3 The construction ofknockout-PTN182-vector
2.4 野生型和Pplbd20型基因表达及配子体观察
进一步提取野生型和敲除后的配子体总RNA进行实时荧光定量PCR,检测的转录水平表达量。小立碗藓野生型的表达量是敲除型配子体的12.35倍,从而表明基因敲除载体转化成功并降低了的基因表达量(图5A)。通过观察野生型和突变体配子体(图5B),可见小立碗藓野生型的茎、叶结构清晰且比较透明,叶片对称结构呈黄绿色,中间叶脉颜色比两侧叶片颜色深,叶片边缘比较平滑无突起,叶片的表皮细胞为长方形状并紧密排列;敲除的突变体无拟茎、叶,呈现树枝状结构,整体呈现黄绿色,各分枝的长度不同,没有规律。因此可以得出,影响配子体的形成。

(A) 原生质体(a)和抗性筛选获得的突变株(b);(B) 3条引物设计区域分布;(C) 野生型和敲除配子体鉴定(1、2和3为野生型,4、5和6为敲除植株)。
Figure 4 The screening and molecular identification of knockoutgametocyte

图 5 PpLBD20的转录水平表达量(A)和野生型(a)和Pplbd20敲除(b)配子体表型(B)
Figure 5 Determination of thetranscriptional level (A) and the observation of wild type(a) and(b) plants(B)
3 讨论与结论
基因敲除指通过在已知基因核酸序列上特定的可译框的特定位点进行缺失突变,获得基因功能缺失突变体来研究其造成的表型效应[18],可以通过插入突变或特定序列被替换。同源重组将外源DNA序列整合到宿主基因组的特定位点来实现基因敲除。由于小立碗藓同源重组率较高,替换型载体是小立碗藓基因敲除载体最常用的载体类型,构建载体时,同源臂大小在1 000 bp左右时转化率最高。在多种载体类型中,PTN182是小立碗藓中常用的敲除载体[19],PTN182质粒是环状DNA分子,可以在细菌细胞中独立存活,而且PTN182质粒含有Ⅱ基因,在大肠杆菌中表现为抗卡那霉素[20],以此为标记特点,可以快速有效得筛选得到的敲除载体。该载体已用于敲除小立碗藓的肉桂醇脱氢酶1、香豆酸-3-羟基化酶基因和胼胝质合酶的敲除[20-22]。
小立碗藓敲除载体成功构建及转入小立碗藓植株为研究在小立碗藓的生长发育功能及是否参与生物胁迫等基因功能建立基础。通过观察敲除小立碗藓的配子体,发现茎叶体结构发生变化,配子体形成丝状,其不能够影响侧生细胞的分化,导致原生质体的细胞只能沿着单一方向生长。已发现玉米调控胚生根和气生根的发育,影响侧生器官的发育[23-24],推测在小立碗藓的侧生器官的分化和形态发育中起作用。前人研究发现LBD参与维管束植物的抗病性。因此可以利用病原菌处理敲除的小立碗藓配子体,检测相关防卫基因的变化,或者构建的超表达植株,通过病原菌侵染后计算菌丝侵染率进一步验证在早期陆生植物种的抗病作用。本文通过构建敲除载体并成功转化小立碗藓的原生质体对研究基因功能以及研究小立碗藓防御机制奠定基础。
[1] BELL E M, LIN W C, HUSBANDS A Y, et al. Arabidopsis LATERAL ORGAN BOUNDARIES negatively regulates brassinosteroid accumulation to limit growth in organ boundaries[J]. PNAS, 2012, 109(51): 21146-21151.
[2] YANG H, SHI G X, DU H Y, et al. Genome-wide analysis of soybean LATERAL ORGAN BOUNDARIES domain-containing genes: A functional investigation of GmLBD12 [J]. Plant Genome, 2017, 10 (1): 1-19.
[3] LU Q, SHAO F J, MACMILLAN C, et al. Genome-wide analysis of the lateral organ boundaries domain gene family inreveals members that differentially impact secondary growth[J]. Plant Biotechnol J, 2018, 16(1): 124-136.
[4] SHUAI B, REYNAGA-PEÑA C G, SPRINGER P S. The lateral organ boundaries gene defines a novel, plant-specific gene family[J]. Plant Physiol, 2002, 129(2): 747-761.
[5] 石玉, 沈诗雅, 张倩茹, 等. LBD基因家族研究进展[J]. 中国细胞生物学学报, 2019, 41(4): 738-745.
[6] MATSUMURA Y, IWAKAWA H, MACHIDA Y, et al. Characterization of genes in the ASYMMETRIC LEAVES2/LATERAL ORGAN BOUNDARIES (AS2/ LOB) family in, and functional and molecular comparisons between AS2 and other family members[J]. Plant J, 2009, 58(3): 525-537.
[7] CHEN W F, WEI X B, RETY S, et al. Structural analysis reveals a “molecular calipers” mechanism for a LATERAL ORGAN BOUNDARIES DOMAIN transcription factor protein from wheat[J]. J Biol Chem, 2019, 294(1): 142-156.
[8] LEWIS M W, BOLDUC N, HAKE K, et al. Gene regulatory interactions at lateral organ boundaries in maize[J]. Development, 2014, 141(23): 4590-4597.
[9] ZHANG Y M, ZHANG S Z, ZHENG C C. Genome-wide analysis of LATERAL ORGAN BOUNDARIES domain gene family in[J]. J Genet, 2014, 93(1): 79-91.
[10] DEYOUNG B J, INNES R W. Plant NBS-LRR proteins in pathogen sensing and host defense[J]. Nat Immunol, 2006, 7(12): 1243.
[11] BA L J, KUANG J F, CHEN J Y, et al. MaJAZ1 attenuates the MaLBD5-mediated transcriptional activation of jasmonate biosynthesis geneMaAOCin regulating cold tolerance of banana fruit[J]. J Agric Food Chem, 2016, 64(4): 738-745.
[12] AGUDELO-ROMERO P, ERBAN A, REGO C, et al. Transcriptome and metabolome reprogramming incv.berries upon infection with[J]. J Exp Bot, 2015, 66(7): 1769-1785.
[13] THATCHER L F, KAZAN K, MANNERS J M. Lateral organ boundaries domain transcription factors[J]. Plant Signal Behav, 2012, 7(12): 1702-1704.
[14] THATCHER L F, POWELL J J, AITKEN E A B, et al. The lateral organ boundaries domain transcription factor LBD20 functions inwilt susceptibility and jasmonate signaling in Arabidopsis[J]. Plant Physiol, 2012, 160(1): 407-418.
[15] SCHAEFER D G, ZRŸD J P. Efficient gene targeting in the moss[J]. Plant J, 1997, 11(6): 1195-1206.
[16] HUANG X L, YAN H Q, LIU Y J, et al. Genome-wide analysis of LATERAL ORGAN BOUNDARIES DOMAIN-inand stress responses[J]. Genes Genom, 2020, 42(6): 651-662.
[17] BUSTIN S A, BENES V, GARSON J A, et al. The MIQE guidelines: minimum information for publication of quantitative real-time PCR experiments[J]. Clinical Chem, 2019, 55(4): 611-622.
[18] 董文, 李卫, 郭光沁, 等. 苔藓植物小立碗藓功能基因组学研究新的模式系统[J]. 遗传, 2004, 26(4): 560-566.
[19] 周莎莎. 基因克隆与表达研究进展[J]. 现代农业科技, 2008(22): 280-281, 284.
[20] 崔彩云, 晏国洪, 姜山. 小立碗藓基因敲除载体的构建及C3H1-npt Ⅱ- C3H2目的片段最佳PCR条件筛选[J]. 北方园艺, 2016 (5): 113-118.
[21] 赵春燕, 晏国洪, 姜山. 小立碗藓基因敲除载体的构建及酶切方法优化[J]. 江苏农业科学, 2016, 44(3): 59-63.
[22] 陈波. 小立碗藓胼胝质合酶6基因功能的初步研究[D]. 贵阳: 贵州师范大学,2019.
[23] 汤玮婧. LBD转录因子家族的起源与进化[D]. 杨凌: 西北农林科技大学, 2013.
[24] 卢寰, 时振英. 高等植物特有的基因的分子生物学功能研究进展[J]. 植物生理学报, 2013, 49(9): 833-846.
Construction and protoplast transformation ofknockout vector
YAN Huiqing, AO Xuanzhen, LIU Lu, ZHOU Qin, SUN Guilian, HUANG Rong
(School of Life Sciences, Guizhou Normal University, Guiyang 550025)
Lateral organ boundaries domain () is a kind of the plant-specific gene family. Previously, we identified thatwas significantly induced inwithinfection, but the function of theis still not known. Here, we extracted the genomic DNA from, and amplified the upstream and downstream homologous fragments ofby PCR and respectively inserted into the PTN182 vector. The knockout vector was constructed using homologous recombination and was verified by enzyme digestion and sequencing. The constructed vector was transformed into protoplast by addition of polyethylene glycol 6000 with the concentration of 20%, and the transformants were selected and identified. We observed that the knockoutgametocyte did not show the leaf-like structure and formed the filamentous branches. The current finding will facilitate to further explore thein regulating.resistance to.
; vector construction;;gametocyte
Q782
A
1672-352X (2021)01-0015-06
10.13610/j.cnki.1672-352x.20210319.025
2021-3-23 12:12:13
[URL] https://kns.cnki.net/kcms/detail/34.1162.S.20210319.1548.050.html
2020-05-26
国家自然科学基金(32060587),贵州师范大学2017年度学术新苗培养及创新探索专项项目(黔科合平台人才[2017]5726号)和贵州师范大学2020年全国大学生生命科学竞赛计划项目(55092)共同资助。
闫慧清,博士,副教授。E-mail:cheerfulness@126.com
