草鱼肠系膜脂肪组织高质量总RNA提取方法的研究
2021-03-24骆丽婷李胜杰樊佳佳马冬梅
姜 鹏,骆丽婷,李胜杰,樊佳佳,马冬梅
(1.中国水产科学研究院 珠江水产研究所,农业农村部热带亚热带水产资源利用与养殖重点实验室,广东 广州 510380;2.仲恺农业工程学院 动物科技学院,广东 广州 510225)
动物脂肪组织不仅是机体能量贮存库,也是活跃的内分泌器官,其分泌的多种激素和细胞因子在机体代谢平衡、免疫应答等方面发挥重要作用[1-3]。在组织功能研究中,分子水平研究必不可少,其中RNA提取纯化是常涉及的分子生物学试验技术。然而,由于脂肪组织含有丰富油脂,RNA丰度低,导致高质量总RNA提取常常存在一定难度,影响到下游试验的高效开展。
目前,RNA提取广泛采用经典的酸性酚—异硫氰酸胍—氯仿一步提取法[4]。该方法具有操作便捷,无需复杂设备等优点,并配备有成熟的TRIzol等即用型商品化试剂[5-7]。因此,针对脂肪组织RNA含量低、难提取等问题,大多数学者选择在一步法基础上进行优化改良[8]。如吴江维等[9]发现,以猪脂肪组织为材料,增加样品量,延长静置裂解时间,去除油脂层污染等操作,可改善RNA提取质量。采用类似优化步骤,在肉牛、绵羊等动物脂肪组织也获得了较高质量的RNA样品[10-11]。
草鱼(Ctenopharyngodonidella)是中国养殖产量较高的淡水经济鱼类,养殖过程中通常出现体脂过度沉积现象[12]。试验发现,常规方法下,草鱼脂肪组织RNA提取易发生降解,干扰到后续分子生物学试验的有序推进。草鱼脂肪组织主要蓄积在腹腔肠道周围,与哺乳动物脂肪组织相比,质地构造存在明显差异。现有的优化改良策略是否适用于草鱼脂肪组织RNA提取,需进一步验证及摸索。因此,笔者以草鱼肠系膜脂肪组织为试验对象,基于常规的TRIzol试剂一步法,拟通过对优化操作步骤的比较分析,建立一种高质量总RNA提取方法。
1 材料与方法
1.1 试验用鱼
试验用草鱼取自中国水产科学研究院珠江水产研究所生物技术养殖基地。从正常饲养的群体中随机挑选健康个体,体质量为80~150 g。
1.2 试剂耗材与仪器
TRIzol试剂选用TaKaRa公司RNAiso Plus产品(Code No.9108),DEPC水购自Beyotime公司,氯仿和异丙醇等试剂为国产分析纯,PRO 200型精密匀浆器(配置∅7 mm×105 mm不锈钢平头转子)购自PRO Scientific公司(美国),高速冷冻离心机5417R购自Eppendorf公司(德国),TGem微量分光光度计(OSE-260)购自天根生化科技(北京)有限公司,Tanon-3500全自动凝胶图像分析系统购自上海天能科技有限公司。离心管和移液枪头等均使用RNase-free产品。
1.3 常规的RNA提取方法
草鱼活体使用间氨基苯甲酸乙酯甲磺酸盐轻微麻醉,剪断脊柱放血后解剖,快速剪取肝脏、脾脏、肠道(不含肠系膜脂肪)、肠系膜脂肪组织,对应样品组编号为T1~T4(表1)。采用常规方法进行组织RNA提取,每组测试3~5尾鱼,具体操作步骤如下:

表1 常规与优化的总RNA提取方法比较Tab.1 Comparison between conventional and optimized total RNA extraction methods (n=3~5)
取8~15 mg新鲜组织样品放入预冷的1.0 mL TRIzol试剂中,迅速均质匀浆约30 s,室温静置5 min;加入200 μL氯仿,剧烈振荡15 s,室温静置5 min后,4 ℃,12 000 r/min离心15 min,小心吸取上层水相转移至另一新离心管中;加入500 μL的异丙醇,颠倒混匀,室温静置10 min后,4 ℃,12 000 r/min离心15 min;弃上清液,得到白色RNA沉淀物,加入1.3 mL DEPC水配制的75%体积分数乙醇溶液,翻转洗脱,4 ℃,7500 r/min离心5 min;弃上清液,室温干燥沉淀物约5 min,加入30~50 μL的DEPC处理水溶解RNA。质检后,-70 ℃超低温冰箱保存备用。
1.4 优化的RNA提取方法
根据预试验结果,将草鱼脂肪组织样品量增加至约30 mg,每样品TRIzol试剂量增至1.3 mL,并对常规RNA提取步骤进行优化(表1),具体优化操作如下:
(1)优化操作1:取新鲜样品至TRIzol试剂中机械匀浆30 s后,4 ℃,12 000 r/min离心5 min,吸弃上层油脂约150 μL,重复操作1次,将大部分余液移至新管,其余步骤与常规方法相同,对应样品组编号为T5。
(2)优化操作2:取新鲜样品至液氮急速冷冻,研钵内人工充分研磨,然后将粉末状样品加入TRIzol试剂中剧烈振荡30 s裂解,吸弃油脂等步骤与优化操作1相同,对应样品组编号为T6。
(3)优化操作3:与优化操作1大体相同,只增加样品机械匀浆时间至约3 min,对应样品组编号为T7。
(4)优化操作4:取新鲜样品至液氮速冻,再转移冻样至TRIzol试剂中机械匀浆约3 min,其余步骤与优化操作1相同,对应样品组编号为T8。
以上每组(T5~T8)各取3~5尾鱼测试。
1.5 RNA质量检测
取2.0 μL RNA样品,利用微量分光光度计检测RNA样品浓度与纯度。配制1.5%非变性琼脂糖凝胶,取1.0~2.0 μL适量RNA,140 V电泳15 min,紫外灯光下观察RNA条带情况,评估提取质量。
从提取质量较好的T8组中,随机取3个样品送金唯智生物科技有限公司进行高通量测序平台质检,主要包括使用NanoDrop 2000分光光度计检测RNA浓度和质量,以及Agilent 2200 Tapestation系统评估RNA完整性。
2 结 果
2.1 常规方法提取草鱼不同组织RNA的质量比较
采用常规方法提取的草鱼不同组织RNA,质量检测情况见表2。试验发现,与肝脏、脾脏、肠道组织相比,草鱼肠系膜脂肪组织总RNA的提取量偏少,表明草鱼脂肪组织RNA含量较低。常规提取方法下,草鱼肝脏、脾脏、肠道和肠系膜脂肪组织总RNA的D260/D280比值约为2.0,符合1.9~2.0的标准范围,表明常规方法提取的不同组织核酸质量均较高。样品的D260/D230比值略小于2.0,说明可能有微量胍盐或有机溶剂等杂质残留,试验检验其对下游分子试验影响轻微。
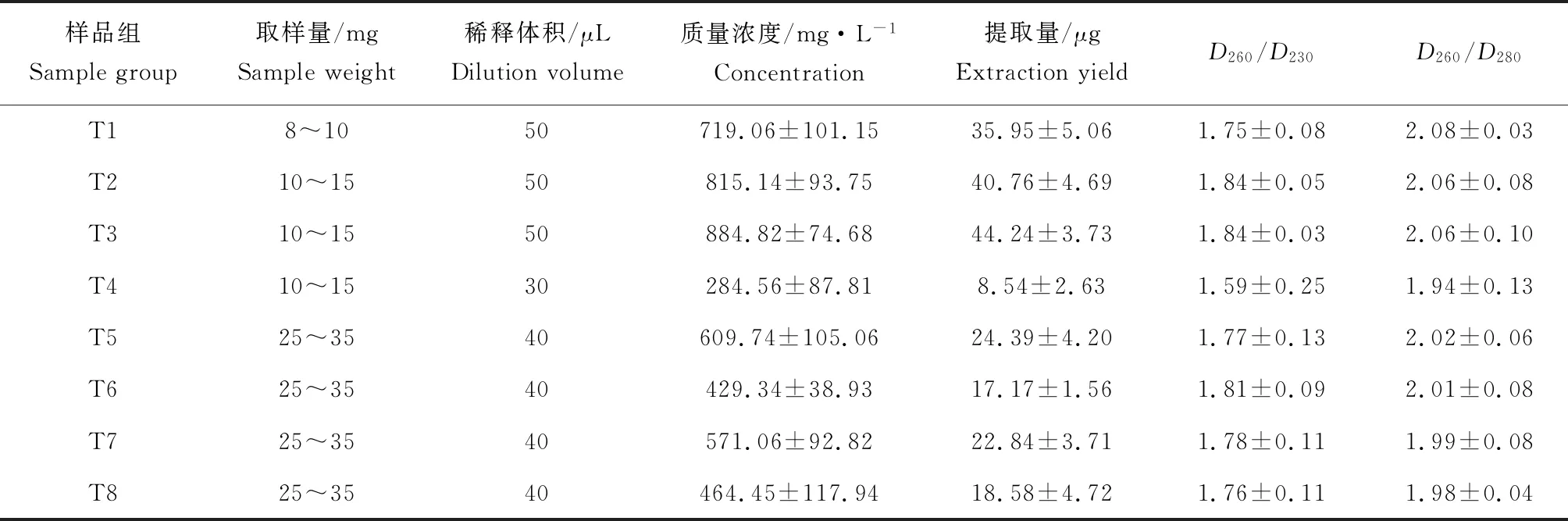
表2 分光光度计检测的总RNA质量情况Tab.2 Quality of total RNA content measured by spectrophotometer (n=3~5)
样品进行琼脂糖凝胶电泳检测(图1)。T1~T3组肝脏、脾脏、肠道组织样品的总RNA在凝胶上显示出清晰的28S和18S rRNA特征性条带,T4组脂肪组织样品则出现严重拖带现象,28S rRNA条带亮度也明显弱于18S rRNA,说明常规方法下提取的草鱼脂肪组织RNA存在降解现象。以上不同组织间的对比试验表明,草鱼肠系膜脂肪组织RNA不仅含量少,而且易发生降解。

图1 总RNA琼脂糖凝胶电泳Fig.1 Agarose gel electrophoresis of total RNAM.DL2000 DNA分子量标准.M.DL2000 DNA marker.
2.2 不同优化操作提取草鱼脂肪组织RNA的比较
在优化操作中,适当增加脂肪组织量(25~35 mg),有助于RNA提取量提升,可满足常规试验用量需求(T6组RNA质量浓度略低,是因研钵里收集粉末状样品时有损耗)。针对草鱼脂肪组织RNA顽固性降解难题,探索可行的优化操作方法。分光光度计检测显示,4种优化方法提取的草鱼脂肪组织总RNA纯度均保持了较高水平。进一步琼脂糖凝胶电泳(图1)后发现,T5组样品经简单去除油脂层操作并未有效改善RNA降解问题,而样品前处理改用液氮速冻研磨方式,T6组样品显示RNA降解有所缓解,但仍有一定程度拖带现象。当T7组样品仅增加了样品机械匀浆时间时,RNA降解问题得以显著改善,尤其是将液氮速冻和增加样品机械匀浆时间步骤一同使用,提取的T8组脂肪组织RNA展示出较高质量的电泳条带特征。
试验结果表明,在TRIzol试剂裂解过程中,增加样品机械匀浆时间(约3 min)可显著降低RNA降解程度,而且匀浆前样品采用液氮速冻处理可减轻取样操作可能带来的降解问题。
2.3 草鱼脂肪组织优化提取RNA的质检分析
为进一步明确优化操作4提取的样品质量,随机挑选T8组3个样品送测序公司进行质检分析。检测结果发现,3个样品RNA纯度较高,关键的RIN值为8.7~9.0(表3),依据公司评级标准,3个样品均为最佳的A级(A级标准:核酸质量≥2 μg,D260/D280≥2.0,RIN值≥7,28S/18S≥1.0,核酸质量浓度≥100 mg/L,体积≥10 μL,5S峰形正常等),表明RNA完整性好,质量满足高通量转录组建库测序要求。此外,由Agilent 2200生物分析仪生成的凝胶图像及电泳色谱峰图(图2)可见,3个RNA样品的质量水平较高,其峰图基线较平整,无明显降解杂峰,28S与18S峰形正常。

表3 改良方法提取草鱼肠系膜脂肪组织总RNA的质量情况Tab.3 Quality of total RNA extracted from mesenteric adipose tissues of grass carp C.idella by the improved method

图2 Agilent 2200生物分析仪评估总RNA完整性Fig.2 Integrity assessment of total RNA by the Agilent 2200 bioanalyzera.采用优化操作4提取的总RNA样品胶样图;b~d.分别为T8-1、T8-2和T8-3样品的电泳图谱;M.电子化分子量标准;Lower.25 bp分子量.a.gel-like images of total RNA of the samples extracted by the optimized-4 method;b—d.electropherograms of T8-1,T8-2 and T8-3 samples,respectively;M.electronic ladder;Lower:25 bp ladder.
3 讨 论
3.1 RNA提取方法及原理
自Chomczynski等[13]提出酸性酚—异硫氰酸胍—氯仿一步提取法,RNA提取才变得简便易行。该方法操作流程主要包括:样品均质与裂解,氯仿分离含RNA水相,异丙醇沉淀和乙醇洗涤。在提取过程中,想要获得高质量RNA需要严格防范内源性和外源性RNA酶的破坏。RNA酶是一种专一水解RNA磷酸二酯键的蛋白酶,在环境中广泛存在,而且可耐受高温煮沸等严苛处理条件[14-15]。因此,为了避免外界环境中RNA酶的干扰,通常需要使用RNase-free耗材,维持清洁的试验环境以及科学规范的操作。除此之外,为了有效抑制样品组织细胞所包含的内源性RNA酶,则需要依赖裂解试剂中异硫氰酸胍等强效蛋白变性剂。
在本试验中,草鱼肠系膜脂肪组织RNA降解问题主要是源于常规提取方法未能有效阻止内源性RNA酶的水解作用。初步分析,RNA降解可能发生在取样起始阶段,也可能在组织裂解过程中,或是二者兼有。为此,在样品取样阶段尝试采用液氮速冻人工研磨方法,期望利用超低温环境来抑制组织内RNA酶的活性,并尽可能缩短样品空气暴露时间。但多次实操发现,液氮速冻研磨操作对样品RNA降解虽有一定缓解,但凝胶电泳仍显示出不同程度拖带现象,甚至时常出现严重降解个例,说明RNA降解在组织裂解过程中也有发生。这意味着TRIzol试剂中的异硫氰酸胍等成分未能高效抑制肠系膜脂肪组织中的RNA酶。考虑到脂肪组织特异性因素,推测脂肪组织所包含的大量油脂很可能影响了蛋白变性剂发挥效能。
3.2 脂肪组织RNA提取方法的优化
如何破除油脂影响,有学者利用液态油滴比重低的特点,采用简单离心去除油脂层方法[16];也有学者通过调整试剂配方来强化脂肪乳化剂分离油脂的效果[17]。以上方法在草鱼肠系膜脂肪中验证效果均不理想。基于上述实际情况,拟尝试在裂解过程中加入剧烈振荡等物理手段。经反复摸索测试后发现,当样品采用机械匀浆,并将匀浆时间由原有约30 s延长至约3 min时,RNA降解得以显著改善,而且批量验证效果也较为稳定。据此推测,长时间机械匀浆所形成的持续剪切冲击力或许有助于TRIzol试剂中的异硫氰酸胍等成分突破油滴阻碍而有效抑制内源性RNA酶。
需要特别说明的是,草鱼肠系膜脂肪组织中实际包裹有难以分离的胰腺组织,这在石蜡切片和转录组测序(数据未展示)中得以证实。通常认为,动物的胰腺组织因富含RNA酶,RNA较难提取[18-19],这也可能是造成草鱼肠系膜脂肪组织RNA易发生降解的原因之一。有研究表明,小鼠胰腺组织样品在液氮低温环境中研磨处理,能有效避免RNA发生降解[20]。在本试验中,或许是由于油脂的复合叠加作用,导致单纯采用液氮研磨未能较好抑制富含RNA酶的草鱼肠系膜脂肪组织。
4 结 论
笔者建立了一种草鱼肠系膜脂肪组织高质量总RNA提取的方法,简要地说,在常规方法基础上,将取样量增至约30 mg,有助于RNA产量提升以满足常规试验需求;样品前处理建议采用液氮速冻方式,并直接将冻样放入TRIzol试剂中机械匀浆,匀浆时间需延长至约3 min,可显著降低RNA降解发生,同时保留离心去除油脂层等操作步骤。该改良方法所获得的总RNA纯度和完整性较高,可满足转录组建库测序等试验要求。
