基于ITS序列的带叶兜兰根内真菌群落组成分析
2021-03-23王晓国张自斌李秀玲周主贵
王晓国,张自斌,李秀玲,周主贵*
(1.广西壮族自治区农业科学院 微生物研究所,广西 南宁 530007; 2.广西壮族自治区农业科学院 花卉研究所,广西 南宁530007)
兜兰属是兰科植物中最具特色的一个类群,分布于东南亚、南洋群岛以及太平洋西南大洋洲岛屿国家[1]。近年来,野生兜兰的过度采集和其自然生境受到严重破坏[2],兜兰分布区和种群数量剧减[3]。目前,兜兰全属植物已被列入“野生动植物濒危物种国际贸易公约”(CITES)附录Ⅰ而受到严格保护[4]。中国是兜兰的主要分布区之一,主要分布于西南地区及华南部分地区[5,6]。广西西北部为我国野生带叶兜兰(Paphiopedilumhirsutissimum)的主要分布区域,近年来广西野生带叶兜兰分布破碎化现象加剧,种群恢复的工作刻不容缓[7]。
根内真菌主要定殖于植物根内,大致分为共生、腐生和寄生真菌3种类型。其中,共生真菌可从土壤中直接吸收植物所需矿质营养,协助根系吸收水分,改善植物养分和水分状况[8,9]。在自然界中,几乎所有的兰科植物均具有菌根,其生活始终必须部分或者全部依靠菌根提供养分,共生真菌对兰科植物的繁殖、生长起着重要的作用[10,11]。在野外环境下,大多数兰科植物种子必须与合适的共生真菌建立共生关系才能够萌发,共生真菌对兰科植物的种群恢复起着重要的作用。
根内真菌的群落结构不仅会影响到共生真菌而且也会对植物的生长和发育起到重要的作用。带叶兜兰多见于地生或者石上浮生,为半附生种类,对共生真菌依赖性较强[12]。采用传统分离培养技术评估植物根内真菌群落多样性是有限的,并非所有根内真菌都能够分离培养[13,14]。采用现代分子生物学技术从植物组织中提取总DNA,设计特异引物通过 PCR扩增和基因组分析,可以避免纯培养分析根内真菌的局限性[15],但这些技术仍然不能全面分析根内所有真菌的物种多样性。高通测量序(high-throughput sequencing)具有测序速度快、通量高的优点,已成为微生物种群结构及多样性分析的有效先进研究手段[16,17]。为此,本研究本着最小化损伤野生带叶兜兰的原则,收集野生带叶兜兰根段,利用Illumina Miseq测序平台对其根内真菌rDNA ITS基因可变区进行测序,结合生物信息学分析野生带叶兜兰根内真菌的群落组成,以期为研究带叶兜兰根内真菌和内生真菌的关系提供理论依据。
1 材料与方法
1.1 样本采集
野生带叶兜兰根样于2017年9月采自广西乐业雅长国家自然保护区内的兰园(LY)、黄琼洞(HJD)、邕木(YM)3个样点。带叶兜兰是濒危兰科植物,因此本研究选取了根系比较发达的植株,每个植株收集1条约5 cm长的根段。每个样点分3个采集处,每处收集4~6条根段混为一个样品处理。根样收集后,装于自封袋,适量填充原生境苔藓或腐殖质,带回实验室置于-80 ℃冰箱短期保存。
1.2 实验方法
1.2.1 根系总DNA的提取 将根系剪成2~3 cm长的小段,先用自来水冲洗干净,再用蒸馏水清洗,放入0.1%的升汞中浸泡5 min 左右;取出后用蒸馏水冲洗干净,再放入75%酒精中浸泡20 s左右,最后用蒸馏水冲洗干净,晾干后备用。
对根段表面消毒后,采用植物DNA提取试剂盒对根样总DNA进行提取,其操作流程按照试剂盒说明书进行。所提DNA的完整性采用1%琼脂糖凝胶电泳进行检测,同时采用Nanodrop紫外分光光度计(Thermo Fisher Scientifific,Wilmington,Delaware,USA)对DNA进行定量分析。
1.2.2 文库构建及测序 以50~100 ng DNA为模板,PCR扩增真菌ITS rDNA上的 ITS2 可变区,真菌通用引物ITS2F(5′-GTGAATCATCGARTC-3′)为前引物,ITS2R (5′-TCCTCCGCTTATTGAT-3′)为后引物,并为每个样本添加特异性Barcode序列,其中Ba-F(5′-ACCTGCGGARGGAT-3′)为前引物,Ba-R(5′-GAGATCCRTTGYTRAA-3′)为后引物;对各处理样本中的真菌ITS1区域进行扩增。使用Agilent 2100 生物分析仪检测文库质量,并且通过Qubit和实时定量PCR检测文库浓度。
DNA文库混合后,按Illumina MiSeq(Illumina,San Diego,CA,USA)仪器使用说明书进行2×300/250双端测序(PE);图像分析和碱基识别是通过MiSeq工具中的MiSeq Control Software (MCS)进行的,最后在Illumina basespace云端计算平台进行初始分类分析。上机测序和分析委托苏州金唯智生物科技有限公司进行。
1.2.3 数据分析 双端测序得到的正反向reads首先进行两两组装连接,过滤拼接结果中含有N的序列,保留序列长度大于200 bp的序列。经过质量过滤,去除嵌合体序列,最终得到的序列用于OTU分析。使用VSEARCH(1.9.6)进行序列聚类(序列相似性设为97%),比对ITS rRNA的参考数据库是UNITE ITS database (https://unite.ut.ee/)。然后用 RDP classifier (Ribosomal Database Program) 贝叶斯算法对OTU的代表性序列进行物种分类学分析,并在不同物种分类水平下统计每个样本的群落组成。基于OTU的分析结果,采用对样本序列进行随机抽样的方法,分别计算Shannon、Chao1等α多样性指数,并作出稀释曲线。
2 结果与分析
2.1 各样品内的OTU水平分析
本次实验利用第二代测序技术对带叶兜兰不同样点根样品中的真菌群落进行了测序研究,测序区域为ITS区段,测序长度在200~500 bp,样本最小平均长度352 bp,最大平均长度407 bp。共测得原始序列条数为1260490条,过滤掉低质量的序列并均一化后,得有效序列数952589条,并在99%相似度下将其聚类为用于物种分类的OTUs,统计得到所有样品在不同OTUs中的丰度信息,共产生1028个OTUs。其中HJD处理中1号样品的内生真菌所得OTUs数量最高,共测得557个OTUs;LY处理中3号样品中最少,仅有143个OTUs (表1)。
稀释曲线可直接反映不同样本的测序数据量的合理性,并间接反映各个样本中物种的丰富程度。从图1中可知,9个样品稀释曲线均基本趋于平缓,说明这些样本基本覆盖了绝大多数的真菌,其真菌群落具有多样性,此次所得序列可基本反映真实环境中真菌群落结构。但稀释曲线仍未达到完全饱和,说明仍有部分真菌种类尚未被发现(图1)
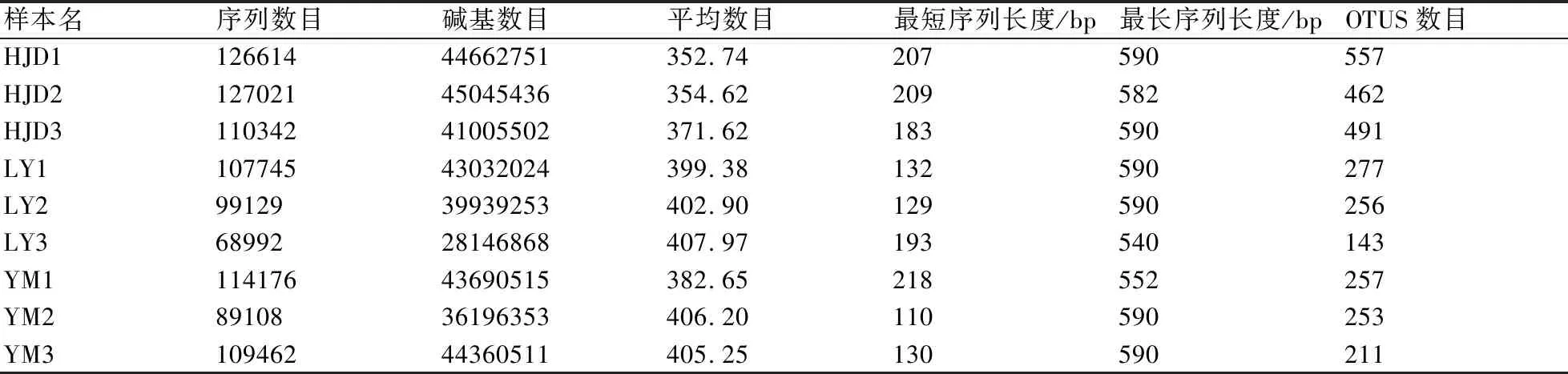
表1 各样品测序数据

图1 各样品的稀释曲线
2.2 各样品中内生真菌群落相关性分析
以各样本点中分布的OTUs数为计算依据,构建韦恩图(图2)。图中每个圆形代表一个样本点中的3个样品的真OTUs总数,中间的共有区域的数字代表所有样品共有的OTUs数目,圆形上的数字代表该样品特有的OTUs数目。如图2所示,带叶兜兰菌根中总共包含2096个OTUs,其中,以HJD处理样品中的OTUs最多,包含1075个OTUs;YM处理样品中的OTUs次之,包含559个OTUs;LY处理样品中的OTUs最少,包含462个OTUs。有271个OTUs同时出现在YM和LY处理中,381个OTUs同时出现在HJD和LY处理中,498个OTUs同时出现在YM和HJD处理中。254个OTUs在所有处理中均有出现,占21.16%,说明实验选取的带叶兜兰根样品中,存在一定的相似内生真菌群落组成。
2.3 各样品中内生真菌多样性及群落结构差异分析
采用Alpha多样性指标中的Coverage指数、Shannon指数和Chao1指数来分析各样品内生真菌的丰富度和多样性。结果表明,同一样本点的不同样品之间Shannon指数存在一定差异,不同样本点之间也存在差异,说明带叶兜兰菌根真菌的群落分化程度较高。从群落丰度Chao1指数和OTUs数量来看,HJD1>HJD3>HJD2>LY1>YM1>YM2>LY2>YM3>LY3,说明HJD样点的带叶兜兰根部的内生真菌群落分化的程度更高。
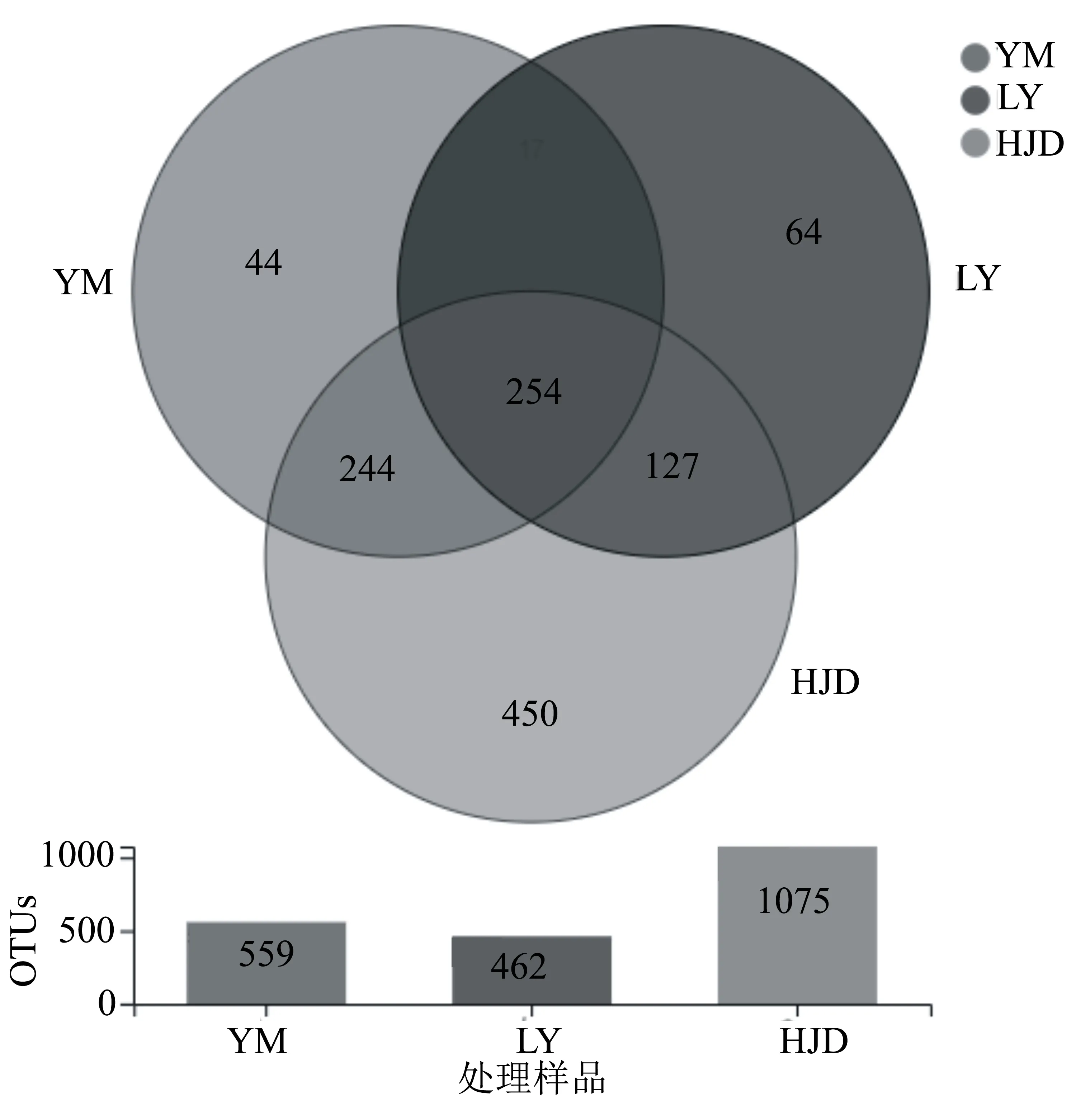
图2 各样本中内生真菌群落相关性分析
PCA的横坐标表示第一主成分,百分比则表示第一主成分对样品差异的贡献值;纵坐标表示第二主成分,百分比表示第二主成分对样品差异的贡献值;图3中的每个点表示一个样品,同一个组的样品使用同一种颜色表示。主成分1(PC1)和主成分2(PC2) 对样品差异性贡献率分别达到38.30%和24.21%,合计达到62.51%。由各样品在主成分中的位置可以看出,LY、YM在一个区域内,且距离极为接近,表明两者之间差异较小;而HJD与LY、YM则存在明显差异。HJD的组内样品分布比较松散,样品间的间隔较大,说明组内样品的差异显著(图3)。以上结果表明,实验选取的带叶兜兰根样品中,虽存在相似的内生真菌组成,但各样本点间仍存在一定差异,且可能与各样本点的带叶兜兰生长环境有关。HJD样点的带叶兜兰多生长在石头上,根系生长环境的均一性较差,更容易受到周围环境、水分等多中因素的影响,从而造成真菌对宿主组织或生长环境的选择偏好性,导致带叶兜兰根样本点间也存在较为明显的差异。
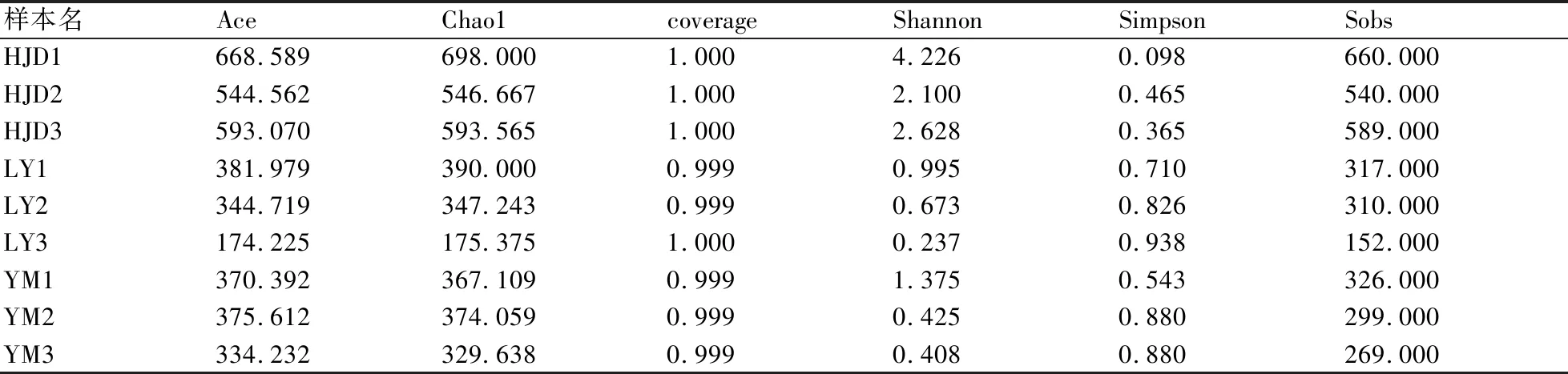
表2 各样点的Alpha多样性
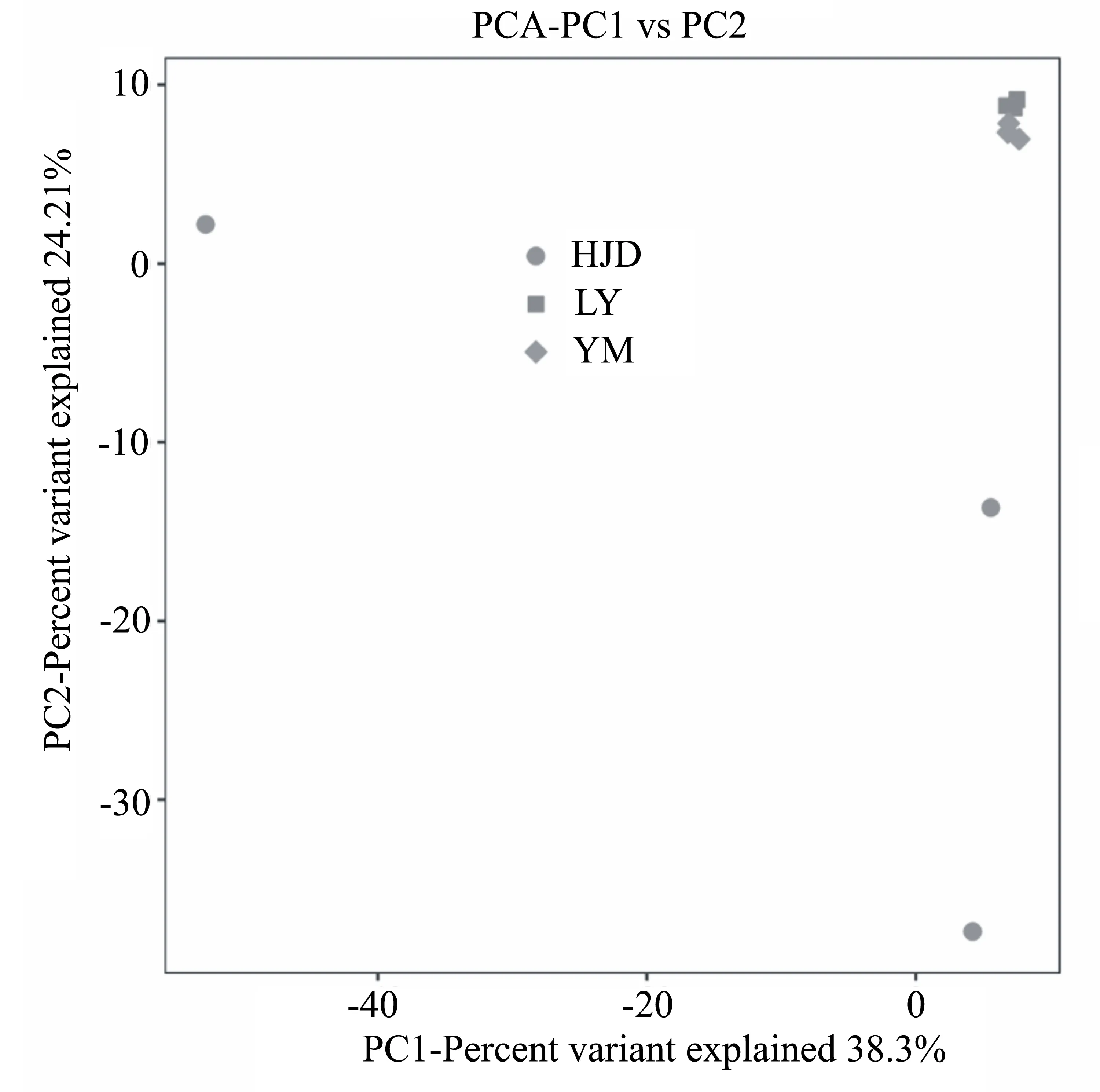
图3 各样点间根内真菌群落主成分分析
2.4 各样品中内生真菌群落结构组成分析
利用RDP classifier对各样品中OTU依次在门(phylum)、纲(class )、目(order)、科 ( family )、属(genus)水平信息上进行真菌物种分析,挖掘样本点间的群落结构组成。结果表明,带叶兜兰根内生真菌群落共涉及6个门、21个纲、46个目、96个科、143个属、217个种及大量未分类种类(表3)。
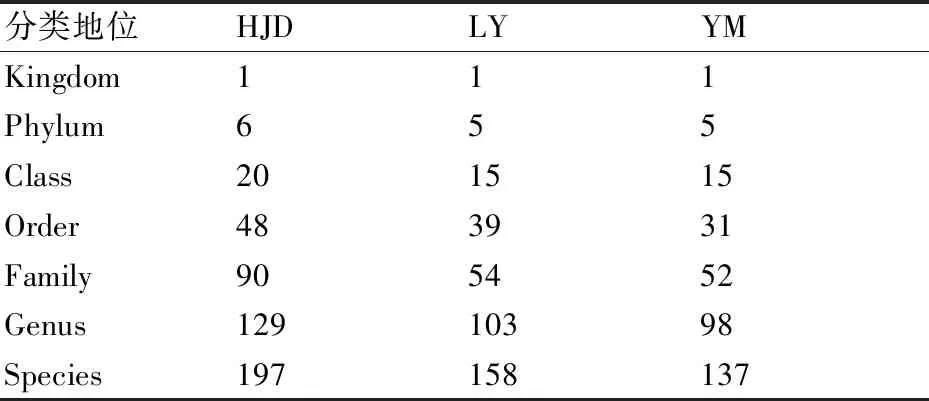
表3 各样点的根内真菌群落组成
在门的水平中,共鉴定出21个纲,对其中20个主要纲进行分析发现,优势菌群包括座囊菌纲(Dothideomycetes)、粪壳菌纲(Sordariomycetes) 、散囊菌纲(Eurotiomycetes)、伞菌纲(Agaricomycetes)、酵母菌纲(Saccharomycetes)、微球黑粉菌纲(Microbotryomycetes)、银耳纲(Tremellomycetes)以及一些未知菌目。座囊菌纲(Dothideomycetes)、粪壳菌纲(Sordariomycetes)、散囊菌纲 (Eurotiomycetes)及伞菌纲(Agaricomycetes)为各样品中的优势菌群。古菌根菌纲(Archaeorhizomycetes)、圆盘菌纲(Orbiliomycetes) 、节担菌纲(Wallemiomycetes)在HJD样品中分布较多(图4)。此外,带叶兜兰根样中存在着大量未分类菌属。
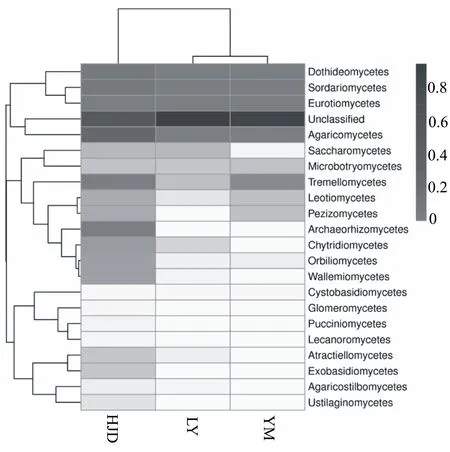
图4 纲水平的带叶兜兰菌根真菌群落组成
在纲水平中共鉴定出46个目,含量最高的前30个目中包括了伞菌目(Agaricales) 、革菌目 (Thelephorales) 、多孔菌目(Polyporales) 、煤炱目(Capnodiales) 、假球壳目(Pleosporales)、葡萄座腔菌目(Botryosphaeriales) 、肉座菌目(Hypocreales)、散囊菌目(Chaetothyriales)、曲霉目 (Eurotiales)等以及一些未知菌纲(图5)。其中,肉座菌目 (Hypocreales)、散囊菌目(Chaetothyriales)、曲霉目 (Eurotiales)在3个样点中均有分布,红菇目(Russulales)在YM样点中分布较多,葡萄座腔菌目(Botryosphaeriales)、炭角菌目(Xylariales)、古菌根菌目(Archaeorhizomycetales)等在HJD样点分布较多(图5)。此外,带叶兜兰根样中还存在着大量未分类菌属。
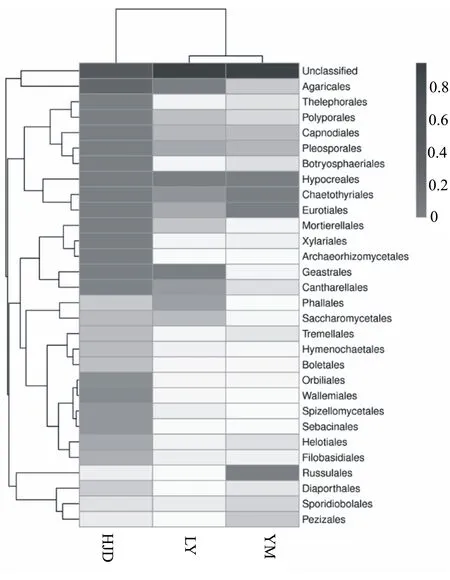
图5 目水平上的带叶兜兰菌根真菌群落组成
进一步分析发现,在属水平上,含量最高的前30个目中包括了木霉属(Trichoderma)、湿伞属(Hygrocybe)、青霉属(Penicillium)、外瓶霉属(Exophiala)、地星属(Geastrum)、散囊菌属(Phaeomoniella)、胶膜菌属(Tulasnella)、镰刀菌属(Fusarium)等以及一些未知菌属。其中大多数菌属都在HJD样点中有分布,裸腹菌属(Gymnomyces)、散囊菌属(Phaeomoniella)在YM样点含量较高(图6)。
由此可见,不同样点的带叶兜兰根内生真菌群落结构组成相近,但各样点间菌群含量则存在明显差异,HJD样点的菌群含量明显与LY和YM的不同,LY和YM样点的菌群含量较为接近。这与上述多样性分析、差异性分析、主成分分析结果一致。
3 结论与讨论
根内微生物组成与植物种类、生长时期、组织类型等密切相关[18,19]。此外,植物生长地理位置、降雨量、日照时间、土壤等环境因素也间接影响内生微生物群落的形成[20,21]。HJD样点3采集处的群落多样性差异较大,该样点的带叶兜兰多为石生,可能与土壤环境的差异较大有关。YM和LY样点采集处的群落多样性也表现出一定的差异,可能是由于此两处的带叶兜兰生长数量较为密集,生长空间的大小可能对群落多样性的贡献更大。HJD样点与YM、LY样点相比,不管是群落多样性还是群落组成都要高,且YM、LY样点带叶兜兰多为成片密集生长,这可能表明在小空间尺度上,带叶兜兰的种群密度可能在一定程度上影响了根内真菌的多样性和群落组成。
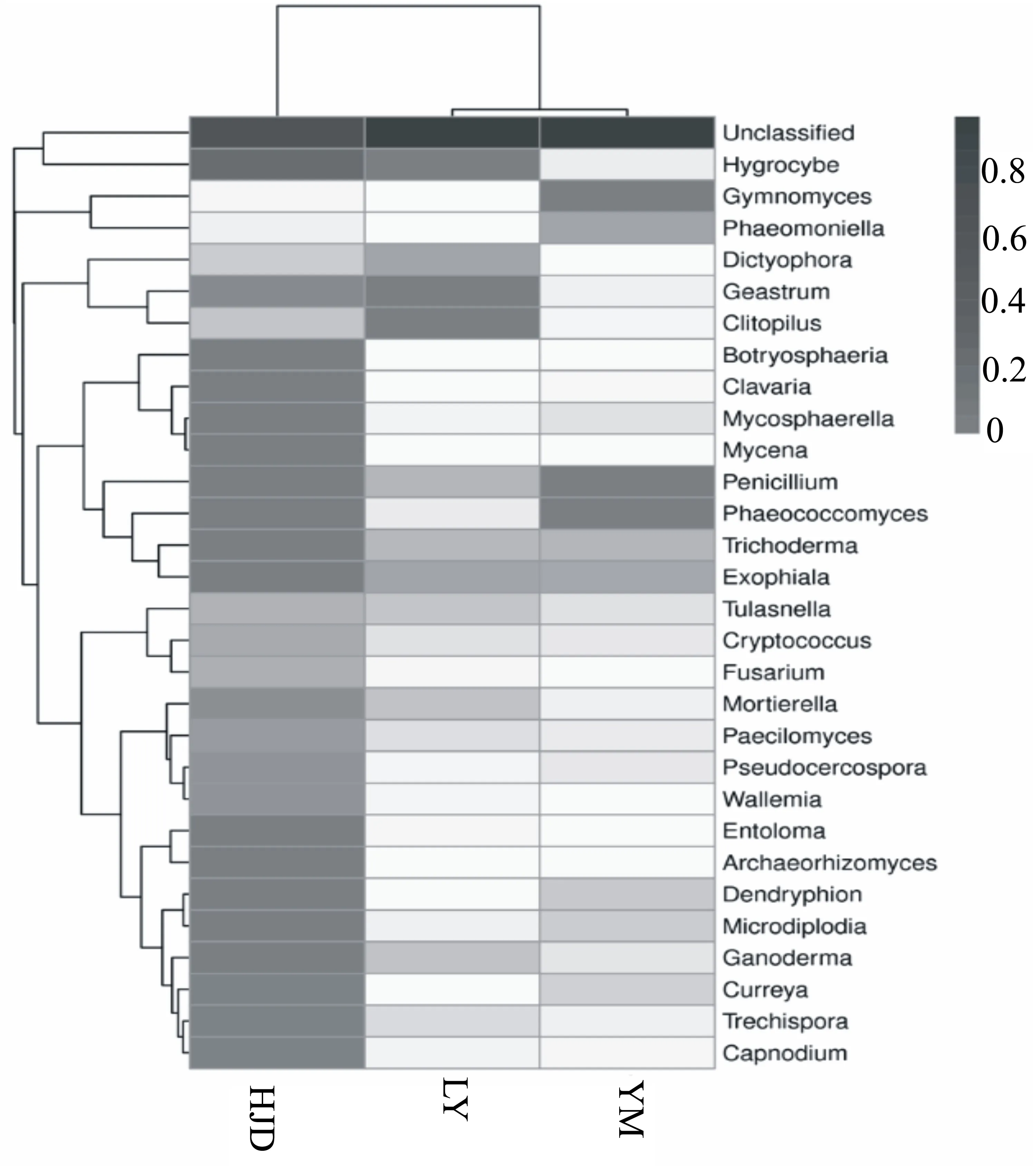
图6 属水平上的带叶兜兰菌根真菌群落组成
带叶兜兰根内真菌群落由一些常见真菌以及部分未知菌属组成。在属水平上,木霉属(Trichoderma)、湿伞属(Hygrocybe)、青霉属(Penicillium)、外瓶霉属(Exophiala)、地星属(Geastrum)、散囊菌属(Phaeomoniella)、胶膜菌属(Tulasnella)、镰刀菌属(Fusarium)等属为优势菌群,这与前人的研究结果[22,23]略有区别,这种区别可能是由生长地域的差异所致[24-26]。与兰科植物形成菌根的共生真菌多为担子菌门的蜡壳菌(Sebacinaceae)、角担菌(Ceratobasidiaceae)、胶膜菌(Tulasnellaceae)、革菌(Thelephoraceae)、锈病菌(Pucciniomycotina)、镰刀菌(Fusarium)等真菌[6]。通过传统组织分离培养法鉴定到的带叶兜兰共生真菌多为胶膜菌(Tulasnellaceae)、角担菌科(Ceratobasidiacea)真菌及少数镰刀菌属(Fusarium)真菌[7]。本研究中优势菌群中仅有镰刀菌和胶膜菌为兰科共生真菌的菌属,其余菌属并未鉴定到,这不代表此次实验中其余兰科共生真菌没有定殖到带叶兜兰中。植物总DNA的提取方法、测序的深度和OTU的丰度值及数据过滤等极有可能将一些含量较少的兰科共生真菌的菌属剔除。分离培养的方法可以分离到部分根内真菌,同时也会低估真菌的多样性。而非培养的方法虽然得到不可培养的真菌,但较培养的方法能够更加全面地了解真菌的多样性和群落结构。共生真菌不管是在种类还是在含量上只占根内真菌的一小部分,除了一些明确的腐生和致病真菌外,其他大多数真菌的种类和含量还是未知,对这些未知真菌确切的生物学功能并不清楚,它们对维持整个根内真菌的多样性及共生真菌种类和数量上的功能也需要深入研究。
