基于WGCNA 挖掘谷子淀粉合成相关基因
2021-03-11胡萌萌张丽玲张雅坤郄倩茹任碧云侯思宇朱喆标高建华王海岗李旭凯韩渊怀
胡萌萌,张丽玲,张雅坤,郄倩茹,任碧云,侯思宇,朱喆标,高建华,王海岗,李旭凯*,韩渊怀*
(1.山西农业大学 农学院,山西 太谷030801;2.山西农业大学 生命科学学院,山西 太谷030801;3.山西省农业科学院 农作物品种资源研究所,山西 太原030000)
谷子属于禾本科狗尾草属,具有耐旱、耐贫瘠、耐贮藏的优点,去皮后称为小米,以其优良的营养价值和口感风味广受欢迎[1]。小麦、水稻、玉米、谷子等谷类作物以淀粉为主要成分,因此成为目前最主要的几种粮食作物。
Waxy基因(简写为Wx)编码植物淀粉合成的关键酶即颗粒结合型淀粉合成酶(Granule-Bound Starch SynthaseⅠ,GBSSⅠ),控制作物籽粒胚乳中直链淀粉的合成。其中直链淀粉含量(Amylose content,AC)是决定作物籽粒食味和蒸煮品质的最重要因素之一[2~5],AC 过高的谷物其食味品质往往较差。研究表明,在水稻驯化过程中,全球范围内存在着从高到低的AC 选择趋势[6]。最新研究发现通过微调Waxy基因可以不同程度上调节水稻的AC,为进一步改良水稻品质提供支撑。李加瑞等人利用RNA 沉默技术对从小麦中分离得到的Waxy基因进行转化,验证了Waxy基因直接影响小麦直链淀粉的含量[7]。目前在多种植物中的Waxy基因 已被克隆[8~10],但对谷子的Waxy基因却缺乏研究。
WGCNA(Weighted Gene Co-Expression Network Analysis,加权基因共表达网络分析),是一种分析多样本基因表达模式的分析方法,可将表达模式相似的基因聚类,并分析模块与特定性状或表型之间的关联关系。在实际应用层面,由于基因共表达网络分析作为一种挖掘和呈现基因在不同样本中表达形式的有效方法,可以鉴定高度共表达的基因模块,模块的特征值或模块中包含的关键基因(hub genes:即与基因网络中的其它基因联系最紧密,在基因网中起关键作用的基因)可用于提炼模块信息,以此深入探讨基因模块或模块中的关键基因和关注的样本特征间的关联关系[11]。
本试验对谷子Waxy基因特征与蛋白质特性进行分析,并与其它5 种禾本科粮食作物的Waxy基因进行聚类比较。对Waxy基因在谷子中的表达模式进行分析,并以名优谷子品种晋谷21 为材料,构建淀粉合成相关基因的共表达网络。本研究结果可为进一步选育优质谷子提供一定的理论参考与依据。
1 材料与方法
1.1 试验材料
转录组数据:2019 年4 月24 日于山西农业大学谷子研究试验基地(山西省太谷区,北纬37°25′13″,东经112°35′26″)种植25 m2晋谷21,2019年8 月27 日采集晋谷21 的灌浆5 个时期籽粒(S1:灌浆初期,外观呈鲜绿色,内含物呈水乳状;S2:灌浆前期,谷子外观呈暗绿色,胚形成且处于乳状内含物中;S3:灌浆中期,谷子外观呈黄绿色,营养物质积累,内含物固化,胚与粉状内含物可分离;S4:灌浆后期,外观呈暗黄色,籽粒质地硬化,内含物较充实,胚与内含物难分离;S5:灌浆末期:外光呈深黄色,谷壳干燥变脆,籽粒成熟),采集方法为,辨别每粒谷子所处灌浆时期,用镊子夹取放入置于液氮中的离心管中,每5 个单株为一个重复,每个重复取3 个离心管。灌浆中期的根、茎、旗叶、倒3 叶及萌发期芽长2 mm 的整颗幼苗各取3 个重复。RNA 提取及转录组测序由诺禾致源公司完成。
1.2 试验方法
1.2.1 谷子Waxy基因获取
在国家水稻数据中心(http://www.ricedata.cn/)下载水稻Waxy基因序列、CDS 序列、蛋白序列和转录起始位点上游2 000 bp 序列。基于谷子多组学数据库(http://sky. sxau. edu. cn/MDSi.htm),比对出谷子的Waxy基因信息并下载相应的序列。
1.2.2Waxy同源基因获取
利用NCBI(https://www.ncbi.nlm.nih.gov)分别获取糜子(Panicum miliaceum)、高粱(Sorghum bicolor)、玉米(Zea mays)、小麦(Triticumaestivum)的蛋白序列文件、注释文件以及基因组文 件,通 过Tbtools[12]将 水稻 的Waxy 蛋白序列比对到该4 个物种蛋白序列文件获取Waxy基因的基因序列、CDS 序列、蛋白质序列、转录起始位点上游2 000 bp 序列。
1.2.3Waxy同源基因聚类分析
利用Clustal X2[13]对6 个物种的Waxy基因蛋白序列进行多重序列比对,并在MEGA 7[14]中以Neighbor-Joining 方法构建系统发育树。以GSDS2.0 工具[15]对Waxy基因的基因结构进行分析。将Waxy 蛋白序列提交至Multiple Em for Motif Elicitation 工具(MEME,Version 5.0.3)[16]分析其蛋白质基序。用PFAM 数据库对Waxy 蛋白的保守结构域进行分析,并使用Tbtools 工具进行可视化。
1.2.4Waxy基因启动子顺式作用元件分析
将Waxy基因转录起始位点上游2 000 bp 序列作为启动子区域提交至PlantCARE(http://bioinformatics. psb. ugent. be/webtools/plantcare/html/)分析启动子作用元件,基于Tbtools 软件进行定位预测。
1.2.5 谷子Waxy基因表达模式分析
将谷子Waxy基因信息输入谷子多组学数据库(http://sky. sxau. edu. cn/MDSi. htm),得到Waxy基因在谷子各组织时期的表达模式。
1.2.6 淀粉基因共表达网络
对转录组数据进行筛选过滤,将得到的clean reads 与参考基因组进行序列比对,基于比对结果进行基因表达量分析,根据各样本中基因差异表达规律来进行聚类,根据无尺度网格确定软阈值(Soft thresholding power)从而划分模块,提取出含淀粉合成基因最多的模块,利用Cytoscape(3.7.1)[17]对该模块进行加权基因共表达网络的绘制。
2 结果与分析
2.1 谷子Waxy 基因及禾本科植物Waxy 基因聚类分析
在phytozome 数据库、NCBI 数据库及MDSi数据库比对共得到13 条Waxy基因,其中包括水稻(LOC_Os06g04200),高粱(Sobic. 010G022600、Sobic. 002G116000),谷子(Si4g02980、Si2g12020),玉米(GRMZM2G024993、GRMZM2G008263),小麦(Traes_2DL_E4B86D0C3、Traes_2AL_06AD35739、
Traes_2BL_25DAD57C9、Traes_4AL_4B9D56131)、糜子(PM09G01630、PM10G01650)。Waxy 蛋白系统进化树如图1(左)所示,主要分为2 部分。多序列比对结果表明,氨基酸序列均在600 aa 左右,13 条氨基酸序列均高度相似。Waxy基因结构(图1左)分析显示,多数基因包含有内含子和外显子结构,呈现断裂基因特征,其中多数基因含有13 个外显子,且基因结构均相似。
Waxy 蛋白质保守基序结果(图1 中)表明,Waxy 蛋 白 共10 个motif,其 中Traes_4AL_4B9D56131和PM10G01650 不含有motif9,其余均含有motif 1-motif 10,且排列高度一致,基序的保守性表明这些Waxy 蛋白在功能上具有较高的相似性。
保守结构域分析结果(图1 右)显示,共有2 个保守结构域,分别为Glyco_transf_5(淀粉合成酶催 化 区,Starch synthase catalytic region)和Glycos_transf_1(糖基转移酶,glycosyl transferase)。保守结构域的排列同样相似,其中Traes_4AL_4B9D56131和PM10G01650不 含 有Glycos_transf_1结构域,但含有Glycos_transf_1_4,与GT_1 为同一家族,具有相同功能。Waxy 蛋白具有如上特征,表明其序列上具有植物颗粒结合型淀粉合成酶特征。综上所述,Waxy 蛋白在各物种间的保守性很强,在进化过程中几乎不受影响。

图1 Waxy 基因结构及蛋白质特征Fig.1 Structure and protein characteristics of Waxy gene
2.2 Waxy 基因启动子顺式作用元件分析
对转录起始位点上游2 000 bp 的启动子顺式元件分析发现(图2),13 个Waxy基因启动子中与淀粉合成相关的元件主要有:参与胚乳表达顺势调控元件(GCN4_motif)、植物激素响应元件(ABRE、P-box、TCA-element)。不同基因的调控元件种类和数目不同,其中水稻LOC_Os06g04200的启动子元件最多,高达20 个,小麦Traes_2AL_06AD35739基因最少,仅含有9 个。此外,每个Waxy基因都包含较多植物激素响应顺式作用元件,表明植物在淀粉合成中存在多种植物激素的参与和调节。
2.3 谷子Waxy 基因表达模式分析
对谷子2 个Waxy基因表达模式分析发现(图3,图4),基因Si4g02980在谷子灌浆期及成熟后的籽粒中表达量很高,说明其在籽粒淀粉合成中可能起重要作用,而基因Si2g12020在抽穗期的倒二叶片中的表达量较高,而在籽粒中的表达较低,说明其可能在谷子功能叶片淀粉合成中起重要作用。
2.4 谷子淀粉合成相关基因的共表达模块分析
2.4.1 基因共表达网络构建
对RNA-Seq 数据进行筛选,过滤掉在超过半数样本中没有表达的低质量的基因后,共获得31659 个有效基因用于创建基因表达矩阵。设置R2为0.9,选取power=9 来计算两两基因间的TOM 矩阵,结果共得到37 个模块,模块中基因个数2~5 586 不等(图1~图4)。
2.4.2 模块的GO 富集分析
选取含淀粉合成基因数目多的2 个模块,对2个模块进行GO 功能富集分析,富集结果涉及到了生物学过程(biological process),细胞组分(cellular component)和分子功能(molecular function)。部分富集结果(表1)显示,bisque4 模块富集到了葡聚糖生物合成的过程(GO:0009250)、丙酮酸生物合成的过程(GO:0042866)、糖酵解过程(GO:0006096)等相关的调控通路。skyblue 模块富集到多糖生物合成的过程(GO:0000271)、丙酮酸生物合成的过程(GO:0042866)和淀粉生物合成的过程(GO:0019252)等相关的调控通路。

图2 Waxy 基因启动子顺式作用元件Fig.2 Waxy gene promoter cis acting element

图3 谷子Si4g02980 基因表达模式Fig.3 Expression pattern of Si4g02980 gene in foxtail millet
图4 谷子Si2g12020 基因表达模式Fig.4 Expression pattern of Si2g12020 gene in foxtail millet
2.4.3 互作网络的构建及核心基因功能注释
利用Cytoscape 软件对bisque4 和skyblue 模块进行网络可视化(图5),选取网络中心权重值较高的基因作为核心基因共得到17 个,在水稻数据库及拟南芥数据库中对其进行功能注释(表1),其中含有8 个淀粉合成酶基因,3 个葡萄糖合成酶基因及一些蛋白激酶和调控蛋白基因。谷子Waxy基因Si4g02980也作为核心基因存在于网络中,证明了WGCNA 方法在对数据分析中具有较高的可靠性。
2.4.4 模块候选基因及功能注释

表1 模块核心基因功能注释Table 1 Functional annotation of modular hub genes
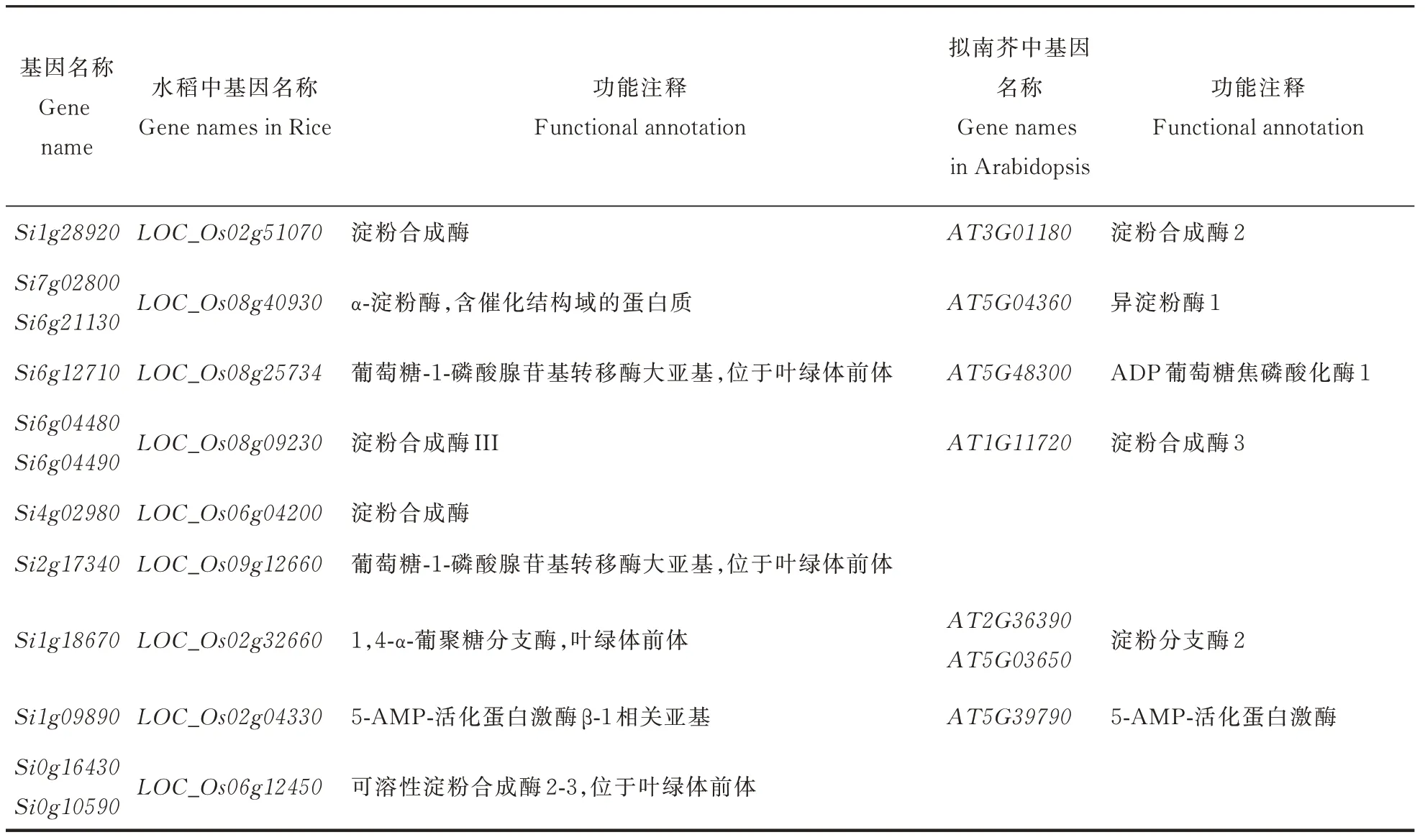
续表

图5 bisque4、skyblue 模块相关基因网络图Fig.5 Genetic network diagram of bisque4 and skyblue module
筛选与核心基因高连通性的基因,得到候选基因,在bisque4 模块中找到10 个候选基因,skyblue 中有7 个,对这17 个候选基因在水稻及拟南芥数据库中进行功能注释。注释结果如表2 所示,这些基因主要编码蛋白质锌指结构域和结合蛋白,其中,谷子Si8g15270基因与水稻中的LOC_Os02g15350基因同源,其编码醇溶谷蛋白框结合蛋白(Rice prolamin box binding factor,RPBF),该蛋白可调节种子贮藏蛋白基因的表达,调节种子中蛋白、淀粉和脂类含量,在水稻籽粒灌浆期中发挥重要作用[18]。其它候选基因的研究报道较少,需进一步功能验证。
3 讨论与结论
通过对谷子及其它5 种禾本科作物Waxy同源基因及同源蛋白质基序的研究发现,虽然基因结构的差异较大,氨基酸序列有所不同,但基本的结构域是保守的,这说明在进化过程中尽管基因结构有一定的变化,但其蛋白质的变化较小。2 个保守结构域与其它物种的研究结果一致[19,20]。
目前对于Waxy基因在水稻种的研究已较为深入。最新研究通过CRISPR/Cas9 技术编辑水稻Waxy基因启动子上的关键顺式作用元件,明确了一个可定量调节Waxy基因表达的启动子靶位点,创建了新的Waxy等位基因,在不同Waxy等位基因中编辑该位点将有机会获得更多微调AC 的新Waxy等位基因。研究也表明编辑基因的核心启动子可能是一种适度调节目的基因表达的通用型方法,同样也可以应用于其他作物中,为分子育种工作提供了新颖的思路。
本文通过对谷子与其它5 种禾本科作物Waxy基因的分析,明确了禾本科作物Waxy基因结构的多样性及其氨基酸序列的保守性;基于不同组织表达模式分析推断Si4g02980是谷子籽粒淀粉合成的主效基因;利用27 份转录组数据进行的WGCNA分别获得一批包括转录因子的关键基因,可为深入探索谷子及禾谷类淀粉合成调控提供一定的理论依据。
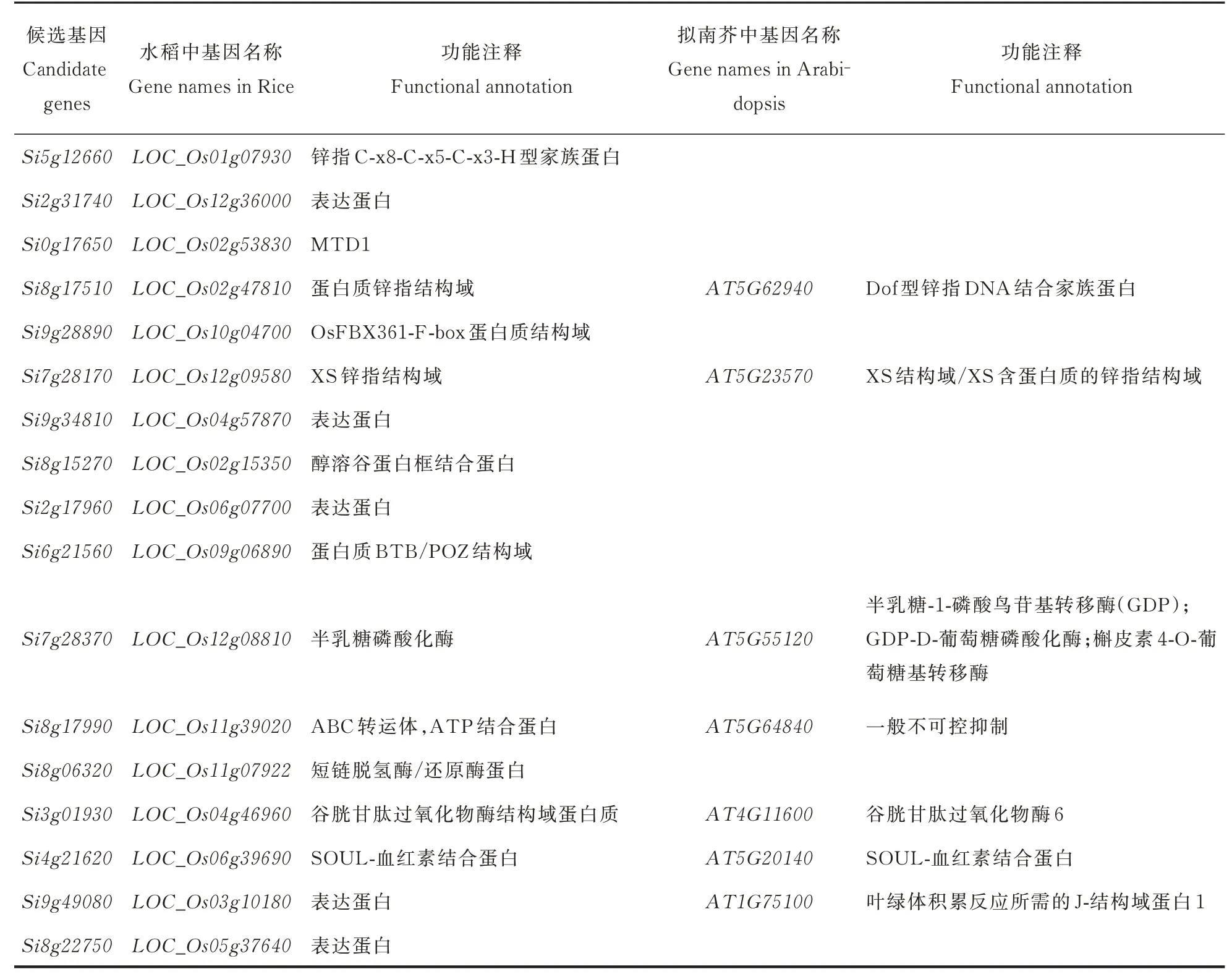
表2 候选基因功能注释Table 2 Functional annotations of candidate genes
