食品中常见病原微生物的DNA条形码识别技术
2021-03-10钟文涛王淑好王芳妹
钟文涛 徐 越 王淑好 王芳妹
(1. 国家农副产品质量监督检验中心,湖南 长沙 410007;2. 湖南省产商品质量监督检验研究院,湖南 长沙 410007)
加拿大动物学家Hebert等[1]于2003年提出“DNA条形码”的概念,希望用“标准化”的基因片段(如COI基因)作为物种快速识别的标记,建立起物种名称和生物实体之间一一对应的关系。经过10余年的发展,科研人员将DNA条形码与基因测序技术相结合,在动植物的相似种识别方面已经做了大量工作[2],在国际上建立了多个与DNA条形码相关的合作组织和大型数据库[3],取得了初步成效。Buddhachat等[4]评估了条形码技术与传统形态学鉴定技术在鱼类分类上的差异,认为DNA条形码更为准确、高效;Xing等[5]使用COI基因对中国市场上销售的各种动物源食品的标签错误问题进行了调查,发现23%的产品标签存在瑕疵;Liu等[6]为识别中国药典中有毒药用植物及其掺假物,对比了4种候选DNA条形码的识别效率,发现ITS2基因可用作通用条形码。
对微生物DNA条形码的研究虽有文献[7-8]报道,但相较动植物而言则显得滞后。究其原因是由于微生物具有物种的多样性和基因的高突变性,找到稳定可靠的保守基因片段作为研究目标具有一定难度[9]。现有文献[10-11]报道发现适用于原核生物DNA条形码研究的目标基因有16SrRNA、COI和cpn60,其中16SrRNA在各大数据库中资源最为丰富,因此对其的研究也相对集中。
在食品检验领域,DNA条形码在肉制品、海产品真实性鉴定上已有应用[12],但对食品中病原微生物的相关研究尚未开展。研究选择16S rRNA的V3~V6区域为目标基因片段,对食品中常见病原微生物及其表型类似株的序列进行分析,目的是建立一种基于DNA条形码技术的快速、准确的识别方法。
1 材料与方法
1.1 材料与试剂
Chelex 100:美国Bio-Rad公司;
2×PCR Master mix、DNA Marker、Agarose Gel DNA Extraction Kit:宝生物工程(大连)有限公司;
电泳级琼脂糖:生工生物工程(上海)股份有限公司;
8种常见病原微生物(见表1)和15种表型类似株(大肠埃希氏菌、奇异变形杆菌、普通变形杆菌、弗氏柠檬酸杆菌、产气肠杆菌、斯氏李斯特氏菌、英诺克李斯特氏菌、伊氏李斯特氏菌、枯草芽孢杆菌、蕈状芽孢杆菌、巨大芽孢杆菌、溶藻弧菌、霍乱弧菌、荧光假单胞菌、表皮葡萄球菌):ATCC、CICC、CMCC菌株库和广东环凯微生物科技有限公司。
1.2 仪器与设备
PCR仪:C1000 Touch型,美国Bio-Rad公司;
核酸蛋白测定仪:SmartSpec plus型,美国Bio-Rad公司;
电泳仪:powerpac universal型,美国Bio-Rad公司;
凝胶成像系统:GelDoc XR+型,美国Bio-Rad公司;
高速离心机:MiniSpin Plus型,德国Eppendorf公司。
1.3 菌株纯化及核酸提取
菌株活化后用非选择性培养基划线分离,挑取单个菌落接种增菌肉汤,(36±1) ℃培养(24±2) h。将菌悬液振荡混匀后,取1.5 mL在8 000 r/min下离心5 min,弃上清液,使用适量的无菌生理盐水洗菌3次,最后一次离心时尽量吸干剩余液体。在沉淀物中加入30 μL 5%的Chelex 100溶液并使其重新悬浮,然后在100 ℃沸水中保持10 min,立即置于冰上冷却1 min,12 000 r/min离心10 min,所得上清液作为扩增模板,-20 ℃保存备用。
1.4 引物设计
16S rRNA包含9个V区(可变区,Variable region)和10个C区(保守区,Conserved region),且V区和C区交替排列[13-14]。设计DNA条形码通用引物时应考虑将上下游引物落在C区,中间包含一个或数个V区。在GeneBank中搜索表1中8种病原微生物的16S rRNA基因组编码序列,选择尽可能完整的序列,目的是包含所研究的V3~V6区域(起止位置为433~1 043,共计610 bp[15])。将序列导入到DNASTAR软件中,通过MegAlign功能分析菌种间16SrRNA基因的同源性和变异性,用Primer Premier 6设计适合在不同菌种间扩增的通用引物。共测试了两对通用引物(表2),其中通用引物1为项目组自行设计,通用引物2为项目组对文献[15]中的引物进行改良所得。引物序列由宝生物工程(大连)有限公司合成。
1.5 PCR扩增
通用引物PCR反应在20 μL体系中进行:2×PCR Master mix 10 μL、10 μmol/L正反引物各0.5 μL,DNA模板1 μL,无菌水8 μL。PCR反应条件为:94 ℃预变性3 min,94 ℃变性30 s,60 ℃退火30 s,72 ℃延伸150 s,循环35次,72 ℃再延伸5 min。取5 μL反应产物进行1%的琼脂糖凝胶电泳,用凝胶成像系统分析扩增产物。将琼脂糖凝胶上的电泳条带切下,按照琼脂糖DNA纯化试剂盒说明书进行提取纯化后,送至宝生物工程(大连)有限公司进行基因测序鉴定。
1.6 数据处理
将测序所得的序列除去首尾各30 bp碱基,选择合适的数据库进行序列比对。GenBank是最为常用的比对数据库,序列资源非常丰富,但其质量良莠不齐;而RDP是基于细菌和真菌的核糖体序列信息建立的专业数据库,最新版本已包含3 356 809条16S rRNA信息,可提供足够可靠的比对信息。采用GenBank的Blastn功能,结合RDP的Classifier和Sequence Match功能,对测序结果进行综合分析。

表1 菌种信息及其16S rRNA序列片段编号
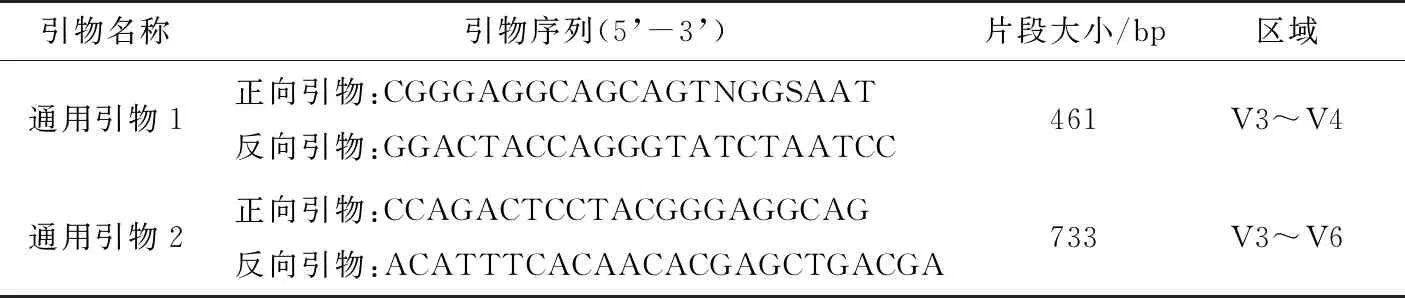
表2 通用引物序列
1.7 干扰菌测试
按照1.3中描述的试验步骤,对15种干扰菌进行纯化并提取核酸。使用表2中的2对通用引物分别进行PCR扩增,后续PCR扩增按照1.5中所述的步骤进行,数据处理按照1.6中所述执行。
2 结果与分析
2.1 电泳图谱
由于所扩增的23种测试菌株分别属于4个“目”、6个“科”及11个“属”,为了实现扩增效率的最大化,在设计通用引物时使用了简并碱基。从图1和图2可以看出,试验条件下,通用引物1和2对所有目标片段均进行了有效的扩增,电泳条带清晰明亮,大小符合预期,没有发生引物二聚体的现象,说明通用引物的适用性较强。
2.2 序列比对
经比对,在GenBank数据库中,通用引物1扩增的序列Percent Identity最低值为97.96%,通用引物2扩增的序列Percent Identity最低值为98.91%;经RDP数据库的Classifier检索,所有序列的“门、纲、目、科、属”信息匹配率为100%,通用引物1扩增的序列在Seqmatch score最低值为0.983,通用引物2扩增的序列在Seqmatch score最低值为0.972。
因此,不论是在GenBank数据库还是在RDP数据库进行检索,序列的匹配率均符合预期值,所有菌种的序列均能准确鉴定到“属”水平,部分能准确鉴定到 “种”水平(见表3)。通用引物1和通用引物2的扩增产物虽然长度相差近300 bp,但是对试验所涉及的目标菌的识别能力基本一致,仅在福氏志贺氏菌的比对结果上存在差异。
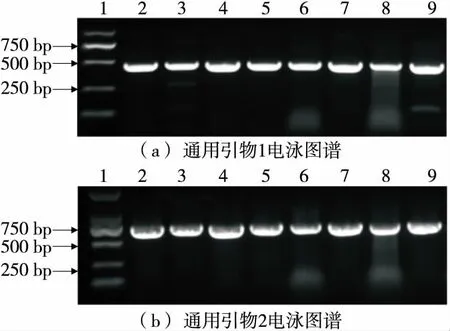
1~9泳道分别为DNA Marker、鼠伤寒沙门菌、阪崎肠杆菌、福氏志贺氏菌、副溶血性弧菌、铜绿假单胞菌、单核细胞增生李斯特氏菌、蜡样芽孢杆菌、金黄色葡萄球菌
2.3 菌种识别
根据表3所列信息,对8种常见的病原微生物及相关15种表型类似菌株按所属“目”“科”进行分类后,统计DNA条形码序列在不同分类水平上的菌种识别能力,如图3、4所示。可以发现,16S rRNA的V3~V6区域能较好地鉴别弧菌目、假单胞菌目和芽孢杆菌目的葡萄球科相关菌种,可明确区分其中的致病菌和非致病菌;而对芽孢杆菌目中的李斯特氏菌科、芽孢杆菌科中的相关菌种只能鉴别到“属”水平,还需要借助其他技术手段才能达到区分的目的;对肠杆菌科目肠杆菌科的菌种识别中,沙门氏菌属和志贺氏菌属不能鉴定到“种”水平,但可以明确区分肠杆菌科的其他属,由于食品中分离出的沙门氏菌属和志贺氏菌属均属于致病菌范畴,因此也能达到病原微生物识别的目的。
部分菌种仅能识别到“属”水平,在“种”水平给出了多个可能的鉴定结果,可能由于这些菌种在V3~V6区域的序列过于近似,引物设计时可考虑扩增其他一个或几个可变区域。
DNA条形码结合基因测序技术极大地提高了物种分类学的准确性和高效性[16-17],其最大优势在于可以实现对未知物种的识别。目前,基因测序技术已经发展到了第三代,每一代技术各具优势,应用场景也存在差异。例如高通量测序可快速实现对环境样品中微生物进行宏观普查,节约了大量时间、人力成本,但同时由于得到的信息量大,干扰数据多,因此针对性不强。而试验建立的方法其成本和技术门槛较低,试验结果无需生物信息学专业人士分析,适用于普通实验室中单一样品或单一菌落分析。
但是,在DNA条形码试验设计及执行过程中有以下几点需要引起重视:① 不同物种间通用引物的设计需要考虑的因素较多;② 对纯化后的核酸质量(如纯度、浓度)要求较高,否则会出现套峰或无法检测的情况;③ 测序长度一般控制在400~800 bp的范围内,且首尾20 bp左右的序列由于准确度较差,需舍去后再进行数据库比对;④ 菌种的16S rRNA数据库信息需进一步更新、完善,以增加菌种识别的范围和准确度。

1~16泳道分别为DNA Marker、大肠埃希氏菌、奇异变形杆菌、普通变形杆菌、弗氏柠檬酸杆菌、产气肠杆菌、斯氏李斯特氏菌、英诺克李斯特氏菌、伊氏李斯特氏菌、枯草芽孢杆菌、蕈状芽孢杆菌、巨大芽孢杆菌、溶藻弧菌、霍乱弧菌、荧光假单胞菌、表皮葡萄球菌

表3 序列比对结果
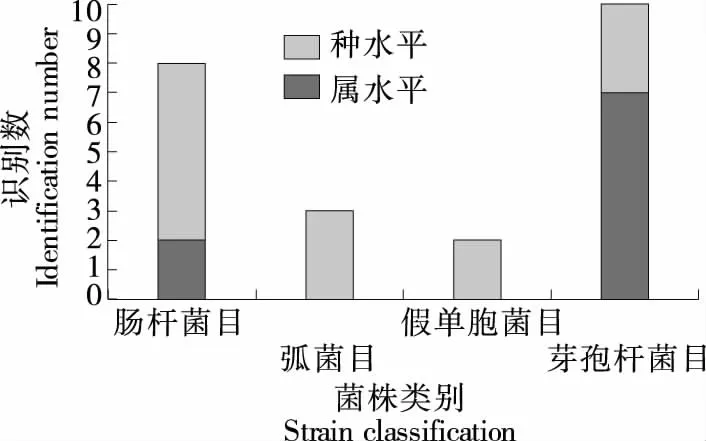
图3 “目”水平的菌种识别

图4 “科”水平的菌种识别
3 结论
中国现行食品安全检验标准体系中涉及病原微生物的检测均为针对某一指定菌种进行分离鉴定,强调检验结果的特异性,而对产品中可能存在的其他微生物风险无法全面覆盖。试验基于细菌的16S rRNA,对DNA条形码在食品生物安全领域的应用进行了探索。依照试验设计,选择V3~V6区域作为目标片段,对变形杆菌纲(肠杆菌目、弧菌目、假单胞菌目)具有足够的分辨率,能准确识别食品中的致病菌和相关干扰菌,而对芽孢杆菌纲(芽孢杆菌目)的鉴定将范围缩小到了“属”水平,减轻了进一步识别的工作量。在食品病原微生物研究范畴内,COI、cpn60或其他基因片段是否能实现更高的识别效率,还有待进一步验证。DNA条形码技术能实现在较低的技术平台上对未知微生物的无靶标识别,极大地方便了企业查因溯源及监管机构开展风险评估。
