基于DNA条形码技术鉴别有毒鹅膏菌属物种
2021-03-01白文明邢冉冉陈丽萍雷红涛
白文明,邢冉冉,陈丽萍,彭 涛,雷红涛,陈 颖,*
(1.华南农业大学食品学院,广东 广州 510642;2.中国检验检疫科学研究院,北京 100176;3.中华人民共和国昆明海关检验检疫技术中心,云南 昆明 650051)
鹅膏菌属(Amanita)属于真菌界(Fungi)、担子菌亚门(Basidiomycotina)、层菌纲(Hymenomycetes)、伞菌目(Agaricales)、鹅膏菌科(Amanitace),是大型真菌中分布较广的大属。全世界报道且被验证的鹅膏菌有近500 种,我国报道的有130多种(亚种、变种及变型)[1]。在这些鹅膏菌中,既有可食用的鹅膏菌,如橙盖鹅膏菌(Amanita caesarea)、卵盖鹅膏菌(A.ovoidea)、红黄鹅膏菌(A.hemibapha)等[2-3],也有含毒的鹅膏菌,如锥鳞白鹅膏菌(A.virgineoides)、小毒绳鹅膏菌(A.melleiceps)、鳞柄白鹅膏菌(A.virosa)[4-7]。据不完全统计,我国2019年共发生了53 起蘑菇中毒事件,其中52.8%的病例是由于食用鹅膏菌属引起的,如致命鹅膏菌(A.exitialis)、灰花纹鹅膏菌(A.fuliginea)、裂皮鹅膏菌(A.rimosa)等。鹅膏菌属物种主要分布在树林边缘地方,在我国主要分布于亚热带地区,如广州、湖南、云南、山东等地;少数分布于我国温带或热带地区,如海南的灰盖粉褶鹅膏(A.griseorosea)、假褐云斑鹅膏菌(A.pseudoporphyria)等。
传统的真菌鉴定及分类主要是形态学鉴定,依据子实体菌盖、菌柄、菌托等不同形态特征以及孢子类型进行宏观的分析和评价[8]。该方法简单直观,但因真菌物种形态特征的有限性,以及形态滞后等特点,仅靠有限的特征很难得出真菌系统的亲缘以及物种特征,因此该方法存在一定局限性和主观性。随着蘑菇中各种毒素的发现,通过检测毒素鉴别蘑菇类型的方法也随之发展起来,如从最开始的汁液显色法[9]、层析法到更加精确的液相色谱二极管阵列检测[10]、电感耦合等离子体质谱[11]、液相色谱-质谱[12-13]等方法。这些检测技术在毒素分子检测方面灵敏度高、可靠性好,且能检测尿液和血浆中的肽类毒素[14],但此类方法前处理时间和成本较高,且毒素提取过程中易混入样品中的磷脂类物质,造成设备离子源的污染,同时产生一定的基质干扰,影响结果的准确性[15-16]。随着分子生物学技术的飞速发展,DNA分子标记技术在真菌分类学和系统进化研究[17]已逐渐成熟,如简单重复序列[18-19]、限制性片段长度多态性[20]等。特别是近年发展起来的DNA条形码技术,现已经成为分子鉴别以及分子分类学的核心方法,在真伪鉴定物种鉴别等方面发挥着重要作用,广泛应用于动植物、微生物的物种鉴定[21]。
理想的DNA条形码区域应该易于扩增,变异程度足以区分物种[22-23]。近年来,已有利用核糖体DNA上的内转录间隔区(internal transcribed spacer,ITS)、核糖体大亚基(large subunit ribosomal,LSU)、RNA聚合酶II的第2大亚基(the second largest subunit of RNA polymerase II,RPB2)、β-微管蛋白(β-tubulin)等对大型真菌成功进行种类鉴定及分子系统发育分析的研究报道[24-29]。对于鹅膏菌属,也有基于ITS、LSU、β-tubulin、RPB2等基因序列对致死鹅膏菌、灰花纹鹅膏菌、淡红鹅膏菌等进行分子鉴定的报道[5,30-33]。
本研究基于ITS、LSU、RPB2、β-tubulin4 条基因对27 种有毒鹅膏菌进行序列测定及分析,评价不同DNA条形码候选序列对有毒鹅膏菌的鉴别能力,旨在为有毒蘑菇分类鉴别、亲缘关系分析提供一定的理论基础,为有毒蘑菇诱发的食源性中毒风险预警和准确鉴定提供技术支撑。
1 材料与方法
1.1 材料与试剂
本研究使用的27 种鹅膏菌属共38 个蘑菇样本,见表1。其中假淡红鹅膏菌由中国疾病预防控制中心赵云峰老师提供,其余鹅膏菌样品由湖南师范大学陈作红教授提供并进行形态学鉴定,所有鹅膏菌均为子实体。
DNAsecure Plant Kit DNA新型植物试剂盒 天根生化科技有限公司;ExoSAP-ITTM聚合酶链式反应(polymerase chain reaction,PCR)产物快速回收试剂盒、BigDye Terminator v3.1 Cycle Sequencing Kit、BigDye XTerminator Purification Kit、Cathode Buffer Container (CBC) 3500 Series、Anode Buffer Container(ABC) 3500 Series和POP-7™ Polymer for 3500/3500xL Genetic Analyzers 北京奥今东方科技有限公司;PCR扩增引物均由英潍捷基(上海)贸易有限公司合成。
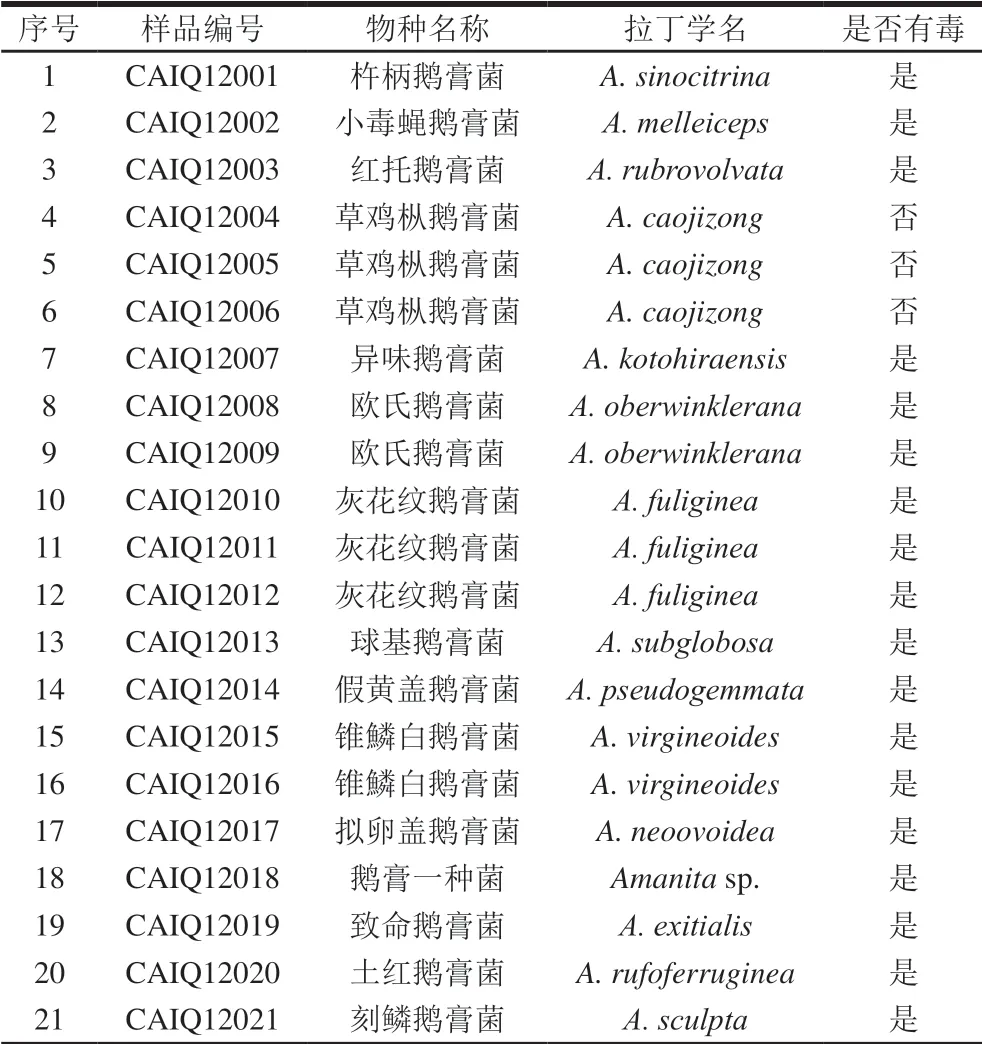
表1 鹅膏菌属样品信息Table 1 Information about the collected Amanita samples

续表1
1.2 仪器与设备
BT323S电子天平 赛多利斯科学仪器有限公司;Sorvall™ ST8台式离心机、Nanodrop超微量分光光度计美国Thermo Fisher Scientific公司;VORTEX-GENIE2涡旋振荡器 美国Scientific Industries公司;电泳仪、Versa Doc凝胶成像仪 美国Bio-Rad公司;Qubit®3.0核酸荧光蛋白定量仪 美国Invitrogen公司;PCR仪、ABI3500测序仪 美国Applied Biosystems公司。
1.3 方法
1.3.1 DNA提取与PCR扩增
鹅膏菌DNA提取方法按照DNAsecure Plant Kit DNA新型植物试剂盒(天根)操作说明进行,提取的DNA样品保存于-20 ℃备用。
选用ITS、LSU、RPB2、β-tubulin4 个DNA片段,分别用通用引物(ITS4/ITS5[34]、LSU-LROR/LSU-LR5[35]、bRPB2-6F/bRPB2-7-1R[36-37]和β-tubulinF/R[38])对鹅膏菌样品进行扩增,引物序列信息及扩增程序见表2。

表2 扩增基因片段引物信息及扩增程序Table 2 Sequences of primers and PCR amplification procedures used in this study
ITS反应体系:2hTaqPCR Master Mix 12.5 μL,10 μmol/L上下引物各0.3 μL,10 ng/μL DNA模板3 μL,加无菌去离子水至25 μL。LSU反应体系:2hTaqPCR MasterMix 12.5 μL,10 μmol/L上下引物各0.2 μL,10 ng/μL DNA模板3 μL,加无菌去离子水至25 μL。RPB2反应体系:5 U/μLTaq酶0.2 μL,dNTP 1.5 μL,10h PCR buffer 2.5 μL,10 μmol/L上下引物各0.25 μL,10 ng/μL DNA模板2 μL,加无菌去离子水至25 μL。β-tubulin反应体系:5 U/μLTaq酶0.2 μL,dNTP 1.5 μL,10h PCR buffer 2.5 μL,10 μmol/L上下引物各0.3 μL,10 ng/μL DNA模板4 μL,加无菌去离子水至25 μL。用2%的琼脂糖凝胶电泳检测PCR扩增产物后,将条带单一且明亮的PCR产物进行纯化回收,进行下一步Sanger测序。
1.3.2 测序
将纯化后的PCR产物进行双向测序,测序反应体系总体积10 μL,包含:PCR纯化产物1 μL,5h Sequencing buffer 1 μL,BigDye Terminator 0.8 μL,引物1.6 μL(1 μmol/L),无菌去离子水5.6 μL。PCR扩增程序:96 ℃预变性2 min;96 ℃变性20 s,55 ℃退火10 s,60 ℃延伸4 min,25 个循环,4 ℃保存。采用BigDye XTerminator Purification Kit对产物进行纯化后,吸取上清液于96 孔测序板中,于ABI 3500基因分析仪进行测序。
1.4 数据处理
对双向测序所得样品结果用DNASTAR软件中的SeqMan程序进行校对拼接,去除低质量区的序列。将拼接后的序列提交NCBI的GenBank数据(http://blast.ncbi.nlm.nih.gov)进行序列比对鉴别物种来源。利用MEGA6.0软件对各基因序列进行多序列比对分析,并基于Kimura-2-Parameter(K2P)双模型[39]和邻位连接(Neighbor-Joining,NJ)法[40]计算遗传距离,构建系统发育树,采用Bootstrap检验法进行可信度测验,自展系数为1 000。
2 结果与分析
2.1 DNA提取与PCR扩增
分别提取38 个鹅膏菌属样品的基因组DNA,用Nanodrop超微量分光光度计测定浓度及纯度,各样品DNA质量浓度在40~584.8 ng/μL之间,表明DNA浓度较高,A260nm/A280nm在1.60~2.06,表明DNA纯度较好,符合后续实验要求。利用通用引物对ITS、LSU、RPB2、β-tubulin4 条序列进行PCR扩增,电泳结果如图1所示。引物ITS4/ITS5、LSU-LROR/LR5、RPB2-6F/7-1R、β-tubulin-F/R扩增成功率分别为89.47%(34/38)、89.47%(34/38)、84.21%(32/38)、97.37%(37/38),表明引物β-tubulin-F/R的通用性较引物ITS4/ITS5、LSU-LROR/LR5、RPB2-6F/7-1R更好。A.sculpta、A.fritillaria未能扩增出RPB2目的条带,A.javanica未能扩增出RPB2、β-tubulin目的条带,A.sphaerobulbosa、A.manginiana、A.gymnopus未能扩增出ITS、LSU、RPB2目的条带,但能扩增出长度约为425 bp的β-tubulin目的条带,其原因是由于实验样品存放时间太长,DNA被降解成短片段,因而扩增片段大于600 bp的长片段ITS、LSU、RPB2等基因难以成功扩增出相应片段[41]。因此结合引物ITS4/ITS5与β-tubulin-F/R则可将本研究中所有鹅膏菌物种扩增出来。
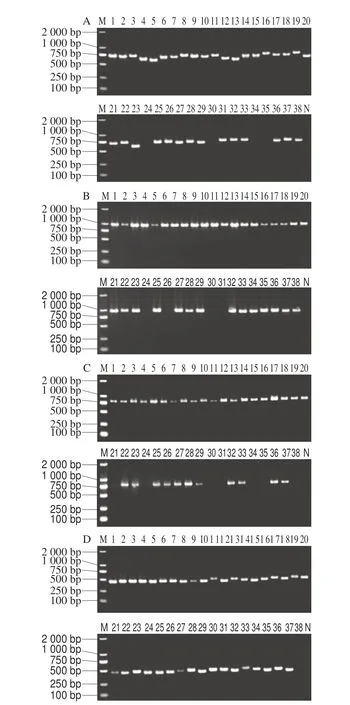
图1 鹅膏菌样品4 条基因序列的PCR扩增产物电泳图Fig.1 Electropherograms of PCR-amplified products of Amanita samples using four barcodes
2.2 PCR产物测序与序列比对分析
将纯化后的PCR产物用ABI3500基因分析仪进行双向测序,对符合标准的测序序列进行拼接和校对。共获得ITS序列34 条,LSU序列34 条,RPB2序列32 条,β-tubulin序列37 条,测序成功率均为100%,不同候选序列在序列长度上存在明显差异,由高至低依次为LSU、RPB2、ITS、β-tubulin。将拼接后的序列提交NCBI的GenBank数据库(http://blast.ncbi.nlm.nih.gov)进行序列比对鉴别物种来源。如表3所示,33 种鹅膏菌的LSU、RPB2、ITS、β-tubulin基因序列与NCBI数据库中的参考序列相似度在99%及以上,与形态学鉴定结果一致;5 种鹅膏菌比对结果与形态学鉴定结果不一致,其中,形态学鉴定结果为A.caojizong的CAIQ12004和CAIQ12006样品,其序列比对鉴定结果分别为A.pseudoprinceps、A.yuaniana;编号为CAIQ12018的样品,形态学鉴定为Amanitasp.,而序列比对鉴定为A.minutisquama;编号为CAIQ12008和CAIQ12037的样品,其扩增片段序列与A.rimosa和A.aspericeps的同源性分别为100%和99%,与形态学鉴定的A.oberwinklerana、A.sinensis不一致。整体而言,4 条DNA条形码的鉴别能力从高至低分别为β-tubulin、ITS、LSU、RPB2,且4 条基因序列在比对结果上具有很好的一致性,表明所得到的序列准确度较高。
2.3 系统发育树构建
K-2-P参数模型是生命条形码联盟(Consortium for the Barcode of Life,CBOL)推荐用于计算遗传距离的模型[42]。在本研究中,采用K-2-P参数模型计算了所用鹅膏菌的种内和种间遗传距离(表4)。数据显示,ITS、RPB2、β-tubulin基因序列的种间最小遗传距离均远大于种内最大遗传距离,存在明显的条码间隔,不存在种内和种间的交叉重叠,这一结果表明ITS、RPB2、β-tubulin片段适合作为DNA条形码。其中,ITS、β-tubulin表现出最高的种间序列差异,可作为优选条形码。LSU序列的种间最小遗传为0.03,种内最大遗传距离为0.034,种间平均遗传距离是种内平均遗传距离的244 倍,但在0.030~0.034区间存在种内和种间遗传距离的交叉重叠,这会造成部分物种的鉴定错误,表明此基因序列在一定程度上能够鉴定物种,不适合作为本研究中用于鉴定鹅膏菌属的DNA条形码。
将ITS、LSU、RPB2、β-tubulin扩增基因序列进行比对对齐,通过建立NJ树以评价候选DNA条形码对鹅膏菌属物种的鉴别效率。NJ树结果显示(图2),基于鹅膏菌ITS、LSU、RPB2、β-tubulin序列构建的系统发育树均可分为两大支。ITS序列系统发育树中A.caojizong、A.subglobosa、A.javanica、A.fritillaria、A.subpallidorosea、A.rimosa聚为一支,其余16 种鹅膏菌聚为一支;LSU序列系统发育树的两大支分别为A.caojizong、A.subpallidorosea、A.javanica、A.subglobosa、A.melleiceps、A.flavipes聚为一支,其余16 种鹅膏菌聚为一支;RPB2序列系统发育树的一支为A.caojizong、A.subglobosa、A.subpallidorosea、A.vaginata、A.flavipes、A.rufoferruginea,其余15 种鹅膏菌聚为一支。β-tubulin序列系统发育树中A.pallidorosea、A.pseudogemmata、A.rufoferruginea、A.gymnopus、A.caojizong、A.minutisquama聚为一支,其余22 种鹅膏菌聚为一支。在NJ树中,如果绝大多数物种都能聚集成单系,且具有较高的节点支持率,则表明所选择的DNA条形码在上述物种鉴定的适用性和可行性较高。

表3 DNA条形码扩增序列比对结果Table 3 Alignment results of DNA barcode amplified sequences
当DNA条形码基因区域遗传变异较少,进化速率较慢时,则易出现不同物种的序列形成内聚性聚类的现象[43]。LSU作为28S rDNA基因上保守区和可变区中片段较长的基因,其遗传变异较少,在LSU序列构建的NJ树中,A.caojizong、A.javanica聚为一支,表明上述鹅膏菌亲缘关系较近;在RPB2序列构建的NJ树中,A.rufoferruginea、A.subglobosa、A.flavipes聚为一支,分析原因可能是由于RBP2序列单拷贝和进化速率慢,较其他基因相对保守[44-45],当物种亲缘关系较近时,RBP2基因具有相似性。这也表明了所选择的DNA条形码能够在一定程度上鉴别遗传变异较小的物种。
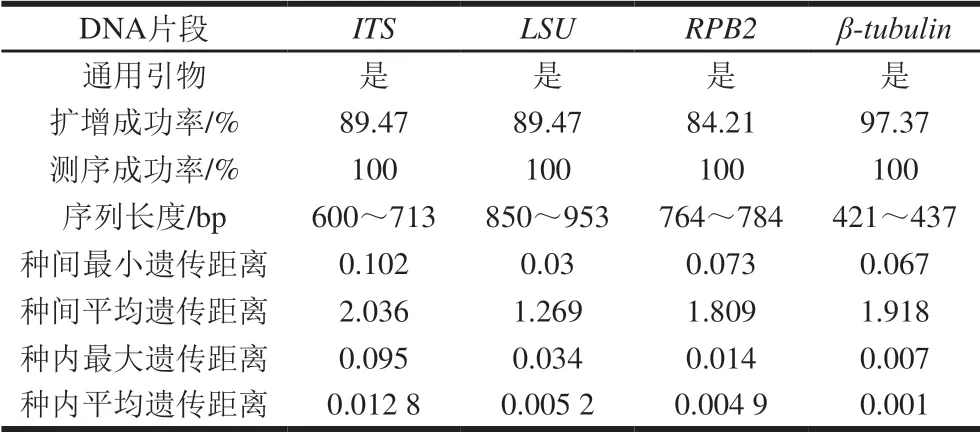
表4 4 种候选序列信息比较Table 4 Comparison of four candidate sequences
通过序列比对分析与系统发育分析结果显示,出现有毒鹅膏菌与无毒鹅膏菌鉴别错误的现象。编号为CAIQ12004、CAIQ006的无毒A.caojizong鉴定为有毒的A.pseudoprinceps以及无毒的A.yuaniana,以ITS、LSU、RPB2、β-tubulin四种序列分析,三者遗传距离分别为0.502~1.567、0.212~1.019、0.287~1.412、0.326~0.923,其遗传距离较小,亲缘相对较近,且在形态学上A.caojizong与A.pseudoprinceps、A.yuaniana极为相似,菌盖幼时半球形,后近平展,褐灰色,中部色深、光滑,这也解释了形态学鉴定会出现误判的原因。编号为CAIQ12037的A.sinensis为无毒鹅膏菌,而鉴定为有毒的A.aspericeps,以ITS、LSU、RPB2、β-tubulin四种序列分析,两者遗传距离分别为1.279、1.084、1.160、0.914,亲缘关系较近。A.sinensis与A.aspericeps在形态上相似度也较高,其子实体边缘有棱纹,灰白色至浅灰色,中部深灰色,表面有泥灰疣状至颗粒状菌幕残余。
另外,编号CAIQ12004的样品与A.pseudoprinceps聚为一支,且与编号为CAIQ12005的A.caojizong分为两支,结合序列比对结果表明该样品不是A.caojizong,而是A.pseudoprinceps,A.pseudoprinceps与A.c a o j i z o n g外观极其相似,常被认为是同物种,从系统发育树上则可证明A.pseudoprinceps与A.caojizong是2 个不同的物种,而非同一物种不同名称;同理,编号为CAIQ006、CAIQ12008、CAIQ12018和CAIQ12037的物种名称应分别为A.yuaniana、A.rimosa、A.minutisquama、A.aspericeps。形态学物种鉴定的参考库需要大量的标本,包括菌盖、菌柄、菌托以及孢子,许多形态特征在不同的发育阶段也会发生一定的变化,因此,偶尔错误识别是不可避免的,这也反映了DNA条形码可以检测到形态学错误识别的情况,可以为大型真菌的物种鉴定提供更多证据。
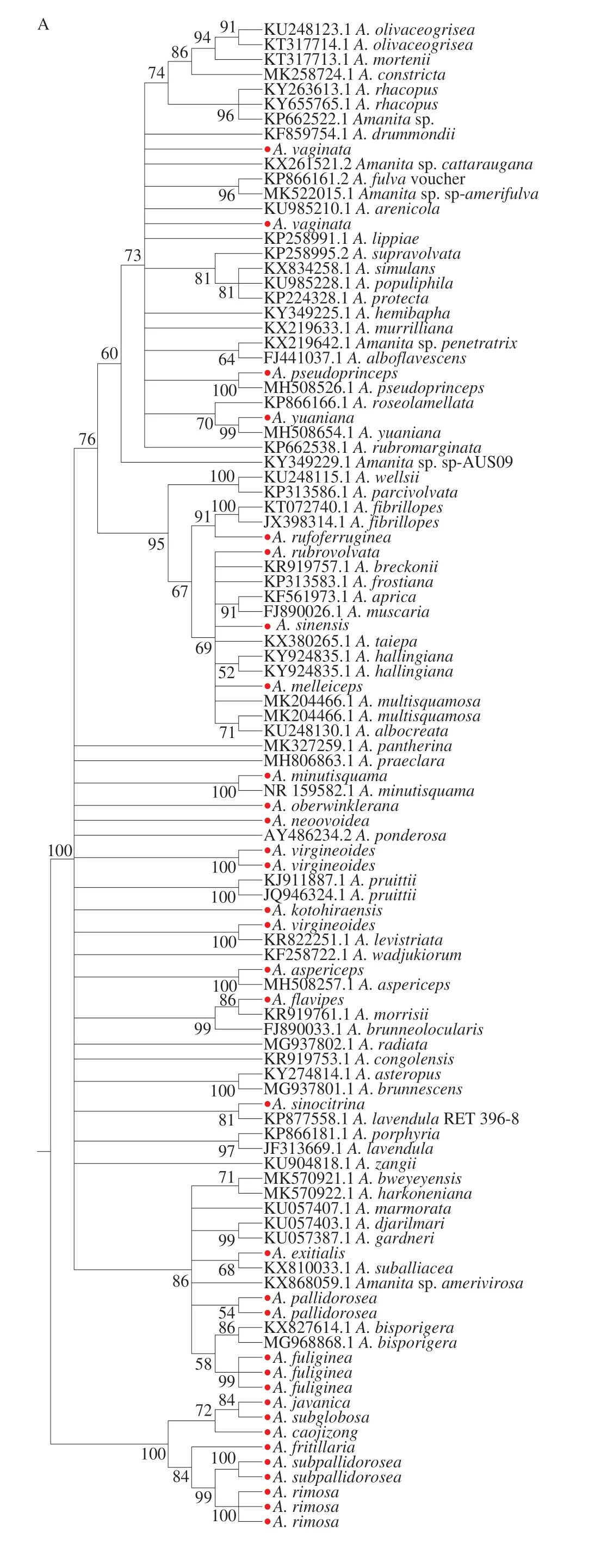
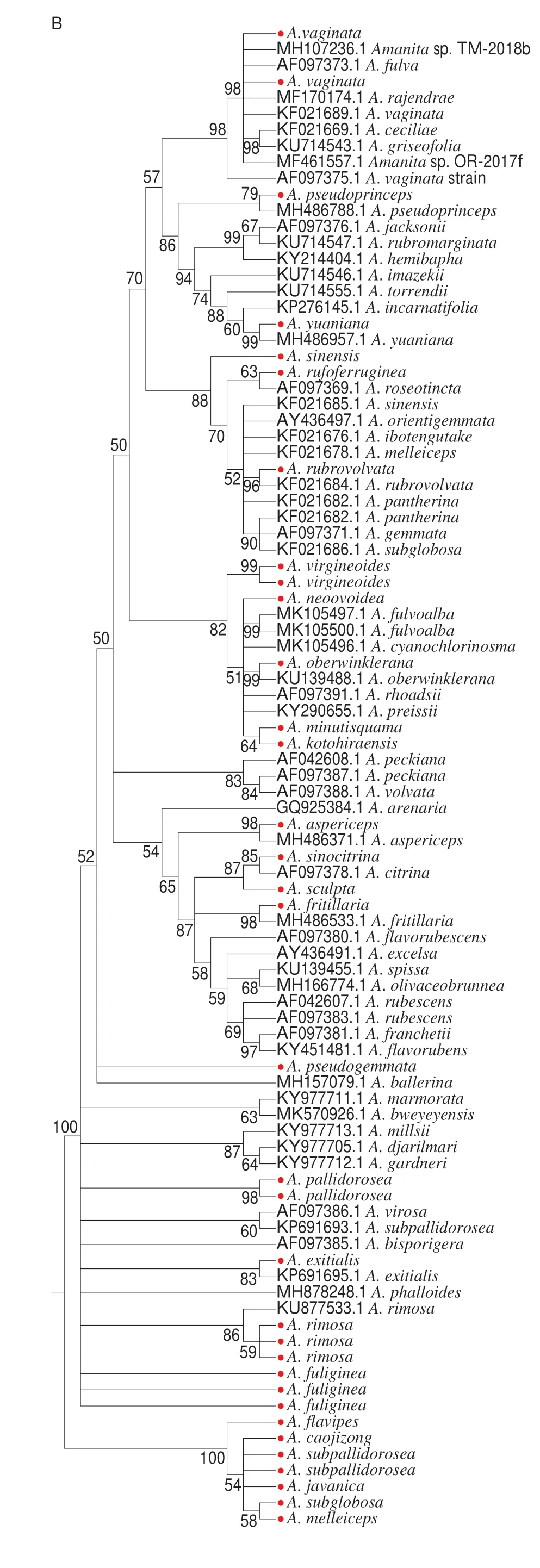

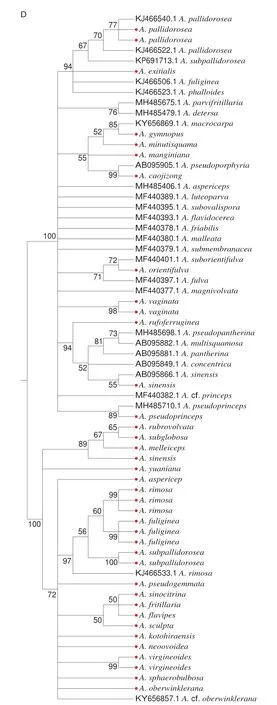
图2 基于ITS、LSU、RPB2序列构建的鹅膏菌物种NJ系统发育树*Fig.2 Neighbor-joining phylogenetic tree of Amanita species based on the sequences of ITS, LSU, RPB2 and β-tubulin
3 结 论
本研究从NCBI序列比对、种内种间变异、系统发育树3 个方面分析不同DNA条形码对于27 种有毒鹅膏菌属物种的鉴别能力。结果表明,4 条DNA条形码序列的扩增成功率从高至低分别为β-tubulin>ITS>LSU>RPB2,β-tubulin、ITS、RPB2三个片段不存在种间和种内遗传距离的交叉重叠,物种分辨率较高,满足DNA条形码的基本要求。鉴于β-tubulin、ITS两个基因结合可鉴别本研究中所有鹅膏菌,因此推荐将两者联合使用用于鹅膏菌属的物种鉴别,为有毒蘑菇诱发的食源性中毒风险进行预警。β-tubulin序列长度在367 bp左右,序列长度较短,适合对深加工的蘑菇制品以及误食毒蘑菇后的呕吐物进行分析。在现实有毒蘑菇中毒事件中很难采集到完整的蘑菇样品,需要从煮熟的残留蘑菇或者是从患者胃抽取物中获得样本用于鉴定,因此,β-tubulin在蘑菇中毒事件中的原因排查环节具有较大的优势,可作为鹅膏菌属中毒事件中物种鉴定的优选条形码。
