GmGSL7c在大豆抗SMV过程中的作用
2021-02-26王梦璇孙希哲孙天杰张洁王冬梅
王梦璇,孙希哲,孙天杰,张洁,王冬梅
(河北农业大学 生命科学学院,河北 保定 071001)
大豆Glycinemax(L.) Merr.是中国主要的粮食作物,而由大豆花叶病毒(Soybeanmosaicvirus, SMV)引起的大豆花叶病毒病在大豆主产区普遍发生.大豆叶片感染SMV后,会出现花叶、皱缩等现象,最终导致大豆品质和产量降低.探索大豆抗SMV机制对保护大豆生产具有指导作用[1].
胞间连丝(plasmodesmata, PD)是由相邻细胞的质膜和内质网膜相互连接形成的纳米通道[2],SMV通过PD这一共质通道来完成胞间转运[3].PD的通透性受到多种因素的调节.Lee等[4]证明在质膜和内质网膜之间存在由肌动蛋白和肌球蛋白形成的辐,这种结构也说明PD的孔径大小具备可调节性. Guseman等[5]在拟南芥中发现,胼胝质的合成一旦受到抑制,PD的通透性也会发生明显变化;Vatén等[6]也证明胼胝质的合成影响根发育过程中的共质运输.随着对PD研究的逐步深入,胼胝质对PD孔径调控的重要性也逐渐被揭示;Sivaguru等[7]在研究重金属与植物的关系过程中发现胼胝质的代谢受Ca2+调节,外源施加铝离子会导致钙稳态的局部变化,使PD因胼胝质沉积而闭合.胼胝质主要由β-1,3-葡聚糖构成,这一多糖的代谢受到胼胝质合酶(callose synthase,GSL)和胼胝质水解酶(β-1,3-glucanases,BG)的共同控制[8]. Chen和Kim[9]提出胼胝质由GSL产生而被BG降解.本实验室前期研究表明,胼胝质在大豆抵御SMV侵染中发挥重要作用.使用胼胝质合成抑制剂(2-DDG)抑制胼胝质积累,可在未接种的上位叶片中检测到SMV外壳蛋白基因SMV-CP.而本文主要关注在大豆抵御SMV过程中GSL的作用.
GSL最早在酵母中发现,随后在植物中也陆续得到GSL与胼胝质合成相关的证据:Hong等[10]在拟南芥数据库中通过酵母GSL的保守结构域FKS1_dom1鉴别了12种GSL,分别命名为AtGSL1-AtGSL12,并发现这些AtGSL还同时具有Glucan_synthase结构域(pfam∶PF02364),在12种AtGSL中仅发现3种与PD上的胼胝质沉积有直接关系的胼胝质合酶,分别为AtGSL8、AtGSL7和AtGSL12. Guseman等[5]发现在gsl8突变体中,PD处的胼胝质变少且通透性增加;Xie等[11]发现在gsl7突变体中筛板和筛孔处胼胝质减少,最终导致茎发育异常;Vatén等[6]研究发现,GSL12的表达可以调控PD上胼胝质的沉积,进而影响细胞间的物质转运.
本实验室的前期工作证明了在大豆抵抗SMV侵染的过程中,一氧化氮(NO)作为重要的信号分子对胼胝质在PD上沉积有正调控作用[12],并建立了抑制NO前后大豆品种‘冀豆7号’与SMV株系N3互作的转录组数据库. 本研究结果表明了大豆胼胝质合酶GmGSL7c是SMV侵染大豆过程中影响PD上胼胝质合成的关键GSL,为揭示PD上胼胝质的动态平衡奠定了基础.
1 材料与方法
1.1 实验材料
大豆品种‘冀豆7号’(J7)与SMV株系N3组成不亲和组合,‘南农1138-2’与N3组成亲和组合,用于SMV的扩繁.‘南农1138-2’由南京农业大学智海剑教授惠赠;J7为河北省农林科学院张孟臣研究员惠赠;SMV株系N3由南京农业大学大豆改良中心提供.
大豆培育在有高压钠灯的防虫温室内培育,温度控制在(25±2) ℃,光照为10 000 lx,光照时间为14 h/d,以灭菌蛭石栽培.大豆接种SMV参考Li等[13]的方法:将带有明显花叶表型的南农1138-2的新鲜叶片加入适量0.1 mol/L磷酸缓冲液和金刚砂进行充分研磨,用灭菌毛刷蘸取研磨液均匀刷在大豆叶片叶脉较少的区域,并在接种后0、4、12、24、48、96 h对接种叶片进行取样,120 h对未被接种的上位叶取样分析.
1.2 载体与菌株
大肠杆菌Top10感受态细胞和根癌农杆菌GV3101感受态细胞均为本实验室保存.克隆载体pEASY-Blunt Zero购自北京全式金生物技术有限公司,TRV-VIGS载体(Tobaccorattlevirus,TRV)由河北农业大学农学院王省芬教授惠赠.
1.3 试剂及耗材
反转录试剂盒PrimeScript RT reagent Kit with gDNA Eraser、高保真酶PrimeSTAR MAX DNA polymerase、限制性内切酶、T4 DNA连接酶购自宝日医生物技术(北京)有限公司(TaKaRa);Taq酶、克隆载体pEASY-Blunt Zero、荧光定量试剂SYBR qPCR Mix购自全式金(北京)生物技术有限公司;表面活性剂Silwet L-77购自北京索莱宝科技有限公司,牛肉浸粉、胰蛋白胨、卡那霉素、利福平、氨苄青霉素、Trizol总RNA提取试剂盒购自生工生物工程(上海)股份有限公司;乙酰丁香酮购自西格玛奥德里奇(上海)贸易有限公司;本文所用其他药品均为国产分析纯试剂.
1.4 大豆叶片药物学注射
选取约2周龄的J7幼苗的第1轮真叶进行药物学实验.对真叶叶片侧脉间隙注射100 μmol/L c-PTIO (NO清除剂),并以注射ddH2O幼苗叶片作为对照.植株恢复培养24 h后,在注射部位进行SMV株系N3的摩擦接种.本实验设置3次生物学重复.
1.5 生物信息学分析
用HMMER软件构建AtGSLs Glucan_synthase保守结构域模型.TBtool软件在大豆蛋白库内检索与HMMER模型相符的蛋白序列,提取E-value < 10-5的基因编号及蛋白序列,提交至NCBI CDD数据库绘制保守结构域对比图.
利用MEGA 7软件构建系统进化树,聚类方法使用Neighbor-Joining (NJ),泊松回归分析选用pairwise deletion,bootstrap设置为1 000次.
1.6 TRV-VIGS载体构建及病毒接种
SoyKB (http://soykb.org)内提取基因序列,并利用NCBI Blast检索基因的特异性区段,Primer6.0软件根据特异性区段设计引物(表1).以J7叶片cDNA作为模版扩增特异性片段(引物见表1).琼脂糖凝胶电泳分离纯化基因片段后,使用限制性BamHI ,EcoRI内切酶处理扩增片段及pTRV2载体,T4 DNA连接酶连接处理后的扩增片段和pTRV2载体,重组载体转化Top10大肠杆菌后送华大基因科技有限公司北京测序部进行检测.对测序正确克隆提取质粒,并转化农杆菌GV3101.
参照Senthil等[14]的方法,在1周龄大豆幼苗第1轮真叶初步形成时,切除大豆顶芽保留子叶.将携带pTRV1和pTRV2载体(或pTRV2重组载体)的农杆菌菌株分别在含有卡那霉素(Kana,50 μg/mL)和利福平(Rif,50 μg/mL)的YEB培养基中28 ℃摇至OD600≈0.5后,转移至50 mL的塑料离心管中5 000g离心3 min,富集菌体弃去上清培养基,无菌蒸馏水漂洗2次后用侵染缓冲液(50 mmol/L MES,2 mmol/L Na3PO4,28 mmol/L葡萄糖,0.1 mmol/L乙酰丁香酮,4.1 mmol/L L-Cys,0.2 g/L Silwet L-77)重悬洗涤后菌体,重悬液OD600≈0.5.将带有pTRV1和pTRV2载体(或pTRV2重组载体)的农杆菌等体积混合,浇灌大豆根茎处,每株浇灌5 mL侵染混合液.此后间隔5 d重复侵染1次,总计3次,最后一次侵染后等待7 d,对植株的第1轮3叶接种SMV株系N3,按需求进行不同时间点样品制备.
1.7 RT-qPCR分析大豆与SMV互作过程中目的基因表达模式
使用Primer6.0设计实时定量PCR引物(表1).RT-qPCR反应按照SYBR premix Ex Taq Ⅱ试剂盒说明书指导在IQTM5多重实时定量PCR仪(Bio-Rad)上运行,样品设3次技术重复.RT-qPCR体系:Mix 5 μL,去离子水3 μL,上、下游引物各0.5 μL,模板1 μL. RT-qPCR程序:94 ℃ 30 s;94 ℃ 5 s,58 ℃ 15 s,72 ℃ 10 s后检测荧光信号,45个循环.得到的CT值采用2-ΔΔCT法进行分析,EF1b(表1)作为内参基因[15].
1.8 RT-qPCR检测基因沉默效率
从GmGSL7c基因的5’UTR特异性区段设计引物(表1).利用RT-qPCR技术检测目的基因的沉默效率,以浇灌携带pTRV1和pTRV2载体的农杆菌植株叶片为对照组,以浇灌携带pTRV1和pTRV2重组载体的农杆菌植株叶片为实验组,本实验设置3次生物学重复.
沉默效率的计算公式为
(对照组基因相对转录水平-实验组基因相对转录水平)/对照组基因相对转录水平×100%.
1.9 胼胝质荧光观察
参照Li等[13]的方法,取接种部位约1 cm2的大豆叶片用2倍乙醇稀释的乳酚油(ddH2O、丙三醇、苯酚和乳酸的质量比为1∶2∶2∶1)煮沸3 min,ddH2O洗涤3次,每次5 min.在0.1 g/L苯胺蓝(pH7.4 0.1 mol/L PBS配制)中染色15 min,ddH2O洗涤3次,每次10 min.将大豆叶片正面向上置于载玻片上,盖玻片轻轻盖压排除气泡.使用OLYMPUS BX-53型荧光显微镜(波长为385 nm激发光)观察胼胝质荧光,每个样品观察30个单侵染点.

表1 引物序列信息
1.10 SMV-CP基因的检测
使用RT-PCR检测大豆叶片中SMV外壳蛋白基因特异性片段SMV-CP. PCR体系为:3 μL样品cDNA,1 μL SMV-CP PCR F/R(表1),5 μL 10× EasyTaq®Buffer for PAGE,4 μL 2.5 mmol/L dNTPs,1 μL EasyTaq®DNA Ploymerase for PAGE,35 μL Nuclease-fress Water. PCR程序:94 ℃ 5 min,94 ℃ 30 s,55 ℃ 30 s,72 ℃ 30 s,72 ℃ 3 min,35个循环.反应结束后加入5 μL 10×Loading buffer(TaKaRa),琼脂糖凝胶电泳分离产物,EB染色后用紫外线照射显示条带位置.
2 实验结果
2.1 大豆胼胝质合酶的分析
利用生物信息学手段在大豆蛋白库中得到24个大豆胼胝质合酶(GmGSL),并对这些蛋白序列进行CDD绘图(图1).图1显示,GmGSL包含Glucan_synthase合酶结构域,多数GmGSL中还包含FKS1_dom1和Vta1结构域.
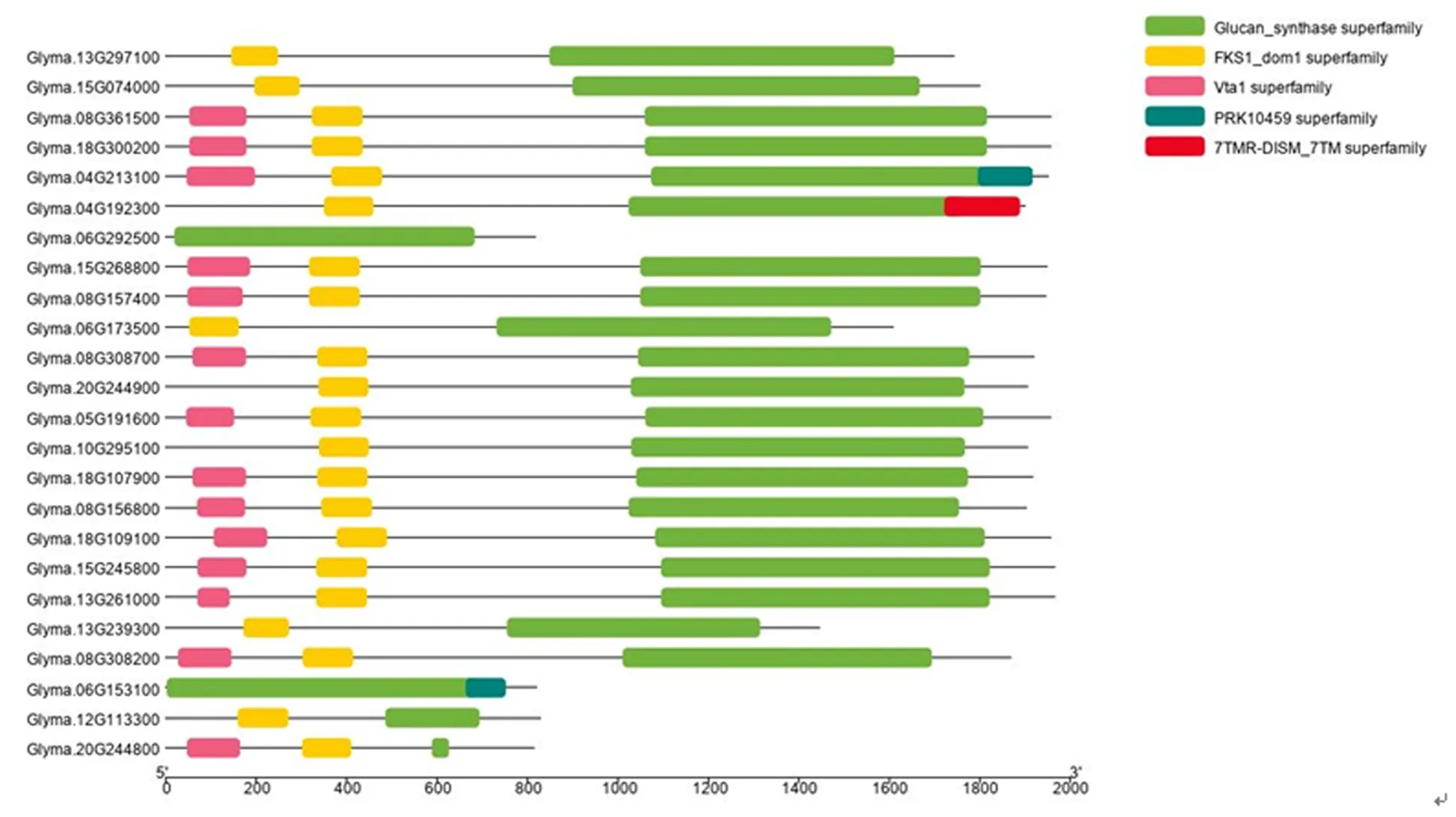
图1 大豆中胼胝质合酶的保守结构域Fig.1 Conserved domains of callose synthase in soybean


图2 拟南芥和大豆中胼胝质合酶的系统进化树Fig.2 Phylogenetic tree of callose synthase in Arabidopsis and soybean
2.2 RT-qPCR分析大豆胼胝质合酶基因的表达
使用RT-qPCR验证GmGSL7c在清除NO前后转录水平上的变化(图3).结果表明,GmGSL7c的表达趋势与数据库表达趋势基本一致.注射H2O和c-PTIO后24 h和48 h检测植株中的GmGSL7c基因表达量,结果如图3b所示:清除NO后GmGSL7c的表达显著降低,说明GmGSL7c在转录水平上受NO的调控.

a.GmGSL7c在预注射H2O和c-PTIO后接种SMV不同时间点转录组数据库;b.RT-qPCR检测得到的表达水平.图3 GmGSL7c基因的转录水平表达模式Fig.3 Expression pattern of GmGSL7c
2.3 GmGSL7c的沉默对胼胝质的影响
按照1.6方法对大豆幼苗进行病毒转染处理,接种SMV株系N3后取48、96 h的叶片,利用RT-qPCR技术分析基因沉默效率(图4)并对接种叶片进行胼胝质苯胺蓝染色. 经过TRV-VIGS处理的植株中GmGSL7c基因在转录水平的表达明显较对照组低,说明GmGSL7c基因得到有效的沉默.
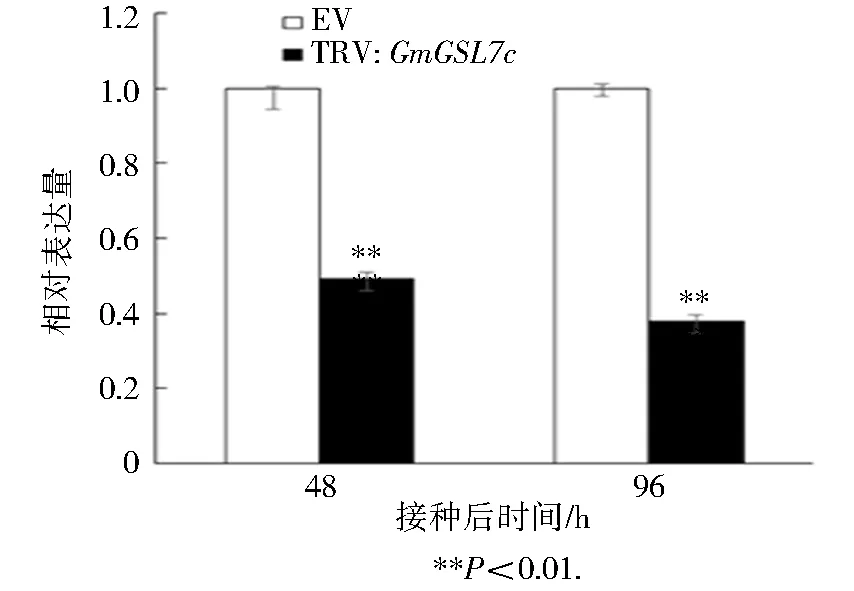
图4 RT-q-PCR检测基因沉默效率Fig.4 RT-q-PCR detection of gene silencing efficiency
为了研究GmGSL7c基因的沉默对SMV诱发胼胝质积累的影响,利用苯胺蓝染色法观察沉默GmGSL7c植株叶片的胼胝质变化,接种SMV株系N3后48 h (图5a),实验组叶片胼胝质面积明显较对照组增加,但荧光强度更弱;接种后96 h(图5b),对照组中单浸染点的胼胝质荧光强度和面积变化不大,而实验组叶片胼胝质荧光强弱不一且面积增加.说明沉默GmGSL7c会影响叶片中胼胝质的合成.

a.接种SMV-N3后48 h胼胝质观察;b.接种SMV-N3后96 h胼胝质观察(箭头所指为单侵染点胼胝质).图5 GmGSL7c沉默后叶片的胼胝质观察Fig.5 Observation of the callose on the GmGSL7c gene silencing leaf
2.4 GmGSL7c的沉默促进SMV在大豆体内的运输
实验室前期研究表明,胼胝质在大豆抵御SMV侵染中发挥重要作用.为了探讨GSL沉默是否影响植株的抗病性,本研究进一步验证沉默GmGSL7c基因的植株PD上胼胝质的合成是否直接影响SMV病毒的细胞间转运.利用PCR技术验证实验组上部未接种的叶片中是否包含SMV外壳蛋白的特异性片段,以此来证明受基因GmGSL7c调控的胼胝质对SMV病毒侵染的影响及相应的沉积部位.如图6显示,实验组上位叶检测到了SMV-CP特异性片段条带(其中Control为以接种SMV-N3病毒的南农1138-2叶片RNA为模版扩增的PCR产物,SMV-CP保守片段为500 bp),而对照组的上位叶并没有检测到类似的条带.持续培养对照组及实验组植株,接种SMV15 d时观察上位叶(图7).可以在实验组植株上位叶观察到花叶表型且叶片有明显皱缩现象,而对照组上位叶没有观察到类似表型且叶片状态良好.进一步证实GmGSL7c基因调控PD上的胼胝质沉积,并在大豆抵御SMV过程中起到关键作用.
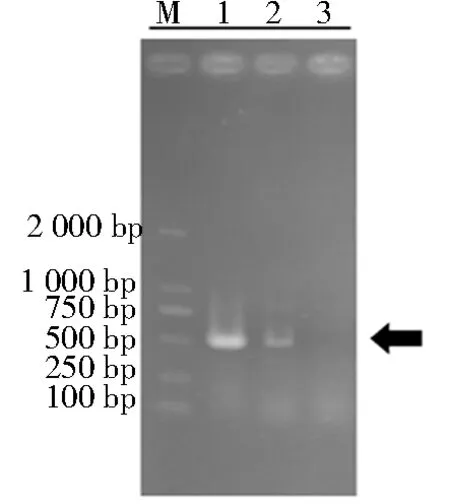
M.Marker;1.Control;2.TRV:GmGSL7c;3.TRV:00.图6 沉默GmGSL7c植株上位叶SMV-CP检测Fig.6 SMV-CP detection of the upper leaves of GmGSL7c gene silencing plants

图7 沉默GmGSL7c的植株上位叶表型Fig.7 Upper leaves phenotype of GmGSL7c gene silencing plants
3 讨论
胼胝质是一种仅在高等植物中发现的多糖,其主要成分是β-1,3-葡聚糖[8]. Levy等[16]的结果显示,PD颈部可以检测到胼胝质的沉积,且PD的通透性受到胼胝质代谢的调节.同时胼胝质的代谢又受GSL和BG的严格调控,GSL参与胼胝质的合成,而BG参与胼胝质的水解过程.其中,GSL只有通过整合到高度特化的蛋白质复合体才能正确合成和沉积胼胝质,这也说明胼胝质的合成过程可能受多种因素的调节[8].
Xiao等[12]的工作已经证明,在由SMV株系N3和大豆‘J7’组成的不亲和组合中,SMC-N3的侵染会引起NO信号的产生,并观察到胼胝质沉积在PD颈部;而在由SMV株系SC-8和大豆‘J7’组成的亲和组合中没有观察到类似的现象.为探求NO信号能否影响GmGSL的表达,并最终影响胼胝质的沉积问题设计实验.首先利用AtGSL中的保守序列构建HMMER保守结构域模型,比对大豆蛋白质组库筛选出24个GmGSL (图1);将这一结果进行系统进化树分析(图2),选取与拟南芥中PD相关的AtGSL相近的分支,同时参照转录组库挑选出受NO信号调控的大豆胼胝质合酶基因GmGSL7c.结果2.1显示胼胝质合酶基因GmGSL7c中包含3个结构域,其中Glucan_synthase结构域参与多糖合成[17];FKS1_dom1为酵母中的保守结构域[18],以该结构域作为参照筛选拟南芥中胼胝质合酶;而GmGSL7c还包含1个非保守的Vta1结构域,这一结构域主要是通过调节ESCRT系统(endosomal-sorting complexes required for transport)中的ATPase,最终影响MVB (multivesicular body)的发生[19].结合De等[8]得出的GSL需要与多种蛋白组装形成胼胝质合酶复合体才能行使功能的结果,推测胼胝质合酶基因GmGSL7c可能与多种蛋白形成的胼胝质合酶复合体启动胼胝质在PD上的合成.利用RT-qPCR检测GmGSL7c基因在不同时间点的表达水平也可以发现,与转录组数据库趋势相近,基因GmGSL7c受NO信号调节,可能参与SMV诱导的PD上的胼胝质沉积.
Li等[13]的结果证明,通过检测未被接种的上位叶中是否含有SMV外壳蛋白的特异性区段的方法来鉴别SMV在植物叶片中的侵染进程;TEM观察亲和组合PD发现,SMV侵染状态下,PD颈区基本观察不到胼胝质的存在,并在未被接种的上位叶中检测到了SMV-CP的保守区段;在不亲和组合中观察到接种叶片中胼胝质被诱导表达,并在上位叶中未能检测到SMV-CP的保守区段.本研究结果显示在不亲和组合中,对沉默GmGSL7c基因且效率较好的植株(图4)接种SMV株系N3,观察沉默组与对照组中接种叶片胼胝质的变化.结果发现,沉默组胼胝质面积随着时间的推移逐步扩展,而对照组中的胼胝质面积变化不大;与对照组相比,沉默组中的胼胝质荧光更弱(图5).以上结果表明,当基因GmGSL7c的表达被抑制时仍有部分胼胝质合成,但合成的胼胝质及其在PD上沉积的量无法阻碍SMV在胞间的扩散,当SMV从一个细胞进入另一个细胞时,胼胝质仍会被诱导合成以进一步限制SMV的运输,最终观察到实验组叶片胼胝质面积不断增加(图5),而顺利逃逸的SMV最终侵入了未被接种的上位叶中,并能在实验组未接种上位叶片中检测到SMV-CP片段(图6),观察到被SMV感染后的花叶症状(图7).
结合这些证据可以得出,SMV诱导NO信号的产生,并影响GmGSL7c基因的表达;而GmGSL7c作为PD上胼胝质沉积的关键酶,当GmGSL7c基因受到抑制时,PD上合成的胼胝质不足以阻断SMV,使得SMV向外逃逸并经韧皮部扩散到上位叶,最终在未被接种的上位叶观察到花叶症状.这一基因功能的揭示说明PD通透性的调控受到多因素影响,为解释植物体内复杂信号网络提供了新证据.
