羔羊瘤胃内容物微生物基因组DNA的提取方法比较
2021-02-24翁秀秀刘婷
翁秀秀,刘婷
(1.兰州大学草地农业科技学院,甘肃 兰州 730020;2.甘肃农业大学动物科学技术学院,甘肃 兰州 730070)
幼龄反刍动物瘤胃发育与其健康和营养关系密切[1].瘤胃组织形态学发育[2-3],瘤胃发酵功能完善[4-5]和瘤胃微生物的定植[6]是衡量瘤胃发育的重要指标.其中,探讨瘤胃微生物区系的定植规律是当前幼龄反刍动物瘤胃发育的研究热点.16S rRNA测序技术已经成为一种普遍方法用于瘤胃微生物区系多样性的研究.前期研究发现,幼龄反刍动物刚出生时,瘤胃内是一个无菌的环境,出生后24 h,来自于初乳和环境的微生物在瘤胃内快速定植.变形菌被拟杆菌逐渐代替;第3天到第12天之间,瘤胃尚未发育成熟,但许多细菌组成的细菌群落已经能被检出,第9天到第15天之间,瘤胃微生物迅速定植,门水平变化的研究结果存在差异;从15 d开始,尽管一些微生物在属的相对丰度发生变化,但在门水平不再表现出明显的时间相关性[7-9].这些研究结果的差异大多归结于饲料组成和饲养方式的差异,却忽略了这些研究中瘤胃微生物基因组DNA提取方法不同引起结果出现差异的可能性.Yu等[10]提出瘤胃微生物群落结构的研究也受DNA提取方法的影响,也有学者研究证实了这一观点[11-12].目前提取瘤胃微生物基因组DNA的方法众多,其中对比成年反刍动物瘤胃微生物基因组DNA的方法也有相关报道[13-15],如隋昶生等[13]的结果表明,冻融法结合CTAB(十六烷基三甲基溴化胺)更适合提取崂山奶山羊瘤胃微生物总DNA;张玲等[14]采用酶解、冻融和SDS(十二烷基磺酸钠)、CTAB相结合的方法提取双峰驼瘤胃微生物总DNA,得到的瘤胃微生物DNA浓度和稳定性更高;Durgadevi等[15]对山羊瘤胃微生物基因组DNA的提取方法做了系统的优化.但适合于羔羊瘤胃内容物微生物基因组DNA提取方法的研究却鲜有报道.综上所述,本试验拟改进传统瘤胃内容物微生物基因组DNA的提取方法,利用q-PCR技术进行瘤胃细菌和古菌拷贝数分析,比较不同提取方法对羔羊瘤胃内容物中微生物多样性的影响,以期筛选出适用于羔羊瘤胃内容物微生物基因组DNA的提取方法.
1 材料与方法
1.1 样品采集
本试验选用6只健康、体质量接近(9.53±0.87 kg)、饲喂同种饲料的56日龄湖羊公羔.羔羊颈静脉放血屠宰后,迅速分割出瘤胃,混匀瘤胃内容物后,用5 mL冻存管(灭菌)收集瘤胃内容物,快速放入液氮罐中带回实验室,-80 ℃超低温冰箱中保存,作为瘤胃微生物基因组DNA提取的试验样品.
1.2 主要试剂与仪器设备
CTAB、NaCl、EDTA(乙二胺四乙酸)、苯酚、氯仿、异戊醇、异丙醇、无水乙醇、GITC(异硫氰酸胍)、SDS均为国产分析纯;Tris-HCl(pH 8.0)、蛋白酶K、PBS(磷酸盐缓冲液,pH 7.2 )购自北京Solarbio生物公司;M5 HiPer pTOPO-TA Cloning Kit(快速克隆试剂盒)购自北京聚合美生物科技有限公司;AxyPrepTMPlasmid Miniprep Kit(质粒DNA提取试剂盒)购自美国Axygen公司;qPCR SuperMix购自浙江博而金科技股份有限公司.
ND-1000微量紫外分光光度计购自美国 Nanodrop公司;q-PCR仪购自美国Bio-Rad公司;MS 3 basic漩涡振荡器购自艾卡(广州)仪器设备有限公司.
1.3 微生物基因组DNA提取
液氮研磨+珠磨+CTAB法:将6份冷冻样品混合于研钵(灭菌)中,加液氮充分研磨并混匀,称取0.5 g研磨后的样品于2 mL离心管中并加入0.3 g玻璃珠,再加入1.0 mL提取液[1.4 mol/L NaCl、20 mmol/L EDTA、2% CTAB和0.1 mol/L Tris-HCl(pH 8.0)],用漩涡振荡器混匀5 min;加入20 μL蛋白酶K,70 ℃水浴15 min,90 ℃水浴5 min,室温下14 000 r/min离心10 min,转移上清液至1.5 mL离心管中,加入等体积苯酚-氯仿-异戊醇溶液(体积比25∶24∶1),剧烈摇匀,室温14 000 r/min离心10 min,可见有3层,将上清移至1.5 mL离心管中,加入0.8倍体积异丙醇,混匀;随后转入-20 ℃冰箱过夜;过夜解冻后室温14 000 r/min离心10 min,弃上清可见有白色DNA沉淀,加入70%酒精冲洗沉淀2次,每次离心10 min,倒置风干至半干,用50 μL双蒸水溶解DNA沉淀,-20 ℃保存.
珠磨+CTAB法:将6份冷冻样品解冻后混合均匀,并称取0.5 g混合样品于2 mL离心管中并加入0.3 g玻璃珠,以下步骤同液氮研磨+珠磨+CTAB法.
液氮研磨+珠磨+SDS+GITC法:将6份冷冻样品混合于研钵(灭菌)中,加液氮充分研磨并混匀,取0.5 g研磨后的样品于2 mL离心管中并加入0.3 g玻璃珠,再加入1 mL提取液(20 mmol/L Tris-HCl(pH 8.0)、1.5% NaCl、4% SDS和0.1 mol/L EDTA),涡旋混匀5 min;加入20 μL蛋白酶K,70℃孵育15 min,90℃孵育5 min;加入200 μL 5 mol/L GITC,混匀后继续 65 ℃水浴15 min,1 4000 r/min离心10 min,转移上清至1.5 mL离心管中,加入等体积苯酚-氯仿-异戊醇溶液(体积比25∶24∶1)500 μL,摇匀后静置5 min,14 000 r/min离心10 min,抽取上清至1.5 mL离心管中,加入0.8倍体积预冷的异丙醇,冰上放置1 h;14 000 r/min离心10 min,移除试管内液体,保留白色沉淀,加入70%酒精冲洗沉淀2次,每次离心10 min,倒置风干至半干,用50 μL双蒸水溶解DNA沉淀,-20℃保存.
液氮研磨+CTAB+SDS法:将6份冷冻样品混合于研钵(灭菌)中,加液氮充分研磨并混匀,称取0.5 g研磨后的样品于2 mL离心管中,加入1 mL提取液 [100 mmol/LTris- HCl(pH 8.0)、1.5% NaCl、1% CTAB、0.1 mol/L EDTA和10 mmol/L PBS(pH 7.2)]和75 μL 10% SDS,涡旋混匀5 min;加入20 μL蛋白酶K,70 ℃孵育15 min,90 ℃孵育5 min,室温14 000 r/min离心10 min,转移上清至1.5 mL离心管中,加入苯酚-氯仿-异戊醇溶液(体积比25∶24∶1)500 μL,摇匀后静置5 min;以下步骤同液氮研磨+珠磨+SDS+GITC法.
将提取的微生物基因组DNA样品置于-20 ℃冰箱中妥善保存,便于后续试验分析.
1.4 基因组DNA浓度、纯度及片段完整性检测
提取到的DNA用1.2%琼脂糖凝胶电泳检测其片段完整性,用微量紫外分光光度计测定微生物基因组DNA浓度(ng/μL)及D260/280和D260/230比值.
1.5 q-PCR分析
1.5.1 q-PCR标准曲线制作 用M5 HiPer pTOPO-TA Cloning Kit快速克隆试剂盒对目的基因进行克隆,具体操作严格参考说明书.用AxyPrepTMPlasmid Miniprep Kit提取微生物质粒DNA.检测质粒DNA浓度,根据各质粒的分子量与质量浓度计算获得拷贝数,并进行10倍的连续梯度稀释,取8个稀释梯度制得标准品作为模板进行q-PCR反应,每梯度3个重复,绘制Ct值与质粒拷贝数间关系的定量标准曲线.各菌所用引物序列见表1.
标准质粒拷贝数计算公式:
质粒拷贝数(copies/μL)=浓度(ng/μL)×10-9(ng/μL与g/μL之间的换算)×6.02×1023(copies/mol) / [660(g/mol/bp) ×质粒碱基对数(bp)][16].
1.5.2 q-PCR检测条件 标准质粒和样品的q-PCR反应体系均采用20 μL体系.反应体系:2×Biogold qPCR SuperMix 10 μL,上下游引物各0.4 μL,质粒DNA模板(样品DNA模板)1 μL,双蒸水8.2 μL.反应条件:95 ℃预变性3 min,95 ℃变性10 s,60 ℃退火20 s,72 ℃延伸10 s,40个循环,72 ℃延伸10 min.
1.5.3 样品中微生物的拷贝数计算 将每种微生物的Ct值代入相应的标准曲线,利用公式计算得到样品中的微生物拷贝数,对结果进行log转换后再进行数据分析.
计算公式:拷贝数(copies/g)=(MQ×C×VD)/(S×V).其中MQ为根据标准曲线Ct值得到的拷贝数(copies),C是每个样品基因组DNA 的浓度(ng/μL),VD是提取DNA时最后溶解DNA 的双蒸水的体积(μL),S为用于q-PCR的DNA量(ng),V为提取DNA时所称量的样品量(g)[23].

表1 微生物引物序列
1.6 数据统计分析
用SPSS 19.0统计分析软件对测得的DNA浓度、D260/280、D260/230比值和不同微生物log(拷贝数)进行单因子方差(one-way ANOVA)分析,差异显著时,采用Duncan’s法进行多重比较,以P<0.05作为差异显著性评判标准.
2 结果与分析
2.1 不同方法提取瘤胃内容物微生物基因组DNA的浓度、纯度及片段完整性比较
从图1可以看出,4种提取方法均能得到微生物基因组DNA,但提取效果存在差异.液氮研磨+珠磨+CTAB法和珠磨+CTAB法条带较暗且条带下部有明显拖尾现象,液氮研磨+CTAB+SDS法条带较液氮研磨+珠磨+SDS+GITC法弱,液氮研磨+珠磨+SDS+GITC法条带最亮,条带无拖尾也无杂质污染,说明该方法所获得的基因组DNA产量较高.
由表2可以看出,4种方法所提取的瘤胃内容物微生物基因组DNA浓度和D260/280、D260/230比值均存在显著差异(P<0.001).珠磨+CTAB法获得的DNA样品浓度最高,液氮研磨+珠磨+SDS+GITC法获得的DNA浓度次之,平均浓度为1 121.15 ng/μL.OD260/280比值反映蛋白质污染情况,测得液氮研磨+珠磨+CTAB法、珠磨+CTAB法和液氮研磨+珠磨+SDS+GITC法所得DNAD260/280>1.9,液氮研磨+CTAB+SDS法所得DNA样品D260/280<1.8.D260/230比值表征DNA样品中碳水化合物、腐殖酸和酚类等污染情况,4种方法所得DNAD260/230比值均小于2.0,其中液氮研磨+珠磨+SDS+GITC法该值最高为1.92,显著大于另外3种方法(P<0.001),液氮研磨+珠磨+CTAB法和珠磨+CTAB法低于液氮研磨+珠磨+SDS+GITC法,液氮研磨+CTAB+SDS法D260/230比值最低.
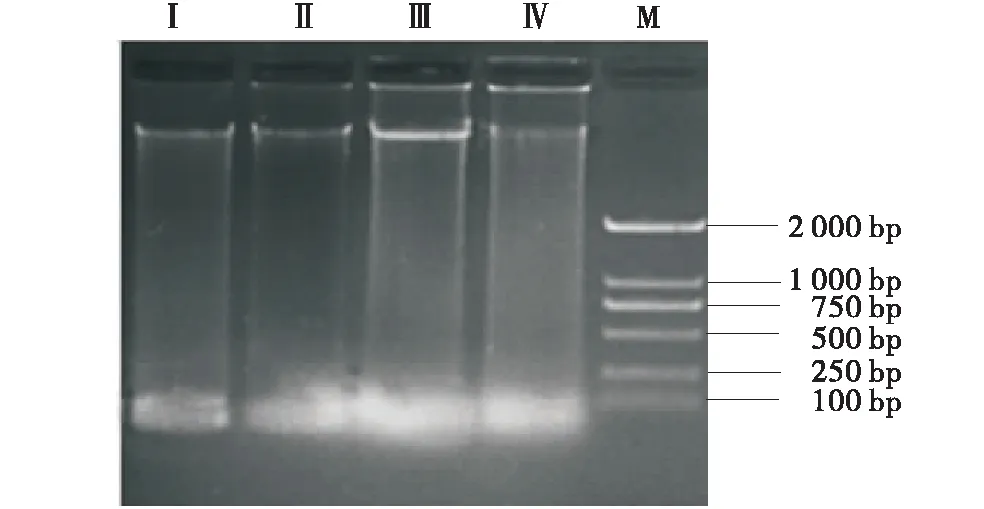
液氮研磨+珠磨+CTAB法(Ⅰ);珠磨+CTAB法(Ⅱ);液氮研磨+珠磨+SDS+GITC法(Ⅲ);液氮研磨+CTAB+SDS法(Ⅳ).Liquid nitrogen grinding+bead mill+CTAB method (Ⅰ);Bead mill+CTAB method (Ⅱ);Liquid nitrogen grinding+bead mill+SDS+GITC method (Ⅲ);Liquid nitrogen grinding+CTAB.SDS method (Ⅳ).图1 羔羊瘤胃内容物微生物基因组DNA琼脂糖凝胶电泳Figure 1 Microbial genomic DNA agarose gel electrophoresis of rumen contents in lambs

表2 不同提取方法获得的羔羊瘤胃内容物微生物基因组DNA浓度(ng/μL)和D值
2.2 重组质粒标准曲线
以已知拷贝数的质粒标准品为模板进行q-PCR反应,10倍为一个稀释梯度.建立反映Ct值与质粒拷贝数对应关系的定量标准曲线,根据曲线和计算公式可得样品中各细菌和产甲烷菌的数量.标准曲线见图3.从图3中可以看出,微生物重组质粒DAN的的标准曲线的斜率为-0.279 5~-0.347 3,相关系数均大于0.98,符合q-PCR标准曲线的要求.

图2 q-PCR检测质粒标准品的熔解曲线Figure 2 Melting curve of plasmid standard substance detected by q-PCR
2.3 羔羊瘤胃内容物微生物拷贝数
对4种方法所获得的羔羊瘤胃内容物微生物基因组DNA样品进行了q-PCR检测,根据q-PCR得到的Ct值及公式计算出4种方法所得DNA样品中各细菌和产甲烷菌的拷贝数.
由表3可知,4种方法提取到的基因组DNA经q-PCR分析,瘤胃细菌和产甲烷菌的数量存在显著差异(P<0.001),其中琥珀酸拟杆菌(Fibrobactersuccinogenes)数量最高,总细菌(Total bacteria)次之,白色瘤胃球菌(Ruminococcusalbus)数量最少.液氮研磨+珠磨+SDS+GITC法所获得的DNA样品经检测瘤胃总细菌的数量显著大于其它方法(P<0.001)、产甲烷菌的丰富度均显著高于其它方法(P<0.001),该方法提取效率较高.液氮研磨+珠磨+SDS+GITC法和液氮研磨+CTAB+SDS法所得羔羊瘤胃内容物微生物DNA样品中琥珀酸拟杆菌(Fibrobactersuccinogenes)、白色瘤胃球菌(Ruminococcusalbus)、嗜淀粉瘤胃杆菌(Ruminobacteramylophilus)、甲烷杆菌(Methanobacteriales)和瘤胃甲烷短杆菌(Methanobrevibacters)的数量近似,且显著高于另外2种方法(P<0.001).液氮研磨+珠磨+SDS+GITC法得到的DNA样品中溶纤维丁酸弧菌(Butyrivibriofibrisolvens)数量显著高于其它方法(P<0.001),液氮研磨+CTAB+SDS法对该菌的提取效率也较高.珠磨+CTAB法获得的DNA样品中瘤胃细菌和产甲烷菌的数量显著低于其他3种方法(P<0.001),且该方法对于瘤胃产甲烷菌的提取效率较差,总甲烷菌的丰富度仅为5.52 ng/μL,该法所获得的羔羊瘤胃微生物DNA多样性较差.液氮研磨+珠磨+CTAB法得到的DNA经q-PCR检测瘤胃细菌和产甲烷菌数量较珠磨+CTAB法多.

图3 微生物重组质粒DNA的标准曲线Figure 3 Standard curve of recombinant plasmid DNA of microorganisms
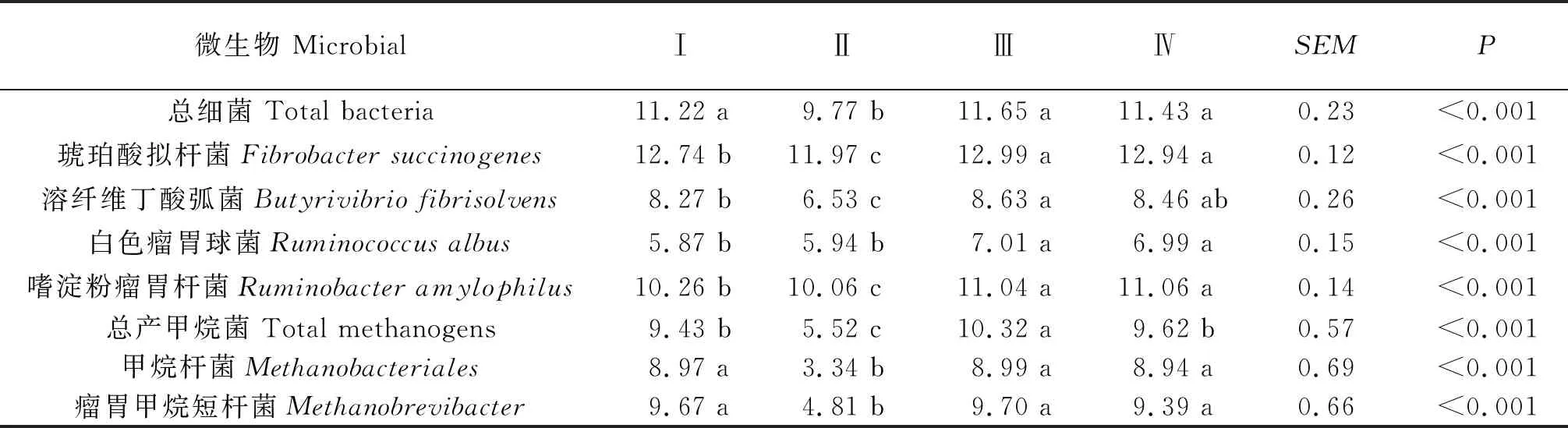
表3 不同DNA提取方法对羔羊瘤胃内容物微生物数量的影响
3 讨论
羔羊瘤胃内容物成分复杂,含有庞大的微生物种群、饲料残渣以及消化道脱落细胞和许多不明生物物质[24],这使得提取高质量的羔羊瘤胃内容物微生物基因组DNA有一定难度.本试验中,4种方法提取得到的基因组DNA经琼脂糖凝胶电泳检测结果显示,4条泳道均有DNA谱带出现,每种方法均加入蛋白酶K,促使微生物细胞破裂而释放更多的DNA[25],虽然方法不同,但最终目的都是有效地使微生物细胞壁裂解,去除非核酸成分且避免核酸的损失而得到高质量的基因组DNA[26].液氮研磨+珠磨+CTAB法和珠磨+CTAB法条带有明显拖尾现象,且泳道下方有亮的核酸短片段,DNA条带也较液氮研磨+珠磨+SDS +GITC法暗.液氮研磨+珠磨+CTAB法和珠磨+CTAB法均使用了CTAB抽提液,CTAB是一种常用的阳离子洗涤剂,可以沉淀多糖和蛋白质,但是不能有效地裂解细胞[27].研究发现,CTAB更适合于提取真菌基因组DNA[28],CTAB法主要用于植物性总DNA的提取[29].本试验中珠磨+CTAB法得到DNA样品浓度较高但q-PCR检测结果显示瘤胃细菌和产甲烷菌的数量显著低于其它3种方法(P<0.001),可能是提取到了部分瘤胃真菌和瘤胃内容物中未完全降解的植物纤维的DNA而导致核酸浓度较高.D260/280、D260/230是评价DNA纯度的常用指标,Henderson等[11]提出用于分子生物学研究的参考标准,基因组DNAD260/280≈ 1.8,D260/230值在2.0~2.2为宜.结合表1分析,这两种方法得到的DNA样品D260/280值均大于1.9(D260/280≈ 1.8,大于1.9表明有RNA污染;小于1.7表明有蛋白质污染),泳道下部短的核酸片段可能是植物细胞的DNA或者RNA.试验中液氮研磨+珠磨+CTAB法同时使用了珠磨和液氮研磨两种物理破壁方法再结合CTAB法提取到的DNA样品经q-PCR检测瘤胃细菌和产甲烷菌的数量均高于珠磨+CTAB法(仅用了珠磨),说明增强前期细胞破壁处理能够弥补CTAB不能有效裂解微生物细胞壁的缺陷而得到高质量的基因组DNA.液氮研磨+珠磨+SDS+GITC法和液氮研磨+CTAB+SDS法主要采用SDS裂解微生物细胞,SDS是一种阴离子清洗剂,它能够破坏蛋白质之间的相互作用,溶解细胞膜[30].SDS对革兰氏阳性菌和革兰氏阴性菌均有较好的裂解效果,能够得到高质量的微生物基因组DNA[31],提取过程中加入SDS裂解液后70 ℃水浴加强了裂解效果.本试验中,液氮研磨+珠磨+SDS+GITC法加入了GITC,其使得蛋白变性,提高了DNA的产量,该方法所得DNA样品浓度为1121.15 ng/μL,D260/280比值为1.94,D260/230比值为1.92,从q-PCR检测结果(表3)可知,革兰氏阳性菌溶纤维丁酸弧菌(Butyrivibriofibrisolvens)和白色瘤胃球菌(Ruminococcusalbus)的数量均显著大于其它方法,这一结果与曾波[32]等的研究结果一致.但Yu等[33]发现,应用蛋白酶K和GITC提取地表水中鞭毛虫DNA的产率较低.Myer等[34]利用反复珠磨结合SDS提取的肉牛瘤胃微生物基因组DNA可用于16S rRNA测序分析.本试验结果表明,液氮研磨+珠磨+SDS+GITC法所获得的羔羊瘤胃内容物微生物DNA样品用于q-PCR检测表明,革兰氏阳性菌溶纤维丁酸弧菌(Butyrivibriofibrisolvens)和白色瘤胃球菌(Ruminococcusalbus)的数量较高,说明SDS和GITC对羔羊瘤胃细菌和产甲烷菌有良好的作用,能够有效地裂解细胞使细胞内和细胞壁上的DNA充分释放出来,该方法所获得的微生物DNA样品能够用于羔羊瘤胃内容物中微生物的多样性分析.液氮研磨+CTAB+SDS法提取得到的DNA样品浓度较液氮研磨+珠磨+SDS+GITC法低,其D260/230比值为1.64,表明所得DNA样品存在碳水化合物或者化学试剂污染,需要进一步纯化.液氮研磨+CTAB+SDS法对于瘤胃细菌和产甲烷菌DNA的提取效果略低于液氮研磨+珠磨+SDS+GITC法.
本试验中,珠磨+CTAB法所得DNA样品浓度较高,利用q-PCR不能高效地检测到瘤胃细菌和产甲烷菌数量,液氮研磨+珠磨+CTAB法得到的DNA样品经q-PCR检测结果优于珠磨+CTAB法,这可能是因为提取过程中DNA降解严重,片段不完整,无法用于分子生物学研究.液氮研磨+珠磨+SDS+GITC法能够得到较纯的DNA样品,利用q-PCR技术能够准确地检测出瘤胃细菌和产甲烷菌.液氮研磨+CTAB+SDS法所获得DNA样品浓度和纯度低于液氮研磨+珠磨+SDS+GITC法,需要进一步纯化,液氮研磨+CTAB+SDS法得到的DNA样品中瘤胃细菌和产甲烷菌数量与液氮研磨+珠磨+SDS+GITC法相近.由此得出,SDS较CTAB更适合于提取羔羊瘤胃内容物微生物基因组DNA.综上所述,液氮研磨+珠磨+SDS+GITC法提取的羔羊瘤胃微生物基因组DNA浓度和纯度较高,且q-PCR检测出瘤胃细菌和产甲烷菌的数量最多,能够提供质量较高的羔羊瘤胃内容物微生物基因组DNA用于分子生物学研究.
4 结论
本研究采用液氮研磨+珠磨+SDS+GITC法所获得的羔羊瘤胃内容物微生物基因组DNA质量较高,可用于研究羔羊瘤胃内容物中微生物的定植.为全面了解羔羊瘤胃健康发育奠定基础.
