基于GenBank中16S rRNA分析的产甲烷菌的地域分布特征
2017-10-31晏磊郎传平王伟东王彦杰
晏磊,郎传平,王伟东,王彦杰
基于GenBank中16S rRNA分析的产甲烷菌的地域分布特征
晏磊,郎传平,王伟东,王彦杰
(黑龙江八一农垦大学生命科学技术学院,大庆 163319)
通过从GenBank中搜集产甲烷菌,探讨产甲烷菌的地域分布特征。目前,收集到的甲烷杆菌纲,甲烷球菌纲和甲烷火菌纲产甲烷菌16S rRNA序列235条,相似性分析后剩余191条。综合分析产甲烷菌16S rRNA的全长、前段、中段和后段的系统进化树可发现,全球不同地域来源的产甲烷菌的分布有一定的特征,甲烷球菌纲的产甲烷菌多来源于海洋沉积物,甲烷微菌纲和甲烷杆菌纲的来源比较广泛,但也有一定的偏好性,沉淀物和动物来源的产甲烷菌具有明显差别,而同为淡水、油田或是稻田的产甲烷菌的相似性却较高。基于此推测产甲烷菌的起源可能是多源的,环境可能作为进化压力在产甲烷菌的长期进化过程中扮演重要的角色。
产甲烷菌;16S rRNA序列分析;地域分布;系统发育
甲烷是一种温室气体,更是新的清洁能源。产甲烷菌是甲烷的主要来源,也是一类极端厌氧古细菌,属水生古细菌门(Euryarchaeota)。甲烷细菌有球形、杆形、螺旋形,八叠球状等,还有的能连成长链状[1]。产甲烷菌属于严格厌氧微生物,常和伴生菌生活在一起,菌体大小形态都十分相似,在一般光学显微镜下不好判明。而且其生长繁殖特别缓慢,故而培养分离比较困难。美国著名微生物学家Hungate曾在上世纪50年代培养分离甲烷细菌并获得成功[2]。其后世界上很多研究者对甲烷细菌进行了培养分离工作,并对Hungate所建立的分离方法进行了改良,使甲烷细菌相比过去更易于培养分离。产甲烷菌存在于严格厌氧环境,如人类的消化系统和反刍动物的瘤胃、动物粪便、厌氧污泥和沼气反应器、湖泊或海底沉积物、深部油层和煤层等环境中均有分布。此外,产甲烷菌还存在于某些极端环境中[3],其数量丰富,种类繁多。根据《伯杰氏细菌鉴定手册》(第九版)截至2016年产甲烷菌已经发展为4纲5目[5],分别为甲烷杆菌纲(Methanobacteria),甲烷球菌纲(Methanococci),甲烷微菌纲(Methanomicrobia),甲烷火菌纲(Methanopyri),尤其是甲烷微菌纲包括产甲烷菌数目最多。通过对其基因组学的研究使得对其代谢特点和分类与其地域分布的联系被了解。
16S rRNA经常被用来作为原核生物系统发育以及多样性研究的标志性基因,用表型方法鉴定有困难的原核生物,用16S rRNA序列分析能进行快速准确的鉴定[4]。目前为止,中国,日本,印度,德国,中国台湾,美国,俄罗斯,太平洋,印度洋以及大西洋等世界各处均有发现产甲烷菌。因而,通过对GenBank中搜索到的235条产甲烷菌序列的16S rRNA的分析,了解产甲烷菌的地域分布特征,进而探寻到产甲烷菌进化起源。
1 数据来源与分析方法
1.1 数据来源
截止到2016年4月15日在GenBank中搜索得到的235条产甲烷菌16S rRNA序列。搜索的关键词有 methanogen 16s,archaeon clone biogas 16s,archaeon methane-producing 16s等,尽可能包括所有产甲烷菌的16S rRNA。由于存在许多长短不一的序列,因而在搜索数据时对其长度要求为>700 bp,并且对其进行甄别,去除非产甲烷菌的16S rRNA序列。
1.2 数据整理与分析方法
将得到的所有产甲烷菌16S rRNA序列信息进行整理,利用BLAST(Basic Local Alignment Search Tool,http://www.ncbi.nlm.nih.gov) 在线比对工具对比,根据Stackebrant等认为16S rRNA相似性>97%,认为属于同一个种[6],对于同一地点,同一批次提交的16S rRNA序列,若有两条序列相似性>97%,则认为两者属于同一种产甲烷菌,只需保留一条即可[7]。对所有序列进行整理,确定其分离源,国家,分类地位等,并且统计不同生境产甲烷菌在各纲中所占比例。在构建系统进化树时,由于所取得的序列长短不一,所作出的进化树可靠性不强,所以利用产甲烷菌的 16S rRNA 的前段(800 bp),中段(500 bp),后段(1 000 bp)和全长分别进行系统进化树分析(利用MEGA4.0软件采用N-J法构建系统发育树)。同时就不同生境,不同分离源,不同特点的产甲烷菌进行分析,以寻求地域规律性。
2 结果与分析
2.1 原始数据整理
对原始数据进行整理后获得剩余产甲烷菌16S rRNA191条,对其分离源进行分类整理,分别为沉积物来源(包括来自海洋,陆地,油田,地下水,红树林等各种生境的沉积物),生物反应器来源(包括UASB反应器,沼气反应器,沼气发电场等各种生物反应器),动物来源(包括反刍动物的瘤胃,动物粪便,食木昆虫的消化道等)。这三种分离源的产甲烷菌16S rRNA数量分别为110,39,42,在各纲中的比例见图1。产甲烷菌最多的是甲烷微菌纲,其次是甲烷杆菌纲,甲烷火菌纲收集的数据较少,结合分离源分析发现产甲烷菌的甲烷微菌纲和甲烷杆菌纲在三种来源中均占有很大比例,说明在自然界这俩纲的产甲烷菌的分布非常广泛,数量领先,因此,甲烷微菌纲是目前产甲烷菌中研究最多的种类。而甲烷菌的动物来源中甲烷杆菌纲的数目高于甲烷微菌纲,这也许和动物体内特殊的微环境有关。
在收集到的191条产甲烷菌的16S rRNA中,有甲烷杆菌纲57条,甲烷球菌纲14条,甲烷微菌纲119条,甲烷火菌纲1条,上述四纲的分离来源以及具体分布情况如表1。GenBank中现有的产甲烷菌主要分布于中国,德国,日本,印度,美国,中国台湾,太平洋,印度洋,大西洋等地。各个地区中大多以甲烷微菌纲的甲烷菌数目最多,而美国和法国却以甲烷杆菌纲的产甲烷菌数量占优势,另外甲烷微菌纲的分布集中于各大海洋。
2.2 产甲烷菌的系统进化分析
产甲烷菌的16S rRNA的系统进化树如图2,由产甲烷菌的前段,中段,后段以及全段所作出的系统进化树虽然细节不同,但大体形状是相似的,分别出现四个分枝,为分类学上的四纲,从进化树中可以看出从总的分类上来看,分离源相同的大多聚类分区明显,但也有少数是例外的。以TR-Z13,NBRC 103675为代表的微菌纲的产甲烷菌形成一个最大的分支,以A3,FS406-22为代表的甲烷球菌形成一个较小的分支,以8-2,ATM为代表的甲烷杆菌形成另一个分支,这些都与实际观察相符合。
2.3 不同分离源的产甲烷菌的比较
2.3.1 沉积物来源与动物来源的产甲烷细菌的比较
沉积物与动物来源的产甲烷细菌比较(见图1),由图可知沉积物来源中甲烷微菌纲(66.4%)占优势,而在动物来源中以甲烷杆菌纲(61.9%)为主,另外还发现球菌纲大多数只来源于沉淀物。图3是动物来源和沉淀物来源的产甲烷菌的16S rRNA的单独构建的系统进化树,对此观察分析得到,沉淀物来源与动物来源的产甲烷菌的相似性较低,系统进化位置差别明显。动物来源的产甲烷菌的16S rRNA,相似性较高(90%以上),而沉淀物来源的产甲烷菌的16S rRNA的相似性较低(最高为97%,最低为72%)。

图1 产甲烷菌16S rRNA各纲的来源比例Fig.1 The classification proportion of source with 16S rRNA for Methanogenic bacteria

表1 产甲烷菌数据统计(据提交GenBank的16S rRNA序列数统计)Table 1 Methanogenic Bacteria statistics(according to the 16S rRNA submitted to the GenBank)
2.3.2 不同的沉淀物来源的产甲烷菌的比较

图2 产甲烷菌16S rRNA的系统进化树Fig.2 Phylogenetic tree based on 16S rRNA of Methanogenic bacteria注:图2中加菱形符号的16S rRNA序列为动物来源
由图3可知,来自淡水沉积物和海水沉淀物的产甲烷菌各自聚类分区较为明显,来自淡水的产甲烷菌属于甲烷微菌纲居多,且球菌纲数目稀少,目前未发现来源于淡水沉淀物甲烷微菌纲的产甲烷菌;来自海水沉淀物的甲烷菌在各纲都有分布,种类丰富,相似性相差较大(最高达99%,最低只有79%),来自淡水和海水的甲烷微菌纲的相似性较高。对于沉淀物中其他来源,如全球各地的水稻田中发现的产甲烷菌的16S rRNA进行两两比对,发现产甲烷菌两两相似性较高(平均85%以上),另外又对沉淀物中全球油田来源的产甲烷菌的16S rRNA进行了两两相似性分析,发现两两相似性也较高(平均88%以上)。
2.3.3 不同的动物来源的产甲烷菌的比较
由图4可知,反刍动物瘤胃和动物粪便来源的产甲烷菌的聚类分区不明显,进化树分叉为甲烷微菌纲和甲烷杆菌纲两种来源的产甲烷菌在上述两纲中的分布较为均匀,对产甲烷菌的16SrRNA进行两两比对,发现两种来源的产甲烷菌在同一纲中的两两相似性较高(平均85%以上)。

图3 海洋沉淀物与淡水沉淀物的来源的产甲烷菌的16SrRNA的系统进化树Fig.3 Phylogenetic tree based on 16SrDNA of Methanogenic bacteria in marine sediments and freshwater sediments注:图3中圆形图标的是淡水沉淀物来源的产甲烷菌16S rRNA序列
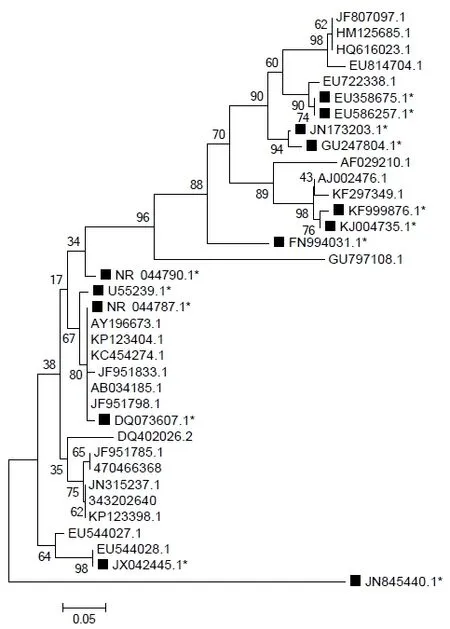
图4 动物粪便和反刍动物瘤胃来源的产甲烷菌的16SrRNA的系统进化树Fig.4 Phylogenetic tree based on 16SrDNA of Methanogenic bacteria in animal manure and rumen in ruminants注:图4带有方形标记的是动物粪便来源的产甲烷菌的16S rRNA序列
3 讨论
对于不同区域来源的产甲烷菌系统发育进行分析,发现相似性高的产甲烷菌多来源于相近的环境中。如已获得纯培养的2株产甲烷菌MT(AY260432.1)和 SMA-21(NR 109423.1),两者的相似性大于97%[7-8],他们分别发现于德国寒冷的陆地和西伯利亚勒拿河三角洲的活性冻土。两株可培养的产甲烷菌的 DMS1(AF120163.1)和 TM(AJ276437.1)的相似性也高于97%[9-10],分别发现于荷兰和坦桑尼亚的淡水湖中。法国脑脓肿病人的脓液中发现的产甲烷菌Vande(LN876656.1)与美国动物的粪便中发现的产甲烷菌GS(NR 044788.1)的相似性达97%[11-12]。此外,甲烷球菌纲的产甲烷菌多发现于海洋,由此表明产甲烷菌的分布可能具有一定的区域性特征。产甲烷菌可能作为土著菌生活在海洋中,经过长期进化,形成了特定的形态。如上文所分析,相似的产甲烷菌多来源于相近的环境中,这也可能是因为长期演化过程中环境条件作为重要的进化压力使得处于相似环境中的产甲烷菌向相同方向进化。然而也有不同的情况,如三株产甲烷菌如H-A4(KF952484.1)、PL-15/HP(DQ136171.1)和 Mic6c09(AB546256.1)分别分离于墨西哥高钠盐碱土的特斯科湖、美国阿拉斯加的海洋沉积物和日本腐蚀的天然气管道中[13-16]。究其原因可能是三株菌在单独进化过程中的趋同进化,可能是进化偶然,也有可能是环境中某个环境因子(如pH)作为进化压力促使产甲烷菌朝同一方向进化。研究受目前GenBank的产甲烷菌的16SrRNA的数目限制,随着对产甲烷菌的更加深入的研究,将得到更多纯培养的菌株,更多更大数量的产甲烷菌的发现,必然能使人们获得更加全面的关于不同地域分布的产甲烷菌的特征,并为产甲烷菌的生态意义和应用前景做出更多贡献。
4 结论
不同区域产甲烷菌的系统进化分析发现产甲烷菌具有明显的生态分布特征。沉淀物来源和动物来源的产甲烷菌区分明显,淡水和海水来源的产甲烷菌也有明显差别。而同为沉淀物和动物来源的产甲烷菌的相似性较高,反映生态环境和产甲烷菌的分布相关性密切。这些观测都暗示在长期演化过程中,环境条件作为重要的进化压力,促使产甲烷菌向同一方向进化(相似的环境形成相似的产甲烷菌类型)或多方向进化(不同环境形成了不同的产甲烷菌类型),由此说明产甲烷菌的起源可能是多源的。
[1] 王保玉,刘建民,韩作颖,等.产甲烷菌的分类及研究进展[J].基因组学与应用生物学,2014,33(2):418-425.
[2] 李煜珊,李耀明,欧阳志云.产甲烷微生物研究概况[J].环境科学,2014,35(5):3295-3301.
[3] 朱晨光,宋任涛.产甲烷古菌[J].能源作物育种,2009,29(1):1-5.
[4] 张之强,孙成权,程国栋,等.可持续发展研究进展与趋向[J].地球科学进展,1999,14(6):589-595.
[5] 崔晓光,范圣第,权春善.沼气池中产甲烷菌的分离鉴定及其分布的研究[D].大连:大连理工大学,2007.
[6] 张文燕,张圣妲,肖天,等.趋磁细菌的地域分布特征[J].环境科学,2010,31(2):450-458.
[7] 郝鲜俊,洪坚平,高文俊.产甲烷菌的研究进展[J].资源环境,2007,35(1):111-113.
[8] 刘艺,邹冬生.甲烷菌的分离纯化和产气效果研究[J].生态学,2014,S181:10537.
[9] Brber R D,Zhang L,Harnack M,et al.Ingram-Smith K.S.Complete genome sequence Menthanosaeta concilii,a specialist in aceticlastic methanogensis[J].Journal of Bacteriology,1993(14):3668-3669.
[10] Sundset M,Edwards J.Molecular Diversity of the rumen microbiome of Norwegian Reindeer on Natural Summer Pasture[J].Microbial Ecology,2008(8):9414-9417.
[11] Hasan S H,Talat M,Rai S.Sorption of cadmium and zine from aqueous solution by water hyaeinth[J].Bioresouree Teehnology,2007,98:918-928.
[12] Hungate R E.Methods in Mcrobiology[M].New York:A-cademic Press Inc,1969:117-132.
[13] Mikucki J A,Liu Y,Delwiche M,et al.Isolation from Deep Marine Sediments That Contain Methane Hydrates,and Description of Methanoculles submarinus sp.Nov[J].Applied and Environmental Microbiology,2003,69:3311-3316.
[14] Tabassum R, Rajoka I.Methanogensis of carbohydrates and their fermentation products by syntrophic Methane producing bacteria isolated from freshwater sediments[J].Biosourse Technology,2007,72(18):199-205.
[15] Maria V,Sizova A,Nicolai S,et al.Isolation and Characterization of oligotrophic acidotolerant methanogenic consortia from a Sphagum peat bog[J].Microbiology Ecology,2003,45:301-315.
[16] Sun W T,Xiao Q M,Lou C R.Production and emission of methane and it’s influencing factor in soil[J].Rain Fed Crops,2000,20(5):44-47.
Geographical Distribution Characteristics of Methanogenic Bacteria Based on the Analytics of 16S rRNA in GenBank
Yan Lei,Lang Chuanping,Wang Weidong,Wang Yanjie
(College of Life Science and Technology,Heilongjiang Bayi Agricultural University,Daqing 163319)
The geographical distribution characteristics of methanogen were discussed through collecting the methanogen from Gen-Bank,235 sequences of 16S rRNA of methanogenic bacteria were collected,191 sequences were left after the filtering.It could be found that the global distribution of methanogens from different regions had certain characteristics after the full-length,front,middle and rear sections of the phylogenetic tree of the 16S rRNA of the methanogens being analyzed comprehensively.The large majority of the Methanococcus are from the ocean sediments.Although,huge numbers of Methanobacteria and Methanomcrobia were widespread throughout the entire earth but had a certain preference,Methanogens isolated from sediments and animals respectively had significant differences,those strains with similar habitats had higher similarity such as the trains from fresh water,oil and paddy.The type of Methanogen was closely related to the habitat condition.Thus,the origin of methanogenic bacteria might be multiple and the environment play an important role in its long evolutionary process.
methanogen;16S rRNA of sequence analysis;geographical distribution;Phylogeny
Q938
A
1002-2090(2017)05-0059-07
10.3969/j.issn.1002-2090.2017.05.015
2016-10-21
秸秆发酵高效产沼气菌系的构建及功能(B2011-13)。
晏磊(1981-),男,副教授,兰州大学毕业,现主要从事微生物方面的教学与研究工作。
