扩展青霉erg4的生物信息学、亚细胞定位及表达分析
2021-02-10韩占红宗元元张学梅王斌PRUSKYDov毕阳
韩占红 宗元元 张学梅 王斌 PRUSKY Dov 毕阳
(1. 甘肃农业大学食品科学与工程学院,兰州 730070;2. Department of Postharvest Science of Fresh Produce,Agricultural Research Organization,Volcani Center,Bet Dagan 50250,Israel)
扩展青霉(Penicillium expansum Link)是重要的采后病原真菌,寄主范围广,可侵染苹果、梨、桃、樱桃等二十多种果实,其所引起的青霉病造成的采后经济损失巨大[1]。此外,P. expansum还会在果实体内产生对消费者健康具有潜在危害的棒曲霉毒素[2]。因此,减轻P. expansum对果实的危害是当前生产中亟待解决的问题。
麦角甾醇(Ergosterol)是真菌细胞膜上的特有组分,在真菌生长发育过程中具有重要作用[3],可确保真菌细胞膜的稳定性、流动性、渗透性以及膜上相关蛋白的活性[4-8]。此外,麦角甾醇还对真菌细胞内外的信号分子转导、膜蛋白的正确定位以及功能的正常发挥具有重要影响[9-10]。麦角甾醇的生物合成受一系列erg基因的调控,其中erg4编码的是一种甾醇C-24(28)还原酶(sterol C-24(28)reductase),催化麦角甾醇合成的最后一步反应,将前体物质ergosta-5,7,22,24(28)-四烯醇转化为麦角甾醇[11-12]。酿酒酵母(Saccharomyces cerevisiae)中仅存在一个erg4基因,该基因的缺失不仅导致细胞生长缺陷,还会改变细胞膜的结构[13-14]。缺失erg4的S. cerevisiae对氧化胁迫的敏感性增加[15]。敲除erg4的红法夫酵母(Red Yeast Xanthophyllomyces dendrorhous)麦角甾醇合成受到抑制,导致前体物质大量积累[16]。禾谷镰孢菌(Fusarium graminearum)中erg4的缺失也会导致菌丝生长缓慢、产孢量降低、分生孢子畸形、分隔数减少、脱氧雪腐镰刀菌烯醇合成量下降、致病性及抗氧化胁迫能力显著降低[17]。烟曲霉(Aspergillus fumigatus)中存在erg4A和erg4B两个erg4同源基因,erg4A和erg4B的共同缺失会完全阻断麦角甾醇合成,并造成严重的产孢缺陷,但其单独缺失既不影响麦角甾醇的合成,也不影响正常产孢[18]。虽然已有erg4调控酵母和其他丝状真菌生长发育、致病性及产毒方面的报道,但该基因编码蛋白是如何参与P. expansum的生物学过程还未见报道。我们前期的生物信息学分析结果显示,P. expansum中存在erg4A、erg4B和erg4C三个erg4基因,但基本生物学功能未知。
本研究通过RT-PCR方法扩增erg4A、erg4B及erg4C的CDS区全长;利用生物信息学方法对这3个基因的序列信息、编码蛋白的跨膜螺旋及亲疏水性进行预测分析;通过构建3个基因的GFP融合表达载体,利用农杆菌介导转化P. expansum后观察3个融合表达蛋白的亚细胞定位。此外,还通过RT-qPCR对这3个基因在P. expansum不同生长发育时期、不同培养基状态以及在黑暗和蓝光培养条件下的表达特性进行分析。以期为全面解析这3种蛋白质的结构和功能研究提供理论依据。
1 材料与方法
1.1 材料
1.1.1 实验材料 扩展青霉Penicillium expansum T01和根癌农杆菌(Agrobacterium tumefaciens EHA105)及携带绿色荧光蛋白基因gfp和筛选标记基因G418的亚细胞定位载体pC-NEO-GFP由中国科学院植物研究所果实逆境应答及生物控制技术研究组惠赠。P.expansum于马铃薯葡萄糖琼脂培养基(PDA)上活化扩繁。
1.1.2 仪器与设备 光学显微镜(CX21FSIC型,奥林巴斯工业有限公司,中国);超净工作台(SW-CJ-2FD型,苏净集团苏州安泰空气技术有限公司,中国);高速冷冻离心机(H-1850R型,长沙湘仪离心机仪器有限公司,中国);立式压力蒸汽灭菌锅(LDZX-30KBS 型,上海申安医疗器械厂,中国);荧光显微镜(BX51DP74型,日本奥林巴斯有限公司,日本);恒温培养箱(DHP-9082型,上海-恒科学仪器有限公司,中国),超低温冰箱(DW-HL218型,中科美菱低温科技有限公司,中国);PCR热循环仪(S1000,BIO-RAD,美国);超微量紫外可见分光光度计(206-26300-48,日本岛津);实时荧光定量PCR系统(QuantStudio®5,美国THERMO FISHER,美国);激光扫描共聚焦显微镜(LSM800,德国Zeiss公司)。
1.2 方法
1.2.1 erg4A、erg4B和erg4C基因序列的获取 根据NCBI(https://www.ncbi.nlm.nih.gov/)中 P. expansum MD-8的erg4基因序列进行Nucleotide BLAST比对,检索获得P. expansum T01中erg4A、erg4B和erg4C的基因全长序列。
1.2.2 总RNA的提取及第一链cDNA的合成 用TRNzol Universal总RNA提取试剂盒(天根生化科技有限公司,北京)提取P.expansum菌丝总RNA,用1%琼脂糖凝胶电泳检测其结构的完整性,用超微量紫外可见分光光度计检测RNA的浓度和纯度,使用PrimeScripTMRT reagent Kit with gDNA Eraser试剂盒(TaKaRa,RR047A)进行gDNA消化以及第一链cDNA的合成,于-20℃保存待用。所有操作均按试剂盒说明书进行。
1.2.3 erg4A、erg4B和erg4C基因CDS的克隆 根据上述分析得到的3个基因全长序列,设计特异性引物,引物序列见表1。以cDNA为模板,用2×Phanta Max Master Mix高保真酶(诺唯赞生物,南京)进行PCR扩增,PCR反应体系如下:0.5 μL F引物(10 μmon/L),0.5 μL R 引物(10 μmon/L),1 μL cDNA,10 μL 2× Phanta Max Master Mix,8 μL ddH2O。扩增产物经1%的琼脂糖凝胶电泳检测后,使用胶回收试剂盒(全式金生物,北京)进行PCR产物的回收,回收产物送北京擎科生物科技有限公司测序。测序结果用DNA MAN软件进行比对分析。

表1 erg4A、erg4B和erg4C基因CDS的克隆引物序列Table 1 Primers sequence for the clone of CDS of gene erg4A,erg4B and erg4C
1.2.4 Erg4A、Erg4B和Erg4C蛋白的生物信息学分析 通过在线分析工具GSDS(Gene Structure Display Server,http ://gsds.gao-lab.org/)分析这 3 个基因的结构,利用NCBI数据库提供的ORF Finder(https://www.ncbi.nlm.nih.gov/orffinder) 对 DNA 序列进行开放阅读框(ORF,Open Reading Frame)预测。利用生物学在线分析工具-TMHMM 2.0(http://www.cbs.dtu.dk/services/TMHMM)和 ProtScale(https://web.expasy.org/protscale/) 对 Erg4A、Erg4B及 Erg4C的跨膜螺旋和蛋白亲疏水性进行预测分析。
1.2.5 Erg4A-GFP、Erg4B-GFP和Erg4C-GFP融合表达载体构建 利用同源重组的方法分别在Erg4A和Eg4B蛋白的N端以及Erg4C蛋白的C端进行GFP的标记。构建GFP融合表达载体的引物见表2。

表2 用于构建GFP融合表达载体的引物序列Table 2 Primer sequences for GFP fusion vector construction
1.2.6 P. expansum的转化 参考Li等[19]农杆菌介导转化P. expansum的方法,通过PCR验证阳性转化子。阳性转化子的验证引物序列见表3。

表3 转化子验证所用引物序列Table 3 Primer sequences for transformant validation
1.2.7 Erg4A、Erg4B和Erg4C的亚细胞定位的观察在10 mL CY液体培养基中接种1 mL P. expansum孢子悬浮液,于25℃、200 r/min条件下培养13-14 h,使其孢子萌发,于8 000 r/min离心5 min,用磷酸盐缓冲液洗涤3遍,在一洁净载玻片中央滴加15 μL菌悬液,将盖玻片扣在其上,轻压后置于共聚焦显微镜下观察。并分别进行目标蛋白与细胞核和内质网的共定位观察。
细胞核染色:使用终浓度为0.6 μg/mL的DAPI荧光染料对样品进行避光染色15 min,用磷酸盐缓冲液洗涤除去染液后进行荧光显微镜观察。
内质网染色:取少量ER-Tracker Red染料按照1∶1 000的比例加入到ER-Tracker Red稀释液中制成工作液,使用前于37℃水浴锅预温育。工作液与待观察样品按1∶1的比例混合,37℃孵育25 min后,用磷酸盐缓冲液洗涤去除染料,随后置于荧光显微镜下观察。
1.2.8 RT-qPCR反应分析基因mRNA水平 总RNA的提取及cDNA的合成参照1.2.2的方法。利用实时荧光定量PCR分析P. expansum在不同发育阶段、不同培养基状态以及黑暗和蓝光培养条件下的erg4的mRNA转录水平。以β-tubulin(GenBank accession number:AF003248.1)作为内参基因。PCR反应体系为 20 μL,包含 :10 μL TB Green Premix Ex Taq II,0.8 μL 正向引物(10 μmon/L),0.8 μL 反向引物(10 μmol/L),0.4 μL ROX Reference Dye II,2 μL cDNA,6 μL ddH2O。PCR 反应程序为 :95℃预变性 30 s,95℃变性5 s,60℃ 34 s,进行40个循环。通过相对定量方法2-ΔΔCt[20]比较目的基因的表达量差异。每个样品均设置3个重复。
1.2.9 孢子悬浮液的配制 参照Tahir等[21]的方法并做修改。P. expansum在PDA培养基上25℃恒温培养5 d后,用无菌水收集孢子,经4层纱布过滤后得到孢子悬浮液,通过血球计数板计数后用无菌水调整浓度为1×106spores/mL备用。
1.2.10 不同发育阶段erg4A、erg4B和erg4C的表达 取500 μL P. expansum孢子悬浮液接种到100 mL CY(3 g/L NaNO3,1g/L K2HPO4·3H2O,0.5 g/L KCl,0.5 g/L MgSO4·7H2O,0.01 g/L FeSO4·7H2O,30 g/L Surose,5 g/L Yeast Extract,pH 5.2)液体培养基中,于25℃,200 r/min条件下培养。分别于0 h、14 h和3 d后收集孢子、萌发的孢子和菌丝,于液氮中速冻,保存至-80℃超低温冰箱待用。
1.2.11 固体和液体培养下erg4A、erg4B和erg4C的表达 将玻璃纸高温灭菌后铺在CYA固体平板上,接种2.5 μL的P. expansum孢子悬浮液于平板中央,25℃培养3 d后收集菌丝保存至-80℃超低温冰箱待用。接种500 μL的P. expansum孢子悬浮液于100 mL CY液体培养基中,于25℃,200 r/min振荡培养3 d,用4层灭菌纱布过滤收集菌丝,再用无菌水冲洗3遍,将菌丝于液氮中速冻,保存至-80℃超低温冰箱待用。
1.2.12 黑暗和蓝光培养条件下erg4A、erg4B和erg4C的表达 将玻璃纸高温灭菌后铺在PDA平板上,接种2.5 μL的P. expansum孢子悬浮液,于25℃培养24 h,然后将玻璃纸连同菌丝一起转移至装有CY液体培养基的24孔板中,分别置于黑暗和蓝光(光照强度在1 600 Lux)条件下,于25℃恒温培养箱中培养2 d,收集菌丝,液氮速冻后保存至-80℃超低温冰箱待用。
1.2.13 erg4A、erg4B和erg4C基因的RT-qPCR引物设计 根据克隆得到的erg4A、erg4B和erg4C 的cDNA序列,使用Primer Express software 3.0(Applied Biosystems)软件设计RT-q PCR引物,引物序列见表4。

表4 实时荧光定量PCR引物序列Table 4 Primers for real-time fluorescent quantitative PCR
1.2.14 数据处理 试验数据以平均值±标准差(mean±SD)表示,采用软件Origin 8.0(Northampton,MA,USA)作图;SPSS20.0统计分析软件进行数据处理,应用单因素方差分析(One-way ANOVA)和邓肯氏(Duncan’s)差异分析,在P=0.05水平上检测差异显著性。
2 结果
2.1 erg4A、erg4B和erg4C的CDS区克隆
以P. expansum cDNA为模板,利用CDS克隆引物erg4A-F/R、erg4B-F/R及erg4C-F/R对其进行PCR扩增,扩增产物通过1%琼脂糖凝胶电泳检测,获得清晰明亮的单条带(图1)。

图1 erg4A、erg4B和erg4C的全长CDS克隆Fig. 1 Full-length CDS cloning of erg4A,erg4B and erg4C
2.2 erg4A、erg4B和erg4C的基因结构及序列信息
基因结构分析结果表明,erg4A基因全长为1 476 bp,编码491个氨基酸,不含内含子;erg4B基因全长为1 619 bp,编码496个氨基酸,含有大小分别为51 bp和77 bp的2个内含子;erg4C基因全长为1 762 bp,编码531个氨基酸,含有大小分别为51 bp、56 bp和59 bp 的3个内含子(图2)。开放阅读框的预测结果表明,erg4A一共有14个ORF,最长的ORF长度是1 476 nt,位于正义链,读码框为1;erg4B一共有12个ORF,最长的ORF长度是873 nt,位于正义链,读码框为3;erg4C一共有13个ORF,最长的ORF长度是1 473 nt,位于正义链,读码框为1。

图2 erg4A、erg4B及erg4C的基因结构示意图Fig. 2 Genetic structures of erg4A,erg4B and erg4C
2.3 Erg4A、Erg4B和Erg4C蛋白跨膜螺旋
跨膜螺旋预测结果显示,Erg4A和Erg4C蛋白均包含9个跨膜螺旋、5个膜内结构和5个膜外结构;Erg4B仅有7个跨膜螺旋、4个膜内结构和4个膜外结构(图3)。这些跨膜螺旋表明Erg4A、Erg4B和Erg4C是典型的膜蛋白,对转运功能的发挥具有重要作用,3个蛋白均定位于细胞质膜或具有膜结构的细胞器上。

图3 Erg4A、Erg4B和Erg4C的跨膜螺旋Fig. 3 Transmembrane helices of Erg4A,Erg4B and Erg4C
2.4 Erg4A、Erg4B和Erg4C蛋白的亲疏水性
蛋白的亲疏水性分析结果表明,Erg4A、Erg4B及Erg4C 3条蛋白序列均存在明确的亲疏水区域,且疏水区域明显多于亲水区域。整条肽链表现出疏水性,通常蛋白质的跨膜区为疏水区,这与利用TMHMM对蛋白跨膜螺旋结构的预测结果相吻合。其中,erg4A基因编码蛋白氨基酸序列第165位氨基酸疏水性最强(3.078),第11位氨基酸为亲水性最强(-2.978)。erg4B基因编码蛋白氨基酸序列第36位氨基酸疏水性最强(2.933),第467位氨基酸为亲水性最强(-3.133),erg4C基因编码蛋白氨基酸序列第396位氨基酸疏水性最强(2.767),第415位氨基酸为亲水性最强(-3.178)。同时,Erg4A、Erg4B和Erg4C蛋白分别有9个、7个和9个疏水区(图 4)。

图4 Erg4A、Erg4B和Erg4C蛋白亲疏水性Fig. 4 Hydrophilicity/Hydrophobicity of Erg4A,Erg4B and Erg4C protein
2.5 GFP融合表达菌株的PCR鉴定及形态观察
将传代培养的阳性转化子提取基因组DNA,以此为模板进行PCR验证,用特异性引物Erg4-GFPF/R和载体引物CX1/CX2对Erg4A-GFP转化子进行验证,发现其条带大小为1 500 bp左右,且野生型无条带出现,与预期结果相同(条带预期大小1 476 bp);用特异性引物Erg4B-GFP-F/R和载体引物CX1/CX2对Erg4B-GFP转化子进行验证,发现其条带大小为1 700 bp左右,且野生型无条带出现,与预期结果相同(条带预期大小1 619 bp);用特异性引物Erg4C-GFP-F/R和载体引物CX1/CX2对Erg4CGFP转化子进行验证,发现其条带大小为1 800 bp左右,且野生型无条带出现,与预期结果相同(条带预期大小1 762 bp)(图5)。以上结果表明GFP融合表达菌株构建成功。对GFP融合表达菌株的生长状况进行观察后发现,荧光标记菌株菌落表型与野生型基本一致(图6)。
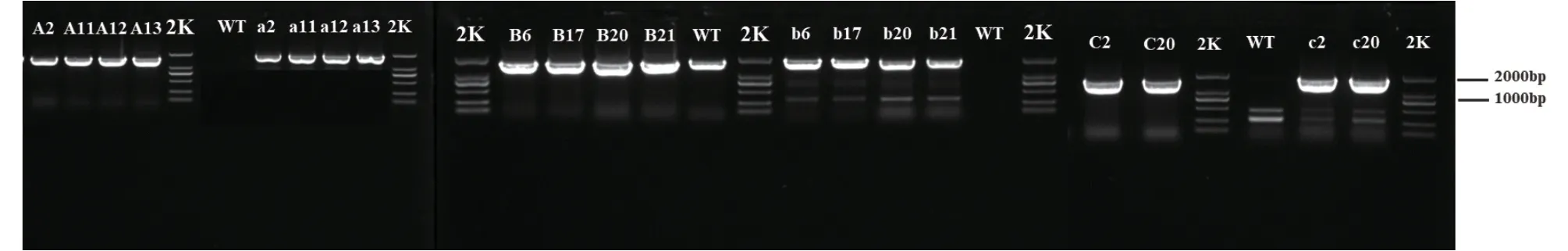
图5 GFP融合表达菌株PCR验证Fig. 5 GFP fusion expression strain verified by PCR

图6 野生型与荧光标记菌株erg4A-GFP、erg4B-GFP及erg4C-GFP的菌落形态比较Fig. 6 Comparison of colony morphologies of erg4A-GFP,erg4B-GFP and erg4C-GFP in wild type and fluoresence labelled strains
2.6 Erg4A、Erg4B和Erg4C的亚细胞定位观察
在激光共聚焦显微镜下观察各蛋白的绿色荧光蛋白信号结果(图7)显示,3个融合表达蛋白GFP的绿色荧光均在细胞内呈现圆圈形的分布。

图7 融合表达蛋白GFP的绿色荧光分布Fig. 7 Distribution of green fluorescence of GFP fusion expression protein
为进一步明确这3个蛋白的亚细胞定位,通过DAPI和ER-Tracker Red染色,进行细胞核与内质网和GFP融合蛋白的共定位结果显示,Erg4A、Erg4B和Erg4C均定位在菌丝细胞内许多圆圈结构上,且DAPI核染色显示这些圆圈结构均存在于细胞核的外围,因此初步判定该结构为内质网。为进一步确认定位结果,我们用内质网红色荧光探针染色,共定位结果显示融合蛋白与内质网的分布一致。因此,我们认为ErgA、ErgB及ErgC蛋白具有相同的亚细胞定位,均定位于内质网膜上(图8-9)。

图8 DAPI核染色与GFP的共定位Fig. 8 Colocalization of DAPI nuclear staining with GFP
2.7 不同发育阶段erg4A、erg4B和erg4C的表达差异
如图10所示,在孢子阶段、孢子萌发阶段以及成熟菌丝阶段,erg4A的表达量无明显变化,但erg4B和erg4C的表达量均显著上升,其中erg4B的上升幅度更为明显。erg4B在孢子阶段的表达量分别高出 erg4A和erg4C 29.5倍和3.1倍(P<0.05),在孢子萌发阶段的表达量分别高出erg4A和erg4C 72.9倍和1.4倍(P<0.05),在成熟菌丝阶段的表达量分别高出erg4A和erg4C 83.6倍和1.72倍(P<0.05)。

图10 扩展青霉中erg4A、erg4B和erg4C在孢子阶段、孢子萌发阶段以及成熟菌丝阶段的相对表达量Fig. 10 Relative expressions of erg4A,erg4B and erg4C in the spore,germinated spore and mycelium stages in P. expansum

图9 内质网红色荧光探针染色与GFP的共定位Fig. 9 Colocalization of endoplasmic reticulum red fluorescent probe with GFP
2.8 CY固体和液体培养条件下erg4A、erg4B和erg4C的表达差异
如图11所示,erg4A在CY固体和液体培养条件下的相对表达量未见显著差异,但erg4B和erg4C在液体培养条件下的相对表达量均显著高于固体培养,分别高出 4.96倍和 1.6倍(P<0.05)(图 11)。由此表明,erg4B和erg4C的表达均受培养基状态的影响。

图11 扩展青霉中erg4A、erg4B和erg4C在CY固体和液体培养条件下的相对表达量Fig. 11 Relative expressions of erg4A,erg4B and erg4C in P. expansum in Czapek yeast extract solid medium and Czapek yeast extract liquid medium
2.9 黑暗和蓝光条件下erg4A、erg4B和erg4C的表达差异
erg4A和erg4B在蓝光条件下的表达量均显著高于黑暗条件,分别高出5.6倍和6.4倍(P<0.05),虽然erg4C在在蓝光条件下的表达量略高于黑暗,但无显著差异(图12)。由此表明,erg4A和erg4B对蓝光信号响应积极,erg4C对蓝光信号不敏感。

图12 扩展青霉中erg4A、erg4B和erg4C在黑暗和蓝光条件下的相对表达量Fig. 12 Relative expressions of erg4A,erg4B and erg4C in P. expansum in dark and blue condition
3 讨论
本研究发现,P. expansum中同时存在3个erg4基因,分别命名为erg4A、erg4B和erg4C。其中,erg4A基因编码491个氨基酸;erg4B基因编码496个氨基酸;erg4C基因编码531个氨基酸,这些同源基因的存在可能是丝状真菌在长期的进化过程中为了适应环境变化、调节细胞膜流动性和通透性等功能表现出的一种适应机制[22]。Erg4A与Erg4B的氨基酸序列相似性为21.86%,Erg4B与Erg4C的氨基酸序列相似性为19.93%,而Erg4A与Erg4C的氨基酸序列相似性高达55.87%。由此可知,Erg4A与Erg4C的同源性较高,预示Erg4A与Erg4C可能存在功能互补现象。Erg4A和Erg4C各含有9个跨膜螺旋,而Erg4B仅含有7个跨膜螺旋,推测这3种蛋白可能定位于质膜。先前的研究表明,S. cerevisiae的 Erg4仅含有7个跨膜螺旋[23],A. fumigatus的Erg4A和Erg4B均含有9个跨膜螺旋[18],推测P.expansum的Erg4B与S. cerevisiae的Erg4可能具有类似的功能,而Erg4A和Erg4C与A. fumigatus的Erg4A和Erg4B功能类似。由于P. expansum的Erg4A、Erg4B和Erg4C均表现出疏水性,可能定位于质膜的磷脂双分子层。
本研究观察到,融合表达蛋白Erg4A-GFP、Erg4B-GFP和Erg4C-GFP的绿色荧光均在细胞内呈现圆形结构,DAPI核染色显示这些环状结构均位于细胞核的外围,且与内质网红色荧光探针染色的荧光信号完全吻合,该结果与Erg4A、Erg4B及Erg4C具有多个跨膜结构域以及表现出疏水性的结果基本一致。对S. cerevisiae的研究发现,麦角甾醇在内质网上经一系列酶促反应合成后转运至质膜等部位发挥其功能[23]。S. cerevisiae中许多与麦角甾醇合成相关的蛋白主要定位于内质网,如Erg1和Erg4[11,23]。此外,通过内质网标记蛋白Erg11对A. fumigatus中Erg4A和Erg4B进行了红色荧光标记发现,这两种蛋白也定位于内质网上[18]。因此,我们观察到的P.expansum的Erg4A、Erg4B和Erg4C蛋白均定位于内质网的结果与前人在S. cerevisiae和A. fumigatus上报道的结果基本一致。
本研究中发现,在孢子阶段、孢子萌发阶段以及成熟菌丝阶段,erg4A的表达量无明显变化,但erg4B和erg4C的表达量均显著上升,其中erg4B的上升幅度更为明显。这可能是由于Erg4A与Erg4C具有相似的蛋白结构和相同的内质网定位,在催化麦角甾醇合成方面存在功能互补。即当某个erg4基因低表达时,另一同源基因的表达量就会相应升高以弥补对菌株造成的损害[24]。推测在正常情况下,这3个基因同时发挥作用,erg4B功能更为明显。这种互补机制可使菌株免受因一种基因突变给菌株带来的伤害,在提高P. expansum的生存适应性方面具有积极的功能。还有研究发现桑黄菌(Phellinus linteus)中erg24的表达在培养前期和中期无显著变化,但在培养后期迅速升高,其原因是培养后期培养基中的营养成分减少,有害次生代谢产物大量累积,导致细胞受到损害,而麦角甾醇能够维持细胞形态和膜结构的完整性[25]。因此,推测P. expansum中erg4B和erg4C在3个阶段的高表达可能与适应培养基中有害次生代谢产物的自我保护有关。至于是否存在该机制尚有待进一步研究。
麦角甾醇生物合成是一个需氧的过程,每合成一分子的麦角甾醇需要消耗12分子的O2[26]。本研究发现,erg4A在CY固体和液体培养条件下的表达量没有显著差异,而erg4B和erg4C在CY液体培养条件下的相对表达量均显著高于固体培养条件。该结果与前人发现的麦角甾醇生物合成需氧的结果类似。由于液体培养条件下O2供应充足,可保证麦角甾醇顺利合成[22]。至于O2条件如何调控P.expansum中erg4A、erg4B和erg4C的表达差异尚有待进一步揭示。蓝光是调控丝状真菌生长发育最重要的环境信号分子[27]。已有研究发现,蓝光可以调控深绿木霉(Trichoderma atroviride)细胞膜的瞬时电位和ATP在细胞内的积累[28],表明蓝光参与深绿木霉的能量供应。本研究观察到,erg4A和erg4B在蓝光条件下的相对表达量均显著高于黑暗条件,而erg4C在蓝光和黑暗条件下没有显著差异,表明erg4A和erg4B的表达受到蓝光信号的调控,推测Erg4A和Erg4B存在可响应蓝光信号的感光结构域,至于Erg4A和Erg4B是否存在响应蓝光信号的感光结构域尚有待进一步验证。
4 结论
本研究通过生物信息学手段分析了扩展青霉中erg4A、erg4B和erg4C的基因结构、跨膜螺旋结构域和亲疏水性。Erg4A、Erg4B及Erg4C三个蛋白均定位于内质网。erg4B和erg4C的表达受扩展青霉不同生长发育阶段以及培养基状态的影响,erg4A和erg4B的表达受蓝光信号的调控。
