基于EfficientNet模型的多特征融合肺癌病理图像分型
2021-02-07叶紫璇肖满生
叶紫璇,肖满生,肖 哲
(湖南工业大学 计算机学院,湖南 株洲 412007)
0 引言
肺癌是一类高发病率的恶性肿瘤,其中的肺腺癌(lung adenocarcinoma,LUAD)和鳞状细胞肺癌(squamous cell carcinoma of the lung,LUSC)是 最常见的亚型[1]。目前,胸部影像学检查如电脑断层(computed tomography,CT)等只能提供初步的检查和评估,临床上的肺癌确诊方式主要是依赖于病理学家在显微镜下观察细胞及细胞间的形态,然而这一过程基于病理学家的主观意见,不仅耗时,而且因医生的能力不同可能导致较大偏差[2]。如何高效且准确地确认肺癌类型是目前临床对病理学专家的迫切要求。随着人工智能的发展,目前该领域的研究方法可分为两类:一类方法是基于传统机器学习对肺癌病理图像进行研究,主要集中于从图像中提取人为设计的特征,如纹理、空间和核分布等相关特征,然后将提取的特征通过传统的机器学习算法进行预测;另一类方法则是基于深度学习的算法,卷积神经网络(convolutional neural network,CNN)模型可以自动获取肿瘤空间信息,并且对组织形态进行量化分析,为解决医学图像领域的困难提供了更多的可能性。
近年来,基于深度学习的肺癌病理图像分类已经有了广泛的研究,A.Teramoto 等[3]通过深度卷积神经网络设计了肺癌细胞病理自动分类模型,其由3 个卷积层、3 个池化层和2 个全连接层组成,该DCNN模型在腺癌、鳞癌和小细胞癌的分类诊断中的准确率分别为89.0%,60.0%,70.3%,总准确率为71.1%,结果与病理学家的诊断准确率相当。N.Coudray 等[4]使用Inception v3 模型,不仅在对肺组织及肺癌区分任务中获得了良好的效果,还根据图像的形态特征,预测了STK11、EGFR、FAT1、SETBP1、KRAS 和TP53 6个突变基因,AUC(area under curve)可达73.3%~85.6%。基于块的肺癌病理图像分类中,宁静艳等[5]采用卷积神经网络,分别在随机取块和滑动取块中取得了86%和81%的总分类精度。以上研究均证明了利用卷积神经网络在肺癌病理图像分类中已取得了一些成果。然而仍存在如下问题:1)卷积神经网络中含有大量的参数,在样本量较少的情况下容易造成过拟合;2)卷积神经网络往往关注最后一层特征而忽略了其它层的特征,准确率有待提高。
针对上述问题,借鉴深度卷积网络EfficientNets(high-efficiency network)的网络结构,课题组提出一种自动对非小细胞肺癌全扫描组织病理图像分型的方法,将网络中低维特征与包含丰富抽象语义信息的高维特征相结合,从而丰富特征表示,解决病理图像区分度较小的问题。另外,Haralick 纹理特征是借助灰度共生矩阵(gray-level co-occurrence matrix,GLCM)得到的,而灰度共生矩阵是一种通过研究图像灰度的空间相关特性来描述纹理的图像处理方法,其对纹理特征的描述能力较强,在病理图像任务中取得了较好的效果[6-7]。为减轻网络模型对训练样本的依赖,本研究还融入了纹理特征,建立适用于病理图像的神经网络模型来实现全尺寸肺癌组织切片图像分型的目的。
1 基于特征融合的EfficientNet 模型
1.1 纹理特征
纹理特征是一种全局特征,反映了图像中同质现象的视觉特征,包含了物体表面具有缓慢变换或周期性变化的组织结构的重要信息,具有旋转不变性。常用的纹理特征包括Haralick 纹理特征、Gabor 特征和局部二值模式(local binary patterns,LBP)特征等。课题组提取了Haralick 纹理特征来描述肺癌病理图像的纹理属性。
灰度共生矩阵[8]作为纹理特征提取的经典算法,其描述的实质是统计一对灰度在某种位置下出现的频率。灰度共生矩阵定义图像I 中,统计灰度值为i的像数点(k,l)与距离为d=(m-k,n-l)、灰度值为j的像素点(m,n)同时出现的概率P(i,j,d,θ):
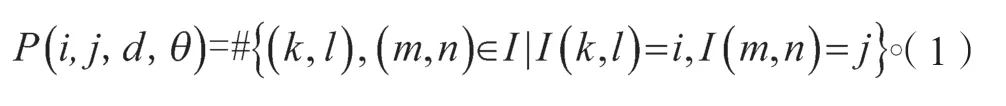
式中:#为该集合中的元素个数;I(k,l)为图像I中坐标为(k,l)的像数灰度值;d表示两像素点的欧氏距离;θ通常考虑水平、对角线、垂直及反对角线(0,45,90,135°)4 个不同方向。
R.M.Haralick[9]基于GLCM 提出了14 种统计特征,即角二阶矩、熵、对比度、逆差矩、相关性、方差、和均值、和方差、和熵、差方差、差均值、差熵、相关信息测度以及最大相关系数。能量、熵、对比度、相关性4 种特征既便于计算又有较好的效果,故本文选取这4个特征代表纹理特征。为使得到的特征向量与方向无关,需在4 个方向上对特征向量取值,并且由于不同的特征的重要程度不同,本文采用加权的方法将上述特征fn合成为16 维纹理特征向量ft:
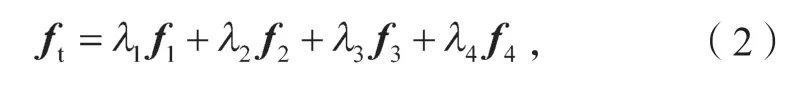
式中λ为特征权值。
1.2 基于EfficientNet 模型的网络特征
在深度学习网络中,各层特征图侧重于不同类型的信息,低层次的特征语义信息较少但具有高分辨率信息,高层次的特征分辨率较低,但具有高语义信息,然而网络通常以最后输出的高层次特征图作为依据进行分类预测,网络低层次的特征信息未得到利用。在计算机视觉任务中需要丰富的特征信息,尤其是在医学图像任务中,图像较为复杂,低层次的特征信息不可忽视,并且有研究表明,结合浅层特征在一定程度上可以提升模型精度[10]。本研究选取近年来表现优异的EfficientNets 网络提取图像多维度的特征。
EfficientNets 是由google 于2019 年使用神经网络架构搜索(neural architecture search)设计的一系列网络模型[11]。在限制目标和存储空间的情况下,通过神经网络架构搜索方式设计一个主干网络,并且将模型进行缩放获取EfficientNet B0~B7 模型。本文以EfficientNet B0 网络为主网络模型,网络参数如表1 所示,其中,size为卷积核或池化核的大小和数目,stride表示步长,MBConv(mobile inverted Bottlenneck conv)表示移动倒置瓶颈卷积模块,k3×3/5×5 分别代表卷积核大小,class 代表类别个数。
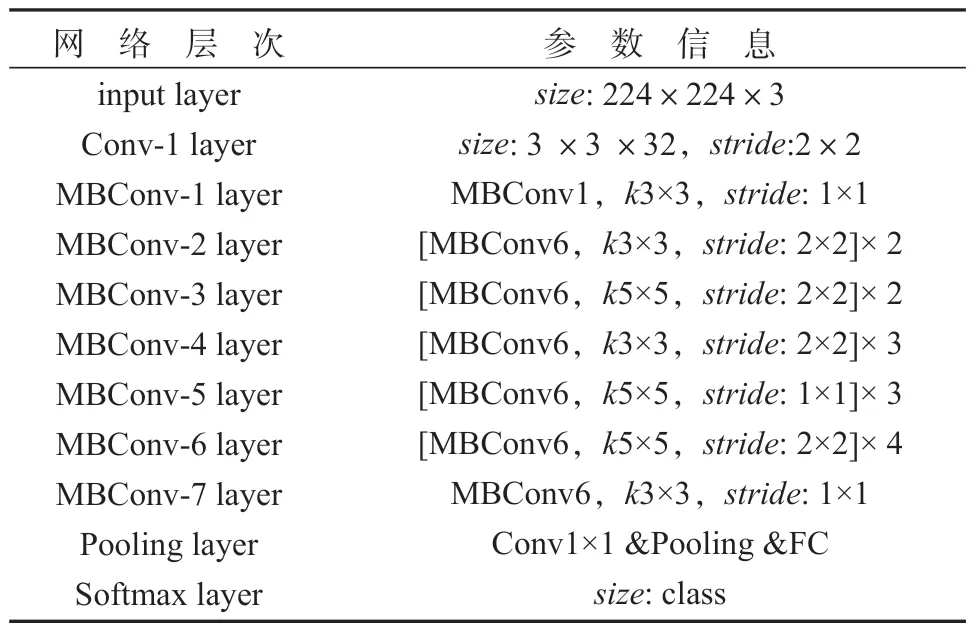
表1 EfficientNet-B0 模型Table 1 EfficientNet-B0 model
由表1 可知,EfficientNet-B0网络由16 个移动倒置瓶颈卷积模块,2 个卷积层,1 个全局平均池化层和1 个分类层构成,其中核心结构为MBConv 模块。该模块结构类似于MobileNetV2[12]的Inverted residual block 模块,其结构如图1 所示。
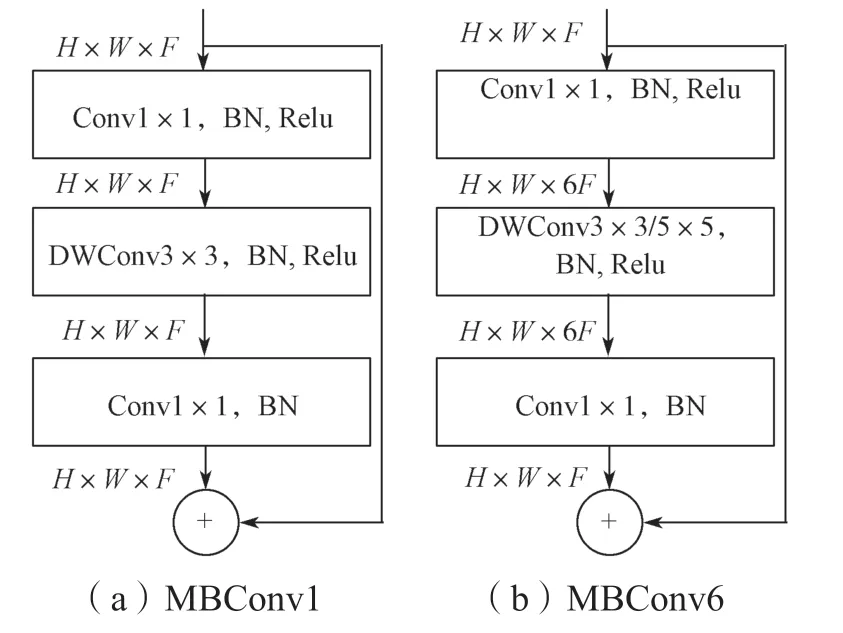
图1 MBConv 模块Fig.1 MBConv block
首先对输入进行1×1 的逐点卷积并根据扩展比例改变输出通道维度,接着进行k×k的深度卷积(depthwise convolution),最后再次使用1×1 的逐点卷积将其维度缩小。同时,该模块还引入了压缩与激发网络(squeeze-and-excitation network,SENet)[13]的注意力思想,更能关注信息量大的通道特征,而抑制不重要的通道特征。值得注意的是,MBConv 模块激活函数使用的是Swish 激活函数,表达式如式(3)所示:
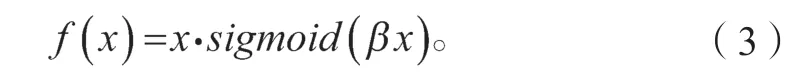
式中:sigmoid函数为;β为可调参数。
如图2 所示,以EfficientNet-B0 为主网络模型,其最后的卷积层输出1×1 的卷积结果,去掉其最后的全连接层与softmax 层,加入一个全连接层,FC1输出1 024 维特征向量作为模型的深层次特征。由于过于底层的特征图不具备较高的参考价值,在原始图像输入后提取不同MBConv 层的特征图fd2、fd3。为保留特征图中的关键信息,在特征跨层连接时仅使用一个全连接层连接,从而获取FC2、FC3 层特征向量,作为本文的深层网络特征。
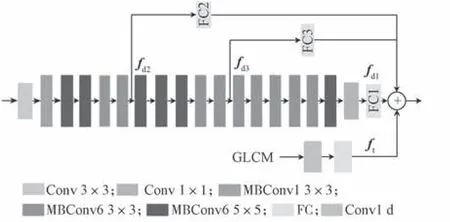
图2 EfficientNet-B0 网络的改进Fig.2 Improved version of EfficientNet-B0 network
1.3 基于EfficientNet 模型的多特征融合算法
特征融合方式主要分为前期融合与后期融合两种,其中前期融合指先融合多层的特征,然后在融合后的特征上训练预测器,后期融合则是通过结合不同层的检测结果改进检测性能。串联融合属于前期融合,简单易行且在特征量不多时效果较好,因此本文选择串联融合方式对上述特征进行融合。
如图2 所示,1×1×B的GLCM 特征向量输入至由卷积层和全连接层构成的纹理特征模型,并将纹理特征ft与EfficientNet B0 网络所提取的多维度特征fd1、fd2、…、fdn级联起来,得到深度数据融合特征f,公式如式(4)所示。
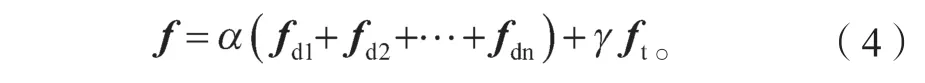
式中,α、γ为可调权重。
实验中,选用concatenate 函数来完成,能将单特征的全部信息保留并融合。接着多个特征融合后的特征向量被送入softmax 算法实现分类。为防止产生过拟合现象,本文采用Dropout 方法,在不同的训练阶段随机丢弃不同的神经元以减少神经元之间的依赖性。同时,用于特征融合的激活函数选择ReLU(rectified linear units)函数,其收敛速度远快于sigmoid 和tanh 函数,且不存在梯度消失问题。
2 基于多特征融合的肺癌病理图像分型实现过程
2.1 H&E 病理图像预处理
对于来自于不同实验室或不同批次的病理图像之间存在较大的差异,在图像进行训练前对其进行预处理使提取的特征更为鲁棒。对于H&E 病理图像预处理包括图像分块处理、颜色标准化图像和数据增强3 个步骤。
全尺寸组织病理图像的尺寸一般为亿兆像素级,难以直接进行分析,并且背景占WSI 的70%~80%。为解决这一问题,本文对全尺寸组织病理图像进行图像分块处理。对于已标记的全尺寸病理图像,为了使图像能在模型中训练,本文首先获取全切片病理图像中被标记的位置;再将全尺寸病理图像按照一定尺寸经过不重叠采样得到若干RGB 三通道的小尺寸病理图像。同时对RGB 空间的图像进行二值化处理,检索所有轮廓获取其面积,选取一阈值对低信息量的小尺寸图像过滤以降低噪音图像对模型性能的影响。
本文使用的肺癌病理图像均为苏木素-伊红(H&E)染色组织病理图像,但是来自不同的医疗中心,不同样本以及组织切片制备或图像采集过程中的不一致导致病理图像之间具有明显的差异[14]。采用适当的预处理可减少这一差异对后续实验的影响。因此,本文运用颜色归一化方法[15]对所有病理图像进行处理。首先,选取一例目标图像,经颜色标准化后的小尺寸图像与目标图像具有相同的高斯分布。本文由病理医师根据主观判断选择染色较好的切片作为目标图像。具体方法借助于HSD(hue-saturationdensity)色彩空间,利用高斯混合模型对数据集进行聚类,通过数据集图像与目标图像的高斯分布转换,训练出可实现无监督的颜色标准化模型。
除此之外,由于已有数据集的样本量有限,本文采用数据增强方法将小尺寸病理图像数据扩充。通过如翻转、仿射变换、对比度扰动、旋转等方法,在增强数据集的同时不改变图像的组织形态和纹理特征,以提高模型的泛化性能。
2.2 模型的迁移学习
对于传统的机器学习,将某问题中有用的信息应用到不同但相关的问题中需要复杂的数学推导。相比之下,深度学习中的迁移学习[16]可以容易地将某个问题所得到的深度学习模型应用到相关问题上,因此在缺乏训练数据的情况下,可以使用迁移学习来解决一些较为困难的预测问题。癌症基因组图谱(the cancer genome atlas,TCGA)作为最大的公开病理图像数据集之一,每个癌种只包含数百张病理图像[17],而ImageNet 数据集包含1 400 万标记图像用于图像识别任务,可见,目前仍然缺乏大型已标记的病理图像数据集。迁移学习可以使用预训练的网络模型和权重参数提取特征来进行病理图像分析,如使用ImageNet 数据集训练模型中的参数。研究表明,与从零开始训练相比,迁移学习收敛速度更快,准确率更高[18]。
本文经过预处理得到数量增大的训练图像样本后,采用Pytorch 深度学习框架对改进的EfficientNet-B0 模型进行参数微调:
1)输入训练图像为224×224×3 的肺癌H&E图像以及1×1×B的GLCM 特征向量,解冻网络层重新进行训练;
2)参数初始化,采用AdamW 优化器,将学习速率从0.01 降低到0.000 1,使得模型的权重不会下降过多过快;
3)开始训练,训练集经前向传播,反向传播,不断计算梯度和误差,并更新参数。
4)重复步骤3),直至满足停止条件。
得到训练好参数的2 分类网络模型,即肺鳞癌和肺腺癌。
图3 为肺癌病理图像分型流程。
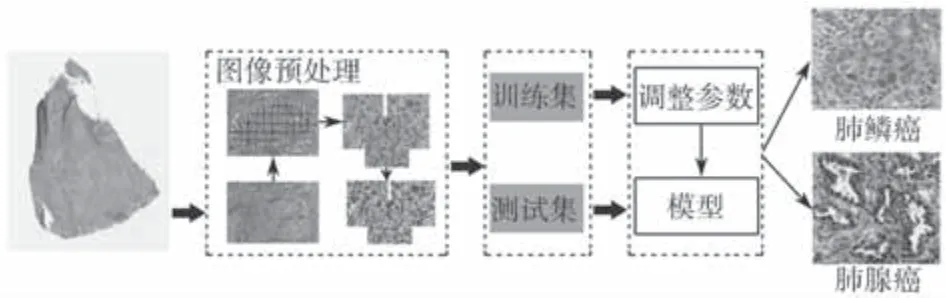
图3 肺癌病理图像分型流程图Fig.3 Pathological image classification process of lung cancer
2.3 肺癌病理图像分型实现
本文模型主要包含图像预处理、模型构建与训练以及模型测试3 个部分,流程图如图3 所示,具体实现步骤如下。
1)图像预处理
输入:数据集L。
输出:训练集T和测试集V。
利用随机函数对数据集进行随机抽取,80%划分为训练集,20%划分为测试集;
对数据集中的每一例全尺寸病理图像进行图像分块,得到224×224 的小尺寸病理图像数据集;
通过颜色归一化模型,将图像分块后的数据集图像进行颜色归一化操作;
随后对颜色归一化后的数据集进行翻转、仿射变换、对比度扰动、旋转等图像增强操作,得到扩展后的数据集。
2)模型构建与训练
输入:训练集T。
输出:训练好的网络模型。
将训练集中每一张小尺寸图像转换为灰度图像,采用2.1 节的方法计算灰度共生矩阵,提取纹理特征;
利用EfficientNet B0 模型对每一张小尺寸图像提取多维度特征;
连接所有特征的全连接层,实现特征融合;
将融合特征输入Softmax 层中,构建肺癌病理图像分型模型;
利用迁移学习策略训练模型,得到训练好的网络模型。
3)模型测试
输入:测试集V。
输出:准确率。
1)将测试集中的所有样本输入训练好的网络模型中进行预测;
2)统计预测结果,得到准确率。
3 实验结果与分析
3.1 实验数据
本文所用的数据集来源于癌症基因组图谱(网址:https://www.cancer.gov/about-nci/organiza-tion/ ccg/research/structural-genomics/tcga)公共数据库,共采用302 例非小细胞肺癌全扫描组织病理图像,其中包含169 例肺腺癌切片和133 例肺鳞癌切片,并且临床病理医生对全扫描组织病理图像进行癌症区域标记。本文按照8:2 的比例将数据集分为训练集和测试集。为了实验的真实性与统一性,随机选取数据,所有的图像都经过了相应的预处理。
3.2 实验环境
实验平台硬件配置:Intel(R)Xeon(R)E5-2640v4@2.40GHz 10 核CPU;64 GB DIMM 2 400 MHz 内存;Matrox G200eR2 独立显卡;64 位Linux 操作系统。软件方面:anaconda3 为开发平台,Facebook 人工智能研究院(Facebook AI research,FAIR)团队的深度学习开源框架Pytorch 为程序框架。
3.3 评价指标
选用准确率(accuracy,ACC),AUC 以及ROC(receiver operating characteristic)曲线对实验结果进行评价。ROC 曲线和AUC 通常用来评价二分类模型的性能,ROC 曲线以真阳性率(true positive rate,TPR)为纵坐标,假阳性率(false positive rate,FPR)为横坐标,曲线下与坐标轴围成的面积定义为AUC值,AUC值越大,模型的分类效果越好。各个指标的含义如式(5)~(8)所示:

式(5)~(8)中:真阳性(true positive,TP)表示样本为阳性且被预测为阳性的个数;假阳性(false positive,FP)表示样本为阴性而被预测为阳性的个数;真阴性(true negative,TN)表示样本为阴性且被预测归为阴性的个数;假阴性(false negative,FN)表示样本为阳性而被错误地预测为阴性的个数;i为预测为阳性概率的样本序号;N为阴性的样本数,M为阴性样本数。
3.4 结果与分析
本文将经预处理的小尺寸病理图像作为输入,送入上述网络架构中。全连接层将EfficientNet B0 提取的3 个特征图与纹理特征融合的特征构成一个特征向量作为softmax 的输入,最后实现非小细胞肺癌病理图像分型目的。网络模型中超参数的取值对模型性能有很大的影响。实验中,α设为1/3,λ为1,λi均为1/4,进行了100 次循环,学习率设置为1e-4,采用 AdamW 优化器。采用分批次训练的方式以减小计算量,每批训练128 张训练图像。肺癌组织切片图像准确率和和损失率变化曲线,如图4a、4b所示。
从图4a 中可以看出在前20 个epoch 模型准确率快速上升,效果较为明显;之后的训练过程中,准确率发生小幅度上升,迭代至30 个epoch 时开始收敛,最终稳定在99%左右。同样从图4b 中可以看出,损失值变化一直呈减小的趋势,震荡幅度在40个epoch 之后速度趋于平稳。相对于应用于同一数据集的其他深度学习神经网络,本文提出的方法具有更快的收敛速度。利用训练后的模型对测试集中小尺寸病理图像进行测试,ROC 曲线的AUC 值为86%,如图4c 所示。
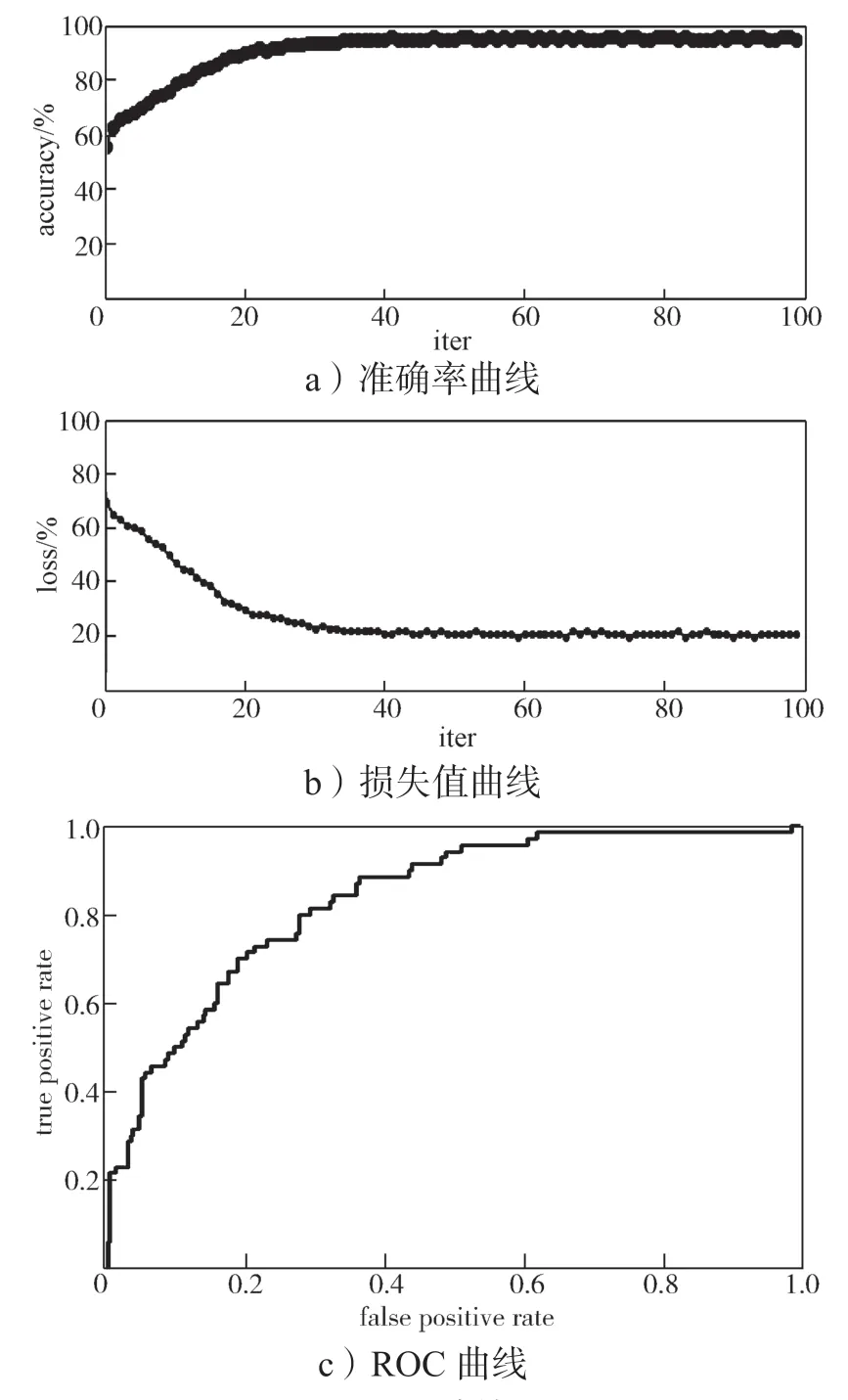
图4 实验结果Fig.4 Illustration of experimental results
在图像预处理过程中,颜色归一化可以降低H&E 染色图像的颜色差异,使模型学习时不受颜色差异的影响。以往研究表明,颜色归一化可以提高计算机视觉任务的性能。本文将其进行对比,结果表明,在肺癌病理图像分型任务中,颜色归一化可以使准确性提高4%~6%,如表2 所示。

表2 不同方法对肺癌数据集的准确率Table 2 Accuracy rate of different methods for lung cancer data set
目前,医学病理图像任务大多采用Resnet 网络进行数据训练[19-20]。为了能更好地评价本文模型的性能,本文选取多种模型及其它方法进行实验对比,即分别利用Resnet50和EfficientNet B0 网络及Resnet50-Fusion、EfficientNet B0+HOG、EfficientNet B0+LBP 模型在相同的数据集上训练网络,其结果如表2 所示。可以看出,在未加特征融合的情况下,相较于Resnet 50 网络模型,EfficientNet B0 模型的准确率更高,且具有更小的计算消耗和时间消耗,这与自然图像分类结果相符。然而,病理图像比自然图像更为复杂,其中包含大量特征信息。在分别加入LBP、GLCM、方向梯度直方图(histogram of oriented gradient,HOG)特征后,本文所建立的模型能够获得更好的预测效果,加入LBP 特征所达到的准确率与本文模型相差不大。与形状特征相比,纹理特征在肺癌病理图像上展示了更好的结果。此外,Resnet50-Fusion和本文所建立的模型的准确率分别达到81.84%和84.29%。因此利用EfficientNet B0 模型在不同层次提取特征与纹理特征相融合,提高了模型对肺癌病理特征的分型效果。
4 结语
本文针对肺癌组织病理图像提出了多特征融合的肺癌组织病理图像自动分型方法。对全尺寸病理图像进行图像分块,颜色归一化等预处理提高训练图像质量,利用预处理后的训练图像对已有的EfficientNet B0 网络进行迁移学习,将网络得到的多维度特征与图像的纹理特征融合并利用Softmax 方法进行分类。实验结果表明,本文方法能够利用肺癌病理图像中的各种特征信息,有效地提高病理图像分类效果。目前,通过深度学习实现癌症的病理分型大多还停留在实验室研究阶段,未能真正进入临床,下一步的研究不仅需要对网络模型进行改进优化,还需要扩大样本数据集,在更多样本以及其他癌种的病理切片上进行进一步的验证,以便于早日帮助医生提出更好的治疗方案,更好地满足临床应用。
