基于多相似度融合的药物重定位推荐算法
2021-01-19鲍天嘉智余肖生
陈 鹏,鲍天嘉智,余肖生
(三峡大学 计算机与信息学院,湖北 宜昌 443002)
0 引 言
药物重定位(drug repurposing),俗称“老药新用”,是指通过现有的技术手段将已经产生适应症的药物重新定位,寻找其新的适应症[1]。药物重定位这一概念自被提出以来,国内外学者对该方向算法的研究投入了巨大的精力[2-3]。张永祥等认为药物重定位是网络药理学的重要应用领域,介绍了基于小分子特征、基于蛋白靶点特征等方法[4]。谢达菲等提出了利用药物-靶标关系、药物-药物关系和药物-疾病关系分析等方法进行药物重定位的计算预测[5]。文献[6]将深度学习的方法应用于药物重定位。Luo等人提出了一种基于综合相似度和随机游走的药物重定位算法[7-8],通过将药物和疾病特征信息与已知的药物-疾病关系相结合,首次提出了药物与疾病相似性的综合相似度计算方法。文献[9-11]提出了基于多数据源融合的药物重定位算法。文献[12-15]提出了一系列基于协同过滤的药物重定位算法研究,从多源数据的角度出发,通过协同过滤来计算药物-疾病对应关系预测值。
由于数据稀疏性对协同过滤的影响较大,所以现阶段基于协同过滤的药物重定位算法大多通过融合多种数据源进行计算的方法来减小影响,但在计算过程中仅使用了药物相似度,忽略了疾病相似度的作用。
综上所述,传统的基于协同过滤的药物重定位算法虽然有一定的效果,但仍有很大的进步空间。该文提出了一种基于多相似度融合的药物重定位推荐算法(MSF),首先由药物-疾病数据源计算出疾病相似度,其他三种数据源计算出的三种相似度融合为药物相似度,再分别利用基于项目和基于用户的协同过滤进行药物与疾病对应关系的预测值计算,并通过融合方法将两种预测值融合为最终的药物-疾病关系预测值。MSF算法在充分利用多源数据的前提下,将计算出的相似度和预测值融合,降低了数据稀疏性对协同过滤的影响。
1 相关理论
1.1 药物重定位
药物重定位已经成为医学领域研究的一大热门。现阶段,由于药物和疾病数据的大量增长,从不同角度进行药物重定位研究的案例也越来越多。为证明本文对药物重定位的研究角度是切实可行的,从药物与疾病的关系、药物化学结构、药物靶蛋白和药物副作用四个方面进行讨论。
Chiang等人[16]提出了一种从疾病的角度看待药物重定位的观点,当两种疾病可以被多种相同的药物治疗时,认为两种疾病是相似的。如果存在一种药物只对其中一种疾病有治疗效果,则认为该药对另一种疾病也存在潜在的治疗关系,可以作为治疗该疾病的候选药物。药物的化学结构被认为可以用来度量药物间相似性,Dudley等人[17]提出药物的化学性质与其治疗效果有密切的关系,药物的化学结构和生物活性之间存在定量关系,所以药物的化学结构可以作为药物重定位的研究方向。药物靶蛋白是药物治疗疾病的关键因素,含有相似靶蛋白的药物也可能会有类似的作用效果[18],因此靶蛋白可以作为药物重定位中度量药物相似性的一个研究角度。同样,与药物对疾病的治疗效果类似,药物产生的副作用提供了人类的表型特征,因此从药物的副作用角度进行药物重定位的研究也是可行的[19]。
通过上述讨论证明从药物与疾病的关系、药物化学结构、药物靶蛋白和药物副作用这四个角度进行药物重定位研究是可行的。但是上述文献提供的方法都是从多个角度中的一类或两类进行药物重定位研究,可能会导致预测值的偏差,因此,该文从四个角度综合考虑并进行研究,减小了预测结果的有偏性,更具有实际应用价值。
1.2 协同过滤
协同过滤算法在近几年被广泛应用于各大电商行业以及影视平台的应用推荐中[20-21]。传统基于邻域的协同过滤算法分为两种,即基于用户的和基于项目的,两种算法的本质都是通过选取k个最相似的邻居进行评分预测。
协同过滤的基本流程为:先通过余弦相似度、皮尔逊相关系数、谷本系数等相似度度量方法计算用户或项目的相似度。再利用计算出的相似度找到相似度最高的k个邻居,由对应的两种公式求出预测分数:
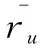
(1)
(2)定义rui为用户u对项目i的评分,D为项目i的邻居集合,simij表示项目i和j的相似度,ruj为用户u对项目j的评分,基于项目的预测值计算如式(2):
(2)
2 基于多相似度融合的药物重定位推荐算法
传统的基于协同过滤的药物重定位算法仅使用药物相似度进行预测值计算,往往忽略了疾病相似度在药物重定位中的作用且研究角度比较单一,而由于单个数据源的稀疏问题,导致计算出的有效药物、疾病相似度较少,许多项目无法找到合适的邻居,因而计算出的预测值偏差较大[22]。该文提出了MSF算法,即:首先由药物-疾病数据源计算出疾病相似度,再通过药物-化学结构、药物-靶蛋白以及药物-副作用数据源计算出三种相似度融合为药物相似度,使用疾病相似度和药物相似度计算两种预测值并融合为最终的预测值,整体流程如图1所示。
2.1 相似度计算
2.1.1 疾病相似度
疾病相似度通过药物-疾病数据进行计算。药物-疾病治疗关系数据从UMLS[23]中的NDF-RT(national drug file-reference terminology)进行采样,其中药物与疾病有作用关系则值为1,若无关系则为0。这种药物-疾病数据集在文献[11]中被认为是药物重定位的“金”标准数据集。基于文献[16]的思想,该文对于疾病相似度的计算只考虑药物与疾病是否有对应的治疗关系,而不考虑治疗效果的优劣,所以疾病相似度通过谷本系数进行,与使用余弦相似度等方法相比,在不影响相似度准确性的情况下,简化了计算的复杂程度。疾病相似度的计算公式如式(3):
(3)
其中,sim(ia,ib)表示疾病a和疾病b的相似度,Ia表示可以治疗疾病a的药物数量,Ib表示可以治疗疾病b的药物数量,|Iab|表示可以同时治疗疾病a和疾病b的药物数量。sim(ia,ib)的值应该在区间[0,1]之间。

图1 算法流程
2.1.2 药物相似度
在文献[17-19]的基础上,该文认为通过药物-化学结构、药物-靶蛋白和药物-副作用三种数据源计算出的药物相似度可以通过一定的权重融合为最终的药物相似度,其中药物-化学结构数据从PubChem[24]进行采样,药物-靶蛋白数据从UniPort Knowledgebase[25]中采样,药物-副作用数据从SIDER[26]数据库中采样。融合相似度的方法有效缓解了单个数据源因为数据稀疏而导致计算出的有效相似度较少的问题,并且通过融合减少了计算最终预测值所需要的步骤。由于采用相似度融合的方法,所以三种数据源应该选用同一种相似度计算方法。与计算疾病相似度类似,用于药物相似度计算的三种数据源中只考虑药物与三种属性的对应关系,所以同样采用谷本系数进行相似度的计算,如式(4):
(4)
其中,sim(da,db)表示药物a和药物b的相似度,(a)在使用药物-化学结构数据源的计算过程中,Da表示药物a包含的化学结构数量,Db表示药物b包含的化学结构数量,|Dab|表示药物a和药物b包含的相同化学结构数量;(b)在使用药物-靶蛋白数据源的计算过程中,Da表示药物a对应的靶蛋白数,Db表示药物b对应的靶蛋白数,|Dab|表示药物a和药物b对应的相同靶蛋白数;(c)在使用药物-副作用数据源的计算过程中,Da表示药物a会产生的副作用数,Db表示药物b会产生的副作用数,|Dab|表示药物a和药物b产生的相同副作用数。同样,sim(da,db)的值也应该在区间[0,1]之间。
simd表示由三种相似度融合得到的最终药物相似度,sims表示由药物-化学结构算出的相似度,simp表示由药物-靶蛋白算出的相似度,simf表示由药物-副作用算出的相似度。使用式(5)进行计算:
simd=αsims+βsimp+γsimf
(5)
其中,α+β+γ=1。
相似度融合的权值计算采用试探法,以0.1为步长进行试探,通过多次实验确定一组能使效果最优的权值。试探法的优点是使用简单且较为准确,缺点是运行效率较低,但在设置合适的步长时可以在一定的时间范围内得到预期的结果。通过试探法得出当α=0.2,β=0.4,γ=0.4时,效果达到最优。
2.2 预测值计算
通过两种相似度计算方法得到疾病相似度和药物相似度后,为了融合两类相似度计算出的预测结果,提出预测值融合的方法,如式(6):
(6)



(7)
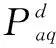
(8)
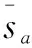
由于两种预测值也要进行融合,为了保持数据的一致性,两类算法中选取的邻居数应该为相同值。根据多次实验结果,设置ω1=0.6,ω2=0.4。
2.3 算法流程
Step1:将四种数据源转换成对应的矩阵,药物-疾病矩阵通过式(3)计算疾病相似度,其他三种数据源得出的矩阵通过式(4)计算出相似度。
Step2:由Step1算出四种相似度,将通过式(4)算出的相似度用式(5)融合成药物相似度,通过式(3)得出的相似度作为疾病相似度。
Step3:得出的疾病相似度通过式(7)计算出药物与疾病对应关系的预测值,药物相似度则通过式(8)计算出预测值。
Step4:用式(6)将两类预测值融合得到最终的药物与疾病对应关系预测值。
3 实验结果及分析
3.1 数据集
为了验证算法的有效性,该文采用的数据集是文献[9]中的数据,该数据集是通过UMLS、PubChem、UniPort Knowledgebase和SIDER中的数据进行人工处理和清洗获得的,其中包括药物-疾病数据、药物-化学结构数据、药物-靶蛋白数据和药物-副作用数据。四种数据均采用二进制表示对应的关系:0表示无相关对应关系,1表示有对应关系。数据集包括536种药物,以及对应的578种疾病、881种药物化学结构、1 385种副作用和775种对应靶蛋白。其对应的数据稀疏程度分别为:0.992 0,0.859 2,0.945 5,0.995 4。稀疏程度主要用于观察数据集中无作用的数据占总数据的比例,从得出的数据可以看出四种数据源的稀疏程度都比较高,无作用数据占总数据的比例较大。
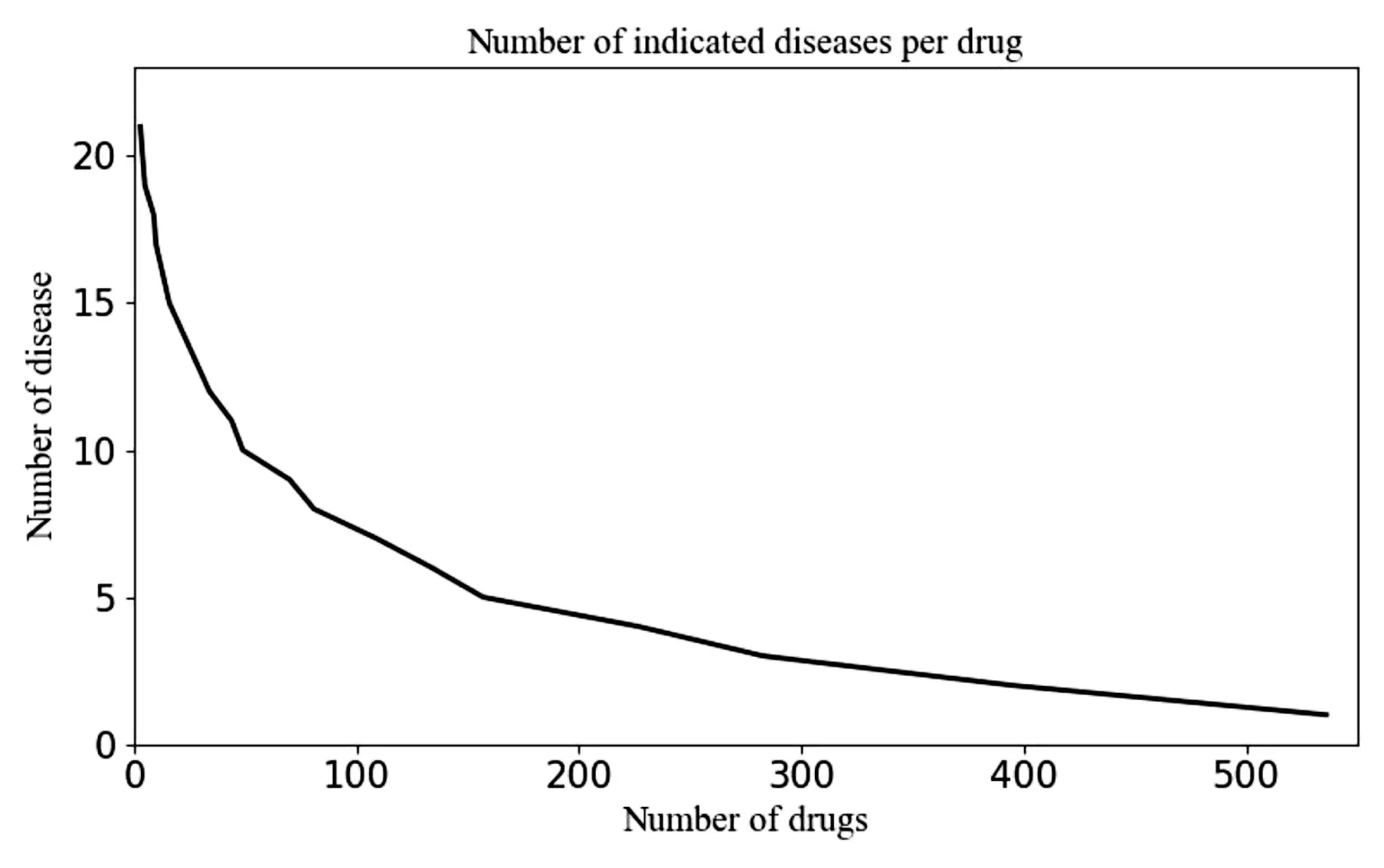
图2 药物-疾病对应数量关系
从药物-疾病数据源中可以算出每一种药物对应可以治疗的疾病数,如图2所示,可以看出大约只有50种(10%)药物可以治疗10种以上疾病,大部分药物(75%)只能治疗5种以下的疾病。
3.2 实验结果
文中将药物重定位算法当作一个二分类问题来看待,对于每种药物,如果疾病可以治疗则为1,反之则为0,为了保证实验的准确性和健壮性,使用十折交叉验证法进行实验。该文采用准确率、召回率、F-score和ROC曲线来进行算法效果好坏的判断。为了给出这四种分类指标的准确定义,首先定义一个二分类问题的混淆矩阵,如表1所示。

表1 混淆矩阵
通过混淆矩阵可以对精确率、召回率、F-score进行定义,如下:
(9)
(10)
(11)
为了确定计算中最相似的邻居数k使预测值最精确,提供了每种数据源在不同邻居数情况下的精确率表现,如图3所示。通过观察每种数据源各自的精确率变化,确定一个使算法效果最好的k值。通过观察可以看出当k=25时,各数据源保持在精确率比较高的稳定状态,虽然在k>25时,药物-化学结构和药物-疾病数据源的精确率会有小幅度的提升,但是药物-靶蛋白和药物-副作用数据源的精确率会有大幅的下降,所以该文将邻居数选取为25。

图3 各数据源在不同邻居数下的精确率
在确定邻居数k后,通过图4、图5对比MSF算法和其他算法的效果。在观察图4(ROC曲线)时发现,所有数据源的AUC值都很高,这是由于药物重定位是一个高度不平衡问题,所有的数据源都是稀疏的,即使假正例的数量变化很大,也会因为真反例的基数大,而假正率只发生极小的变化[9]。所以在ROC曲线和AUC值对于区分算法效果表现较差的情况下,采用了P-R曲线(见图5)和精确率、召回率、F-score(见表2)来进一步证明MSF算法优于传统药物重定位算法。

图4 基于单个数据源的传统药物重定位算法和MSF算法的ROC曲线比较

图5 单个数据源的传统药物重定位算法和MSF算法的P-R图比较
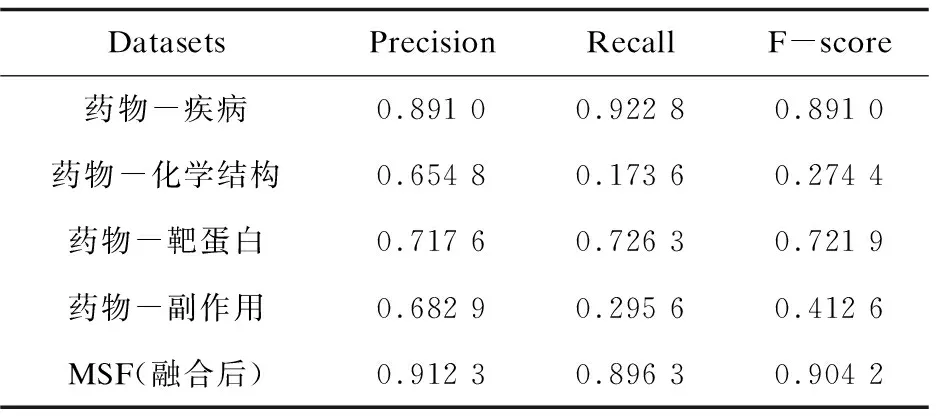
表2 单个数据源与MSF算法指标对比
通过P-R曲线可以看出,MSF算法的P-R曲线包裹了所有单个数据源的P-R曲线。通过表2可以看出,只有通过药物-疾病数据源得出的召回率比MSF算法略高,但是在精确率和F-score方面MSF算法都表现得更好,据此可以得出MSF优于基于单个数据源的药物重定位推荐算法。
表3展示了MSF算法与其他两种算法(SLAMS算法[9]和DRCFFS算法[14])的比较。可以看出与SLAMS算法和DRCFFS相比,MSF算法在三种指标上都有较大的提升。
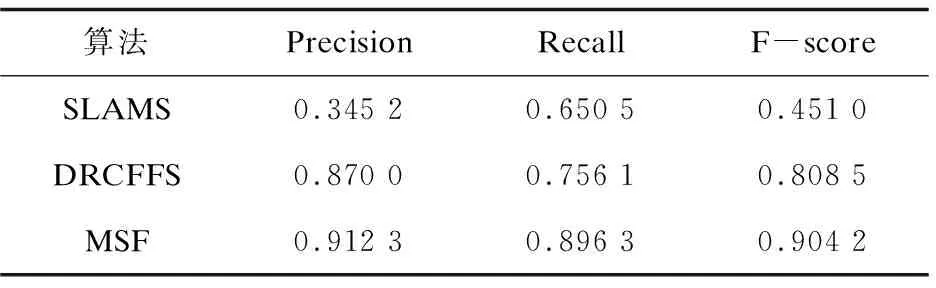
表3 算法效果比较
通过上述几种不同指标的对比表明,MSF算法在P-R曲线以及各项指标上都有良好的表现,可以得到更精确的药物重定位预测结果。
4 案例分析
药物重定位算法的目的是预测旧药物的新用途,为了证明提出的算法是有实效的和现实意义的,表4给出了利用MSF算法预测的一些药物-疾病组合,其中头孢西丁临床证明可以治疗由敏感菌导致的呼吸道感染等疾病,这与可以治疗支气管炎的预测相符合。厄他培南对肺炎的治疗作用在文献[27]中已经得到证实。文献[28]中进行了头孢克洛治疗鼻窦炎的临床实验,治愈率达到15.5%。文献[29]中给出了利用美罗培南治疗皮肤软组织感染的实例。
表5给出了甲状腺肿大治疗预测值前五的药物,其中甲硫咪唑和丙硫氧嘧啶已经得到临床验证[30],剩下三种药物尚未得到临床证明,但在文献[31]中双硫仑被证明对甲状腺治疗有一定作用。
综上所述,MSF算法可以预测出一些已经得到临床认证的药物-疾病组合,并且也能发现一些尚未证明有临床意义的药物-疾病组合,但是药物的治疗效果与疾病相关症状对应。通过一系列的案例证明,MSF算法具有可行性,对于临床药物重定位具有一定的辅助作用。

表4 预测药物-疾病治疗

表5 甲状腺肿大治疗预测值前五的药物
5 结束语
提出了一种基于多相似度融合的药物重定位推荐算法(MSF算法),实验结果显示MSF算法与SLAMS算法和DRCFFS相比,在三种指标(精确率、召回率和F-score)上的表现更好。综上所述,提出的MSF算法优于传统的药物重定位算法,可以预测有治疗效果的药物-疾病组合,为更好地发挥药物治疗效果、提升药物利用价值起到一定的作用。但是,MSF算法也有一定的缺陷,比如:通过试探法计算相似度融合的权值耗时较长,计算疾病相似度所采用的数据源较少。在后续的研究过程中应当对相似度融合的方法进行改进并且在疾病相似度的计算上使用多种数据源。
