基于SLAF-seq 技术构建栽培草莓高密度遗传图谱
2021-01-08张建军范婧芳宋世佳李海山
董 辉,杨 莉,李 莉,张建军,范婧芳,宋世佳,杨 雷,李海山
(1.河北省农林科学院 石家庄果树研究所,河北 石家庄 050061;2.河北省农林科学院,河北 石家庄 050031;3.河北省植保植检总站,河北 石家庄 050035)
栽培草莓(Fragaria×ananassa, 2n=8X=56)是蔷薇科草莓属的多年生草本植物,是重要的经济作物,在世界范围内广泛栽培。中国是世界草莓第一生产国,种植面积和产量均居世界第一[1]。栽培草莓起源于智利草莓(Fragariachiloensis)和弗州草莓(Fragariavirginiana)的偶然杂交[2],为异源八倍体,相较于二倍体作物其遗传背景复杂,相关的遗传育种研究具有一定难度。近年来,AFLP、SCAR、SSR等分子标记技术被广泛用于栽培草莓遗传图谱的构建和数量性状的QTL定位、遗传进化分析及亲缘关系鉴定[3-5],Weebadde等[6]构建了429个标记定位的总图距1 541 cM的AFLP图谱,van Dijk等[7]构建了508个标记定位的总图距1 846 cM的SSR图谱,此外还包括多个标记相结合绘制的整合图谱[8],但是由于标记本身的特性和数量限制,构建的图谱密度和饱和度较低。
随着新一代测序技术(NGS)的发展以及SNP标记的开发利用,使得利用SNP标记构建遗传图谱成为可能,图谱从质量和清晰度上都有了很大的提高。近年来,这项技术也逐渐应用于栽培草莓中。2015年,Bassil等[9]在栽培草莓中获得了6 594个标记定位的总图距2 050 cM的SNP图谱。2017年,Nagano等[10]在栽培草莓中获得了11 574个标记定位的总图距2 816.5 cM的SNP和SSR的整合图谱。
本研究利用石家庄果树研究所自育高抗白粉病草莓品种红星为母本,日本高感白粉病草莓品种红颜为父本构建F1群体,通过SLAF-seq技术对栽培草莓F1群体进行高通量测序和SNP标记的开发,构建了高密度SNP遗传图谱,并对其特征和应用进行了分析研究。该图谱的构建有助于后期开发分子实用性标记,为栽培草莓的数量性状基因定位和基因克隆奠定基础。
1 材料和方法
1.1 试验材料
2015年以石家庄果树研究所自育高抗白粉病草莓品种红星为母本,日本高感白粉病草莓品种红颜为父本杂交,2016年2月播种杂交种子,2016年9月将实生苗移栽进温室大棚内,进行常规管理。
1.2 试验方法
采集红星×红颜杂种分离群体F1中200株子代及双亲的幼嫩草莓叶片,采用CTAB法提取基因组DNA,检测合格后,运用SLAF-seq技术和HighMap软件开发高密度分子标记,构建遗传图谱。
1.2.1 酶切方案设计及测序 根据草莓基因组大小、GC含量等信息,进行酶切预测,最终选择最适内切酶Hpy166II,定义SLAF标签酶切片段长度为314~394 bp,预计可得到126 427个SLAF标签。目的片段的筛选采用最适酶切方案进行酶切试验,SLAF标签加A处理、连接Dual-index[11]测序接头等,文库质检合格后用Illumina Xten进行PE150 bp测序,同时选用日本晴水稻(OryzasativaL.japonica)作为对照,用来评估建库试验的准确性。
1.2.2 SNP开发及编码 为了确保SNP的准确性,根据测序数据在栽培草莓参考基因组(ftp://ftp.kazusa.or.jp/pub/strawberry/genome/)上的定位结果,综合GATK和Samtools 2种方法得到的交集变异位点。为了便于遗传分析采用遗传学通用的等位编码规则,对多态性标签进行基因型编码。
1.2.3 图谱构建 将多态性SNP标记按照相应步骤进行过滤,用以保证遗传图谱质量:首先,去掉亲本测序深度小于4X的标记,选择基因型覆盖所有子代60%以上个体的标记;其次,参考Huang等[12]方法将筛选出的SNP标记分为28个连锁群,计算两两标记之间的MLOD值[13],去掉与其他SNP的MLOD值低于25的标记;最后,采用HighMap[14]软件以连锁群为单位分析估算标记的线性排列及相邻标记间的遗传距离,最后,参考Sun等[15]方法对遗传图谱质量进行评估。
2 结果与分析
2.1 SLAF文库建立及高通量测序
2.1.1 SLAF文库建立 使用Hpy166II内切酶对基因组进行酶切。SLAF标签长度为314~394 bp。预测得到126 427个在基因组上分布均匀的SLAF标签。以水稻测序数据为对照,用来评估酶切方案实施的有效性和酶切效率,本次试验中,对照酶切效率为95.22%,双端比对效率为93.41%,说明SLAF建库正常。根据标签的实际长度绘制对照序列插入片段的长度分布图,发现大部分对照序列的插入片段长度在预期的范围之内,建库质量良好(图1)。
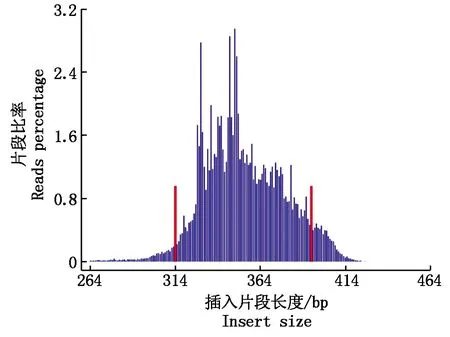
图1 对照序列插入片段分布图
2.1.2 测序质量值验证 为了检验测序质量,对测序质量值Q30进行了散点图图示验证。通过母本测序质量值作图(图2),发现在Q30>40的质量值范围内分布着绝大部分碱基,说明测序精确度高。
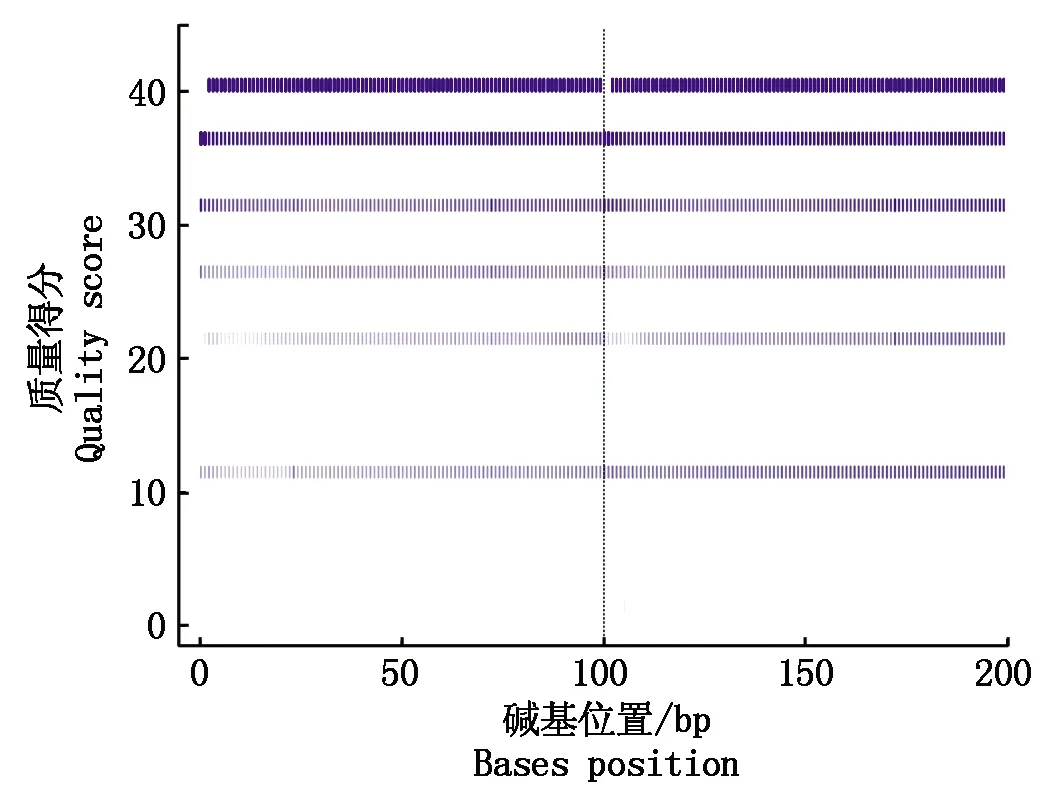
横坐标为碱基位置,纵坐标为质量值。前后100 bp分别为第一端和另外一端测序数据的Q30分布。同一个位置对应的测序数据质量越高,颜色越深。
2.1.3 碱基分布检查 通过检测有无AT、GC分离现象,对母本测序碱基分布情况进行评估(图3)。从分布图可以看出,A与T、G与C分布一致,符合碱基配对原则,说明得到了有效的SLAF序列,可以进行后续试验。
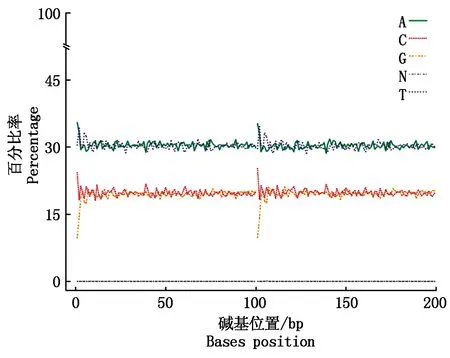
横、纵坐标分别为碱基位置和比率;碱基A、C、G、T和识别不出的N分别用绿色、红色、橙色、蓝色和灰色表示。前后100 bp分别为第一端和另一端测序数据的碱基分布。
2.1.4 测序数据统计与评估 通过SLAF文库的建立和高通量测序,共获得565 919 066 reads,(表1),测序量值Q30为95.36%,GC含量为39.68%,分布正常;其中父母本分别获得16 140 629,12 601 802 reads,Q30分别为96.28%和96.41%,GC含量分别为40.24%和39.25%;子代获得2 685 883 reads,平均Q30为95.35%,平均GC含量为39.68%。

表1 样品测序数据统计
2.2 标记开发
本研究共获得2 136 939个SNP标签,能成功分型的数目为152 515个,过滤掉aaxbb后得到可用于作图的SNP标记56 237个;同时还开发了717 881个SLAF标签。
2.3 遗传图谱构建及评估
2.3.1 遗传图谱构建 过滤掉与其他SNP标签的MLOD值均低于25的标签,共上图14 412个SNP,构建了28个连锁群,遗传图谱的总图距为4 022.16 cM,标记完整度为99.84%(上图标记中,确定基因型的标记占总标记的比例),标记间平均距离为0.28 cM(表2、图4)。
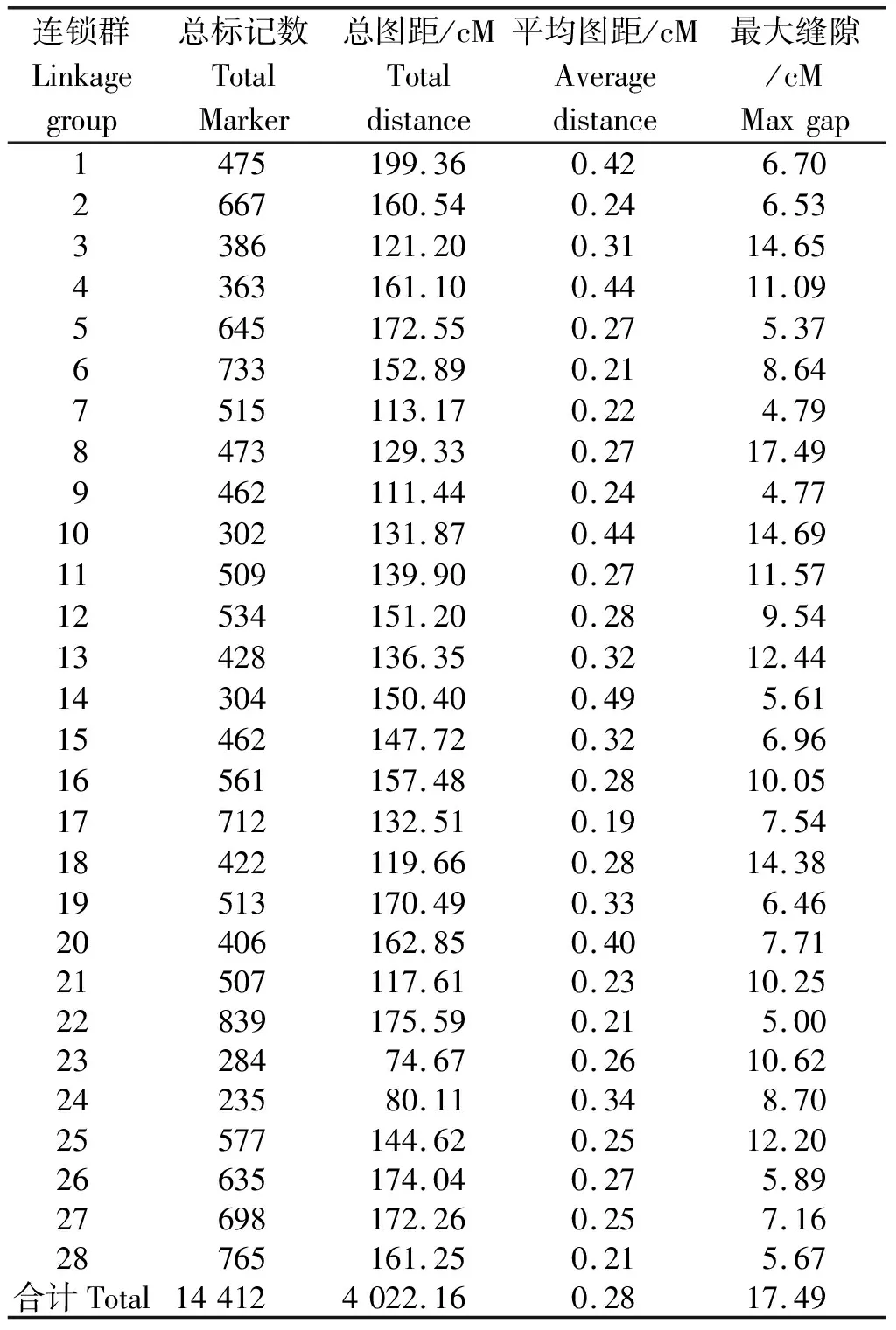
表2 栽培草莓遗传图谱基本信息统计
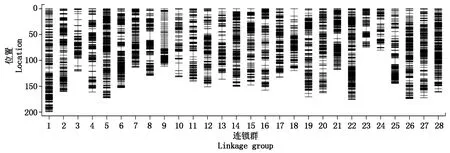
图4 栽培草莓遗传图谱
最大缝隙越小表示图谱越均匀,遗传图谱上最大和最小缝隙分别为17.49,4.77 cM,分别位于LG8和LG9,说明SNP的分布不是完全均匀的。由475个标记组成,长度为199.36 cM的LG1为28个连锁群中最大的,最小的连锁群为LG23,由284个标记组成,长度为74.67 cM。
2.3.2 遗传图谱评估
2.3.2.1 单体来源评估 连锁群LG1单体来源主要都是蓝色和绿色,说明本研究每个个体中较大区段均来源于亲本,而无法判断的白色区域和代表缺失的灰色区域占的比例很小,图谱质量高(图5)。

每个标记用一个横行表示,样品中的每条染色体用一个竖列表示,每个个体的父、母本染色体分别用第一、二列表示,个体之间用空白列隔开,来自亲本第一个、第二个、无法判断和缺失的等位基因分别用绿色、蓝色、白色和灰色表示。
2.3.2.2 标记连锁评估 遗传图谱实质上是多点重组分析,标记间的距离与重组率有着密切的联系,距离越近,重组率越小。为了了解染色体交换重组的规律,可以通过热图来分析相邻标记间的重组关系。连锁关系由强到弱分别用黄色、红色和紫色表示,由图可知标记顺序正确(图6)。
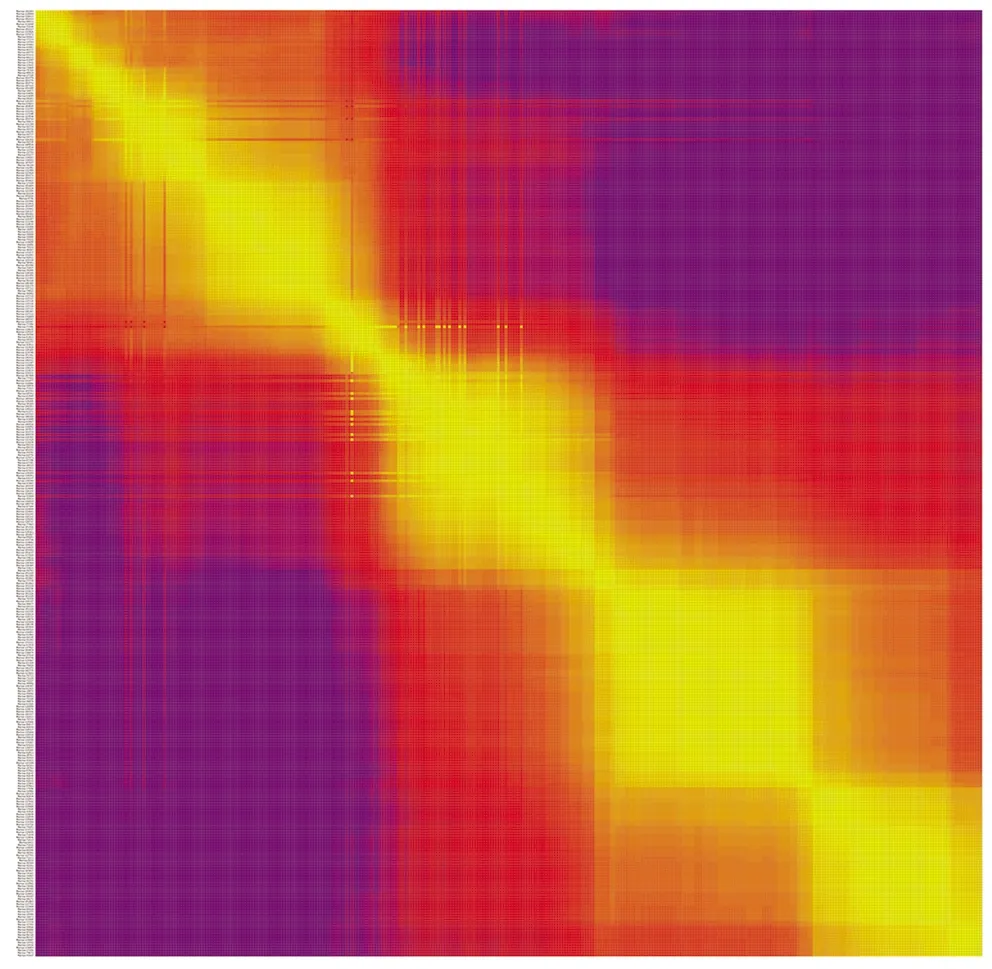
每一行和每一列都是标记,2个标记间的重组率用小方格表示,重组率从小到大体现在颜色上是从黄到红到紫的变化。
3 结论与讨论
遗传图谱对遗传研究、分子标记辅助育种以及QTL定位提供了重要的基础,遗传图谱的通用性和适用性决定于所包含的标记类型和数量。SNP标记作为高密度的单核苷酸多态性标记,为许多物种构建高密度遗传图谱提供了条件[16-18]。
SLAF-seq技术适合于快速、大量的开发SNP标记,是位点特异性扩增和高通量测序技术的结合,为了确保高效的开发高密度,分布均匀的标记,其根据研究对象参考基因组特点设计测序方案,目前已经在许多作物上得到应用[19-23]。近年来,栽培草莓SNP标记遗传图谱的构建研究也取得了一定的进展。2015年,Bassil等[9]在栽培草莓中获得了6 594个标记定位的总图距2 050 cM的SNP图谱,Davik等[24]获得了902个标记定位的总图距1 581.5 cM的SNP图谱。2017年,Nagano等[10]在栽培草莓中获得了11 574个标记定位的总图距2 816.5 cM的SNP和SSR的整合图谱,Lee等[25]获得了208个标记定位的总图距800.8 cM的SNP图谱。2019年,Hossain等[26]在栽培草莓中获得了1 268个标记定位的总图距2 581.57 cM的SNP图谱。
本研究获得了565 919 066 reads,717 881个SLAF标签和2 136 939个SNP标记,其中有56 237个SNP为高质量标记,可用于栽培草莓遗传图谱标记的定位和构建。本研究用HighMap软件绘制了高密度栽培草莓遗传图谱,共28个连锁群,总长度为4 022.16 cM,定位到图谱上的SNP标记为14 412个,标记完整度为99.84%,标记间平均距离为0.28 cM,图谱质量明显优于近期报道的栽培草莓遗传图谱,研究结果为栽培草莓研究提供了大量的分子标记,也为后期基因的精细定位、分离以及分子育种提供了重要依据。
