基于荧光原位杂交的藜属植物核型分析
2021-01-07权有娟李想袁飞敏刘博陈志国
权有娟 李想 袁飞敏 刘博 陈志国



摘 要: 為精确地识别藜属植物染色体组的核型特征,该文研究了4种来自青海高原的野生藜属植物(灰绿藜、藜、菊叶香藜及杂配藜)和1种从美国引进的栽培藜麦品种PI614932-HX(3)基于染色体荧光原位杂交(rDNA FISH)的核型。利用5S rDNA和45S rDNA对5种藜属植物有丝分裂中期的染色体进行FISH研究。藜属植物的核型分析结果表明:(1)藜属植物中存在二倍体(2n=2x=18)和四倍体(2n=4x=36)两种倍性,藜麦和灰绿藜为四倍体,其余3种为二倍体。(2)藜麦、灰绿藜、藜、菊叶香藜及杂配藜的核型公式分别为2n=4x=36=34m (2AST)+2sm,2n=4x=36=32m(4AST)+4sm,2n=2x=18=16m(4AST)+2sm,2n=2x=18=18m及2n=2x=18=16m+2sm。(3)染色体由大部分的中部着丝粒染色体(m)和少部分近中部着丝粒染色体(sm)组成。(4)核型类型除了菊叶香藜为1B以外,其余均属于2B类型。(5)在藜麦、灰绿藜及藜中具有分布位置不同、数量不等的双随体。5S rDNA、45S rDNA FISH结果表明:(1)藜麦和灰绿藜的染色体上存在2对5S rDNA位点和1对45S rDNA位点,藜、杂配藜的染色体上存在1对5S rDNA位点和1对45S rDNA位点,菊叶香藜的染色体上只存在1对5S rDNA位点。(2)5S rDNA和45S rDNA位点均位于染色体的短臂上。该研究首次获得了藜属植物基于5S rDNA和45S rDNA荧光原位杂交核型,为藜属植物亲缘关系研究和细胞生物学研究提供了分子细胞遗传学依据。
关键词: 藜属, rDNA, 荧光原位杂交, 核型
中图分类号: Q943
文献标识码: A
文章编号: 1000-3142(2021)12-1988-08
收稿日期: 2020-04-17
基金项目: 中国科学院种子创新研究院项目(INASEED);海西州财政支持农业项目(HXNM001);青海省种子工程项目(2019016);青海省重点研发与转化计划项目(2020-NK-122) [Supported by the Innovative Academy of Seed Design (INASEED); Financial Support Agriculture Project of Haixi Prefecture(HXNM001); Qinghai Seed Engineering Project(2019016); Key R & D and Transformation Program of Qinghai Province(2020-NK-122)。]
作者简介: 权有娟(1994-) ,硕士研究生,主要从事植物遗传育种研究,( E-mail)quanyoujuan828@163.com。
通信作者: 陈志国,研究员,博士生导师,主要从事作物遗传育种研究,( E-mail)zgchen@nwipb.cas.cn。
Karyotype analysis of Chenopodium species based on fluorescence in situ hybridization
QUAN Youjuan1,2, LI Xiang1,2, YUAN Feimin3, LIU Bo1,2, CHEN Zhiguo1*
( 1. Northwest Institute Plateau of Biology, Chinese Academy of Sciences/Key Laboratory of Adaptation and Evolution, Chinese Academy of Sciences/Key Laboratory of Crop Molecular Breeding in Qinghai Province, Xining 810008, China; 2. University of Chinese Academy of Sciences, Beijing 100049, China; 3. Northwest Agriculture and Forestry University, Yangling 712100, Shaanxi, China )
Abstract: In order to get much more precise information on the karyological characteristics of Chenopodium L. species, the karyotypes of four wild Chenopodium species from Qinghai Plateau, including C. glaucum, C. ablum, C. foetidum and C. hybridum, and one cultivated C. quinoa PI614932-HX(3) introduced from the United States were analyzed by using chromosome fluorescence in situ hybridization (rDNA FISH). 5S rDNA and 45S rDNA were mapped on the metaphase chromosomes of the five species by FISH. The results of the karyotype analysis were as follows: (1) There were two kinds of ploidies in these Chenopodium species, including a diploid (2n=2x=18) and a tetraploid (2n=4x=36), C. quinoa and C. glaucum were tetraploids, and the other three species were diploids. (2) The karyotype formulas of C. quinoa, C. glaucum, C. ablum, C. foetidum and C. hybridum were 2n=4x=36=34m (2AST)+2sm, 2n=4x=36=32m(4AST)+4sm, 2n=2x=18=16m(4AST)+2sm, 2n=2x=18=18m and 2n=2x=18=16m+2sm, respectively. (3) The chromosomes of Chenopodium were mainly composed of metacentric chromosomes (m) and a few submetacentric chromosomes (sm). (4) Except for C. foetidum belonging to 1B type, the others belonged to 2B type. (5) There were double satellites distributed at different positions with different numbers on the chromosomes of C. quinoa, C. glaucum and C. ablum. The results of 5S rDNA and 45S rDNA FISH were as follows: (1) There were two pairs of 5S rDNA loci and one pair of 45S rDNA loci on the chromosomes of C. quinoa and C. glaucum, one pair of 5S rDNA and one pair of 45S rDNA on the chromosomes of C. ablum and C. hybridum, and only one pair of 5S rDNA on the chromosomes of C. foetidum. (2) 5S rDNA and 45S rDNA loci were all located on the short arm of the chromosomes. It is the first report on karyotype analysis with 5S rDNA and 45S rDNA loci in Chenopodium species and the results will provide a cyto-moecular genetic basis for phylogeny and cell biology research of Chenopodium species.
Key words: Chenopodium L., rDNA, fluorescence in situ hybridization (FISH), karyotype
藜属(Chenopodium L.)按原来的分类系统属于藜科(Chenopodiaceae),APGⅡ植物分类系统将其划分到苋科(Amaranthaceae)藜亚科(Chenopodioideae)中(Angiosperm Phylogeny Group,2003),為一年生或多年生草本,全世界约有250种,部分种类具有食用、药用和饲用、保护生态环境等功能(张薇薇等,2015)。在我国,有19个野生藜属植物种(刘尚武,1997)及1个引进栽培种藜麦(Chenopodium quinoa),主要分布于西北、华北、西南和东北等地。该属植物常生长在海拔较高、土地贫瘠、盐碱化较重的干旱、半干旱地带,大多具有较强的耐旱性、耐盐性(Eisa et al., 2001)。
关于藜属植物,前人在资源调查(张薇薇等,2015)、系统发育与分类(孔宪武和简焯坡,1979;Fuentes-Bazan et al., 2012;王春海,2015)、生理特性(Karssen, 1976;陈莎莎等,2009;王璐等,2015;袁飞敏等,2018;张乐等,2019)、营养特性(Abugoch & Lilian, 2009;王艳萍等,2019)、植物化学(杜华,2007;张洪慎等,2018)、分子生物学(刘艳霞等,2016;付苏宏等,2016;Jarvis et al., 2017;宋娇,2018)、遗传育种(杨发荣等,2015;林春等,2019)等方面进行了广泛的研究。由于藜属植物的染色体较小,染色体数目较多,导致藜属植物染色体制片难度大,传统的核型分析方法很难明确识别其染色体,其核型研究尚处于初级阶段。据前人研究,藜属植物内部存在多倍化现象,主要有二倍体(2n=2x=18)、四倍体(2n=4x=36)和六倍体(2n=6x=54)3种倍性(Aellenet al., 1943),单倍染色体基数为9(x=9)(Kolano et al., 2001),但Ambrina组的染色体基数为8,如C.ambrosioides的染色体数目为2n=2x=16(Giusti, 1970)。在国内,何燕等(2019)利用常规压片法仅对一个栽培型藜麦品系进行了核型研究,至今未见其他野生藜属植物核型研究的报道。
rDNA FISH是细胞遗传学研究中常用的手段之一,具有灵敏度高、分辨率高、安全性好、实验周期短、直观可见等优点,可为核型分析提供一个稳定有效的细胞学可识别标记,也为植物外源染色体鉴定、遗传育种、系统进化及亲缘关系等研究提供重要的信息(Soltis et al., 1999;Volkov et al., 2007)。Maughan et al.(2006)利用FISH对藜麦和其野生近缘种C. berlandieri的45S rRNA基因间隔区序列(IGS)和5S rDNA间隔区(NTS)进行了分析,证实了两者之间的亲缘关系非常近,推测它们可能来源于异源多倍体藜麦的两个亚基因组,其中一个和现在C. berlandieri中的NTS序列相似,这一发现表明这两个异源四倍体物种至少有一个共同的二倍体祖先。Kolano et al.(2012)对34种藜属植物的中期染色体进行5S rDNA和45S rDNA的物理定位,通过位点数目及分布特点的观察,可以为识别34种藜属植物的染色体提供明确有效的分子细胞学标记。Kolano et al.(2015)分析了23个二倍体藜属植物染色体上5S rDNA和45S rDNA分布的数量,推测了二倍体植物基因组进化的模式和方向。
本研究以5种藜属植物为材料,系统研究了藜属植物染色体荧光原位杂交技术体系,获得了基于FISH技术的藜麦、灰绿藜、藜、菊叶香藜及杂配藜的染色体核型图,并分析了5S rDNA、45S rDNA在5种藜属植物中期染色体上的分布特点。研究结果为藜属植物基因在染色体上的物理定位提供了技术基础,也为进一步在分子细胞学水平探讨藜属植物亲缘关系奠定了基础,这也将是首次对国内野生藜属植物的研究提供分子细胞遗传学资料。
1 材料与方法
1.1 供试材料
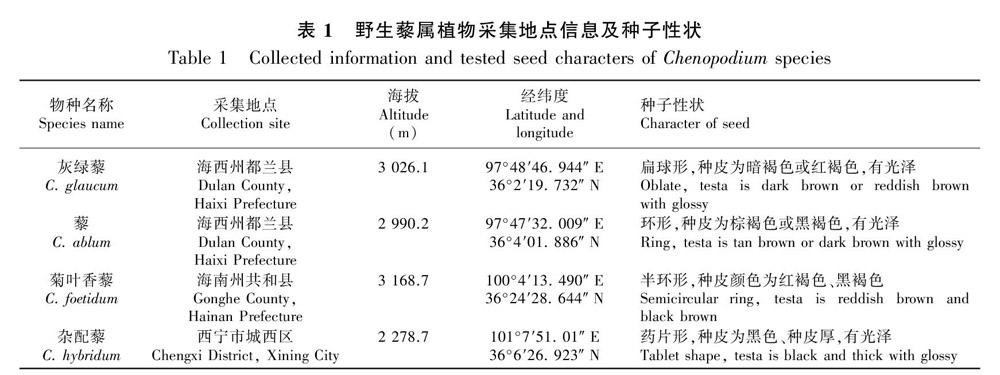
供试藜麦栽培品种引自美国,编号为PI614932-HX(3),野生藜属植物于2016年10月分别采自柴达木盆地、共和盆地及西宁周边荒地,每个物种分别采集20余个个体,由中国科学院西北高原生物研究所卢学峰研究员鉴定,单独脱粒后种子保存在中国科学院高原生物适应与进化重点实验室中。采集地点及种子性状详细信息见表1。
1.2 方法
1.2.1 中期分裂相染色体制备
挑选成熟度好、饱满、籽粒大小一致的供试种子,用70%的酒精消毒后,用蒸馏水漂洗3次,分别放置于铺有两层滤纸的发芽盒(12 cm × 6 cm)中。再用蒸馏水浸润后放置于种子培养箱中。种子培养箱参数条件设置为25 ℃,昼长16 h,夜长8 h,湿度65%。待根尖长至1~1.5 cm时,将整个发芽的种子放到4 ℃左右的冰水混合物中预处理20~24 h。取出发芽的种子后用乙醇∶冰醋酸(3∶1)固定至少30 min。从根尖上切取1~2 mm的分生区,45%乙酸火焰干燥压片,在相差显微镜下镜检, 将一个视野下有多个清晰的中期染色体分裂相的片子放于-80 ℃冰箱中冷冻保存。
1.2.2 标记探针 探针为通用探针,购自上海生物工程有限公司。
1.2.3 荧光原位杂交 荧光原位杂交实验参考自本课题组喻凤等(2017)的方法。将制备好的染色体制片从-80 ℃冰箱中拿出后迅速用刀片揭掉盖玻片,干燥后放置于0.2 mol·L-1溶于70%酒精的氢氧化钠溶液中变性8~10 min;取出后直接放置于预冷的70%乙醇中做脱水处理约40 min;干燥后将配制好的杂交液10 μL(5 μL 50%甲酰胺,2 μL 20×SSC,1 μL 鱼精DNA,2 μL 10% 硫酸葡聚糖)中加入5S rDNA和45S rDNA各1 μL,混匀后滴至干燥的片子上,盖上较大的盖玻片;将片子放于潮湿密闭盒中,置于37 ℃下杂交8 h,过夜。
1.2.4 镜检 待时间到后用2×SSC或ddH2O冲洗几次,干燥后向制片上滴10~12 μL含有荧光猝灭剂的DAPI染液,盖上较大的盖玻片。用Leica荧光显微镜在低倍镜下找到染色体,在高倍镜下快速拍照,并标记后保存。
1.2.5 图像处理及分析 图片用软件Photoshop cc 2015进行调整及优化并完成配对,用软件Emage J、软件Excel 2019进行后期的图像处理及染色体长度、臂比等的计算。核型分析及核型分类的依据分别采用李懋学等(1985)和Stebbin et al.(1971)的劃分标准,核型不对称系数参照Arano et al.(1962)的方法。
2 结果与分析
2.1 5种藜属植物的核型分析
基于rDNA FISH技术得到了5种藜属植物中期染色体5S rDNA和45S rDNA FISH制片,测定核型参数,并进行核型分析(表2)。藜麦和灰绿藜都是四倍体,染色体数目均为2n=4x=36,藜、菊叶香藜及杂配藜都是二倍体,染色体数目均为2n=2x=18,没有发现其他倍性或非整倍体存在的现象。除了菊叶香藜全部由中部着丝粒染色体组成外,其余4种藜属植物的染色体主要由中部着丝粒染色体(m)和少数近中部着丝粒染色体(sm)组成。藜属植物染色体绝对长度不足2 μm,属于小染色体。不同种类藜属植物的核型公式和染色体相对长度存在差异。根据染色体分类系统,菊叶香藜的核型类型为1B,核型形态较对称,相较于其他4种核型类型为2B的藜属植物在进化上较原始。
藜属植物部分种中具有随体。在藜麦中发现了1对双随体,位于2号染色体上的其中一条同源染色体的短臂上。在灰绿藜和藜中各发现2对双随体,均位于两条同源染色体的短臂上, 在灰绿藜中为12号染色体,在藜中为9号染色体。
2.2 藜麦rDNA FISH信号位点检测结果
rDNA FISH检测显示,藜麦染色体上检测到2对5S rDNA和1对45S rDNA信号位点:1对5S rDNA信号位点位于12号染色体的两条同源染色体的间隙位置,另1对位于17号染色体短臂(S)末端,信号强度一致;45S rDNA信号位点位于5号染色体短臂(S)末端,并且1个同源染色体杂交信号较另1个强(图1:B,C)。
在信号强度上和前人的研究结果存在微小差异,虽然本研究中45S rDNA与Kolano et al.(2012)发现的45S rDNA位点的信号强度一致(图1:A),但本研究中45S rDNA的信号明显强于5S rDNA位点信号。
2.3 灰绿藜rDNA FISH信号位点检测结果
灰绿藜染色体上检测到2对5S rDNA和1对45S rDNA信号位点:1对5S rDNA信号位点位于3号染色体的短臂(S)末端,另1对位于7号染色体两个同源染色体的间隙,3号染色体上的信号杂交比7号染色体强;45S rDNA信号位点位于12号染色体短臂(S)末端的随体上,杂交信号强度一致(图2)。Kolano et al.(2008a)研究结果显示,二倍体灰绿藜(2n=2x=18)中存在1对5S rDNA位点和1对45S rDNA位点。本研究中四倍体灰绿藜中多1对5S rDNA位点。
2.4 藜rDNA FISH信号位点检测结果
藜染色体上检测到1对5S rDNA和1对45S rDNA信号位点:5S rDNA位点位于1号染色体的短臂(S)末端,杂交信号强度一致;45S rDNA位于9号染色体短臂(S)末端的随体上,杂交信号强度一致(图3:A,B)。
藜是一个复合体,有二倍体、四倍体、六倍体(Bhargava et al., 2006)。已有的研究结果显示,六倍体藜中存在4对5S rDNA位点和2对45S rDNA位点,5S rDNA位点和45S rDNA的杂交信号强度均不同(Kolano et al., 2008b)。二倍体藜中存在2对5S rDNA位点和1对45S rDNA位点,均位于染色体末端,5S rDNA位点在信号强度和大小方面存在明显差异(图3:C,D)。本研究中二倍体藜中丢失了信号较强的1对 rDNA位点。
2.5 菊叶香藜及杂配藜的rDNA FISH信号位点检测结果
菊叶香藜染色体上检测到1对5S rDNA信号位点,未检测到45S rDNA信号位点,5S rDNA位于2号染色体的短臂(S)末端,杂交信号强度一致,信号较强(图4:A,B)。
杂配藜的染色体上检测到1对5S rDNA位点和1对45S rDNA位点:5S rDNA位点在5号染色体的两个同源染色体的间隙位置,杂交信号强度一致;45S rDNA位点在7号染色体短臂(S)末端,1个杂交信号较另1个强很多(图4:C,D)。此结果和Kolano et al.(2012)的研究结果一致。
3 讨论与结论
3.1 藜属植物的染色体数目及类型
本研究中所有藜属植物的染色体基数为9,存在多倍化现象,和Mehra & Malik(1963)、Pal & Shukla(1990)等的研究结果一致。有些物种其种内存在多种倍性,不同倍性的植物核型不同,有些物种在相同倍性的不同亚种(品种)中,核型也存在差异。Bhargava et al.(2006)首次对藜麦、藜、菊叶香藜等几个藜属植物进行了全面的细胞遗学研究,用常规核型分析法获得几种藜属植物的核型数据。其中,7份藜麦材料中,核型基本相似,均为四倍体,推翻了早期藜麦中存在混倍性的研究结果(Wang et al., 1993),并以染色体的最长臂和最短臂的比值划分核型类型,比值小于2的为1A类型,比值大于2的为1B类型,核型不对称指数变化范围在43.9%~47.4%之间,不同染色体的最长染色体不是中部着丝粒染色体(m)就是近中部着丝粒染色体(msm),臂比值在1.18~1.56之间,其中第4对、第9对、第18对的中位数(M或m)最为保守。第10对和第13对变异最大,臂比值分别在1.0~1.86和1.0~1.78之间。在藜复合群体中,三种倍性的藜之间核型差异较大,四倍体藜的核型不对称性程度比二倍体和六倍体高,已经具有杂交不亲和性。二倍体藜之间核型也存在明显的分化,最长染色体和最短染色体的比值相对较高,为2.63,核型类型有1A和2A两种类型,有3对近中部着丝粒染色体(sm),核型不对称系数变化范围在43.6%~47.9%之间。其菊叶香藜的核型公式为2n=2x=18=4M+1m+3msm+1sm,核型类型为1A,核型不对称系数为44.1%。除了与相应倍性物种的染色体数目一致外,在核型公式、核型类型及核型不对称系数等方面和该研究中的藜属植物不同,对于栽培型的藜麦,可能由于异源起源(Wilson, 1990)、自花授粉(Risi & Galwey, 1984)导致染色体形态变化(主要是倒位和易位)从而引起染色体核型上的微小差异。相同倍性的野生藜属植物核型也存在差异,这可能是由环境、中期染色体制片技术及方法、测量工具等造成。
3.2 藜属植物的随体
本研究中,藜麦、灰绿藜及藜中出现了数目不等、位置不同的双随体。藜麦中具有1对双随体,位于2号染色体的短臂上,和已报道的C. quinoa PI587173、C. quinoa PI584524及C. quinoa CHEN33/84中双随体分布的位置相同,可能与其属同一种类型或来源自相同的地方,在3号短臂、3号长臂、8号短臂、12号短臂及15号短臂也有分布(Bhargava et al., 2006;何燕等,2019)。在藜中发现了2对双随体,位于9号染色体的短臂上,和已报道的印度二倍体藜C. album ‘Chandanbathua’ 中随体的分布一致(Bhargava et al., 2006)。先前的研究中没有关于灰绿藜中存在随体的报道,我们首次发现了2对双随体。迄今,在藜属植物的研究中,只有在藜麦、四倍体灰绿藜、二倍体和四倍体藜中具有双随体,此3种物种之间是否具有较近的亲缘关系及能否作为藜属植物识别的标记需进一步研究。
3.3 5种藜属植物的rDNA FISH差异
已有研究表明,在二倍体、四倍体、六倍体的藜属植物中5S rDNA和45S rDNA不存在共定位,45S rDNA总是位于染色体的末端,5S rDNA位置的分布有两种,一种分布在染色体的末端,一种分布在两个同源染色体的间隙,具体的位置分布因物种而异,rDNA位点的数量有3~6个(Kolano et al., 2012),此5种藜属植物中除菊叶香藜具有2個(1对)rDNA位点外,藜麦、灰绿藜、藜和杂配藜的研究结果与前人的结果基本一致。本研究中发现二倍体藜属植物中,分布有1对(2个)5S rDNA位点和1对(2个)45S rDNA位点(菊叶香藜除外),四倍体藜属植物中具有2对(4个)5S rDNA位点和1对(2个)45S rDNA位点,六倍体藜属植物中具有4对(8个)5S rDNA位点,2对(4个)45S rDNA位点,5S rDNA的数量随着染色体倍性的增加而成倍增加,Prokopowich et al.(2003)和Long et al.(2013)也在其他动植物中发现了这种现象,但Kolano et al.(2015)发现在二倍体藜属植物中rDNA的数量跟基因组的大小没有相关性,如C. ficifolium 486 具有2对5S rDNA位点和1对45S rDNA位点,C. ficifolium 147 具有1对5S rDNA位点和2对45S rDNA位点。在多倍体藜属植物,尤其是在四倍体和六倍体植物中,45S rDNA的数量比5S rDNA少。Kolano et al.(2011)发现在异源多倍体物种形成后,至少有一个祖先亚基因组经历了45S rDNA基因位点的丢失。目前,藜属植物中关于rDNA位点及数量的研究,都存在45S rDNA位点的情况,本研究中菊叶香藜中没有45S rDNA位点,猜测有可能和5S rDNA位点存在共线性,Kolano et al.(2015)就认为在C. standleyanum中,5S rDNA位点和45S rDNA位点位于同一对染色体上。
致谢 窦全文研究员提供了实验平台和条件,在此谨对窦老师的悉心指导和帮助表示衷心感谢!
参考文献:
ABUGOCH J, LILIAN E, 2009. Quinoa (Chenopodium quinoa Willd.): Composition, chemistry, nutritional and functional properties [J]. Adv Food Nutr Res, 58: 1-31.
AELLEN P, JUST T, 1943. Key and synopsis of the American species of the genus Chenopodium L. [J]. Am Mid Nat, 30(1): 47-76.
ANGGIOSPERM PHYLOGENY GROUP, 2003. An update of the Angiosperm Phylogeny Group classification for the orders and families of flowering plants: APG Ⅱ [J]. Bot J LinnSoc, 141(4): 399-436.
ARANO H, 1962. Cytological studies in subfamily Carduoideae (Compositae) of Japan VII [J]. Bot Mag in Tokyo, 75(892): 401-410.
BHARGAVA A, RANA TS, SHUKLA S, et al., 2005. Seed protein electrophoresis of some cultivated and wild species of Chenopodium [J]. Biol Plant, 49(4): 505-511.
BHARGAVA A, SHULKA S, OHRI D, 2006. Karyotypic studies on some cultivated and wild species of Chenopodium (Chenopodiaceae) [J]. Gen Res Crop Evol, 53(7): 1309-1320.
CHEN SS, YAO SX, YUAN JW, et al., 2010. Seed germination characteristics of halophyte Chenopodium glaucum L. and its adaptability to the natural desert habitats in Xinjiang [J]. J Plant Physiol, 46(1): 75-79. [陈莎莎,姚世响,袁军文,等,2010. 新疆荒漠地区盐生植物灰绿藜种子的萌发特性及其对生境的适应性 [J]. 植物生理学报, 46(1): 75-79.]
DU H, ZHOU LG, LI C, et al., 2007. Recent studies on the chemistry and bioactivity of Chenopodiaceous Plant [J]. Nat Prod Res Dev, 10(19): 884-889. [杜华,周立刚,李春,等,2007. 藜科植物化学成分与生物活性的研究进展 [J]. 天然产物研究与开发,10(19): 884-889.]
EISA S, HUSSIN S, GEISSLER N, et al., 2012. Effect of NaCl salinity on water relations, photosynthesis and chemical composition of quinoa ( Chenopodium quinoa Willd.) as a potential cash crop halophyte [J]. Aust J Crop Sci, 6(6): 357-368.
FU SH, LEI M, ZHANG YQ, et al., 2019. Mining and analysis of genic single nucleotide polymorphism(SNP) information in transcriptome of Chenopodium foetidum [J]. SW Chin J Agric Sci, 32(4): 734-740. [付蘇宏,雷鸣,张勇群,等,2019. 菊叶香藜转录组单核苷酸多态性(SNP)信息挖掘及功能注释分析 [J]. 西南农业学报, 32(4): 734-740.]
FUENTES-BAZAN S, MANSION G, BORSCH T, 2012. Towards a species level tree of the globally diverse genus Chenopodium(Chenopodiaceae) [J]. Mol Phylogenet Evol, 62(1): 359-374.
GIUSTI L, 1970. EI genero Chenopodium en Argentina 1: Numeros de cromosomas [J]. Darwiniana, 16: 98-105.
HE Y, DENG YX, LI MH, et al., 2019. Chromosome number and karyotype analysis of quinoa(Chenopodium quinoa Willd. ) [J]. J SW Univ (Nat Sci Ed), 41(1): 27-31. [何燕,邓永辉,李梦寒,等,2019. 藜麦品系的染色体数目及核型分析 [J]. 西南大学学报(自然科学版) [J]. 41(1): 27-31.]
JARVIS DE, HO YS, LIGHTFOOT DJ, et al., 2017. The genome of Chenopodium quinoa [J]. Nature, 542(7641): 307-312.
KARSSEN CM, 1976. Two sites of hormonal action during germination of Chenopodium album seeds [J]. Physiol Plantarum, 36(3): 264-270.
KONG XW, JIAN ZP, 1979.Flora Reipublicae Popularis Sinicae (Vol. 25 2nd) [M]. Beijing: Science Press: 79-98. [孔宪武,简焯坡,1979. 中国植物志(第25卷第2分册) [M]. 北京: 科学出版社: 79-98.]
KOLANO B, PANDO LG, MALUSZYNSKA J, 2001. Molecular cytogenetic studies in Chenopodium quinoa and Amaranthus caudatus [J]. Acta Soc Bot Pol, 70(2): 85-90.
KOLANO B, PLUCIENNICZAK A, KWASNIEWSKI M, et al., 2008a. Chromosomal localization of a novel repetitive sequence in the Chenopodium quinoa genome [J]. Bot Mag in Tokyo, 75(892): 401-410.
KOLANO B, SIWINSKA D, MALUSZYNSKA J, 2008b. Comoarative cytogenetic analysis of diploid and hexaploid Chenopodium album Agg. [J]. Acta Soc Bot Pol, 77(4): 293-298.
KOLANO B, GARDUNIA BW, MICHALSKA M, et al., 2011. Chromosomal localization of two novel repetitive sequences isolated from Chenopodium quinoa genome [J]. Genome, 54: 710-717.
KOLANO B, TOMCZAK H, MOLEWSKA R, et al., 2012. Distribution of 5S and 35S rRNA gene sites in 34 Chenopodium species (Amaranthaceae) [J]. Bot J Linn Soc, 170(2): 220-231.
KOLANO B, SIWINSKA D, MCCANN J, et al., 2015. The evolution of genome size and rDNA in diploid species of Chenopodium s.l.(Amaranthaceae) [J]. Bot J Linn Soc, 179(2): 218-235.
LI MX, CHEN RY, 1985. A suggestion on the standarization of karyotype analysis in plant [J]. J Plant Sci, 3(4): 297-302. [李懋学,陈瑞阳,1985. 关于植物核型分析的标准化问题 [J]. 植物科学学报, 3(4): 297-302.]
LIN C, LIU ZJ, DONG YM, et al., 2019. Domesticated cultivation and genetic breeding of Chenopodium quinoa [J]. Hereditas, 41(11): 1009-1022. [林春,刘正杰,董玉梅,等,2019. 藜麦的驯化栽培与遗传育种 [J]. 遗传, 41(11): 1009-1022.]
LIU YX, LAN XX, CAO J, et al., 2016. Screening of qRT-PCR reference genes for Chenopodium album and C.glaucum of Chenopodiaceae [J]. Guihaia, 36(12): 1511-1518. [刘艳霞,兰欣欣,曹婧,等,2016. 藜科植物藜和灰绿藜实时荧光定量PCR内参基因的选择 [J]. 广西植物,36(12): 1511-1518.]
LIU SW, 1997. Qinghai Flora (Volume I) [M]. Xining: Qinghai People’s Publishing Press: 185-233. [刘尚武,1997. 青海植物志(第I卷) [M]. 西宁: 青海人民出版社: 185-233.]
LONG Q, RABANAL FA, MENG DZ, 2013. Massive genomic variation and strong selection in Arabidopsis thaliana lines from Sweden [J]. Nat Genet, 45: 884-890.
MAUGHAN PJ, KONLANO BA, MALUSZYNSKA J, et al., 2006. Molecular and cytological characterization of ribosomal RNA genes in Chenopodium quinoa and Chenopodium berlandieri [J]. Gene, 49(7): 825-839.
MEHRA PN, MALIK CP, 1963. Cytology of some Indian Chenopodiaceae [J]. Caryologia, 16(1): 67-84.
PAL M, SHUKLA S, 1990. A hexaploid grain chenopod from Eastern Himalayas [J]. Newslett Himalayan Bot, 8: 12-14.
PROKOPOWICH CD, GREGORYT R, CREASE TJ, 2003. The correlation between rDNA copy number and genome size in eukaryotes [J]. Gene, 46: 48-50.
RISI J, GALWEY NW, 1984. The Chenopodium grains of the Andes: Inca crops for modern agriculture. in advances in applied biology [M]. Academic Press: London: 145-216.
SONG J, 2018. Genetic diversity of germplasm and the variation analysis of quinoa [D]. Xining: Qinghai University: 1-52. [宋嬌,2018. 藜麦种质资源遗传多样性研究及藜麦品种(系)变异率分析 [D]. 西宁: 青海大学: 1-52.]
SOLTIS DE, SOLTIS PS, 1999. Polyploidy: recurrent formation and genome evolution [J]. Trends Ecol Evol, 14(9): 348-352.
STEBBINSG L, 1971. Chromosome evolution high plant [M]. London: Edward Aronld Ltd: 85-104.
VOLKOV RA, KOMAROVA NY, HEMLEBEN V, 2007. Ribosomal RNA in plant hybrids: inheritance, rearrangement, expression [J]. Syst Biodivers, 5(3): 2661-276.
WANG S, TSUCHIYA T, WILSON HD, 1993. Chromosome studies in several species of Chenopodium from North and south Americas [J]. Indian J Genent Plant Breed, 47: 163-170.
WANG CH, 2015. Systematic studies on Chenopodium and related genera of China [D]. Qufu: Qufu Normal University: 1-42. [王春海,2015. 中国藜属及近缘属植物的系统学研究 [D]. 曲阜: 曲阜师范大学: 1-42.]
WANG L, CAI M, LAN HY, 2015. Comparison of salt tolerance between Chenopodium album and C. glaucum [J]. Acta Phytophysiol Sin, 51(11): 1846-1854. [王璐,蔡明,兰海燕,2015. 藜科植物藜与灰绿藜耐盐性的比较 [J]. 植物生理学报, 51(11): 1846-1854.]
WANG YP, REN T, WANG TT, 2019. Determination and comparison of nutritive components of quinoa and other grains [J]. Modern Food, (22): 164-166. [王艳萍,任婷,王童童,2019. 藜麦与燕麦、糙米等5种谷物营养成分的测定与比较 [J]. 现代食品,(22): 164-166.]
WILSON H, 1990. Quinoa and relatives(Chenopodium sect. Chenopodium subsect. Celluloid) [J]. Econ Bot, 5: 253-263.
YANG FR, 2015. Breeding and application prospects of new variety Chenopodium quinoa cv. Longli 1 [J]. Gansu Agric Sci Technol, (12): 1-5. [杨发荣,2015. 藜麦新品种陇藜1号的选育及应用前景 [J]. 甘肃农业科技, (12): 1-5.]
YU F, 2017. Comparative cvtogentics on Medicago sttiva L. and related species [D]. Beijing: University of Chinese Academy of Sciences: 1-89. [喻凤,2017. 苜蓿近缘种属的比较细胞遗传学研究 [D]. 北京: 中国科学院大学: 1-89.]
YUAN FM, QUAN YJ, CHEN ZG, 2018. Effects of sodium stress on seed germination of Chenopodium quinoa Willd. [J]. J Arid Land Resour Environ, 32(11): 182-187. [袁飞敏,权有娟,陈志国,2018. 不同钠盐胁迫对藜麦种子萌发的影响 [J]. 干旱区资源与环境, 32(11): 182-187.]
ZHANG HS, LIU YX, LI W, 2018. Study on the extraction method of total alkaloids from C. foetidum [J]. High Indus Sci Technol, 34(8): 41-42. [张洪慎,刘义行,李伟, 2018. 菊叶香藜总生物碱的提取方法探究 [J]. 轻工科技, 34(8): 41-42.]
ZHANG L, GUO H, BAO AK, 2019. The unique salt-secreting structures of halophytes: salt bladders [J]. Acta Phytophysiol Sin, 55(3): 232-240. [张乐,郭欢,包爱科2019. 盐生植物的独特泌盐结构—盐囊泡 [J]. 植物生理學报, 55(3): 232-240.]
ZHANG WW, MENG XP, WAN WX, et al., 2015. Resources for wild plants of Chenopodiaceae in Hebei Province [J]. Hebei J For Orchard Res, 30(4): 419-423. [张薇薇,孟祥普,万五星,等,2015. 河北省藜科(Chenopodiaceae)野生植物资源研究 [J]. 河北林果研究, 30(4): 419-423.]
(责任编辑 周翠鸣)
