基于不同测序技术的微生物群落结构比较
2021-01-05岳正波李玉龙
黄 纷, 岳正波, 李玉龙, 谭 娟, 王 进
(合肥工业大学 资源与环境工程学院,安徽 合肥 230009)
20世纪90年代,不依赖于纯培养的DNA测序技术得到了迅速的推广,变性梯度凝胶电泳(denaturing gradient gel electrophoresis,DGGE)、温度梯度凝胶电泳(temperature gradient gel electrophoresis,TGGE)、DNA克隆文库(clone library,CL)等分子生物学技术得到快速发展,能够直接从分子水平上研究微生物资源,避免了传统微生物分析方法的局限性[1]。DGGE方法在同一条带位置上表示相同的菌种,能直观地反映微生物群落的变化情况。CL技术操作简单,在单个或2个检测样品中具有省时、经济的优点[2]。文献[3]用聚合酶链反应变性梯度凝胶电泳(polymerase chain reaction-denaturing gradient gel electrophoresis,PCR-DGGE)的方法分析合肥市塘西河表层沉积物的细菌群落结构,表征塘西河流域城镇化程度以及生态恢复效果;文献[4]用CL方法分析镇江香醋醋醅中乙偶姻合成途径的关键酶,为解析多菌种醋酸发酵过程中乙偶姻微生物合成机理奠定了研究基础。然而,传统的分子指纹图谱,如DGGE和CL,获得的微生物DNA序列通常低于100条,检测限低、工作量大,故对研究样品量少的污泥来说有较大的局限性。
21世纪以来,新一代高通量测序(high-throughput sequencing,HS)技术快速发展,能够对16S rRNA基因的PCR产物进行直接测序,每次分析获得的基因序列数以百万甚至亿万计,不仅通量高,而且能够同时分析上百个不同的样品,成为解析复杂环境中微生物群落物种组成和相对丰度的重要工具[5]。文献[6]用HS技术分析了乐安江从上游至下游水体细菌群落结构的组成变化。
事实上,HS微生物多样性的基本原理与传统的分子指纹图谱基本一致,两者都以样品DNA作为模板获得微生物群落16S rRNA基因的PCR扩增产物后,开展下游分析[7]。文献[5,8]比较了16S rRNA HS、DGGE、CL在环境微生物分析中的应用效果。关于DGGE、CL及HS之间的差异,相关研究很少。
本文作者所在课题组构建了厌氧-缺氧-好氧(anaerobic-anoxic-oxic, A2/O)反应器系统对垃圾渗滤液和酸性矿山排水进行混合处理。A2/O反应器连续运行450 d后,化学需氧量(chemical oxygen demand,COD)去除率达到98.0%以上,硫酸根(SO42-)去除率约为91.0%,氨氮去除率高达99.6%[8]。本文采集A2/O稳定运行阶段各反应器样品,利用DGGE、CL及16S rRNA HS研究微生物群落多样性,并分析比较了3种分析方法的优缺点。
1 材料与方法
1.1 样品来源
垃圾渗滤液取自安徽省某垃圾填埋场,酸性矿山废水取自某尾矿库,将两者混合并调整其pH值为中性后作为厌氧反应器的进水。A2/O反应器流程如图1所示,进入厌氧反应器的混合调节废水ρ(COD)、ρ(SO42-)分别为(7 500±500)、(2 500±65) mg/L,在稳定运行阶段检测到出水ρ(COD)、ρ(SO42-)分别为(1 089±45)、(580±40) mg/L,去除率分别达到89.0%、76.8%,同时检测到甲烷(CH4)生成量为(280±18) mL。在反应器运行稳定阶段采集厌氧、缺氧、好氧3个反应器单元的泥水混合液进行微生物群落结构分析。

图1 A2/O反应器流程
1.2 污泥样品DNA提取
针对3个反应器的污泥样品,分别称取0.5~1.0 g污泥,重复3次,利用OMEGA公司的E.N.Z.A土壤DNA抽提试剂盒提取DNA。对得到的DNA采用1%琼脂糖电泳,电泳电压100 V,电泳时间45 min,利用紫外凝胶成像系统成像,保存图谱,观察所提取DNA的完整性和浓度,DNA 保存于-20 ℃冰箱待用。
1.3 3种测序方法
3种测序方法的细菌PCR引物见表1所列,古菌PCR引物见表2所列。

表1 细菌PCR引物

表2 古菌PCR引物
(1) DGGE测序。采用传统的巢式PCR,细菌引物为F338-GC/R518,大约的扩增片段为230 bp;古菌引物为F787-GC/R1 059,大约的扩增片段为320 bp。引物由上海生工生物工程技术服务有限公司(简称“上海生工”)合成。细菌PCR扩增程序为:① 94 ℃变性5 min;② 30个循环扩增(94 ℃变性30 s,56 ℃退火60 s,72 ℃延伸 30 s);③ 72 ℃延伸5 min。古菌PCR扩增程序为:① 94 ℃变性4 min;② 31个循环扩增(94 ℃变性30 s,56 ℃退火30 s,72 ℃延伸 30 s); ③ 72 ℃延伸7 min。
按照TaKaRa Taq酶说明书配制PCR反应体系50 μL。 取PCR产物5 μL,与1 μL上样染料混匀,1%琼脂糖凝胶电泳检测,恒压100 V 45 min,紫外凝胶成像系统成像,保存图谱。
采用Bio-Rad公司D-code Universal Mutation System DGGE电泳系统对PCR产物进行电泳分析。具体步骤如下:① 凝胶制备,凝胶均采用质量分数8%,梯度为40%~60%;② 上样及电泳,上样体积25~30 μL,75 V电泳14 h;③ 染胶;④ 使用凝胶成像系统拍照;⑤ 切胶后送上海生工进行克隆测序。
(2) CL构建并测序。CL采用的细菌引物为8F/1 492R, 大约的扩增片段为1 500 bp;古菌引物为21F/958R, 大约的扩增片段为1 500 bp。细菌扩增程序为:① 94 ℃变性5 min;② 31个循环扩增(95 ℃变性35 s,55 ℃退火45 s,72 ℃延伸90 s);③ 72 ℃延伸5 min。古菌扩增程序为:① 94 ℃变性5 min;② 34个循环扩增(95 ℃变性30 s,58 ℃退火40 s,72 ℃延伸60 s);③ 72 ℃延伸10 min。
利用Peasy-T3载体和E.coliDH5α菌株建立16S rDNA文库。克隆菌株经蓝白斑筛选后,选取阳性菌株过夜摇菌培养,菌液送至上海生工测序。测序序列与GenBank中的序列比对,得到该片段的系统发育地位及其分类。通过其分类把所有的克隆子分为多个操作分类单元(operational taxonomic unit, OTU), OTU反映了所分析样品中微生物群落的种类,而每个OTU所占的比例反映了该种类在群落中的相对丰度,从而反映了整个群落的微生物结构。
(3) 16S rRNA HS。对于HS的PCR引物,细菌引物为515F/806R,大约的扩增片段为300 bp;古菌引物为349F/806R,大约的扩增片段为460 bp。使用 Illumina Hiseq2500 平台对构建的扩增子文库进行 PE250 测序(广东美格基因科技有限公司),对测序结果进行基本的质控,去除低质量的数据,然后将得到的序列按照97%的相似度进行聚类,筛选出代表序列,同时比对RDP(Ribosomal Database Project)、Greengene等数据库,得到代表序列的分类信息。
2 结果与讨论
2.1 门水平微生物群落分析
门水平细菌群落组成如图2所示。
在厌氧反应器中,DGGE的测序结果共得到6个门水平的微生物,其中Proteobacteria(34.60%)和Thermotogae(39.00%)是优势菌,其次是Firmicutes(7.70%)、Planctomycetes(7.70%)及Bacteroidetes(6.50%);CL方法共得到4个门水平的微生物,优势菌为Proteobacteria(33.82%)和Firmicutes(62.5%),其次是Planctomycetes(2.20%)和Thermotogae(1.47%);16S rRNA HS共检测到8个门水平微生物,其中Firmicutes(23.91%)、Proteobacteria(23.54%)及Thermi(29.70%)为优势菌,此外还检测到了Bacteroidetes(5.84%)、Planctomycetes(8.10%)、Acidobacteria(3.362%)及Chloroflexi(2.08%)。
缺氧反应器中,DGGE测序共得到5个门水平的微生物,分别为Proteobacteria(48.50%)、Thermotogae(18.60%)、Bacteroidetes(15.50%)、Spirochaetes(11.10%)及Planctomycetes(6.30%);CL方法共得到2个门水平的微生物,分别为Proteobacteria(58.82%)和Bacteroidetes(41.18%);16S rRNA HS共检测到10个门水平微生物,包括DGGE和CL检测到的所有门类,此外还有Actinobacteria(2.91%)、Chloroflexi(8.12%)及Firmicutes(14.32%)、Synergistetes(1.32%)。
好氧反应器的门水平微生物优势菌种类用3种方法测得的结果与厌氧和缺氧反应器的结果一致。
由此可知,DGGE和CL技术都能检测到污泥样品中的优势微生物,两者结合分析能更全面地概括菌种信息,而HS技术在读取序列方面具有信息量大的优点,不仅能将DGGE和CL技术分析到的微生物检测到,还能检测到丰度较低的物种,因此运用HS技术能更加全面地获得物种信息。

图2 门水平细菌群落组成
2.2 属水平厌氧反应器微生物群落分析
厌氧反应器中微生物的作用是COD去除的主要途径,硫酸盐还原菌(sulfate-reducing bacteria,SRB)将SO42-还原为硫化物,能与酸性矿山废水中的铜、铁等金属离子形成硫化物沉淀从而固定在污泥中。属水平厌氧反应器细菌和古菌微生物群落组成如图3所示。用3种测序方法分析厌氧反应器中细菌和古菌的群落组成。
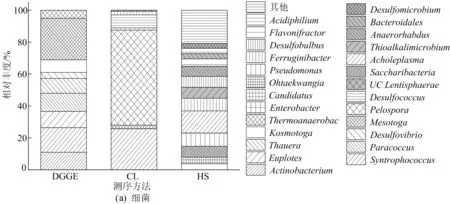
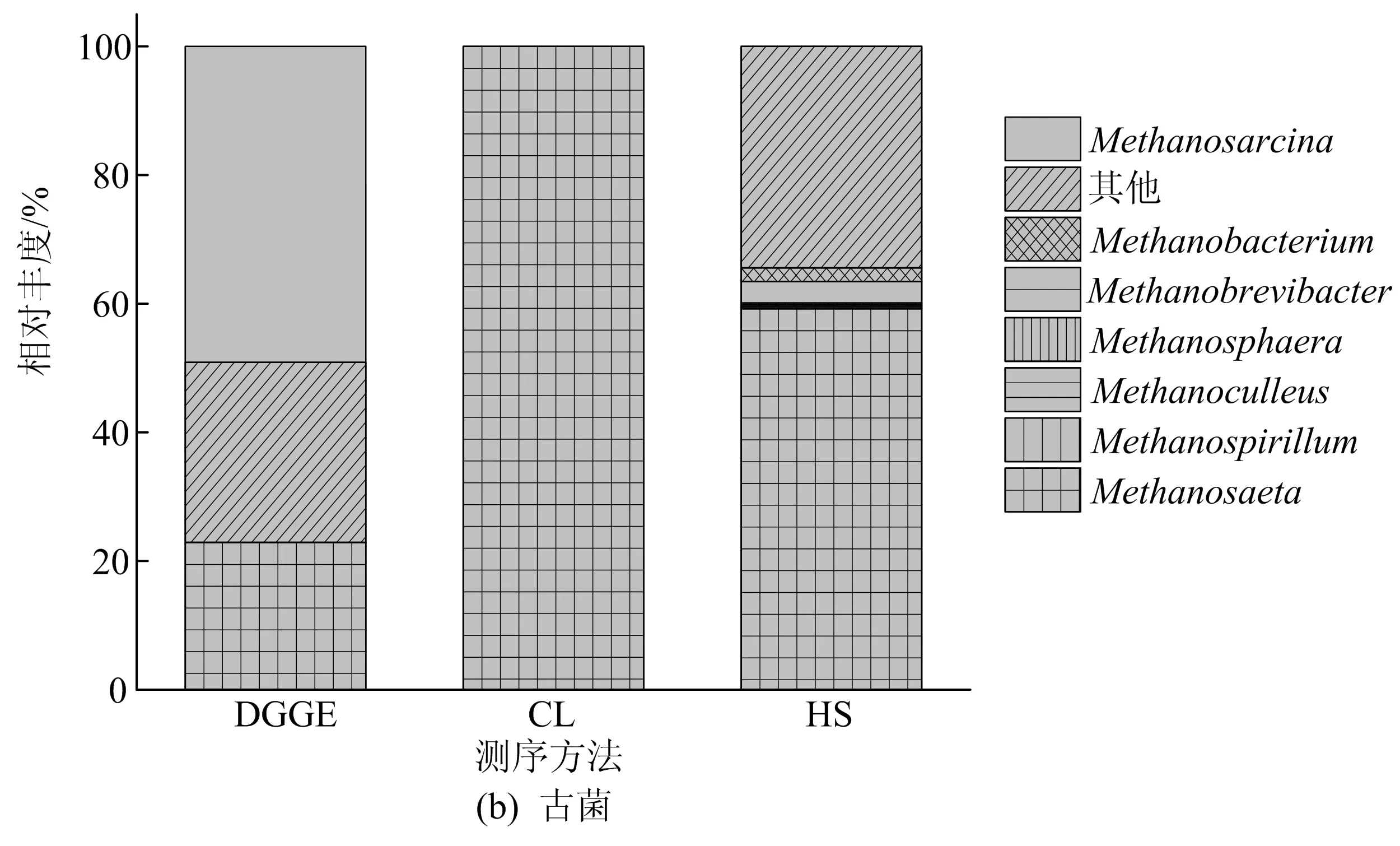
图3 属水平厌氧反应器细菌和古菌群落组成
2.2.1 DGGE测序的细菌与古菌群落结构
由图3a所示厌氧反应器中细菌的测序结果可知:
(1)Mostoga相对丰度为26.00%。该属细菌严格厌氧,生长周期较长,并可以将碳水化合物和蛋白质类物质用于自身菌体生长[9]。
(2)Kosmotoga、Actinobacterium、Euplotes相对丰度分别为7.90%、4.50%、10.20%。该属细菌具有水解发酵功能,主要发酵产物为H2、CO2、乙酸及少量的乙醇和丙酸[10]。
(3)Syntrophococcus相对丰度为7.70%。该属细菌是一种厌氧产酸细菌[11],能将Mostoga等水解菌产生的小分子有机物质进一步分解为乙酸、CO2等。
(4) SRB菌Desulfovibrio相对丰度为3.70%。该属细菌能利用产酸菌产生的H2、CO2等将SO42-还原为硫化物。
由图3b所示厌氧反应器中古菌的测序结果可知,2种产甲烷古菌Methanosaeta和Methanosarcina相对丰度分别为22.90%、49.10%。这2种古菌均能利用乙酸产生CH4。
2.2.2 CL测序的细菌与古菌群落结构
通过CL技术检测到多种微生物,包括Proteobacteria门的大量细菌(如Pelospora属、Syntrophococcus属)以及Desulfococcus属。
(1)Pelospora(59.56%)、Syntrophococcus(25.74%)可以降解多种复杂的有机物。
(2)Desulfococcus(8.09%)是典型的SRB菌。
(3)Candidatus(2.21%)具有厌氧氨氧化功能,是广为人知的厌氧氨氧化菌。
(4)Methanosaeta是检测到的唯一一种产甲烷古菌。
在厌氧反应器中检测到的菌种和其中进水水质情况基本相符,且达到了一定的去除率。
DGGE和CL方法检测到的微生物互为补充,能大致反映厌氧反应器中的微生物群落组成。
2.2.3 HS测序的细菌与古菌群落结构
由16S rRNA HS可知:
(1)Pseudomonas、Kosmotoga相对丰度分别为8.22%、3.76%。该菌属为水解细菌,能够使大分子复杂有机物分解成易被功能微生物利用的小分子物质。
(2)Acholeplasma、Ferruginibacter、Flavonifractor及Saccharibacteria相对丰度分别为13.89%、7.92%、4.61%、6.91%。该菌属为发酵产酸细菌,产物主要有甲酸、乙酸等[12-13]。
(3)Desulfobulbus与Desulfomicrobium相对丰度分别为6.84%、3.01%。该菌属是SRB菌,能利用有机物作为电子供体将废水中的硫酸盐还原成硫代硫酸盐等中间产物和硫化物。
(4) 产甲烷古菌主要有Methanosaeta(59.21%)、Methanobacterium(2.11%)及Methanobrevibacter(3.32%)。
2.2.4 3种方法测序结果分析
垃圾浸出液与矿山废水混合后有助于强化微生物间的协同作用并促进污染物的去除。如在水解发酵菌Mostoga、Kosmotoga、Pelospora及Syntrophococcus属的作用下,不溶性有机物、复杂有机物分解为小分子的有机物,其中一部分被产甲烷菌作为底物产CH4。Methanosaeta是3种方法检测到的优势菌种,因此在厌氧反应器中以乙酸型产甲烷菌为主导。这些微生物的种属组成和数量分布决定了反应器的运行性能。另外一部分有机物则被SRB菌利用,DGGE检测到的SRB菌为Desulfovibrio(3.70%),CL检测到的SRB菌为Desulfococcus(8.09%),而HS检测到的SRB菌为Desulfobulbus(6.84%)和Desulfomicrobium(3.01%),由此可知,针对相对丰度低的菌种(SRB菌),3种分子生物学方法可结合分析,能更加准确地反映污泥样品的微生物群落组成。厌氧反应器中存在的SRB将SO42-还原为硫化物,混合液中的铜、铁等金属离子可与硫化物形成沉淀,因而为重金属离子和硫酸根离子的去除提供了一种生物处理法。
2.3 属水平缺氧反应器微生物群落分析
根据图1可知,缺氧反应器进水是由厌氧反应器出水与好氧反应器回流液混合组成,进水的氨氮质量浓度为83~100 mg/L,在有机碳源的条件下进行反硝化作用,同时去除部分COD。缺氧反应器中硫酸盐的降幅很小,可忽略不计。
缺氧反应器属水平细菌群落组成如图4所示。
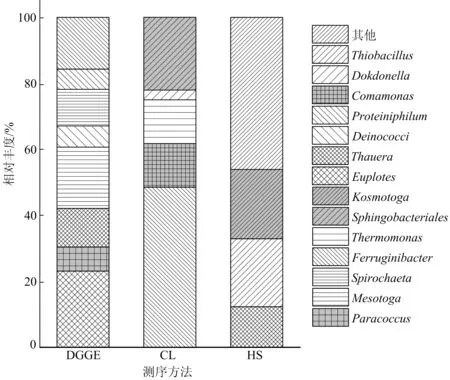
图4 缺氧反应器属水平细菌群落组成
(1) 缺氧反应器中PCR-DGGE方法结果。Bacteroidetes门Proteiniphilum属的相对丰度为6.00%,Thermotogae门Mesotoga属的相对丰度为18.60%,Spirochaetes门的相对丰度为12.90%。还存在部分兼性细菌,如Proteobacteria门的Deinococci属,Bacteroidetes门的Ferruginibacter属,但其总量只占总菌群的15.80%。反硝化细菌Thauera(11.70%)和Paracoccus(7.30%),在硝酸盐作为受体存在时可以进行厌氧生长并具有反硝化功能[14]。
(2) CL分析结果。缺氧反应器检测到Sphingobacteriales(22.06%)、Ferruginibacter(19.12%)、Comamonas(13.24%)及Thermomonas(13.24%)。Ferruginibacter也具有水解有机物的作用,是缺氧反应器中COD去除的承担者。Comamonas有反硝化功能,在文献[15]的硝化-反硝化反应器中也发现了一定量的Comamonas属细菌。
(3) 16S rRNA HS结果。Thauera(12.29%)、Thiobacillus(20.61%)及Kosmotoga(21.01%)为缺氧反应器中的优势菌种。Thauera和Thiobacillus已被研究证实具有异养反硝化功能,因此可认为是反硝化脱氮的主要微生物;推测Kosmotoga为缺氧反应器水解有机物质的主要承担者。缺氧反应器可以代谢氮的细菌主要为Comamonas、Thauera、Paracoccus、Thiobacillus及Pseudomnas,可以降解有机物的微生物有Mesotoga、Ferruginibacter及Kosmotoga。
由此可知,对于缺氧反应器中微生物群落组成,用3种方法分析能更加全面地掌握微生物的信息;对废水进行反硝化脱氮处理可以富集Comamonas和Thauera等细菌,提高反硝化反应器的功能。
2.4 属水平好氧反应器微生物群落分析
好氧反应器出水ρ(COD)稳定在85 mg/L,整个A2/O反应器中COD的去除率达到98.0%;好氧反应器通过活性污泥在曝气的作用下将氨氮氧化成硝态氮,完成硝化作用,最终出水氨氮质量浓度为(1.01±0.36) mg/L,去除率达到99.0%。
好氧反应器属水平细菌群落组成如图5所示。
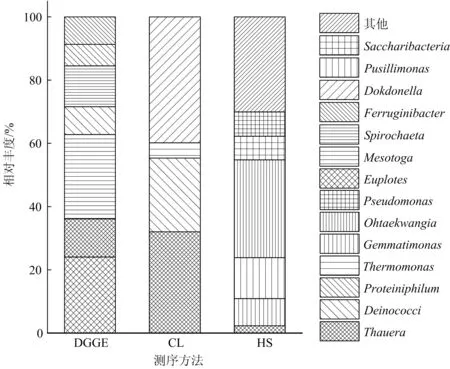
图5 好氧反应器属水平细菌群落组成
(1) 好氧反应器DGGE测序得到的细菌群落组成与缺氧反应器基本相同。
(2) 通过CL技术,在好氧反应器中检测到Dokdonella(39.81%)、Deinococci(23.30%)及Thauera(8.74%)。Dokdonella、Thauera及Deinococci属都有水解有机物的作用。
(3) 16S rRNA HS结果表明,好氧反应器中主要是好氧微生物,如Pseudomnas(7.67%)、Gemmatimonas(8.59%)、Pusillimonas(12.94%)及Ohtaekwangia(30.94%)。Pseudomnas能以硫酸铵作为氮源进行生长,Gemmatimonas是生物除磷固氮过程中的重要菌属。
好氧反应器中大部分为好氧微生物,如Dokdonella、Deinococci及Ohtaekwangia;还有一部分兼性厌氧菌,其主要功能是去除残留的COD;代谢氮的细菌为Pseudomnas及Thauera,这2种细菌可能参与好氧条件下的硝化作用。
2.5 3种测序方法比较
3种分子生物学方法检测结果表明,Proteobacteria门、Firmicutes门、Bacteroidetes门等微生物为主要优势菌群,还原硫酸盐的种群主要包括Proteobacteria门。CL和HS方法同时检测到厌氧反应器污泥样品中含有具有厌氧氨氧化功能的Planctomycetes门细菌,HS还检测到了Thermi和Chloroflexi等门类微生物。DGGE和CL都能检测到污泥样品中的优势微生物,两者结合分析能更全面地概括菌种信息,而HS还能检测到丰度较低的门类,因此HS能更加全面地反映物种信息。
厌氧反应器降解复杂有机物的菌种为Mostoga、Kosmotoga、Pelospora及Syntrophococcus属等,硫酸盐还原菌主要为Desulfovibrio、Desulfococcus、Desulfobulbus及Desulfomicrobium,产甲烷古菌主要是Methanosaeta。细菌分析中,HS比DGGE多检测出了21.42%,比CL多检测出了50.00%;古菌分析中,HS比DGGE多检测出了50.00%,比CL多检测出了83.00%。由此可知,HS相比于其他2种方法灵敏度较高,HS能够较为全面和准确地反映反应器污泥微生物群落结构;而DGGE和CL仅能够反映有限的优势微生物类群,一定程度上可能低估反应器中微生物的物种组成并高估其丰度。因此,在属水平上HS与其他2种方法所测到的物种数相差较大。
在缺氧反应器和好氧反应器中,3种方法测得的微生物种类数目差别不大,DGGE和CL分析得到的反硝化菌分别为Comamonas和Thauera,HS分析得到的反硝化功能细菌为Thiobacillus及Pseudomnas,缺氧-好氧反应器氨氮去除率达到99.0%,水解微生物为Mesotoga、Ferruginibacter及Kosmotoga,因此缺氧-好氧反应器的功能是去除残留部分的COD和氨氮。
综上所述,3种测序方法结合分析能更加真实地反映污泥样品的微生物群落组成。
3 结 论
(1) 厌氧反应器中主要的功能细菌是水解发酵菌和硫酸盐还原菌。前者主要为Mostoga、Kosmotoga、Pelospora及Syntrophococcus属;后者主要为Desulfovibrio、Desulfococcus、Desulfobulbus及Desulfomicrobium属;缺氧和好氧反应器中可以代谢氮的细菌主要为Comamonas、Thauera、Thiobacillus及Pseudomnas属。
(2) 厌氧反应器细菌分析中HS比DGGE和CL分别多检测出21.42%、50.00%;古菌分析中HS比DGGE和CL分别多检测出50.00%、83.00%。HS相比于其他2种方法灵敏度较高,能更全面和准确地反映反应器污泥微生物群落结构。
(3) 宏观上A2/O反应器的处理功能与微观上微生物的功能菌群有很强的相关关系。厌氧反应器中可富集Mostoga等细菌,达到去除有机物的目的;缺氧和好氧反应器中可富集Thauera等菌种进行硝化和反硝化作用。
(4) CL操作简单,适用于样品量少的情况;DGGE适用于样品较多的情况,它能直观地反映微生物群落的变化情况。对于相对丰度低的菌种,3种方法结合分析能更加真实地反映污泥样品的微生物群落组成。
