hsa-miR-221调控非小细胞肺癌分子网络的生物信息学分析
2020-12-31李璟波周少华李少军
李璟波,周少华,夏 晖,李少军,刘 阳
肺癌是一种常见的肺部恶性肿瘤,是目前对人类健康和生命威胁最严重的肿瘤之一。非小细胞肺癌(non-small cell lung carcinoma,NSCLC)约占肺癌总数的80%以上,是肺癌最常见的组织学类型[1]。miRNA是近年来发现的一类非编码小分子RNA,其主要通过与靶基因3'UTR的完全或不完全配对,降解靶基因mRNA或抑制其翻译,从而参与调控个体发育、细胞凋亡、增殖及分化等生命活动。余东虎等[2]运用生物信息学方法研究肺腺癌相关的靶基因,miRNA的异常表达与肿瘤的发生和发展也有重要联系。多种miRNA包括miR-486、miR-30d、miR-499、miR-137、miR-375、miR-182*、miR-221和let-7a等的异常表达与NSCLC的发生和发展有关[3-6]。
hsa-miR-221最初从斑马鱼中克隆获得并得到证实,其与多种肿瘤存在密切关系,包括肝癌、胶质母细胞瘤、前列腺癌、甲状腺乳头状癌和NSCLC等[7]。有研究表明,在肺癌组织中hsa-miR-221表达上调,可以通过靶向抑制肿瘤抑制基因TIMP3的表达促进NSCLC的生长[8]。吕俊杰等[9]研究了117例NSCLC患者的hsa-miR-221表达情况,发现hsa-miR-221高表达的患者预后较差,可以作为一个潜在的肺癌预后预测的标志物。另外,在HBEC4肺癌细胞系中,hsa-miR-221能诱导上皮细胞间充质细胞转换(EMT)[10]。越来越多的证据表明hsa-miR-221在NSCLC的发生和发展中起重要作用,但其具体的调控机制尚不明确。本研究通过运用生物信息学的方法,研究hsa-miR-221上游转录因子、下游调控基因及其与长链非编码RNA(long non-coding RNA,lncRNA)之间的调控关系,推测其在NSCLC的发生与发展中的分子调控网络,为进一步研究其调控NSCLC的分子机制提供线索。
1 材料与方法
1.1 材料 生物信息学预测工具软件加州大学圣克鲁斯分校(University of California,Santa Cruz,UCSC)基因组浏览器(http://genome.UCSC.edu/);人类miRNA疾病数据库(http://202.38.125.151/hmdd/mima/md/);TF-miRNA调控数据库(http://202.38.126.151/hmdd/mima/ttY);miRNA靶基因预测验证数据(http://www.ulnm.uni-heidelberg.de/apps/zm/mirwalk/micronapredietedtarget.htm1);Genecards在线数据库(http://www.geneeards.org/index.php?path=/HTML/page/gcPeople);肺癌相关因子整合数据库IHLDB.rf(http://www.lungcancerdatabase.com/);ConSite数据库(http://consite.genereg.net)。美国国立生物技术中心的基因表达谱(GEO)数据库(https://www.ncbi.nlm.nih.gov/geo/)。
1.2 方法
1.2.1 分析miR-221在人类基因组中的位置、保守性及相关疾病 UCSC基因组在线浏览工具包含有人类及动物的基因组相关数据。采用UCSC分析hsa-miR-221在人类基因组中的位置及保守性,在人类基因组hg19版本中得到hsa-miR-221的注释信息。人类miRNA疾病数据库(the Human microRNA Disease Database,HMDD)通过对遗传学、表观遗传学、循环miRNA和miRNA与靶基因相互作用分析得到miRNA与疾病的关系,目前其版本为v3.2更新版,在其中查询与has-miR-221相关的疾病。
1.2.2 预测hsa-miR-221转录因子 利用TransmiR在线预测hsa-miR-221的转录因子。
1.2.3 查询hsa-miR-221靶基因 MiRwalk综合了多种miRNA靶基因预测软件的结果,提供来自人类、小鼠和大鼠的miRNA的预测信息,利用该预测软件预测hsa-miR-221靶基因。
1.2.4 筛选与NSCLC相关的hsa-miR-221靶基因 在Genecards数据库中查找已证实与NSCLC相关的基因,并与预测软件预测得到的结果取交集筛选出与NSCLC相关的hsa-miR-221靶基因。
1.2.5 搜索与NSCLC相关的lncRNA 肺癌相关因子整合数据库IHLDB.rf收录了69种与肺癌相关的lncRNA,并提供了536对lncRNA与miRNA的相互作用。因此在该数据库中找到与NSCLC相关的lncRNA并筛选出于hsa-miR-221相互作用的lncRNA。
1.2.6 预测lncRNA转录因子 ConSite是两序列间保守区域转录因子结合部位预测软件,运用ConSite软件输入前面预测所得lncRNA的启动子序列,预测得该lncRNA的转录因子,汇总并查阅相关文献,最后筛选出与NSCLC相关的lncRNA的转录因子。
1.2.7 推测hsa-miR-221在NSCLC中分子调控网络 综合上述结果,分析各基因间的关系,绘制hsa-miR-221在NSCLC中的分子调控网络图。
1.2.8 验证hsa-miR-221在NSCLC中的作用 为了评估hsa-miR-221在肺癌中的作用,本研究研究了hsa-miR-221过表达对肺癌细胞系影响。通过R软件及affy和ggplot2等软件进行差异基因分析。选择差异倍数2倍(|logFC|≥1)以上,P<0.05的差异基因找出差异基因,进而与推测hsa-miR-221在NSCLC中分子调控网络中相关基因进行关联,进一步确认hsa-miR-221调控的主要基因,对这些主要基因进行GO及KEGG功能注释。
2 结果
2.1 hsa-miR-221在人类基因组中的位置、保守性及相关疾病 根据人类基因组hg19的结果发现has-miR-221定位于人类第X号染色体上,处在P11.3区域的染色体45 605 584~45 605 693 bp位置,长度共107 bp,其在人、大鼠、小鼠、大象、狗、鸡和斑马鱼等动物中呈高度保守,图1。
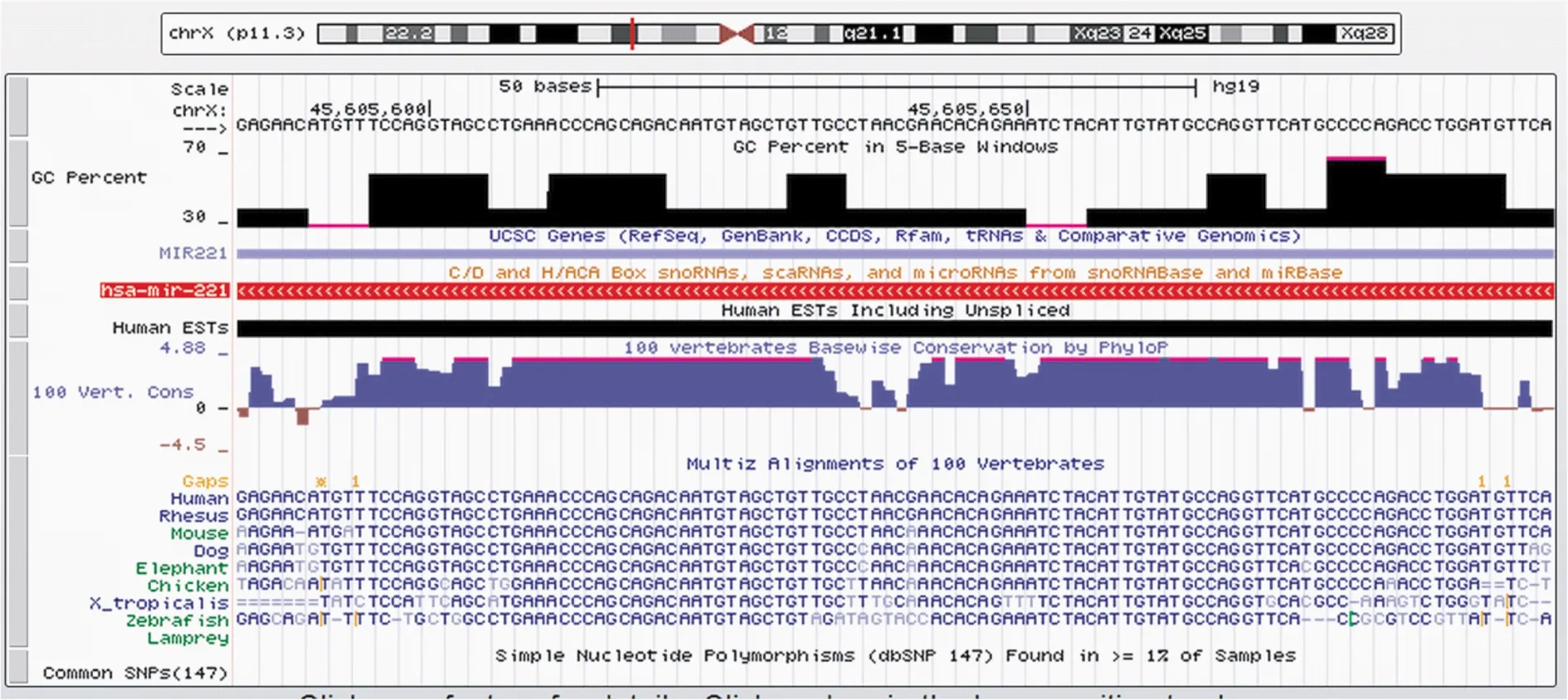
图1 hsa-miR-122在人类基因组中的位置
对HMDD分析发现,hsa-miR-221与多种疾病相关,包括多种肿瘤、心血管疾病以及哮喘和秃头症等(表1)。在肿瘤疾病中包括NSCLC,暗示其可能与NSCLC的发病机制有关。

表1 HMDD中与hsa-miR-221相关的疾病
2.2 hsa-miR-221的转录因子 运用TransmiR软件预测的hsa-miR-221的转录因子,发现其启动子上游可以被多种转录因子如MYC、MYCN、PDGF、ERS1、SF2/ASF、p27和FOS1所识别。其中MYC、MYCN、PDGF、ERS1、SF2/ASF、p27被证实与NSCLC有关。例如,C-MYC被证实与NSCLC的转移有重要联系[11]。MYCN(V-myc avian myelocytomatosis viral oncogene neuroblastoma derived homolog)是一个癌基因,在多种恶性肿瘤中过表达,其过量表达也与NSCLC的增殖有关[12]。
2.3 hsa-miR-221的靶基因 用miRwalk软件预测hsa-miR-221的靶基因为817个。在GeneCards数据库中查找与NSCLC相关的基因共86个,对2个数据库进行比较取交集,得到26个基因。其中包括调节细胞周期的相关酶类CDK2、CDK6、CDKN1B和CDKN2B等。同时包括转录因子E2F2和E2F3以及多种整合素基因。也包含磷酸肌醇3-激酶(phosphoinositide 3-kinase,PIK3)家族基因PIK3CD、PIK3CG和PIK3R1等。另外包含原癌基因BCL-2和抑癌基因PTEN和RB1等。
2.4 hsa-miR-221与lncRNA 利用肺癌整合因子数据库IHLDB.rf查找与肺癌相关的lncRNAs,发现有GAS5、CCAT1、H19、HOTAIR、MALAT1、MEG3、PVT1、TP73-AS1、TUG1和XIST共10个lncRNAs与miRNA有相互作用的数据,这10个lncRNA都与NSCLC有关。继续在IHLDB.rf数据库中查找发现,GAS5、HOTAIR和TUG1预测的靶miRNA包含hsa-miR-221,推测这3个lncRNA与hsa-miR-221的相互调控在NSCLC发生发展中起着重要作用。
2.5 预测lncRNA的转录因子 运用ConSite软件预测上游1 kb启动子序列可能与lncRNAGAS5、HOTAIR和TUG1的结合的转录因子,采用cutoff值为95%。在GAS5中预测得到5个转录因子可以与之结合,HOTAIR中有5个转录因子可以与之结合,TUG1中有5个转录因子可以与之结合(图2)。其中3个lncRNA都可以被SOX-5识别。全基因组芯片分析发现,在NSCLC组织中SOX-5的基因组区域发生了较大改变[13]。E74A可以识别GAS5和TUG1,其与NSCLC的关系尚不清楚。其它转录因子如Snail、n-MYC、ARNT均与NSCLC相关[13]。

A.GAS5
2.6 hsa-miR-221在NSCLC中的分子作用机制网络图 根据以上结果,综合识别hsa-miR-221启动子的MYC、MYCN和PDGF等6个上游转录因子及其下游靶基因PTEN、RB1和BCL2等,和预测所得的与其相关的lncRNA GAS5、HOTAIR和TUG1及它们的转录因子Snail、n-MYC、ARNT和SOX5,这些因子均已证实与NSCLC相关,最后可以推测得hsa-miR-221在NSCLC中的分子调控网络图。hsa-miR-221在表达过程中受到多种途径的调控,在每个途径中都有多种因子的参与,暗示其在调控NSCLC的发生和发展时处于一个十分复杂的分子调控网络中,图3。
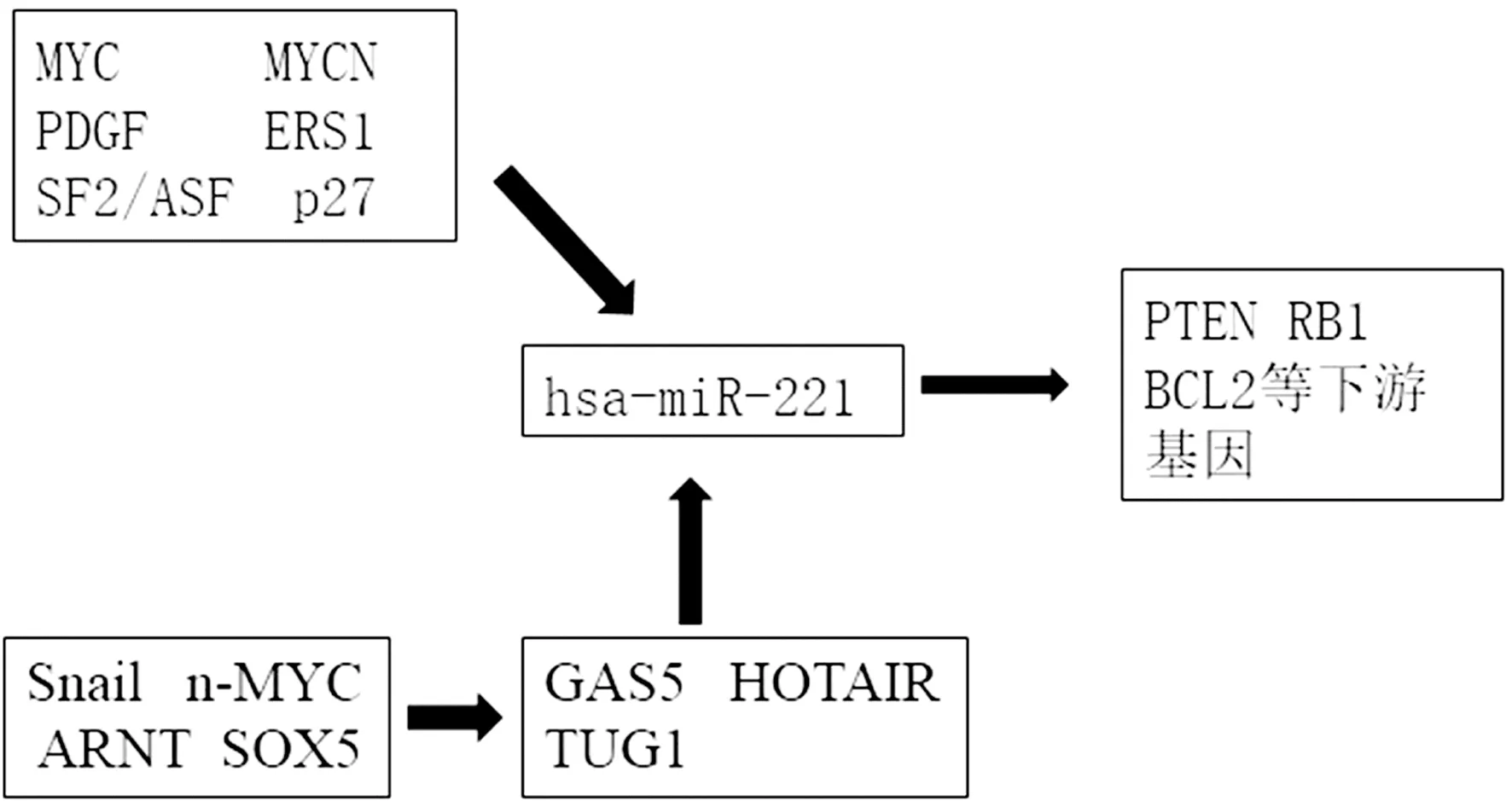
图3 hsa-miR-221在NSCLC组织中表达的分子调控网络示意图
2.7 hsa-miR-221过表达转录组差异基因分析 根据筛选标准筛选出364个差异基因,其中上调274个,下调90个,图4A。
将差异基因与预测的hsa-miR-221在NSCLC组织中表达的分子调控网络中的基因取交集,最终确定5个基因,这5个基因分别为APAF1、COL4A4、ITGA6、CCNE2、E2F2,图4B。GO功能注释发现这些基因主要调控DNA分解代谢过程、凋亡信号通路的调控、DNA复制起始及凋亡执行阶段的调控等功能。KEGG分析发现主要调控细胞周期信号通路、P53信号通路及PI3K-AKT信号通路等。
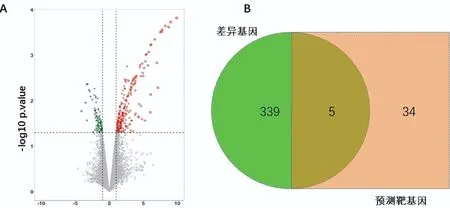
图4 A:hsa-miR-221在NSCLC细胞系中过表达差异
3 讨论
随着研究技术的发展,越来越多的数据表明miRNA在肿瘤的发生和发展中起重要作用。NSCLC是肺癌的主要类型,受多种miRNA的调控,因此研究NSCLC中miRNA的调控网络对阐明癌症发生和发展的分子机制具有十分重要的意义。应用生物信息学手段,通过预测miRNA的调控因子、靶基因及其相互作用的lncRNA和相关的转录因子,张皓旻等[14]研究了kras基因突变对人肺腺癌转录组的影响,韩泽平等[15]绘制了hsa-miR-221在肝癌中的分子调控网络,吕燕妮等[16]绘制了hsa-miR-181a在脑卒中发病中的分子调控网络,明确了这些miRNA在疾病发生中的重要调控作用。
最近研究表明,hsa-miR-221在NSCLC中起重要作用,然而其具体机制并不清楚,因此本研究用生物信息学的方法预测其在NSCLC中的作用。hsa-miR-221在脊椎动物甚至斑马鱼中保守,暗示其有十分重要的意义。其与多种癌症包括NSCLC的发生发展有关。通过预测启动子的转录因子结合发现,其可以被多种与NSCLC相关的转录因子所识别,其中MYC和MYCN都被证实与NSCLC的转移和增殖有重要联系。在受到与NSCLC相关的转录因子调节的同时,hsa-miR-221也可以结合到许多与NSCLC相关的基因上,调节它们的表达。结合miRWalk预测得到的靶基因及Genecards数据库中与NSCLC相关的基因,发现其下游有26个基因与NSCLC有关,包括PTEN、RB1和BCL2等。hsa-miR-221的升高能抑制PTEN的表达[8]。在NSCLC中,抑癌基因PTEN的表达受到抑制,PTEN表达的抑制与NSCLC患者的预后较差有很重要的联系[17]。
同时,本研究还发现NSCLC中,hsa-miR-221的表达还受到lncRNA的调节,这些lncRNA已被证实与NSCLC的发生和发展有重要关系,包括GAS5、HOTAIR和TUG1。这些lncRNA自己也受到多种与NSCLC相关的转录因子的调控。因此通过上游转录因子,lncRNA以及下游靶基因,构成了NSCLC中以hsa-miR-221为核心的分子调控网络。在每个步骤中都有多种分子的参与,暗示了该调控网络十分复杂,hsa-miR-221可以通过多种途径对NSCLC的发生和发展起调控作用。
通过以上生物信息学的分析,本研究绘制了hsa-miR-221调控NSCLC的分子调控网络,为以后进行相关的实验研究提供了十分重要的线索,同时为阐明其在NSCLC中的发病机制提供了新的理论依据。
