基于CRISPR/Cas9系统的拟南芥ugt84a1/ugt84a2双突变体制作及突变位点分析
2020-12-14李星坤潘慧李攀
李星坤 潘慧 李攀

摘要:CRISPR/Cas9基因编辑技术相对于锌指核酸酶(ZFNs)和转录激活因子效应物核酸酶(TALENs)而言,具有操作简便、突变效率高等优点,已经被广泛应用于医学、动物科学、植物科学等领域。拟南芥糖基转移酶UGT84A1、UGT84A2参与植物次生代谢及外源毒物反应,并且为同工酶。本研究以拟南芥糖基转移酶同工酶基因UGT84A1和UGT84A2为靶向基因,构建CRISPR/Cas9双突变体表达载体,并转化到农杆菌浸染拟南芥,从而同时定向敲除靶向基因。根据拟南芥转基因后代的测序结果,对获得的42株阳性转化植株进行突变位点分析,结果表明,有2株阳性植株发生双突变,由此成功构建了ugt84a1/ugt84a2双突变体。试验结果可为加快ugt84a1/ugt84a2功能基因资源的开发利用提供有力的理论与方法支持。
关键词:拟南芥;双突变体;CRISPR/Cas9;基因敲除;位点分析
中图分类号:S184 文献标志码: A 文章编号:1002-1302(2020)20-0049-07
植物体内小分子的糖基化修饰是一种普遍存在的生理现象,并且是植物细胞维持体内代谢平衡的主要机制之一[1-2]。研究表明,糖基化修饰参与调节植物生长发育的各个方面。植物中最早被鉴定的糖基转移酶基因是诺贝尔奖获得者McClintoc于1997年发现的控制玉米种子黑色素沉积的Bronze1基因,后证实该基因编码产生黄酮糖苷的糖基转移酶。Fraissinet-Tachet等研究番茄的防御机制时发现,糖基转移酶基因Twi1的表达能够使植株对病原菌侵染做出快速应答[3]。Jackson等通过对拟南芥中糖基转移酶UGT84B1的研究,发现其对生长素吲哚乙酸有活性,过表达后植物莲座叶数目增加[4]。Tognetti等鉴定了拟南芥生长素糖基转移酶UGT74E2,研究其过表达体表型发现UGT74E2参与调节植物的形态建成和干旱、盐胁迫的应答[5]。Wang等研究表明,UGT87A2参与调控拟南芥开花途径进而影响开花时间[6],这些研究表明,糖基化修饰参与植物生长发育的诸多方面。
而针对同一小分子的糖基化修饰,往往需要几个不同的糖基转移酶基因。例如,在拟南芥糖基化修饰研究中,Hou等通过离体生物化学分析发现,拟南芥细胞分裂素糖基转移酶是由5个不同的成员组成,包括催化细胞分裂素N-糖基化的糖基转移酶UGT76C1和UGT76C2,以及催化细胞分裂素O-糖基化的糖基转移酶UGT85A1、UGT73C5和UGT73C1[7]。Poppenberger等在拟南芥中发现,UGT73C5可以催化油菜素内酯形成23-O-油菜素内酯糖苷物[8],随后他们又发现UGT73C6同样能够糖基化修饰油菜素内酯[9]。此外,拟南芥中已证明的生长素IAA的糖基转移酶包括UGT84B1和UGT74D1这2个成员[10-11],而水杨酸糖基转移酶包括UGT74F1和UGT74F2这2个成员[12]。在玉米糖基化修饰研究中,同样发现针对同一激素存在多个糖基化修饰的同工酶。例如,玉米中细胞分裂素包括cis-ZOG1和cis-ZOGT2这2个成员[13-14]。在水稻中,细胞分裂素糖基转移酶则包括cZOGT1、cZOGT2、cZOGT3这3个成员[15]。这些同工酶的存在也导致了基因的功能冗余,单单敲除某个基因往往不会使植株出现明显表型。
体外试验表明,拟南芥糖基转移酶UGT84A1、UGT84A2都能够作用于外源毒物2,4,5-三氯苯酚[16],Meiner等通过体外试验证明拟南芥糖基转移酶UGT84A1、UGT84A2同时对羟基肉桂酸具有活性[17],表明拟南芥糖基转移酶UGT84A1、UGT84A2与植物脱毒反应及植物的次生代谢有关,并且为同工酶。目前对拟南芥糖基转移酶ugt84a1、ugt84a2仅仅停留在单突变体植株的研究,但是随着基因编辑技术的飞速发展以及生物信息学的广泛应用,对拟南芥糖基转移酶ugt84a1/ugt84a2双突变体的构建已经成为今后科研工作者所必须的工作。锌指核酸酶(zinc-finger nucleases,ZFN)技术[18]、类转录激活因子效应核酸(transcription activitor-like effector nucleases,TALEN)技术[19]以及全新的人工核酸酶CRISPR/Cas9(clustered regulatory interspaced short plindromic repeat/Cas9)系统[20]是目前被广泛应用的三大基因编辑技术。其中CRISPR/Cas9系统由于制作成本低、操作简便,只需合成目标基因的sgRNA靶向序列就能实现对基因特定位点进行特异性编辑,因此更广泛地运用于分子生物学试验中,这一技术短短几年就广泛应用于世界各地各个领域的分子实验。
本研究是利用最新的CRISPR/Cas9基因编辑技术,同时对拟南芥糖基转移酶ugt84a1/ugt84a2进行雙位点的定向敲除,使糖基转移酶的同工基因同时突变,从而获得ugt84a1/ugt84a2双位点突变体植株,并详细分析了其突变位点,为验证该糖基转移酶在体内发挥的功能奠定基础,为加快ugt84a1/ugt84a2功能基因资源的开发利用提供有力的理论与方法支持。
1 材料与方法
1.1 试验材料
野生型拟南芥(Arabidopsis thaliana)及突变体株系种植于临沂大学药学院药用植物实验室植物培养室,温度(22±2) ℃,光照度100 μmol/(m2·s)。CPISPR/Cas9载体,购于百格基因科技(江苏)有限公司,大肠杆菌E. coli DH5α感受态细胞、农杆菌GV3101感受态细胞、T4 DNA Ligase试剂盒、DNA回收试剂盒、质粒提取试剂盒、Taq酶、DNA Maker、PCR耗材等材料与试剂为笔者所在实验室现有保存。gRNA引物由生工生物工程(上海)股份有限公司合成,基因序列由铂尚生物技术(上海)有限公司测定,试验开展时间为2018年9月至2020年1月。
1.2 gRNA引物设计
从拟南芥信息资源网站(The Arabidopsis Information Resource,TAIR)查找到已公布的拟南芥ugt84a1和ugt84a2的基因组序列号分别为AT4G15480和AT3G21560。利用sgRNA设计软件分别设计拟南芥ugt84a1和ugt84a2的sgRNA靶点序列:2个都按照正向引物5′-TGATT+正向靶点序列进行序列的合成,按照反向引物5′-AAAC+反向靶点序列进行序列的合成。
1.3 CPRSPR/Cas9载体的构建
首先根据ugt84a1、ugt84a2靶点序列制备Oligo二聚体(表1)。加入Anneal Buffer 18 μL、SG6977-UP(10 μmol/L)1 μL、SG6977-LW(10 μmol/L) 1 μL,先在95 ℃条件下变性3 min,然后以约 0.2 ℃/s 缓慢降至20 ℃进行退火。另一个Oligo二聚体同法可得。
将Oligo二聚体与CRISPR/Cas载体连接。加入Vector-016B(线性化载体)2 μL、Enzyme Mix 1 μL、Oligo二聚体1 μL、ddH2O 6 μL,在25 ℃条件下反应1 h。取5 μL上述反应液,加入20 μL DH5α感受态细胞,通过42 ℃热激法转入到DH5α。转化子通过引物PUV4-R和PUV3-F(表1)进行菌落PCR验证,验证正确的阳性转化子再进行摇菌,提取质粒后送铂尚生物技术(上海)有限公司进行测序。将验证100%正确的阳性转化子放于 -80 ℃ 保存。取10 μL验证正确的质粒转入农杆菌GV3101感受态细胞中,涂布于含有50 μg/mL利福平和50 μg/mL卡那霉素的LB平板上,28 ℃倒置暗培养2 d后备用。
1.4 农杆菌转化拟南芥
挑取含有目的基因质粒的单克隆阳性农杆菌菌体置于离心管中培养。取培养后的菌液10 μL接种于10 mL含有利福平和卡那霉素的新鲜LB液体培养基中,28 ℃、160 r/min条件下培养2~4 d,直到其D600 nm值达到0.8左右。取该菌液1 mL放于离心管中,离心后保留沉淀,用5%蔗糖溶液和Sitwet-77(万分之二加入)的混合液重新悬浮。随后用悬浮液浸染未开放的花序,浸染后的拟南芥用黑色塑料袋覆盖,暗培养24 h后移入正常光照下继续培养。为了增加浸染的成功率,每隔20 min浸染1次,共浸染3次,并且1周后再次浸染。收集拟南芥成熟种子,先用70%乙醇消毒2 min,迅速用01%的HgCl2清洗种子90 s,最后用无菌水对种子润洗3次。处理后的种子用琼脂水溶液均匀铺在含有潮霉素(HYR)的LB平板上,4 ℃暗处理 2~3 d 后移入正常光照下继续培养,将长成绿苗的拟南芥移入浇过营养液的土壤中继续生长,用于筛选阳性苗。
1.5 拟南芥双突变体的检测
将得到的42株T1代植株进行编码,分别记为T1-1、T1-2、T1-3、…、T1-40、T1-41、T1-42。剪取拟南芥T1代幼苗叶片2~3张,提取其基因组DNA,利用引物84a1-F和84a1-R(表1)进行PCR扩增目的基因,PCR产物用1%琼脂糖凝胶进行初步检测,初步检测正确后送公司进行测序验证。以野生型拟南芥序列作为对照,分析该基因片段靶序列是否突变。
将验证好的84a1发生突变的拟南芥继续用潮霉素筛选获得T2代,提取阳性绿苗基因组DNA,首先利用引物84a1-F和84a1-R进行PCR扩增,与T1代的检测结果进行比对,检测其突变是否已经纯合。将这些84a1突变体纯合体再利用引物84a2-F和84a2-R(表1)进行PCR扩增,用1%琼脂糖凝胶进行初步检测后送铂尚生物技术(上海)有限公司进行测序验证,以野生型拟南芥作为对照,通过序列比对确定其突变基因类型。
将突变的84a2但未纯合的拟南芥T2代继续用潮霉素筛选获得T3代,同上提取其基因组DNA,利用84a1-F和84a1-R进行PCR扩增获得ugt84a1目标序列,利用84a2-F和84a2-R进行PCR扩增获得ugt84a2目标序列,将2种扩增产物送公司进行测序,ugt84a1、ugt84a2同时发生突变的株系则为ugt84a1/ugt84a2双突变体。
2 结果与分析
2.1 sgRNA靶点序列的选择
从TAIR数据库查找到已公布的拟南芥ugt84a1和ugt84a2的基因组序列(分别为AT4G15480和AT3G21560)。利用sgRNA设计软件搜索到可能的拟南芥sgRNA序列,通过进一步筛选最终选定2条sgRNA序列,分别为GCTCGTTACCTTCGTTACAA和GACGAAGAAGTGGATTAACG。利用该靶点序列设计引物,其中拟南芥ugt84a1靶點序列的引物为Oligo(5′→3′) UP(TGATTGCTCGTTACCTTCGTTACAA)和Oligo LW(AAACTTGTAACGAAGGTAACGAGCA);拟南芥ugt84a2靶点序列的引物为Oligo UP(TGATTGACGAAGAAGTGGATTAACG)和Oligo LW(AAACCGTTAATCCACTTCTTCGTCA)。
2.2 载体的构建及其验证
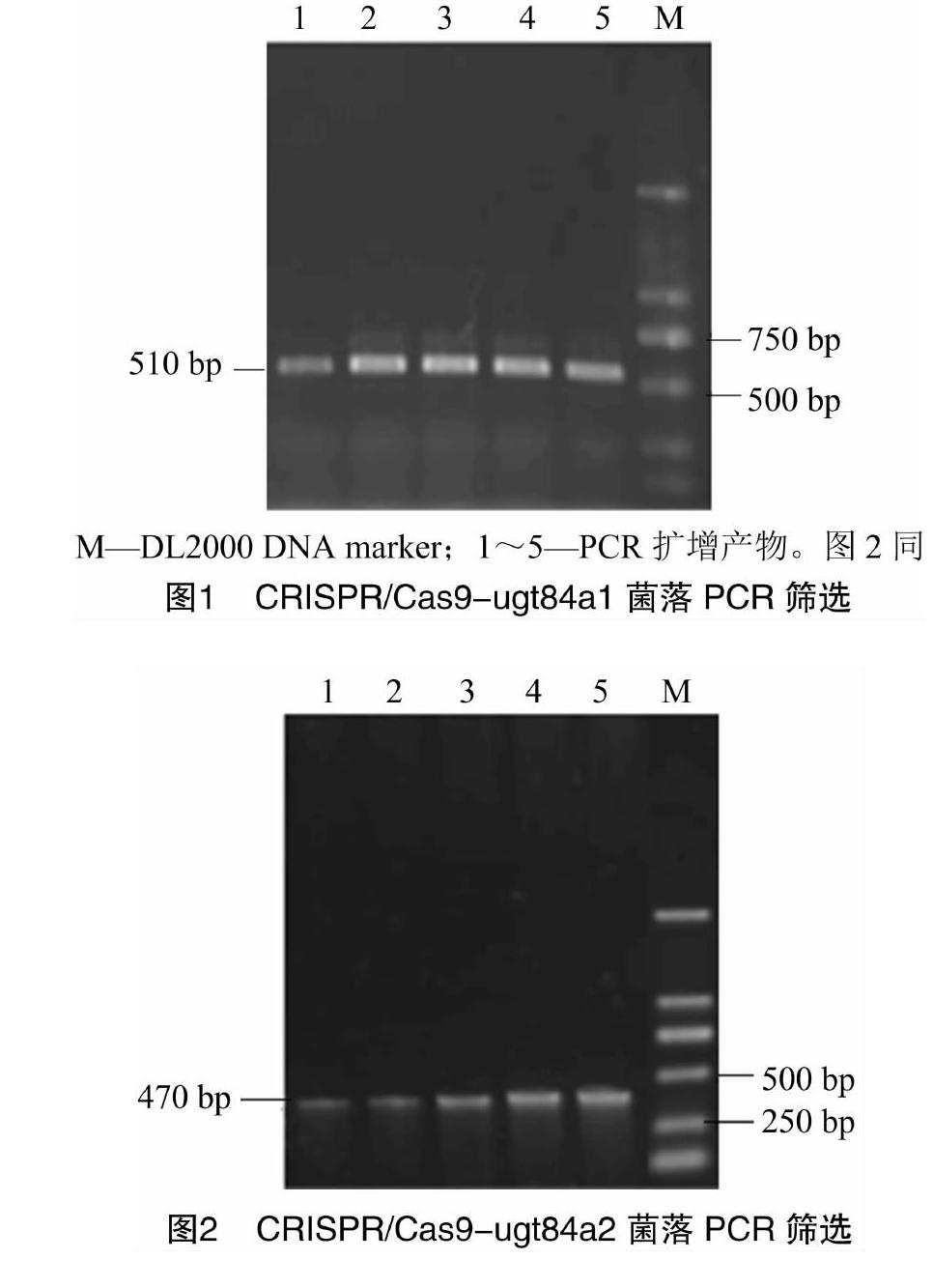
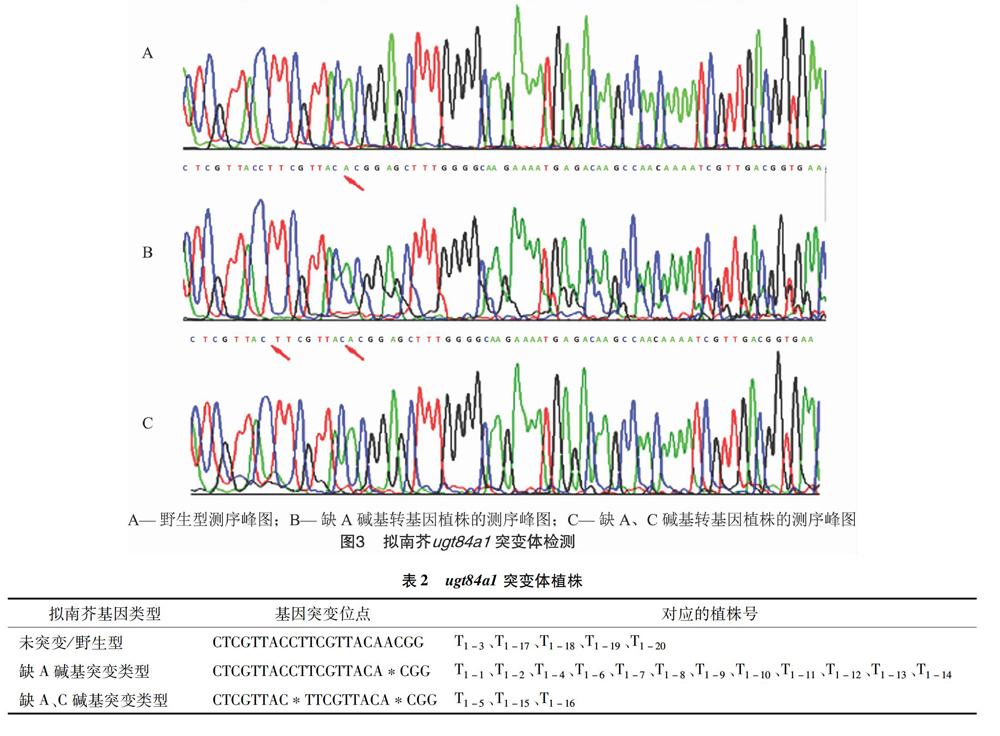
先将sgRNA靶点序列的引物Oligo二聚体在 95 ℃ 条件下变性3 min,然后以约0.2 ℃/s缓慢降至20 ℃进行退火配对连接。然后与线性化的CRISPR/Cas载体在25 ℃反应1 h进行连接。反应结束后首先进行PCR验证,ugt84a1阳性转化子的PCR条带大小为510 bp(图1),ugt84a2阳性转化子的PCR条带大小为470 bp(图2)。验证条带大小正确的转化子,提取其质粒进一步送铂尚生物技术(上海)有限公司测序验证,选取测序序列100%正确的重组载体转入农杆菌并浸染拟南芥。
2.3 拟南芥双突变体的筛选与检测
2.3.1 ugt84a1突变体植株嵌合体序列分析 携带有重组双突变体载体的农杆菌浸染拟南芥获得T0代植株,通过潮霉素筛选拟南芥T0代得到20株T1
代阳性苗,分别编号为T1-1、T1-2、T1-3、…、T1-18、T1-19、T1-20。提取T1代20株幼苗基因组DNA,并利用引物84a1-F和84a1-R(表1)进行PCR扩增,扩增产物送铂尚生物技术(上海)有限公司进行测序分析。将测序序列与野生型拟南芥植株序列进行比对,在20株阳性转基因植株中有12株缺失A碱基,引起基因的移码突变;3株缺失A、C 2个碱基,引起基因的移码突变;5株未发生突变。其中,缺失A碱基的12株拟南芥突变基因类型相同,缺失A、C 2个碱基的3株拟南芥突变基因类型相同(图3),从而得到ugt84a1突变体植株(表2),从图3可以看出,测序序列中有嵌合峰,表明T1代突变植株为嵌合体。
2.3.2 ugt84a1突变体纯合体筛选 选取T1代已经验证好的ugt84a1突变体T1-2、T1-16继续经潮霉素筛选得到T2代植株,每个号选取8株,分别编号为T2-2-1、T2-2-2、…、T2-2-7、T2-2-8;T2-16-1、T2-16-2、…、T2-16-7、T2-16-8。对这些T2代植株进行基因组DNA提取,利用引物84a1-F和84a1-R进行PCR扩增,扩增产物送铂尚生物技术(上海)有限
公司进行测序分析。观察并分析测序结果,与T1代84a1突变体植株的突变位点进行比对,其中T2-2-1、T2-2-5、T2-2-6的T2代与T1代突变位点一致,缺少A碱基;T2-16-4的T2代与T1代突变位点一致,缺少A、C碱基(表3),并且为纯合突变体(图4)。经过T1代与T2代的验证,得到了ugt84a1纯合体植株。缺C、A碱基突变类型 CTCGTTAC*TTCGTTACA*CGG T2-16-4
2.3.3 ugt84a1/ugt84a2双突变体筛选 将检测的ugt84a1纯合体T2-2-1、T2-2-5、T2-2-6和T2-16-4进行ugt84a2基因突变检测(图5)。首先进行基因组DNA提取,利用引物84a2-F和84a2-R进行PCR扩增,扩增产物送铂尚生物技术(上海)有限公司进行测序分析。分析测序结果,与野生型植株的84a2相应基因位点进行比对,检测结果发现,T2-2-1、T2-2-5、T2-2-6缺失A碱基,T2-16-4的A碱基突变为T碱基,插入1个T碱基(表4),由此得到了2种不同类型的ugt84a1/ugt84a2双突变体,分别为ugt84a1/ugt84a2 T2-2-1(ugt84a1缺失A,ugt84a2缺失T)、ugt84a1/ugt84a2 T2-16-4(ugt84a1缺失C、A,ugt84a2缺失T、A突變为C、G突变为T)。
3 讨论与结论
CRISPR/Cas9 是一种能够对基因组的特定位点进行精确编辑的技术,其原理是核酸内切酶Cas9蛋白通过导向性RNA识别特定基因组位点并对双链DNA进行切割,细胞随之利用非同源末端连接或者同源重组方式对切割位点进行修复,实现DNA水平基因敲除或精确编辑[22-24]。CRISPR/Cas9基因编辑技术比起ZFNs和TALENs基因编辑技术而言,有着无法比拟的优点[25]。CRISPR/Cas9系统操作较为简便,只需设计sgRNA靶向序列便可对基因进行编辑[21],由于其独特的优势,CRISPR/Cas9基因编辑技术已经在医学、动物科学、植物科学等领域广泛应用[ 26-28]。
植物体内含有大量的小分子化合物,这些小分子物质的糖基化在植物体内普遍存在[29-30]。糖基化修饰是植物小分子代谢修饰的一种重要形式,通常会引起小分子化合物的失活,从而调节细胞内小分子稳态,影响植物生长发育的诸多方面[31-32]。研究表明,拟南芥糖基转移酶UGT84A1、UGT84A2都能够糖基化外源毒性物质2,4,5-三氯苯酚[16]以及植物内源物质对羟基肉桂酸[17],表明UGT84A1和UGT84A2为同工基因。目前这2个基因的功能研究都停留在单基因突变体研究,目前的研究结果对于解析同源基因的功能和互相作用还缺少有力的证据。虽然拟南芥糖基转移酶UGT84A1和UGT84A2基因早已被发现并克隆,但由于体内代谢平衡调控是一个复杂的过程,其中涉及的相关基因的有序时空表达与基因之间的相互作用还没有具体研究透彻。基于以上原因,本研究利用CRISPR/Cas9系统成功获得了拟南芥ugt84a1/ugt84a2双突变体,此研究为UGT84A1和UGT84A2基因功能研究提供了试验材料,为其他物种中UGT84A1和UGT84A2同源基因的功能研究提供了借鉴。
快速高效筛选双突变体材料对于科学研究是必要的。本研究中,将UGT84A1与UGT84A2靶序列同时构建到CRISPR/Cas9双突变体载体,从而同时敲除同源基因ugt84a1/ugt84a2,通过分析突变体突变位点时发现,针对单一基因突变体往往会筛选到2~3种不同位点突变,其中多数突变的碱基是一致的,说明构建的CRISPR/Cas9载体系统工作稳定性很高。在筛选双突变体过程中,由于2个基因同时突变的概率很低,所以首先筛选ugt84a1单突变体,找到其突变体纯合体后,再筛选另一个基因ugt84a2是否同时突变,这样不仅工作效率高,需要测序的样品数也大大减少,从而提高了突变体筛选效率。综上,本研究的开展对于加快ugt84a1/ugt84a2功能基因资源的开发利用提供有力的理论与方法支持。
参考文献:
[1]Weis M,Lim E K,Bruce N C,et al. Engineering andkinetic characterisation of two glucosyltransferases from Arabidopsis thaliana[J]. Biochimie,2008,90(5):830-834.
[2]Lim E K,Ashford D A,Hou B K,et al. Arabidopsis glycosyltransferases as biocatalysts in fermentation for regioselective synthesis of diverse quercetin glucosides[J]. Biotechnology and Bioengineering,2004,87(5):623-631.
[3]Fraissinet-Tachet L,Baltz R,Chong J L,et al. Two tobacco genes induced by infection,elicitor and salicylic acid encode glucosyltransferases acting on phenylpropanoids and benzoic acid derivatives,including salicylic acid[J]. FEBS Letters,1998,437(3):319-323.
[4]Jackson R G,Kowalczyk M,Li Y,et al. Over-expression of an Arabidopsis gene encoding a glucosyltransferase of indole-3-acetic acid:phenotypic characterisation of transgenic lines[J]. Plant Journal,2002,32(4):573-583.
[5]Togenetti V B,van Aken O,Morreel K,et al. Perturbation of indole-3-butyric acid homeostasis by the UDP-glucosyltransferase UGT74E2 modulates Arabidopsis architecture and water stress tolerance[J]. Plant Cell,2010,22(8):2660-2679.
[6]Wang B,Jin S H,Hu H Q,et al. UGT87A2,an Arabidopsis glycosyltransferase,regulates flowering time via FLOWERING LOCUS C[J]. New Phytologist,2012,194(3):666-675.
[7]Hou B K,Lim E K,Higgins G S,et al. N-glucosylation of cytokinins by glycosyltransferases of Arabidopsis thaliana[J]. Journal of Biological Chemistry,2004,279(46):47822-47832.
[8]Poppenberger B,Fujioka S,Soeno K,et al. The UGT73C5 of Arabidopsis thaliana glucosylates brassinosteroids[J]. PNAS,2005,102(42):15253-15258.
[9]Husar S,Berthiller F,Fujioka S,et al. Overexpression of the UGT73C6 alters brassinosteroid glucoside formation in Arabidopsis thaliana[J]. BMC Plant Biology,2011,11(1):51.
[10]Jackson R G,Lim E K,Li Y,et al. Identification and biochemical characterization of an Arabidopsis in-dole-3-acetic acid glucosyltransferase[J]. Journal of Biological Chemistry,2001,276(6):4350-4356.
[11]Jin S H,Ma X M,Han P,et al. Correction:UGT74D1 is a novel auxin glycosyltransferase from Arabidopsis thaliana[J]. PLoS One,2013,8(4):e61705.
[12]Lim E K,Doucet C J,Li Y,et al. The activity of Arabidopsis glycosyltransferases toward salicylic acid,4-hydroxybenzoic acid,and other benzoates[J]. Journal of Biological Chemistry,2002,277(1):586-592.
[13]Martin R C,Mok M C,Habben J E,et al. A maize cytokinin gene encoding an O-glucosyltransferase specific to cis-zeatin[J]. Proceedings of the National Academy of Sciences of the United States of America,2001,98(10):5922-5926.
[14]Veach Y K,Martin R C,Mok D W S,et al. O-glucosylation of cis-zeatin in maize.Characterization of genes,enzymes,and endogenous cytokinins[J]. Plant Physiology,2003,131(3):1374-1380.
[15]Kudo T,Makita N,Kojima M,et al. Cytokinin activity of cis-zeatin and phenotypic alterations induced by overexpression of putative cis-Zeatin-O-glucosyltransferase in rice[J]. Plant Physiology,2012,160(1):319-331.
[16]Jones P,Messner B,Nakajima J I,et al. UGT73C6 and UGT78D1,glycosyltransferases involved inflavonol glycoside biosynthesis in Arabidopsis thaliana[J]. Journal of Biological Chemistry,2003,278(45):43910-43918.
[17]Meiner D,Albert A,Bttcher C,et al. The role of UDP-glucose:hydroxycinnamate glucosyltransferases in phenylpropanoid metabolism and the response to UV-B radiation in Arabidopsis thaliana[J]. Planta,2008,22:663.
[18]Fauser F,Roth N,Pacher M,et al. In planta gene targeting[J]. Proceedings of the National Academy of Sciences of the United States of America,2012,109(19):7535-7540.
[19]Li T,Liu B,Spalding M H,et al. High-efficiency TALEN-based gene editing produces disease-resistant rice[J]. Nature Biotechnology,2012,30(5):390-392.
[20]Shan Q W,Wang Y P,Li J,et al. Targeted genome modification of crop plants using a CRISPR-Cas system[J]. Nature Biotechnology,2013,31(8):686-688.
[21]Li J F,Norville J E,Aach J,et al. Multiplex and homologous recombination-mediated genome editing in Arabidopsis and Nicotiana benthamiana using guide RNA and Cas9[J]. Nature Biotechnology,2013,31(8):688-691.
[22]Razzaq A,Saleem F,Kanwal M,et al. Modern trends in plant genome editing:an inclusive review of the CRISPR/Cas9 toolbox[J]. International Journal of Molecular Sciences,2019,20(16):4045.
[23]Xu X J,Wan T,Xin H H,et al. Delivery of CRISPR/Cas9 for therapeutic genome editing[J]. Journal of Gene Medicine,2019,21(7):e3107.
[24]Gaj T,Gersbach C A,Barbas C F. ZFN,TALEN,and CRISPR/Cas-based methods for genome engineering[J]. Trends in Biotechnology,2013,31(7):397-405.
[25]Bannikov A V,Lavrov A V. CRISPR/CAS9,the king of genome editing tools[J]. Molekuliarnaia Biologiia,2017,51(4):582-594.
[26]Zhang J F,Chen L,Zhang J,et al. Drug inducible CRISPR/Cas systems[J]. Computational and Structural Biotechnology Journal,2019,17:1171-1177.
[27]Ziegler H,Nellen W. CRISPR-Cas experiments for schools and the public[J]. Methods,2019,172:86-94.
[28]Bradford J,Perrin D. A benchmark of computational CRISPR-Cas9 guide design methods:datasets[J]. PLoS Computational Biology,2019,15(8):e1007274.
[29]Wang J,Hou B K. Glycosyltransferases:key players involved in the modification of plant secondary metabolites[J]. Frontiers in Biology in China,2009,4(1):39-46.
[30]Bowles D,Isayenkova J,Lim E K,et al. Glycosyltransferases:managers of small molecules[J]. Current Opinion in Plant Biology,2005,8(3):254-263.
[31]Lim E K,Bowles D J. A class of plant glycosyltransferases involved in cellular homeostasis[J]. EMBO Journal,2004,23(15):2915-2922.
[32]Sieber P,Schorderet M,Ryser U,et al. Transgenic Arabidopsis plants expressing a fungal cutinase show alterations in the structure and properties of the cuticle and postgenital organ fusions[J]. Plant Cell,2000,12(5):721-738.呂 东,李丹丹,徐汝聪,等. 粳稻蔗糖转运蛋白基因OsSUTs在灌浆期的表达特点[J]. 江苏农业科学,2020,48(20):56-61,74.
