傅里叶变换红外光谱的土壤团聚体有机碳和全氮含量估测
2020-12-04刘翠英张津瑞樊建凌
刘翠英,张津瑞,曾 涛,樊建凌*
1. 南京信息工程大学应用气象学院,江苏省农业气象重点实验室,江苏 南京 210044 2. 南京信息工程大学环境科学与工程学院,江苏省大气环境监测与污染控制高技术研究重点实验室,江苏 南京 210044
引 言
土壤团聚体是土壤的基本单位,是土壤矿物通过胶结团聚过程形成的具有一定大小的土壤基本结构单元,具有水稳性、力稳性、多孔性等特点,对土壤的理化性质有着重要的影响[1]。 此外,土壤团聚体的形成过程及其稳定机制受土壤有机质,特别是其中碳氮的影响,团聚体与有机质含量是土壤结构状况和肥力水平的重要评判依据。 然而,影响土壤有机碳氮在团聚体内的含量与分布的因素非常复杂[2],快速、精确地了解土壤有机碳氮含量在团聚体内部的分布与变化将有助于了解土壤碳氮交换量、储量变化及土壤养分的管理。
近几十年来,人们对红外光谱法预测土壤性质进行了大量的尝试,利用红外光谱可以根据样本的光谱特征确定其中特定成分的含量,整个测量过程简单、快速且无破坏性。 红外光谱法已被用于对土壤有机碳、粘粒含量、全氮含量、pH、可提取态P、K、Fe、Ca、Mg及CEC等指标的分析[3]。 由于光谱数据的复杂性,主成分回归、多元线性回归(MLR)、偏最小二乘法回归(PLSR)等多元数据分析技术常被用于红外光谱预测土壤性质分析建模,随着数据挖掘技术的发展,支持向量机(SVM)、随机森林(RF)、人工神经网络(ANN)等机器学习方法也逐渐被应用于红外光谱数据建模。 然而,针对多元数据处理算法的比较研究还较缺乏,各种算法的优劣及适用范围尚不清楚。 最近,Moura-Bueno等[4]基于红外光谱,采用PLSR、MLR、SVM和RF方法建立了土壤有机碳的预测模型,结果表明,预测效果最好的是PLSR模型; 基于随机森林建立的模型预测效果不够突出。 另一方面,已有研究多针对全土性质进行建模分析,用红外光谱对土壤团聚体有机碳(SOC)和全氮(TN)进行预测的研究还不多见。 本研究以我国内蒙古淡栗钙土为研究对象,采用遗传算法(genetic algorithm, GA)进行特征波长选择,采用PLSR、SVM、ANN和RF等方法建立了不同粒径团聚体SOC、TN和红外光谱吸光度之间的关系模型,进而评价不同模型预测土壤团聚体中SOC和TN含量的潜力,探寻有效预测土壤团聚体性质的最佳算法,为实时、快速分析土壤团聚体有机碳和全氮含量提供技术支撑。
1 实验部分
1.1 土壤样本
土壤样品采自内蒙古自治区乌兰察布市四子王旗,是典型的内蒙古短花针茅荒漠草原带,共采集24个土壤样品,土壤类型为淡栗钙土。 样品运回实验室后在4 ℃保存,采用湿筛法[5]对土壤团聚体进行分级,共分为>2,0.25~2,0.053~0.25及<0.053 mm四级团聚体,共得到96个团聚体样品。 所得各级团聚体样品经冷冻干燥后研磨过100目筛,用稀HCl溶液(1 mol·L-1)去除土壤样品中的无机碳。 将土壤再次研细,过100目,用元素分析仪(Vario EL IIII, Elementar, Germany)测定团聚体土壤有机碳(SOC)和全氮(TN)含量,每个样品重复测定三次并求平均值。
1.2 红外光谱测定与光谱数据处理
团聚体样品红外光谱用傅里叶变换红外光谱仪(Nicolet iS5, Thermo Fisher Inc.)测定,光谱范围为400~4 000 cm-1,分辨率为4 cm-1,扫描次数为32次。 将所得光谱数据转换为吸光度[log(1/反射率)],并校正基线。 采用Savitzky-Golay平滑法对光谱数据进行平滑处理,然后取一阶微分,预处理后的光谱如图1所示。

图1 经S-G平滑(a)及一阶微分(b)处理后的团聚体FTIR光谱图
1.3 校正数据集与预测数据集的划分
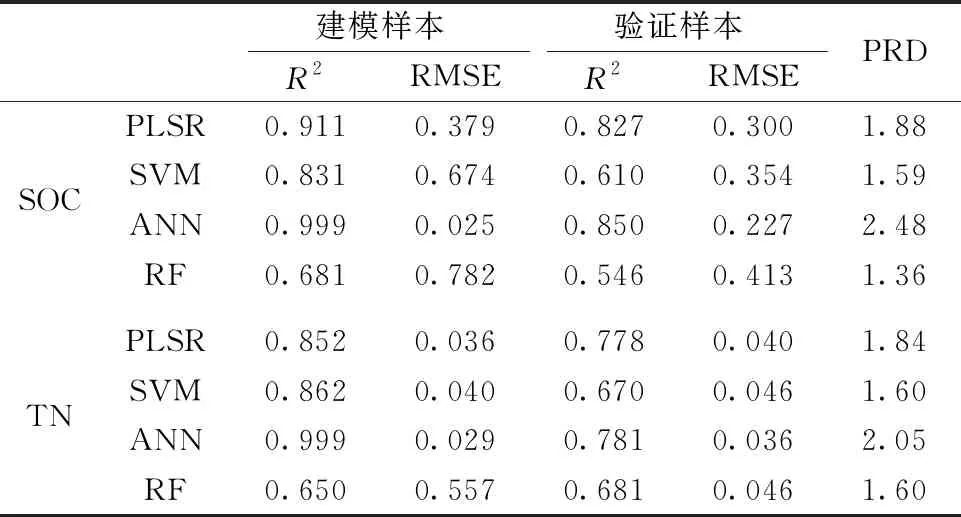
在建立模型前,需要将样品红外光谱及对应的SOC、TN数据划分为建模数据集和验证数据集,采用Kennard-Stone算法进行划分,通过计算样本光谱变量之间的欧氏距离,在样本特征空间里均匀地选取建模样本。 划分所得建模数据集与验证数据集的统计结果如表1所示。

表1 建模数据集与验证数据集的样本统计结果Table 1 Statistic summary of the modeling and validating data subsets
1.4 光谱变量选择与建模方法
1.4.1 确定区间大小
采用遗传算法(genetic algrothim)[6]进行特征波长选择,由于FTIR光谱波长数目较大(每一光谱包含7 468个数据),直接采用原始数据会使GA的优化搜索空间过大,因此需要按一定波长区间对光谱数据进行划分。 按适宜波长间隔将整个光谱均分为x个区间,然后求取各区间数据的标准偏差,平均标准偏差值越小说明区间数据越相近,因此在保障区间数据相近性的前提下应该尽量减少变量数目以优化计算效率。
1.4.2 特征光谱的确定
经S-G平滑及一阶微分处理后的数据,按适宜的波长区间对光谱进行均分,以各区间的平均谱数据为自变量,以交叉校验均方根误差RMSECV为适应度函数,采用遗传算法(GA)进行最优光谱波长的选择,设定种群大小为30,最大繁殖代数为100,交叉概率为0.5,变异概率为0.01[7],五次重复遗传算法后,确定特征光谱。 本研究使用“caret”包进行GA分析。
1.4.3 偏最小二乘法回归(PLSR)
偏最小二乘回归(PLSR)是一种广泛用于土壤光谱定量分析的线性回归模型,使用潜变量方法对预测变量和观察变量的两个投影空间中的协方差结构进行建模。 因此,PLSR模型克服了变量之间的共线性问题。 此外,PLSR模型在选择特征向量时,强调自变量对因变量的解释和预测,消除了无用噪声对回归的影响,并最大程度地减少了模型中包含的变量数量。 本研究使用“pls”包进行PLSR建模。
1.4.4 支持向量机(SVM)
支持向量机(SVM)是一种对数据进行二元分类的模型,SVM的基本模型是找到一个可以分隔正反数据的超平面,选择的超平面需要离训练集数据尽可能远; 支持向量机的关键在于核函数,这是一个非线性的分类器。 它的学习策略就是间隔最大化,找到距离超平面间隔最小的样本点,然后将其间隔最大化。 本研究使用“e1071”包进行SVM建模。
1.4.5 人工神经网络(ANN)
人工神经网络是基于生物学中神经网络的基本原理,模仿生物的神经网络来对复杂的外界信息进行处理,它是对生物神经元网络的简易化模仿。 人工神经网络模型可以并行分布地去处理问题,拥有很高的容错性,把信息的加工和记忆能力结合一起。 它实际上是一个复杂网络,连接大量的简单元件,因此ANN具有很高的非线性,能够进行复杂的逻辑操作,处理非线性关系的样本数据。 本研究使用“neuralnet”包进行ANN建模。
1.4.6 随机森林(RF)
随机森林模型通过自助重采样技术,连续生成训练样本和测试样本,并从训练样本中生成多个分类树以形成随机森林。 它通过对决策树进行平均来降低过拟合的风险。 即使新的数据点出现在数据集中,整个算法也不会受到太大的影响,只会影响一个决策树,并且很难影响所有决策树。 本研究使用“randomForest”包进行RF建模。
1.5 模型评价
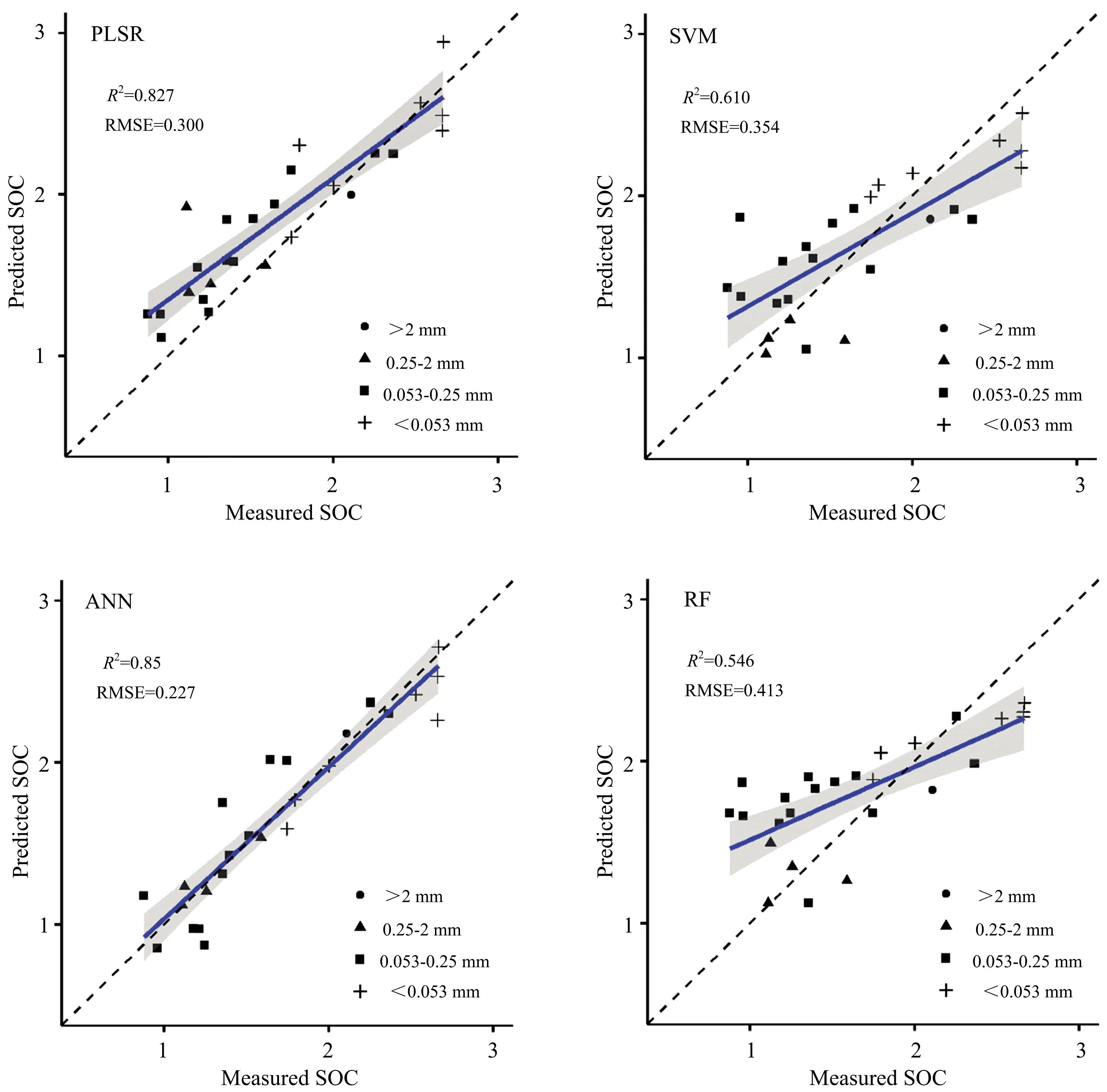
主要采用决定系数(R2)、均方根误差(RMSE)以及相对分析误差(RPD)对模型进行评价。R2越大且RMSE越小说明模型精度越好、其预测效果越好。R2的判断标准为:R2>0.90表示预测结果出色;R2在0.81~0.90之间表示预测结果很好;R2在0.66~0.80之间为预测结果一般;R2<0.66表示预测结果很差。 PRD的判断标准为: RPD>2表明模型具有极好的预测能力; 1.4 由图2可见,波长间隔越小,样本的平均标准偏差越低,但同时区间数目越多。 在波长间隔大小为6 cm-1时最大标准偏差出现一个低谷,此时区间数为600个,因此在兼顾数据相近性与变量少量性的前提下,确定6 cm-1作为划分区间的适宜大小,将整个光谱分为600个区间,然后将区间的平均光谱值作为自变量形成光谱曲线。 图2 平均标准偏差和最大标准偏差与波长区间大小的关系Fig.2 Relations between the mean SD, maximumSD and wavelength interval 采用GA算法并重复5次后,筛选出团聚体SOC特征光谱区间共计303个。 由团聚体SOC特征光谱区间的分布(图3)可以看出,SOC的特征光谱覆盖范围较广,但在2 200~2 700及>3 700 cm-1以上波长范围内选取的特征光谱区间较少。 此外,在GA筛选过程中1 132~1 138,1 372~1 378,1 630~1 636,1 810~1 816,1 864~1 870cm-1这五个光谱区间在每次重抽样中均被选为特征光谱,表明这些光谱区间对团聚体SOC含量较为敏感。 图3 团聚体SOC和TN特征光谱区间分布Fig.3 Histrogam of charasteristic wavelengthsof SOC and TN in soil aggregates 对于土壤团聚体TN,GA算法共筛选出特征光谱161个。 由团聚体TN特征光谱区间的分布(图3)可以看出,TN的特征光谱覆盖范围较平均,但在>3 200 cm-1以上波长范围内选取的特征光谱区间较少。 在GA筛选过程中1 114~1 120,1 258~1 264,1 264~1 270,1 276~1 282和1 408~1 414 cm-1这五个光谱区间在每次重抽样中均被选为特征光谱,表明这些光谱区间对团聚体TN含量较为敏感。 基于特征光谱区间,采用PLSR或ANN对团聚体SOC的建模结果均非常出色(R2>0.90,表2),同时这两种模型对验证样本的预测结果也很好(R2>0.80)。 然而PLSR模型对验证样本SOC含量较低时出现了较明显的高估(图4),导致模型预测结果在低值区与实测值偏差较大,RMSE值较高,使得PLSR整体预测能力较为一般(PRD<2)。 采用SVM模型对团聚体SOC的建模结果也较好,但模型对样本的预测能力却较差(R2<0.66,PRD<2)。 然而,RF对团聚体SOC的建模及预测结果均较差,基本无法对样本进行预测(PRD<1.4)。 总之,四种模型中ANN的建模及预测结果均是最好的,其对验证样本的预测除有少数点偏离外,其余点基本均在1∶1线的附近(图4),其RMSE值在四种模型中最低(RMSE=0.227)。 此外,ANN在对团聚体SOC预测时并没有受团聚体粒径的影响,整体表现出极好的预测能力(RPD>2)。 然而,本研究样品数量相对较少,若增加建模样品数量可能会建立效果更好的模型。 表2 不同模型对SOC和TN预测效果比较Table 2 Comprison of SOC and TN predictingresults by different models 图4 不同模型对团聚体SOC的预测值与实测值对比Fig.4 Comparison of predicted SOC and measured SOC in soil aggregates by different models 基于特征光谱区间,采用PLSR或SVM对团聚体TN的建模结果均较好(R2>0.80,表2),然而这两种模型对验证样本的预测结果却一般(0.66 图5 不同模型对团聚体TN的预测值与实测值对比Fig.5 Comparison of predicted TN and measured TN in soil aggregates by different models 为了验证特征光谱选择对团聚体SOC和TN估测的影响,运用上述结果中表现最好的ANN模型对FTIR全谱数据进行了建模预测。 结果表明,采用全谱数据对验证样本SOC的预测结果较好,对TN的预测结果却一般(图6)。 此外,采用全谱数据对SOC和TN的预测效果均低于基于GA选择的特征光谱区间的ANN模型(图4和图5)。 可见,采用GA进行特征光谱区间的选择不仅可以简化模型的结构,剔除光谱中无关的信息,而且可以提高模型的精度和预测效果。 特征波长选择是红外光谱预测土壤性质的一个重要步骤,通过对特征波长的选择可以剔除无关的信息,提高模型的预测性能及计算效率。 由于红外光谱数据量大,常采用相关系数法、连续投影算法、遗传算法、粒子群算法、蚁群算法等[5-6]来选取特征波长。 如刘振尧等[9]基于随机森林优选信息波长,选取了215个波长信息,建立了土壤有机质的预测模型。 本研究选用遗传算法对特征光谱区间进行筛选,充分利用了土壤的光谱信息,采用人工神经网络建模对团聚体SOC和TN的预测也取得了较满意的结果。 因此,遗传算法从整体最优化的角度筛选了土壤团聚体SOC和TN的特征波段,简化了模型的结构并提高了模型的精度,结合人工神经网络建模,更适用于团聚体土壤有机碳和全氮的光谱特征分析与估测模型的构建。 红外光谱由于测量过程简单、快速且无破坏性,已被广泛用于对土壤有机碳、粘粒含量、全氮含量、pH、可提取态P、K、Fe、Ca、Mg及CEC等指标的分析[3, 10]。 Erktan等[11]采集了法国南部75个荒地土壤样品并将其分为<1,1~2和3~5 mm三级团聚体,利用中红外-近红外光谱对团聚体稳定性进行了预测。 Shi等[12]采集了83个比利时土壤样本并将其分为>250,63~250和<63 μm三级团聚体,利用可见-近红外光谱建立了团聚体稳定性的偏最小二乘法定量模型。 可见,已有研究往往针对不同粒径组分分别建立模型进行预测,该过程要求样本量大且很难对所有组分同时进行合理预测。 此外,不同研究者所选用的土壤团聚体粒径分级方案存在差异,获得的团聚体粒径也不尽相同,不易针对不同粒径进行建模分析,严重制约了红外光谱法在分析预测土壤团聚体理化性质中的应用。 本研究发现,虽然不同粒径土壤团聚体在不同波长的吸光度存在较大差异,但其FTIR光谱的整体趋势是一致的,因此可以考虑将不同粒径土壤团聚体FTIR数据混合建模。 通过遗传算法筛选特征光谱,我们发现人工神经网络模型可以很好地对土壤团聚体碳氮含量进行预测,且不会受团聚体粒径的影响。 这可能由于在遗传算法选择特征光谱时已将某些反映土壤矿物、粘粒等特征的波长区间包含在内(图3),如780~800 cm-1处是石英矿物中Si—O键伸缩振动、910 cm-1为高岭石和三水铝石等粘土矿物中O—H的弯曲振动、1 034 cm-1为高岭石矿物中Si—O键的伸缩振动、3 600~3 700 cm-1为粘土矿物中O—H键的伸缩振动[10, 13]。 由于人工神经网络的复杂性和高度的非线性,其所建立的模型可能已包含了不同粒径对土壤碳氮含量的影响。 因此,我们认为基于遗传算法筛选特征波长区间并采用人工神经网络可以将不同粒径土壤团聚体统一建模,用于团聚体土壤有机碳和全氮含量的估测。 图6 基于全谱数据对团聚体SOC和TN的预测值与实测值对比Fig.6 Comparison of predicted TN and measured TN in soil aggregates by different models (1)采用Savitzky-Golay平滑并取一阶微分对FTIR光谱数据进行预处理后,在波长间隔大小为6 cm-1时最大标准偏差出现一个低谷,同时平均标准偏差较小,区间数量适中,可以用于划分FTIR光谱区间。 (2)基于遗传算法筛选的特征光谱区间构建的土壤团聚体SOC和TN含量的ANN模型建模及预测能力均是最好的(RPD>2),显著优于PLSR、SVM及RF模型。 (3)基于全谱数据的ANN模型对土壤团聚体SOC和TN的预测效果均低于基于GA选择的特征光谱区间的ANN模型。 结果表明,采用GA进行特征光谱区间的选择不仅可以简化模型结构,剔除无关的信息,而且可以提高模型的精度和预测效果。 (4)将不同粒径土壤团聚体FTIR数据混合建模。 通过遗传算法筛选特征光谱,发现人工神经网络模型不仅可以很好地对土壤团聚体SOC和TN含量进行预测,而且不会受团聚体粒径的影响,表明该方法可以用于团聚体土壤有机碳和全氮含量的估测。2 结果与讨论
2.1 适宜波长区间

2.2 土壤团聚体特征光谱区间
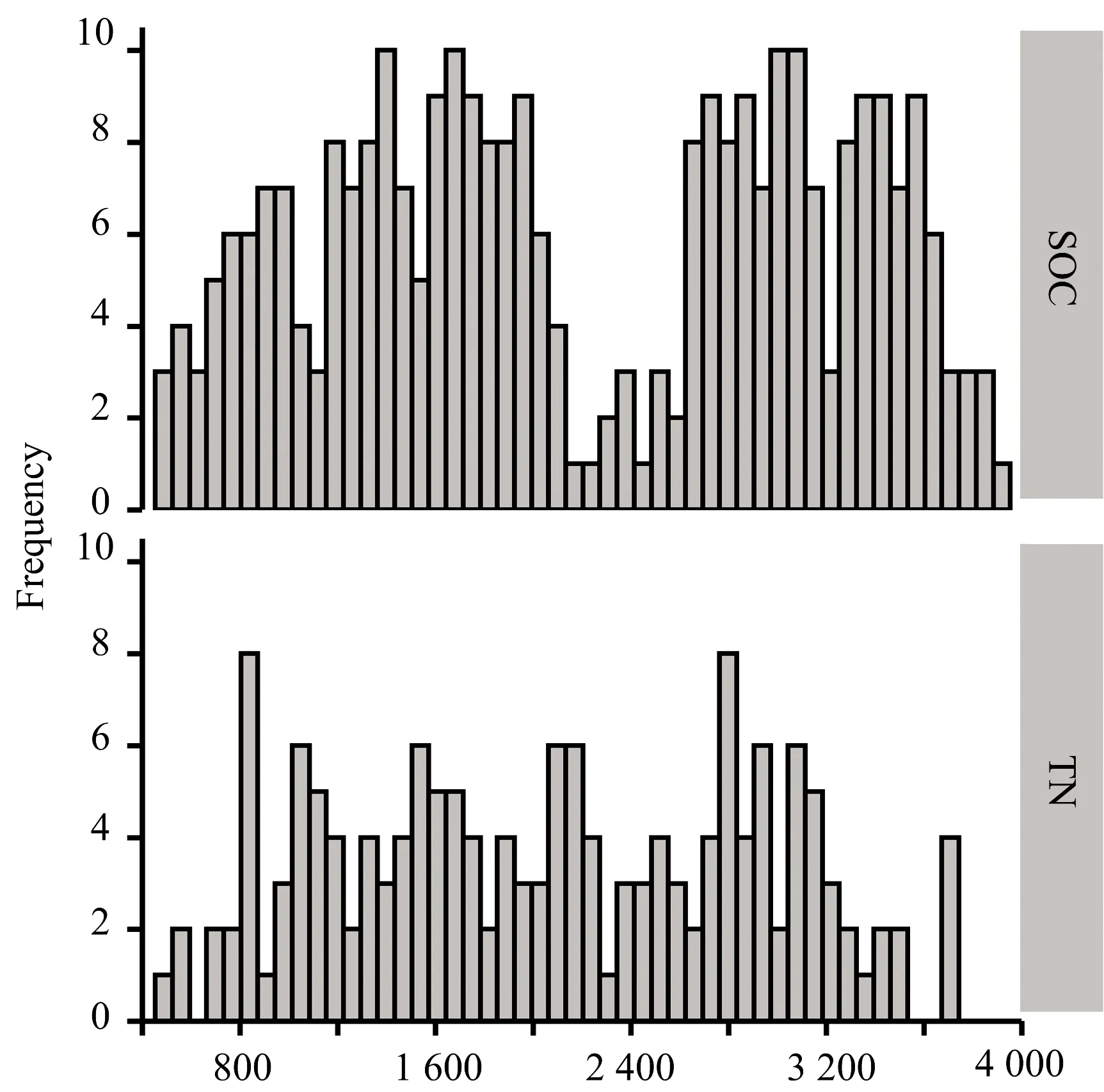
2.3 不同建模方法对团聚体SOC预测结果比较
2.4 不同建模方法对团聚体TN预测结果比较

2.5 基于全谱的团聚体SOC和TN估测

3 结 论
