基于线粒体12S rRNA基因的大鹏半岛海域石珊瑚的系统发育分析
2020-11-06肖宝华廖宝林谢子强谢勇琪覃业曼
肖宝华,廖宝林,王 琼,谢子强,2,谢勇琪,覃业曼
(1.广东海洋大学 深圳研究院,广东 深圳518000;2.深圳市碧海蓝天海洋科技有限公司,广东 深圳518000)
0 引言
造礁石珊瑚(刺胞动物门Cnidaria珊瑚纲Anthozoa六射珊瑚亚纲Hexocorallia石珊瑚目Scleractinia)组成的珊瑚礁生态系统是海洋环境中物种最丰富的生态群落之一[1],主要分布在地球南北纬30°以内的海域[2]2.以往的研究中,多采用珊瑚骨骼形态、颜色、触手等依据构建珊瑚分类体系并对珊瑚进行分类[3].其中,石珊瑚(Scleractinian)多以珊瑚骨骼形态为依据进行分类[4],[5]1893.但是,珊瑚骨骼变异性和同源性[6]640等研究中尚未攻克的难关阻碍了人们对珊瑚分类进化的进一步认识.随着分子生物学的快速发展与珊瑚多样性研究的不断深入,分子生物学方法逐渐应用于珊瑚的系统发育研究中,为珊瑚研究提供了新的思路.其中,基于不同算法模型建立的系统发育进化树也为研究珊瑚进化关系提供新方法.20世纪90年代,科学家开始了石珊瑚的分子生物学研究[7]407.根据相关学者的分子生物学研究积累并结合已有的形态学研究,可以将造礁石珊瑚分为坚实型类群(Robust)和复合型类群(Complex)[6] 641.坚实型珊瑚被认为是钙化程度较高,具备坚实的骨骼结构,珊瑚外观主要以碟片状和团块状为主的类群,包括蜂巢珊瑚科(Faviidae)、石芝珊瑚科(Fungiidae)和褶叶珊瑚科(Lobophyllidae)等[6] 641.复合型珊瑚骨骼钙化程度较低,形态复杂多变,包括鹿角珊瑚科(Acroporidae)、菌珊瑚科(Agaricidae)和滨珊瑚科(Portidae)等[6] 641.同时,科学家在研究的过程中发现珊瑚虫的线粒体基因进化速度慢,相对保守[8].线粒体的12S rRNA基因、16S rRNA基因和细胞色素b基因、细胞色素c氧化酶亚基I基因(COI)等通常作为研究珊瑚系统发育进化的标记基因,其中,rRNA基因结构保守,进化速度缓慢,是研究生物进化关系的典型特征基因[9] 460.
深圳市东部海域受亚热带季风气候的影响,年平均海水表面温度(SST)为21.6 ℃,平均盐度32,地表径流较少且短,呈放射状入海,海岸曲折,滩涂面积少,海洋底质多为粘土和粉沙[10].优越理想的自然条件使大鹏半岛海域成为石珊瑚生长发育的温床.大鹏半岛海域的石珊瑚不但种类丰富,并且珊瑚覆盖率较高,由石珊瑚形成的珊瑚礁生态系统是深圳生态基石的重要组成.2007年对深圳海域珊瑚资源进行调查,共记录了47种石珊瑚[11].近年来,由于受到气候变暖、海洋酸化以及人类活动的影响,全球珊瑚资源呈现退化的趋势[12-13],大鹏半岛海域的石珊瑚覆盖率也逐年下降.因此,本研究通过聚合酶链式反应技术(PCR),以石珊瑚线粒体12S rRNA基因为分子标记,对大鹏半岛海域的16个石珊瑚样品种间关系和系统进化关系进行研究,旨在为深圳东部海域珊瑚资源保护工作提供相应理论基础.
1材料与方法
1.1 采样站位及方法
供试的石珊瑚样品采自深圳东部大鹏半岛海域的6个站位(图1),具体经纬度见表1.珊瑚样品采于2018年7~8月,挑选风平浪静,海水透明度高的时候出海采样.实验人员准备好潜水和水下拍摄设备后,在相应的站位下潜,剪取珊瑚边缘断枝(1~2 cm)并拍摄珊瑚样品图片和视频,以便后期样品的形态学鉴定.所采集的珊瑚样品截取小部分于装有无水乙醇的离心管中保存,用于后续提取样品基因组总DNA.

图1 深圳东部大鹏半岛海域采样站位图

表1 深圳东部大鹏半岛海域采样站位信息表
1.2 样品的形态学鉴定
样品的形态学鉴定参照邹仁林[2]19-235的《中国动物志-腔肠动物门·珊瑚虫纲·石珊瑚目·石珊瑚》和Veron[5]1893-1894六射珊瑚骨骼形态特征分类系统,将所采集的珊瑚样品依据珊瑚骨骼形态分为5科7属9种,各样品的形态学分类情况如表2所示.
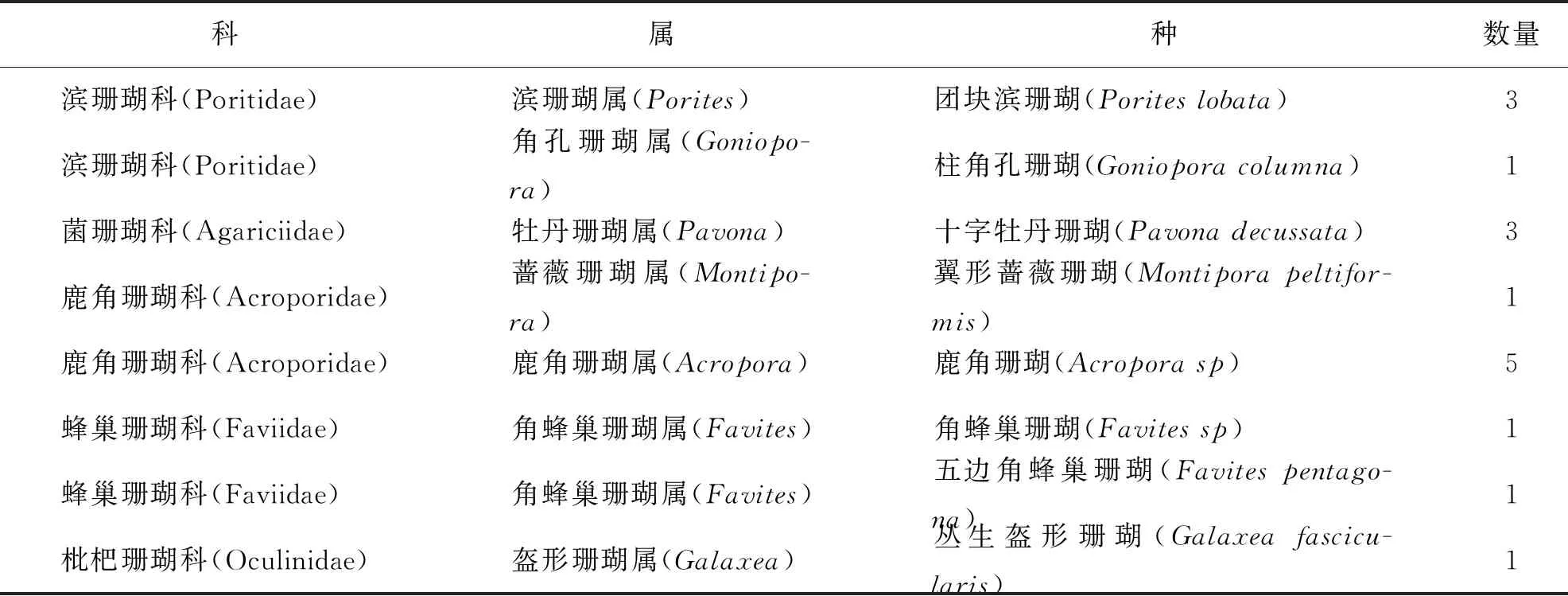
表2 珊瑚样品的形态学分类
1.3 样品的基因组总DNA提取
采集的样品使用滤纸吸附表面黏液并用工具捣碎,捣碎后的样品使用DNA提取试剂盒(北京天根生物有限公司)提取细胞组织基因组.提取获得的DNA样品在经过凝胶电泳检测后,置于4 ℃冰箱中贮藏备用.
1.4引物设计合成
12S rRNA基因的PCR引物采用文献[14]的12S基因通用引物,并委托上海生工生物技术服务有限公司合成.引物序列为:f-AGC CAC ACT TTC ACT GAA ACA AGG;r-GTT CCC YYW CYC TYA CYA TGT TAC GAC.采用升温PCR扩增,先以50 ℃退火5个循环接56 ℃退火的30个循环,产物大小为703~887 bp.
1.5 PCR扩增和DNA测序
PCR反应体系反应(总体积为25 μL):0×Buffer 2.5 μL;dNTP 2 μL;上下游引物各1 μL;Taq DNA聚合酶1 U;DNA模板2 μL;其余用去离子水补至25 μL.
PCR反应条件:5 min预变性(95 ℃),46 s变性(95 ℃),46 s退火(50 ℃),1 min 30 s延伸(72 ℃),共35个循环,最后72 ℃延伸10 min.扩增产物在10 g·L-1的琼脂糖凝胶中检测,选取扩增条带单一、浓度较高的PCR产物送至上海生工生物技术公司进行测序,获取各实验样品的序列信息.
1.6 序列分析
测序所得序列用DNASTAR中的Editseq软件进行编辑、剪切、校正,再将校正好的序列与NCBI非冗余数据库进行同源性比对,以确保所获得的序列为目标序列.
1.7 系统进化树的构建
采用MEGA6.0软件进行基因序列分析,获取样品12S rRNA基因组序列长度、碱基组成、简约信息位点等相关信息,同时根据简约信息位点,计算出种间遗传距离.本研究中系统进化树是使用MEGA6.0软件基于Kimu-ra双参数模型,采用邻位连接法(Neighbor-Joinning,NJ)、最小进化法(Minimum-Evolution,ME)和最大似然法(Minimum-Likelihood,ML)构建出来的.系统进化树中节点的自举置信度水平由自引导值(Bootstrap value)估计,重复次数为1 000.
2 结果与分析
2.1 序列碱基组成分析
通过扩增测序,分别获得16个石珊瑚样品的12S rRNA的基因序列.从表3可知,本研究中的16条基因序列长度在692~830 bp之间.从碱基组成比例来看,16个石珊瑚样品的12SrRNA序列的C含量在12.3%~15.5%之间,G含量在20.5%~24.3%之间,T含量在29%~31.8%之间,A含量最高,在31.8%~35.5%之间.所有序列碱基A、T的含量均高于G、C,具有明显的AT偏倚.其中,Poriteslobata1与Poriteslobata2、Poriteslobat3序列碱基组成相同;Pavonadecussata1和Pavonadecussata3序列碱基组成相同;Acroporasp1、Acroporasp2、Acroporasp3、Acroporasp4和Acroporasp5序列碱基组成相同.
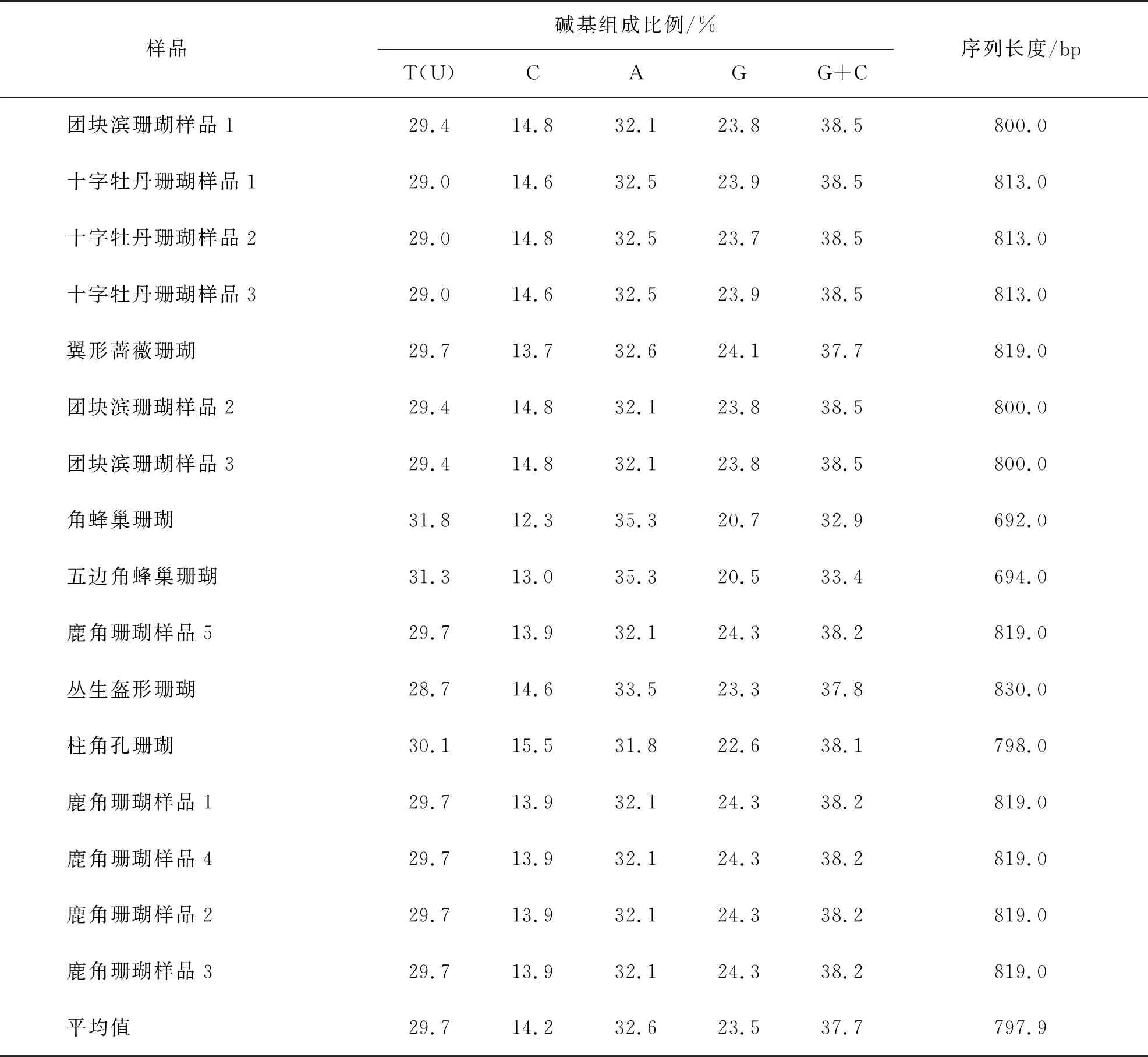
表3 16个石珊瑚样品的12S rRNA基因片段的序列组成
2.2 序列变化情况及种间遗传距离
通过MEGA6.0计算16个石珊瑚样品的种间遗传距离.从表4可知,16个样品的平均距离为0.094 3.遗传距离在科、属、种间呈逐级递减趋势.其中,五边角蜂巢珊瑚(Favitespentagona)和十字牡丹珊瑚第2个样品之间(Pavonadecussata2)的种间遗传距离最大,为0.256 8.团块滨珊瑚的3个样品(Poriteslobate1、Poriteslobate2、Poriteslobata3)间遗传距离为0,根据形态学初步判定这3个样品为同一种石珊瑚,序列碱基组成分析和种间遗传距离分析数据也证实了这一点;十字牡丹珊瑚样品1(Pavonadecussata1)和十字牡丹珊瑚样品3(Pavonadecussata3)遗传距离为0,结合序列碱基组成和形态学分析,初步判定两个样品为同一种十字牡丹珊瑚;鹿角珊瑚的5个样品(Acroporasp1、Acroporasp2、Acroporasp3、Acroporasp4、Acroporasp5)之间遗传距离为0,通过序列碱基组成和形态学分析也可以判定这5个样品为同一种鹿角珊瑚.据此,判定本研究的16个石珊瑚样品来源于9种石珊瑚.排除遗传距离为0的样品,十字牡丹珊瑚样品1(Pavonadecussata1)和十字牡丹珊瑚样品2(Pavonadecussata2)间的遗传距离最小,为0.001 5.

表4 基于12S rRNA基因的16个石珊瑚样品种间遗传距离
2.3 基于12S rRNA对九种石珊瑚的系统进化分析
使用MEGA6.0软件将9种石珊瑚的12S rRNA通过邻位连接法、最大似然法和最小进化法构建系统发育进化树,结果显示3种方法构建的系统发育进化树拓扑结构基本一致,只是在节点置信度上略有差异.由系统发育进化树(图2)可知,这9种石珊瑚聚为2个大的分支:滨珊瑚科(Poritidae)、菌珊瑚科(Agariciidae)、鹿角珊瑚科(Acroporidae)和枇杷珊瑚科(Oculinidae)等复合型珊瑚聚为一簇;蜂巢珊瑚科(Faviidae)等坚实型珊瑚聚类为一簇,这个结果与已有的珊瑚分类学研究结果相符合[7]397,[15],上文种间遗传距离分析中也能看出2个分支间的珊瑚种间距离值相较同一分支的科间、属间、种间距离值较大.从聚类结果可见,根据传统形态学方法划分的各科亲缘关系较近:同为滨珊瑚科(Poritidae)的柱角孔珊瑚(Gonioporacolumna)和团块滨珊瑚(Poriteslobata)亲缘关系较近,聚为一支;同为菌珊瑚(Agariciidae)的两种十字牡丹珊瑚(Pavonadecussata1、Pavonadecussata2)的12SrRNA序列存在细微差异,聚为一支;同为蜂巢珊瑚科(Faviidae)的角蜂巢珊瑚(Favitessp)和五边角蜂巢珊瑚(Favitespentagona)聚为一支.鹿角珊瑚(Acroporasp)与翼形蔷薇珊瑚(Montiporapeltiformis)聚为一支,丛生盔形珊瑚(Galaxeafascicularis)和其他复合型珊瑚亲缘关系较远.

图2 最大似然法构建九种石珊瑚12SrRNA基因序列系统发育进化树注:分枝上数值表示最大似然法、邻接法与最小进化法1 000次重复抽样检测的bootstrap值.
3 讨论
本研究的石珊瑚样品12S rRNA基因片段长度在692~830 bp之间,G+C含量在32.9%~38.9%之间,呈现明显的AT偏倚.通过碱基组成分析、种间遗传距离分析和形态学分析初步将本研究中的16个石珊瑚样品分为9种,系统发育进化树则将9种石珊瑚进一步分为坚实型珊瑚和复合型珊瑚两簇.结合系统发育进化分析和碱基组成分析可见,本研究中的蜂巢珊瑚的12S rRNA序列长度在692~694 bp之间,G+C含量在32.9%~33.4%之间;而复合型珊瑚这一分支的12S rRNA序列长度在798~830 bp之间,G+C含量在37.7%~38.5%之间,因此,本研究中的坚实型珊瑚的蜂巢珊瑚和其余复合型珊瑚的12S rRNA在序列长度和G+C含量间有较为显著的差别.但由于本研究中的实验样品较少,此结论还有待进一步验证.由本文研究结果可知,不同种属间的石珊瑚12S rRNA序列差异较为明显,同一种属的石珊瑚也存在细微差异.十字牡丹珊瑚的2个样品(Pavonadecussata1、Pavonadecussata2)根据形态学鉴定为同一种,但两者的12S rRNA基因存在这差异,说明石珊瑚12S rRNA基因具备用于石珊瑚种间分化研究的潜力.
在石珊瑚分子生物学研究中多采用线粒体COI基因和16S rRNA等作为分子标记基因.COI基因长度适宜、进化速率慢并且富含系统发育信号,能够较好地进行遗传进化分析[9]459.但也由于石珊瑚COI基因缓慢的进化速率,导致石珊瑚COI基因较低的种内分化,在种内鉴定方面存在不足[16].16S rRNA基因也具备较为保守、进化速率较慢的特点,适宜用作种以上分子分类的标记,但在部分珊瑚科属内也存在着种间分化较低的问题[17].相较而言,12S rRNA的基因序列的长度较长、差异性较大并且能够较好地鉴别珊瑚的种间的差异,能够弥补COI基因和16S rRNA在分子鉴定方面的不足,可以较好地作为其他分子标记研究的补充和佐证.
