采水量对寡营养海域浮游真核微生物分子多样性评价的影响
2020-10-09赵荣杰徐奎栋
赵荣杰 , 赵 峰 , , 徐奎栋 ,
(1. 中国科学院海洋研究所海洋生物分类与系统演化实验室, 山东 青岛 266071; 2. 中国科学院大学, 北京100049; 3. 中国科学院海洋大科学研究中心, 山东 青岛 266071)
真核微生物狭义上指原生生物, 广义上还包括单细胞真菌和微型后生动物[1-3], 其种类繁多、分布广泛, 在海洋初级生产、微食物网和生物地球化学循环等生态过程中发挥着重要作用[4-5]。海洋真核微生物多样性的研究对于理解海洋生态环境的变化过程以及人类活动和气候变化对海洋生态系统的影响等至关重要。
近年来, 环境DNA 结合高通量测序技术被广泛应用于真核微生物多样性研究中[6-8], 极大地拓展了对真核微生物多样性及其分布的认知, 检获了大量潜在新类群, 并为微型生物地理分布这一国际热点争论, 提供了大量新鲜素材和佐证[9-14]。近海因陆源输入[15-17]等原因具有丰富的营养盐, 真核微生物丰度和生物量较高, 已有的关于近海水体中的真核微生物多样性研究中, 采样量为300 mL~40 L不等[9,12,18-19]。热带大洋多为寡营养环境, 真核微生物的丰度和生物量明显较近海低, 检获足够的生物多样性所需要的采样量远大于近海。受限于采样方法、采样时间和成本, 大洋生物调查的采样量差异巨大, 范围为10~500 L[6,11,13]。
采样量是影响海洋真核微生物多样性评估的重要因素。 有关采样量对真核微生物多样性影响的研究多集中于沉积物。Fonseca 等人[20]关于海底沉积物中后生动物多样性的研究发现, 8 个采样站共24 个沉积物样品所获得的OCTU(Operational Clustering of Taxonomic units, 可操作聚类分类单元)稀释曲线未达到饱和。由于底栖生物斑块分布的特性, 即使高通量测序技术佐以大量的样品, 真实的物种多样性仍未可知。水体与沉积物不同, 潮汐和洋流等海洋现象增强了水体的连通性, 不同海域之间或不同深度水层之间得以产生化学和生物因素的交流[21], 从而使得水体中真核微生物的分布均匀度指数高于底栖群落, 但水体中真核微生物多样性仍与采样量存在正相关的关系。因此, 探究水体采样量对检获的真核微生物多样性的影响, 对真核微生物多样性研究具有重要的指导意义。
吕宋海峡北起台湾岛南至菲律宾吕宋岛, 是连接中国南海和西太平洋的重要通道[22]。吕宋海峡属于寡营养海域, 作为黑潮在沿太平洋西岸北上及进入南海的过程中的必经之地, 其水文环境和生物多样性深受黑潮携带的西太平洋海流信号的影响[23-26]。该海区真核微生物群落交流密切, 且处于大洋与近岸的交汇区, 基于该海区的研究, 对未来近岸和远洋的真核微生物多样性分布研究均具有重要的参考价值。
本研究旨在探究不同采样量对水体中真核微生物多样性评估的影响, 回答水体中真核微生物多样性是否随采样量的增加而增加, 多大采样量能够充分反映水体中的真核微生物群落结构与多样性分布特点等科学问题。
1 材料与方法
1.1 采样点的选择和样品的采集
实验所用样品系2018 年6 月搭载“华务号”科考船, 于吕宋海峡处获取, 共计5 个采样点, 其中C08(124°E, 22.333°N)、C10(122.667°E, 21.667°N)和C12(124°E, 21.667°N)站位位于吕宋海峡东侧, 处于菲律宾海盆中; C14(120.5°E, 21°N)和C19(120.5°E,19.6°N)站位位于吕宋海峡及其西侧, 处于南海(图1)。
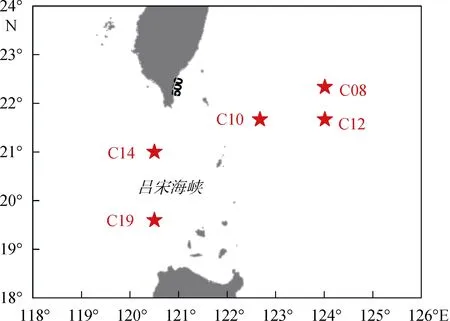
图1 采样站位图Fig. 1 Map of sampling sites
每个采样点设4 个重复, 每个重复采10 L 表层海水。海水预先用孔径为200 µm 的筛绢过滤, 再用孔径为0.22 µm 的聚碳酸酯滤膜过滤, 将滤膜保存于无RNA 酶的2 mL 冻存管, 并立即放置到液氮中保存, 带回实验室后转移至–80℃冰箱保存。采水及过滤所用的水桶、筛绢、塑料管等在每次使用后均用无菌蒸馏水冲洗2~3 遍, 排除样品之间的交叉污染。
1.2 DNA 的提取和真核微生物18S rDNA的扩增
所有样品均采用凯杰 DNA/RNA 提取试剂盒(AllPrep DNA/RNA Mini Kit, Qiagen, Germany)并按照使用说明进行DNA 的提取。最终每个样品得到1 份DNA(100 µL)。以DNA 为模板利用真核生物特异性引 物 F (5′-CCAGCA(G/C)C(C/T)GCGGTAATTCC-3′,565bp-584bp) 和 R (5′-ACTTTCGTTCTTGAT(C/T)(A/G)A-3′, 964bp-981bp)对样品进行18S V4 区的扩增, 扩增程序见文献[27], 每份DNA 设置3 个重复。为减少 PCR 过程中产生的误差, 采用高保真酶Q5(BioLabs INC., NEW ENGLAND)进行扩增。扩增产物采用1%的琼脂糖凝胶电泳进行浓度及扩增片段准确度的检测, 对在480bp 处有明显亮带的样品采用Qubit 2.0 荧光定量仪(Thermo Scientific)测定准确浓度, 并用GeneJET Gel Extraction Kit (Thermo Scientific, USA)进行纯化, 3 个重复进行合样, 用于后续测序。
1.3 高通量测序与数据处理
扩增样品由北京诺禾致源科技股份有限公司利用Illumina HiSeq 测序平台进行测序, 并采用NEB Next Ultra™ DNA Library Prep Kit for Illumina (NEB,USA) 建立样品序列库, 添加barcode 序列。序列库的质量通过Qubit 2.0 荧光定量仪 (Thermo Scientific,USA) 和Agilent Bioanalyzer 2100 system 进行测定。
对Illumina HiSeq 测序平台得到的序列信息进行拼接得到原始数据(Raw Data), 经过质控和去嵌合体得到可用于后续分析的有效数据(Clean Data)。利用USEARCH(version9.2/10.0http://drive5.com/uparse/)对有效数据进行去冗余、去单一序列和97%相似度的OTU(Operational Taxonomic Units, 可操作分类单元)聚类分析, 根据OTU 聚类结果, 对每个OTU 的代表序列与SILVA 数据库比对作物种注释, 得到对应的物种信息和基于物种的丰度分布情况, 即OTU Distribution Table(以下简称OTU Table)。
利用EstimateS (EstimateS 9.1)对OTU Table 中各OTU 丰度信息进行4 次随机重抽样, 重抽样标准为10 000 条序列, 得到各站位在10 L、20 L、30 L和40 L 时的OTU 数目和ACE 指数, 并以此为基础在 Excel 中绘制物种累积曲线、在 SPSS 中利用ANOVA 功能计算各组之间的单因素方差值。
以各个站位4个重复的OTU 丰度信息为基础利用R 语言的VEGAN 包的vegdist 功能(https://CRAN.Rproject.org/package=vegan)计算各个站位4 个重复和重复总和之间的Bray-Curtis 距离, 并以此为基础利用R 语言的ggplot2 包(https://CRAN.R-project.org/package=ggplot2)作物种组成和物种丰度的主坐标分析(PCoA, Principal Co-ordinates Analysis)。
2 结果
2.1 物种丰富度与采样量的关系
自5个站位共计20个样品的分析中获得2 066 528条有效序列, 97%相似度聚类后得到2 737 个OTU。5 个站位真核微生物的OTU 数量与采样量之间均存在正相关关系, 且每个站位的物种累积曲线均未达到饱和。分析发现, 假设各站位40 L水所获得的OTU数量为100%,则10 L 水所获得的平均OTU 数量为64.1%, 20 L 水为81.7%, 而30 L 水为92.3%(图2, 表1)。采样量越大, 可能获得的OTU 数量就越多。

图2 各站位真核微生物物种累积曲线Fig. 2 The accumulation curve of microeukaryotes collected from each station
所有站位不同采样量下所获的OTU 数量的单因素方差分析(one-way ANOVA, 方差齐性检验 P=0.108, 符合正态分布)显示, 采样量对OTU 数量存在显著影响。10 L 水所获得的OTU 数量显著少于其他各组(P=0.00~0.02); 20 L 水所获得的OTU 数量显著少于40 L 水组(P=0.017), 但与30 L 组没有显著差异; 30 L 组与40 L 组没有显著差异, 采样量对OTU数量存在显著影响。

表1 各站位10 L、20 L 及30 L 水的OTU 数量占40 L水OTU 数量的百分比/%Tab. 1 Percentage of OTU number obtained from 10 L,20 L, and 30 L seawater samples in that obtained from 40 L seawater samples
所有站位的不同采样量下的ACE 指数的单因素方差分析(方差齐性检验P=0.930, 符合正态分布)显示, 采样量对ACE 指数存在显著影响(表2)。10 L组的ACE 指数显著少于30 L 和40 L 组(P=0.001~0.006); 20 L 组的ACE 指数与30 L、40 L 组没有显著差异; 30 L 组与40 L 组没有显著差异, 采样量对ACE 指数存在显著影响。
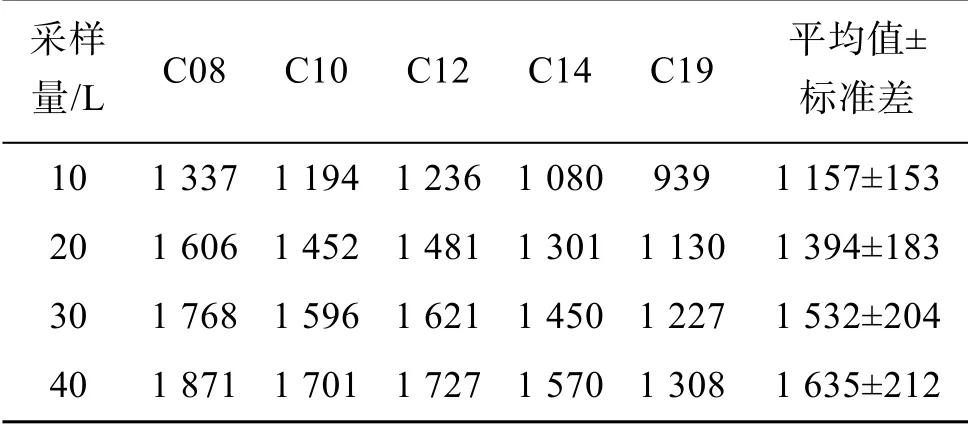
表2 各站位10 L、20 L、30 L 及40 L 水的ACE 指数Tab. 2 ACE index based on the sample sizes of 10 L, 20 L,30 L, and 40 L seawater samples
2.2 群落结构与采样量的关系
对每个站位4 个重复(即4 个10 L 组)和重复相加得到的总和(即40 L 组)的真核微生物群落结构进行比较, 以 Bray-Curtis 距离为基础的主坐标分析(PCoA)显示, 10 L 组与40 L 组之间在物种组成上重合度很高, 无明显差异(图3)。多元方差分析(Adonis)显示, 4 个10 L 组与40 L 组之间的群落差异P 值均大于0.05, 不存在显著差异。
在 5 个站位的样品中, OTUs 主要包括甲藻(Dinophyta)、纤毛虫(Ciliophora)、不等鞭毛类(Stramenopiles)、有孔虫类(Rhizaria)、Hacrobia (包括隐藻类Cryptophya 和定鞭藻类Haptophyta)、后鞭毛类(Opisthokonta)和原始色素体类(Archaeplastida)等主要类群以及其他丰度较低的OTUs(图4, 图5), 其中Dinophyta 和Ciliophora 同属于囊泡类(Alveolata)。Alveolata、Stramenopiles 和Rhizaria(SAR 类群)为主要类群, 在所有样品中的平均OTU 数量依次占总数量的63.3%、10.8%和10.0%, 平均序列数依次占总数量的70%、4.0%和11.4%。
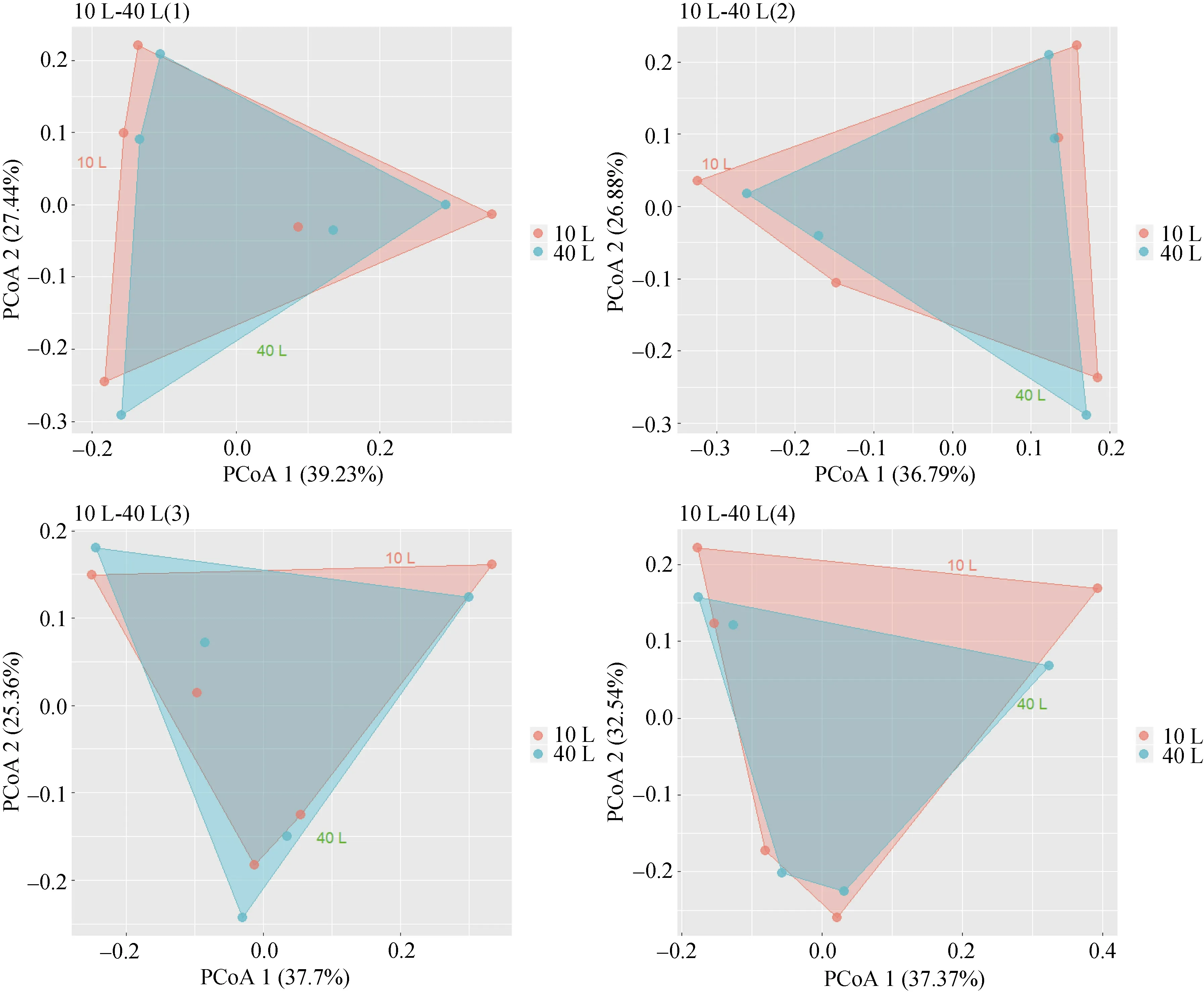
图3 四个10 L 重复和40 L 之间的群落相似性Fig. 3 Similarities between communities obtained from the 40 L seawater and each of 10 L seawater samples
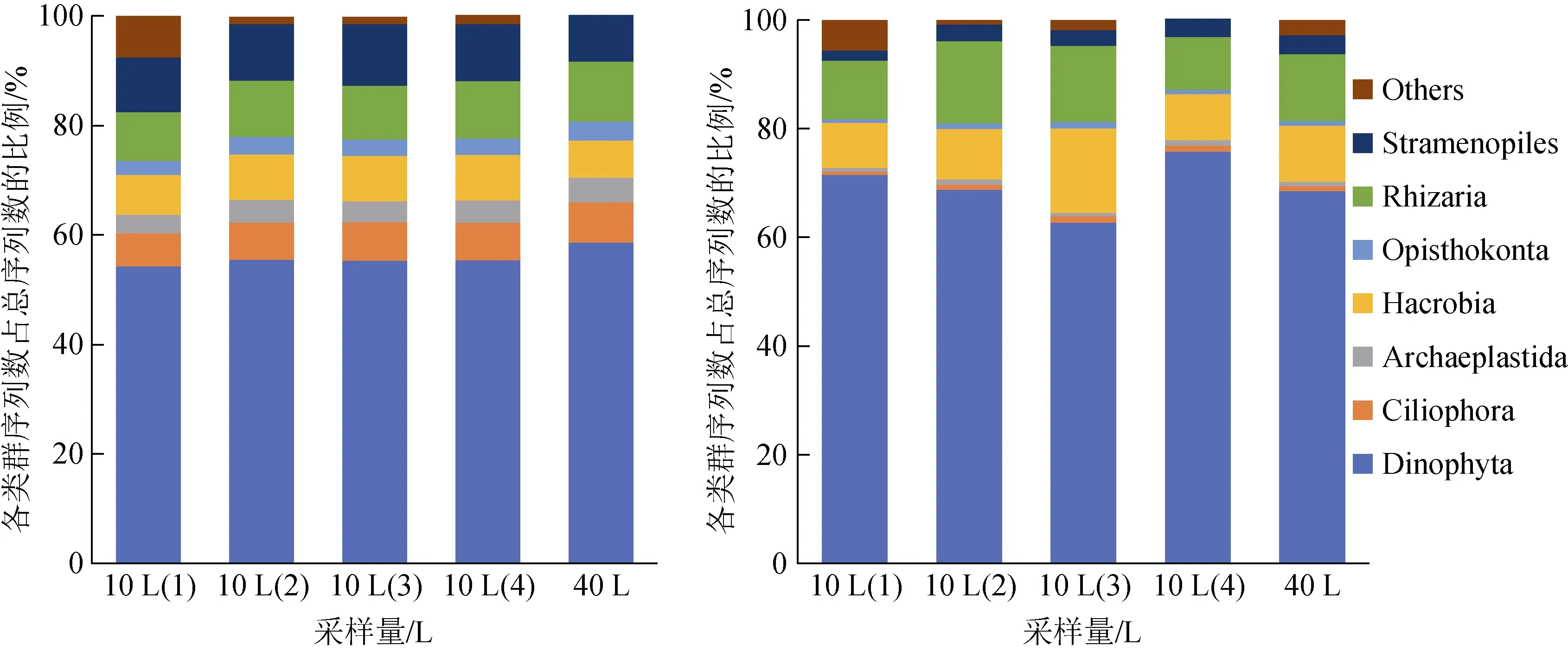
图4 所有站位主要类群的相对丰度Fig. 4 The proportions of the OTUs and sequences of major taxonomic groups
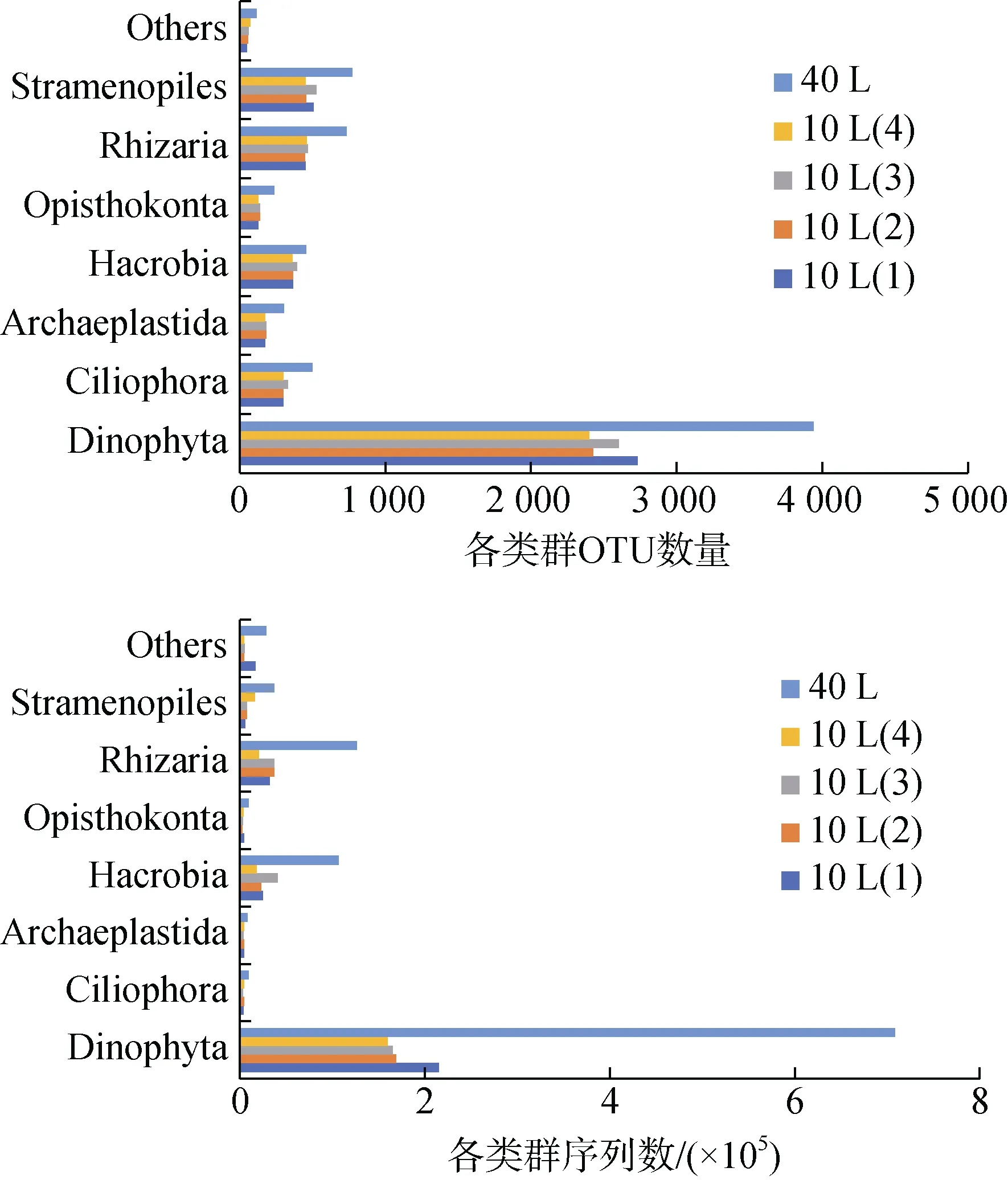
图5 所有站位主要类群的丰度Fig. 5 Number of OTUs and sequences of major taxonomic groups
2.3 优势种和稀有种与采样量的关系
序列数占该样品总序列数1%及以上的OTU 被划为优势OTU, 序列数占该样品总序列数0.1%及以下的OTU 被划为稀有OTU。
在数量上, 优势OTU 数量随采样量的增加无明显变化, 稀有OTU 的数量随采样量的增加而增加(表3)。在数量占比上, 总和(40 L)中优势OTU 占OTU 总数目的比值小于各个重复(10 L), 且优势OTU 占OTU 总数目的比值在各站位不同重复之间无显著差异, 在各个重复与总和之间存在显著差异(P=0.01~0.04); 总和中稀有OTU 占OTU 总数目的比值大于各个重复, 且稀有OTU 占OTU 总数目的比值在同一站位不同重复之间无显著差异, 但不同重复与总和之间均有显著差异(P=0.00~0.02)。
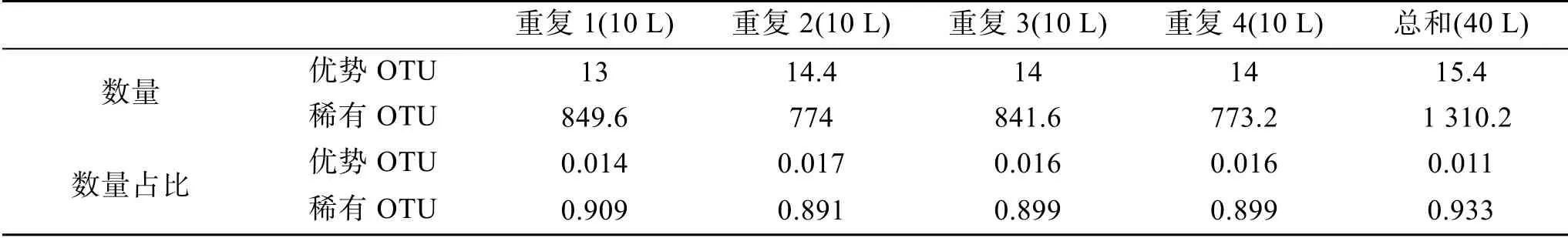
表3 优势OTU 和稀有OTU 数量和数量占比Tab. 3 Number and proportion of abundant and rare OTUs among the total OTUs
2.4 采样量对不同粒级生物多样性的影响
选取鉴定到种且匹配度为100% 的OTUs, 根据对应物种的个体大小对其分组(表4)。分析各组丰度与采样量之间的关系, 发现不同个体大小的 OTU在不同重复和重复总和中均有分布且数量无明显变化(图6), 未检获到采样量对不同个体大小的OTU 丰度的影响。

表4 OTU 及其对应真核微生物类群(属水平)粒径Tab. 4 Body sizes of each OTU based on the best BLAST hit at the genus level
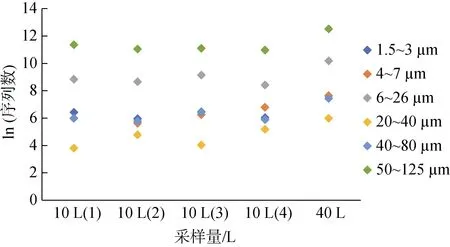
图6 四个10 L 重复和40 L 不同粒级生物序列数Fig. 6 Sequence number of taxa with different body sizes obtained from the 40 L seawater and each of the 10 L seawater samples
3 讨论
关于采样量影响微生物多样性的研究多集中于异质化明显的海底沉积物[28-29], 研究发现, 采样量对沉积物中的细菌、真菌以及小型底栖动物的多样性和群落组成有显著影响[30-32]。在Nascimento 等人的研究中, 随着采样量的增加, 原生生物的群落组成趋于稳定, 表明在评估沉积物中的生物 β-多样性时, 采样量是关键影响因素[28]。
与沉积物不同, 关于采样量对异质性低的水体中真核微生物的影响的研究较少。已有的海洋水体真核微生物多样性研究中, 近岸站位的采样量多至40 L[19], 少则300 mL[9], 这与采样地的水体浑浊度和真核微生物丰度有关[33]。通常情况下, 采样量越大,可能获得的真核微生物物种越多。本研究发现, 随着采样量增加, 吕宋海峡水体中的真核微生物OTU 数量和ACE 指数显著增加, 但当采样量达到20 L 时,ACE 指数与采样量之间不再存在显著相关性, 20 L组的OTU 数量与30 L 组也无显著差异, 而且随采样量增加, 主要类群的种类和相对丰富度比例即真核微生物群落结构并无显著变化。
除了考虑采样量的大小, 野外采样时还需考虑采样时间对样品质量的影响。当采样地水体较混浊时, 滤膜易被堵塞, 将极大地延长过滤时间。而一旦过滤时间过长, 会造成水体中DNA 和RNA 的过多降解, 因此, 野外采样过滤时间一般控制在30 分钟之内[34]。在实际采样过程中, 应同时考虑样品过滤时间, 根据采样地水体的浑浊程度和真核微生物丰度确定合适的采样量。
在本研究中, 各站位不同重复的丰富OTU 数量稳定, 各重复中丰富OTU 数目占全部数目的比例随采样量的增加而降低, 而稀有种OTU 数占全部OTU数量的比例随着采样量增加而增加, 表明优势种更易检获, 小采样量时已基本检获了全部的优势种, 随着采样量增加, 更多的稀有种被检获。微生物群落一般由少数优势种和大量稀有种构成[19], 稀有种因其庞大的数量和高度的多样性很难被全部检测到[35]。
与传统分类学方法相比, 高通量测序能够高效地获取环境中的微生物多样性, 可检测到因丰度较小而难以观察到的稀有种[36]。鉴于其高度的灵敏性,PCR 过程和数据处理的细微差别能够造成截然不同的分类结果, 因此, 在评估样品的真核微生物多样性时, 选择一个可信的实验流程和分类标准尤为重要[20]。首先, 本研究选用的扩增引物为真核生物通用引物, 扩增区域为编码真核生物核糖体小亚基RNA V4 区的DNA, 该区域较为保守, 能够保证属级水平的精确鉴定[27]。其次, 为了获得更全面的真核微生物物种信息, 每个重复样品设置三个PCR 重复, 扩增完成后合并PCR 产物并送测。在后期数据处理时, 选择 97%相似度聚类[37], 以保证获得真实物种信息,同时减少因更高相似度产生的单一序列比例, 由此提高了分类效率。
4 总结
增加采水量可检获更多的OTU, 但当采水量达到20 L 时, OTUs 增加的速率减缓, 与更大体积水样检获的群落多样性指数无显著差异。随着采水量增加, 更多的稀有OTUs 被检获, 而丰富的OTUs 数量变化不显著, 采水量对真核微生物群落结构没有显著影响。综合考虑样品的可得性和现场处理时间的影响, 采集20 L 水所获得的物种丰富度和群落构成可基本反映这一寡营养海域的真核微生物多样性。
致谢: 中国科学院海洋研究所南峰、任强和黄平平协助样品采集, 谨致谢忱。
