乙酸胁迫下东方伊萨酵母的蛋白组学分析
2020-10-09李英迪吴祖芳翁佩芳
李英迪 吴祖芳 翁佩芳
(宁波大学食品与药学学院,浙江 宁波 315800)
果酒是由水果、花卉和草药等原料酿造而成的酒精饮料。由于酵母菌在果酒发酵中发挥不同的作用,现代酿酒工业已采用多菌种发酵研究果酒发酵进程[1]。酿酒酵母(Saccharomyces cerevisiae)普遍应用于果酒发酵和酒精生产,而非酿酒酵母(Non-Saccharomyces cerevisiae)通常通过产生影响葡萄酒香气和结构的代谢副产物来增加葡萄酒的感官特性[1-2]。近年来,非酿酒酵母在果酒发酵中的影响已引起学者的广泛关注[3-5]。酿酒酵母和非酿酒酵母的混合发酵能够提高葡萄酒的品质[6-7]。其中,东方伊萨酵母(Issatchenkio orientalis)是一种非传统的酿酒酵母,最初从热带水果和食物中分离出来。I. orientalis具有多种耐受特性(如乙酸、乙醇、钠盐、高温),而且可以在高胁迫条件下生产低成本生物乙醇[8-10]。前人研究报道了I. orientalis在生物乙醇和琥珀酸生产中对多种胁迫条件的耐受性[11],但在乙酸胁迫下分子水平上的研究较少,其潜在的耐受机制尚不清楚。
前期果酒发酵研究获得了一株风味优良的非酿酒酵母I.orientalis(简写Io 166),并从转录组水平上对Io 166 响应乙醇压力的分子机制进行研究[12]。但鲜见Io 166 对胁迫(如低乙酸和盐耐受性)相关的蛋白质研究。基于串联质谱标签(tandem mass tags,TMT)的定量蛋白质组学已广泛使用,并被证明是量化蛋白质表达水平的可靠方法[13]。本试验采用TMT技术研究乙酸胁迫条件下Io 166 的耐受机制,讨论一系列功能类别和代谢途径中差异表达的蛋白质,以期了解其在蛋白质水平上的耐受机制,在发酵过程中指导传统的酿酒酵母,提高耐受乙酸胁迫的能力。
1 材料与方法
1.1 材料与试剂
东方伊萨酵母Io 166,由宁波大学食品生物技术实验室保存。
胰酶,美国Promega 公司;乙腈,美国Fisher Chemical 公司;三氟乙酸,上海Sigma-Aldrich 公司;甲酸,美国Fluka 公司;碘代乙酰胺、二硫苏糖醇、尿素、三氯乙酸、三乙基碳酸氢铵,美国Sigma 公司;蛋白酶抑制剂,上海Calbiochem 公司;BCA 试剂盒,上海碧云天公司;TMT 试剂盒购于美国Thermo 公司。
1.2 主要仪器与设备
300 Extend C18 色谱柱,美国Agilent 公司;Easy nLC 1000 液相、NanoDrop 2000C、Acclaim PepMap 100反相预柱、Acclaim PepMap RSLC 反相分析柱、Q ExactiveTMPlus 质谱仪,美国Thermo Scientific 公司。
1.3 试验方法
1.3.1 菌种培养及生长曲线的测定 取实验室甘油保藏的Io 166 菌种,在酵母膏胨葡萄糖培养基(yeast extract peptone dextrose medium,YPD)培养基(1%酵母提取物、2%琼脂、2%蛋白胨和2%葡萄糖,pH 值5.5)上划线分离,于培养箱中30℃恒温培养48 h。挑一环单菌落接种到50 mL YPD 液体培养基(去掉2%琼脂,pH 值5.5)中,30℃条件下150 r·min-1摇床培养24 h 后保存于4℃冰箱。取1 mL 已活化的Io 166 种子液接种到100 mL 新鲜的YPD 培养基中,30℃条件下150 r·min-1摇床培养8 h 至生长指数期中期,然后加入乙酸并使其最终浓度为110 mmol·L-1,再继续培养4 h。以未添加乙酸为对照组(CK),每处理进行3 次生物学重复。每隔2 h 测定培养液的OD600,绘制处理组和对照组Io 166 的生长曲线。
1.3.2 蛋白质的提取 称取适量样品至液氮预冷的研钵中研磨成粉末,然后转移到5 mL 离心管中。将4倍体积的裂解缓冲液(8 mol·L-1尿素、1%TritonX-100,10 mmol·L-1二硫苏糖醇和1%蛋白酶抑制剂,pH值8.0)加入各试验组样品中,使用高强度超声处理器在冰上超声处理3 次,然后4℃条件下20 000×g离心10 min 以除去剩余的碎片,再用20%冷的三氯乙酸在4℃下沉淀2 h,最后于4℃条件下12 000×g离心3 min,弃去上清液,即得发酵粗蛋白质。提取的粗蛋白质用预冷的丙酮洗涤3 次后重新溶解于8 mol·L-1尿素中,并根据BCA 试剂盒的说明测定蛋白质浓度。
1.3.3 胰酶酶解 参照王炼炼[14]、李正秀[15]的方法。添加二硫苏糖醇使蛋白质溶液浓度达到5 mmol·L-1,然后于56℃水浴30 min,室温下用碘乙酰胺避光烷基化15 min,使蛋白质溶液最终浓度达到11 mmol·L-1。然后加入100 mmol·L-1四乙基溴化铵稀释蛋白质样品至尿素浓度小于2 mol·L-1。最后以50∶1的蛋白质与胰蛋白酶质量比加入胰蛋白酶,酶解过夜后再以1∶100 的胰蛋白酶与蛋白质质量比,继续酶解4 h。
1.3.4 TMT 标记 参照綦丹丹[16]的方法。胰蛋白酶酶解后,将肽段用Strata X C18 SPE 柱(Phenomenex)脱盐并冷冻真空干燥。将冷冻干燥后的肽在0.5 mol·L-1四乙基溴化铵中重构,并按照TMT 试剂盒说明书进行标记(表1)。将TMT 试剂解冻并溶解在乙腈中,然后在室温下与肽混合温育2 h,最后脱盐并冷冻真空干燥。

表1 样品标记信息Table1 Sample marker information
1.3.5 高效液相色谱法(high performace liquid chromatography,HPLC)分级分离 反相HPLC 高pH值(9.0)条件下将肽段分级,使用300 Extend C18(5 μm×4.6 mm,250 mm)色谱柱。具体操作如下:首先将肽段用8%~32%乙腈(pH 值9.0)梯度在1 h 内分级成60 个级分,然后将肽段合成18 个级分并冷冻真空干燥,备用。
1.3.6 液相色谱-质谱联用分析 参照徐王磊[17]的方法。将肽段溶解在HPLC A 相流动相[0.1%∶2%(v/v) 的甲酸和乙腈水溶液]中,分离采用超高效液相EASY-nLC 1000 系统操作。B 相流动相为甲酸和乙腈水溶液(1∶9,v/v)。HPLC 洗脱梯度参数见表2。肽段经超高效液相色谱系统分离、NSI 离子源电离后,进入Q ExactiveTMPlus 质谱分析。

表2 高效液相色谱洗脱梯度参数Table2 HPLC elution gradient parameters
1.3.7 数据统计及差异表达蛋白的生物信息学分析使用Maxquant 搜索引擎(v.1.5.2.8)处理得到的二级质谱数据。数据库为转录组数据库(NCBI accession number: GCA_000764455.1),增添反库用来算出任意匹配导致的错误发现率(false discovery rate,FDR),同时在数据库中增加常见的污染库,目的是为了排除污染蛋白对检测结果造成的差异。相关检索参数见表3。使用Blast 2 GO 程序和Pathway Tools(E-value≤10-5)对Io 166 蛋白分别进行GO(Gene Ontology)功能注释和KEGG(Kyoto Encyclopedia of genes and genomes)代谢途径注释。以差异倍数(fold change,FC)≥1.2 且P≤0.05 作为标准来筛选差异表达蛋白。用STRING 10.5进行蛋白互作(protein-protein interaction,PPI)网络分析差异表达蛋白质之间的关系。

表3 检索参数Table3 Search parameters
2 结果与分析
2.1 乙酸处理对酵母菌生长的影响
正常培养到8 h 时添加110 mmol·L-1乙酸为处理组,以未加乙酸为对照组,比较在YPD 培养基中的细胞生长。由图1可知,乙酸加入初期,Io 166 生长受到抑制,但随着培养时间的延长,Io 166 细胞逐渐适应了乙酸胁迫,生长逐渐恢复,说明Io 166 对乙酸胁迫产生了一定的抵抗。
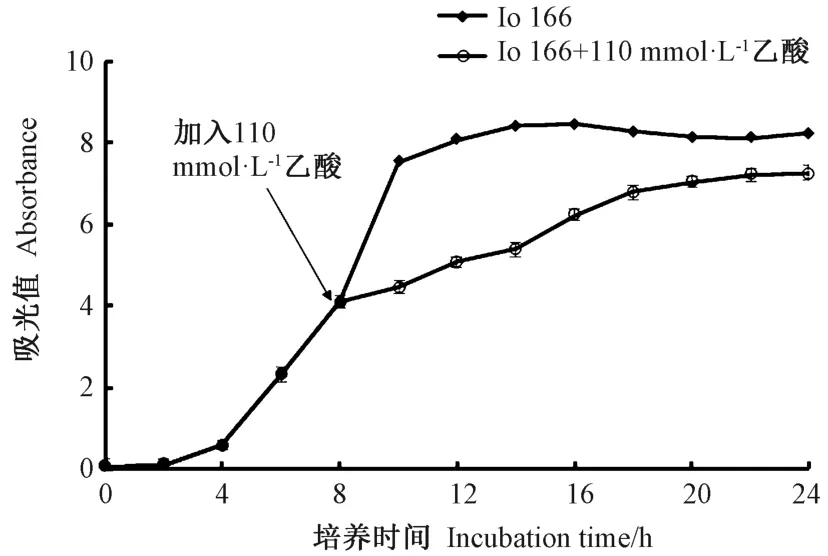
图1 不同培养条件下Io 166 的生长曲线Fig.1 Growth curves of Io 166 under different culture conditions
2.2 质谱质控检测结果
检验所有定性肽段的质量偏移(mass error)(图2-A),结果显示,质量偏移以0 作为中轴,且聚集在低于10×10-6的范围内,表明质量误差符合要求。此外,肽段长度大多分布在8 ~20 个氨基酸残基之间(图2-B),符合胰酶消化肽段的规律,由此说明样品制备符合要求。对分级脱盐合并后的组分进行质谱分析搜库,以PSM FDR≤0.01 和Protein FDR≤0.01 为标准进行数据筛选,共得到25 407 个肽段,标记效率为98.44%。
2.3 基于定量信息的蛋白质聚类分析

图2 质谱数据质控检测结果Fig.2 Quality control test results of mass spectrometry data
蛋白质依据其定量信息(ratio 值)均分为4 个蛋白组(图3):Q1(0 ~25%)、Q2(25%~50%)、Q3(50%~75%)和Q4(75%~100%)。根据这种分类方法,进行下游的聚类分析,包括基于GO 富集的聚类分析,基于蛋白结构域及KEGG 通路的聚类分析。

图3 蛋白质定量信息的分布情况Fig.3 Distribution of protein quantitative information
2.4 乙酸胁迫下Io 166 差异表达蛋白的生物信息学分析
前期研究表明,FC 是基于1.2 ~1.5 倍的变化阈值[18-19],本试验使用1.2 倍定义蛋白质丰度的显著变化。根据标准(FC≥1.2,P≤0.05),461 个蛋白质显示差异表达,包括327 个上调蛋白质和134 个下调蛋白质。GO 注释(图4)和KEGG 分析(图5)结果表明,差异表达蛋白主要分布在代谢过程、对刺激的反应、细胞器和催化活性中。
图4显示了GO 在生物进程、细胞组分和分子功能中的分布,以及每个类别中分布的差异蛋白比例。其中,生物进程中代谢进程34%、细胞进程27%、单一生物进程24%;细胞组分中细胞37%、细胞器23%、细胞膜19%、大分子复合物18%;分子功能中催化活性44%、结合41%。GO 分类表明,乙酸胁迫明显影响了代谢进程和催化活性。

图4 Io 166 差异表达蛋白GO 分类注释Fig.4 GO classification annotation of differentially expressed proteins in Io 166
差异表达蛋白KEGG 注释用于直系同源分配,KOBAS 软件检测KEGG 通路中差异蛋白的统计富集。KEGG 代谢通路(图5)显示了代谢/生物合成途径中差异蛋白的分布。次生代谢物生物合成、抗生素生物合成、氨基酸生物合成和碳代谢是差异表达蛋白KEGG 主要途径。其中,氨基酸的生物合成及某些氨基酸的代谢对耐受乙酸起关键作用。过氧化物酶(Ko03781)、糖酵解(Ko00129)和柠檬酸循环(Ko00164)中的差异表达蛋白存在显著变化。
2.5 响应乙酸胁迫的差异表达蛋白的互作网络分析
由图6可知,部分关键的差异表达蛋白构成一个结构复杂的互作网络,通常具有相似功能的蛋白之间进行调节。蛋白质节点之间的彩色线表示各种类型的相互作用,与其他蛋白质相互作用的蛋白在交互网络中很重要。热激蛋白HSP70 及其伴侣蛋白MDJ1、SSA3、FES1、JAC1、SSC1、SSQ1,HSP40 及其伴侣蛋白YDJ1、APJ1,以 及HSPA1、HSP78、HSP12、HSP60、HSP10、HSP104 显著上调,均在响应乙酸胁迫中起到关键作用。一些重要的差异蛋白表达信息与谷胱甘肽、抗氧化、硫氧还蛋白、色氨酸、热激蛋白等相关,如表4所示。
3 讨论
乙酸胁迫后的一个显著现象是含硫氨基酸生物合成途径的变化,观察到谷胱甘肽(GSH)如GSTY 的合成显著增加,并且诱导了几种具有抗氧化特性的蛋白质的合成。作为细胞内一种重要的调节代谢的物质,GSH 参与三羧酸循环(tricarboxylic acid cycle,TCA)及糖代谢,并激活多种酶,从而促进糖类、脂肪和蛋白质代谢。此外,GSH 在预防细胞氧化应激方面发挥主要作用,可与蛋白质的反应性巯基(蛋白质谷胱甘肽化)结合,保护其免受氧化损伤[20]。Rikhvanov 等[21]、Meng 等[22]、Kitichantaropas 等[23]指出硫氨基酸途径不会被诱导,而是在大多数应激条件下(包括氧化应激,渗透压和热休克反应)被抑制。而本研究中GSH 表达与含硫氨基酸(蛋氨酸、半胱氨酸和甲硫氨酸)生物合成途径的酶调节显著增加,表明在乙酸诱导下其存在共同作用机制。硫氧还蛋白和硫氧还蛋白还原酶作为两种Yap1p 依赖性蛋白在乙酸胁迫条件下表达量显著增加,Yap1p 已被证明是酵母适应氧化应激反应的转录调节因子[24-26],在乙酸耐受性中起重要作用。此外,几种抗氧化剂如PXP9、HYR1、CCP1 也显著上调。综上推断,GSH(SPE3、HYR1、GSTY2)和硫氧还蛋白(GRX8、AIM32、APD1)对于细胞耐受乙酸起到关键作用。
本研究结果表明,氨基酸作为主要代谢产物参与许多细胞生物合成和代谢过程;NADH 氧化、精氨酸生物合成和氨基酸代谢途径显著变化,与乙酸耐受相关的代谢物,如脯氨酸、天冬氨酸,在细胞内积累保护细胞膜免受乙酸胁迫,这与Greetham 等[27]的研究结果一致。在酿酒酵母中,Nugroho 等[28]发现脯氨酸是一种应激保护剂,能够使细胞免受冷冻、干燥、低pH 值和氧化应激损害;Liang 等[29]通过敲除有关脯氨酸生物合成的基因,发现突变型菌株较野生型菌株对胁迫更敏感,补充脯氨酸或相关基因可以恢复耐受性,说明脯氨酸在胁迫条件下对细胞有保护作用。本试验中脯氨酸表达量并未发生显著上调,这可能是由于细胞内脯氨酸量需要控制在一定水平内以耐受乙酸胁迫并避免副作用,如抑制细胞生长[26]。据报道,乙酰辅酶A的增加可以使TCA 循环活化并影响天冬氨酸和谷氨酸的积累[30-31]。本研究中与乙酰辅酶A 相关的蛋白显著上调,同时TCA 循环活跃,天冬氨酸和谷氨酸代谢增强。此外,Ohta 等[32]研究表明色氨酸是与乙醇抗性相关的代谢物,本研究结果发现色氨酸(如PXP9、KGD1)显著上调,说明色氨酸耐受乙酸与耐受乙醇的机制一致。
本试验中,HSP40、HSP78、HSP60、HSP104 和HSP70 及其伴侣蛋白在乙酸胁迫下显著上调。Pukszta 等[33]和Uzarska 等[34]指出HSP70 与伴侣蛋白SSQ1 结构类似但功能不同;Sichting 等[35]和Gowda等[36]分别指出HSP70 与伴侣蛋白SSC1、FES1 之间的相互作用,并且蛋白质的正确折叠或某些蛋白质的稳定性需要两个及以上的分子伴侣参与发挥作用。因此,需要诱导多种伴侣蛋白以响应乙酸胁迫。由于乙酸扰乱蛋白质构象并导致变性蛋白质的积累,因此多聚蛋白质介导的蛋白质修复功能对酵母耐受乙酸至关重要。此外,Von 等[37]研究表明HSP78 是线粒体基质伴侣蛋白,可形成寡聚体并在线粒体耐热性方面与HSP70 协同作用。HSP78 可防止错误折叠蛋白质的组合和蛋白质聚集体的再溶解。Doyle 等[38]指出HSP104 热休克蛋白,作为解聚酶起作用,与HSP40 和HSP70 结合,重新折叠并重新激活变性或成簇蛋白。Song 等[39]指出线粒体基质中的HSP60 是必不可少的,对于许多线粒体酶复合物的正确形成是必要的。对于某些线粒体靶向蛋白质,HSP60 在进入线粒体基质后与多肽相互作用并催化这些多肽重折叠。本研究中由乙酸胁迫诱导的许多HSP 基因存在于细胞质和线粒体中,上调的HSP 相关基因影响多个位置的细胞功能,包括促进转录因子的核功能,改善代谢过程中ATP 能量的产生,维持涉及生物合成的酶功能和分解代谢。

图6 乙酸胁迫下Io 166 蛋白的互作网络图Fig.6 The interaction network of proteins under acetic acid stress in Io 166
4 结论
本研究结果表明,乙酸胁迫初期对Io 166 生长具有一定的抑制作用,但随着培养时间的延长,抑制作用减弱,表明Io 166 对乙酸产生了一定的耐受性。蛋白质组学分析发现,乙酸胁迫下,Io 166 细胞中GSH 的合成增加,具有抗氧化特性的几种蛋白质上调,氨基酸作为主要代谢产物,其生物合成和代谢过程差异表达;HSP 在蛋白质折叠和重折叠中发挥重要作用。本研究结果从蛋白质组学角度解释了Io 166 耐受乙酸的原因,不仅对理解其分子机制具有重要意义,而且对提高果酒酿造微生物耐受乙酸发酵具有潜在的应用价值。
