基于盐胁迫转录组信息的蚕豆F-box基因家族分析
2020-09-12郝树琳陈宏伟廖芳丽李莉刘昌燕刘良军万正煌沙爱华
郝树琳,陈宏伟,廖芳丽,李莉,刘昌燕,刘良军,万正煌,沙爱华
基于盐胁迫转录组信息的蚕豆F-box基因家族分析
郝树琳1,陈宏伟2,廖芳丽3,李莉2,刘昌燕2,刘良军2,万正煌2,沙爱华1
(1长江大学农学院/主要粮食作物产业化湖北省协同创新中心,湖北荆州 434025;2湖北省农业科学院粮食作物研究所/粮食作物种质创新与遗传改良湖北省重点实验室,武汉 430064;3荆州市种子管理局,湖北荆州 434020)
【】通过生物信息学方法分析蚕豆F-box基因家族成员的分布、结构及进化,研究家族成员在不同处理时间条件下的表达模式及对盐胁迫的响应,为该类基因生物学功能和盐胁迫机制的研究提供参考。基于蚕豆盐胁迫转录组测序(RNA-seq)数据,利用NR、Swiss-prot和PFAM 3个数据库和NCBI网站,对蚕豆F-box基因进行筛选注释;利用Web Logo 3、Prot Comp 9.0、MEGA-X和MEME等软件进行保守结构域、亚细胞定位、系统进化树和Motif等生物信息学分析。基于盐胁迫转录组数据分析蚕豆(yz17134耐盐和yz17078不耐盐)F-box基因家族在盐胁迫下的差异表达模式,并采用实时荧光定量PCR技术(qRT-PCR)检测部分家族成员在16和24 h的表达情况。基于盐胁迫转录组测序数据,注释得到161个蚕豆F-box基因,均含有F-box保守结构域。根据C端结构域的不同,将其分成11个亚族:FBX、FBXFBA、FBXLRR、FBXPP2、FBXKelch、FBXTUB、FBXFBD、FBXDUF、FBXACTIN、FBXWD40和FBO。保守结构域分析表明,F-box保守基序中包含1个极度保守的色氨酸残基。比较分析蚕豆F-box家族和拟南芥F-box家族共同构建的进化树,发现同一C端结构域的基因大多聚集在一起。亚细胞定位预测结果显示,124个F-box基因定位于细胞外,37个定位于细胞核中。基因结构分析表明,蚕豆F-box家族基因的DNA序列中均无内含子,且均由UTR区和CDS区组成。基于盐胁迫转录组数据的F-box差异表达模式分析表明,蚕豆F-box基因在2个不同处理时间点上的表达各不相同,在盐处理16 h的表达较为明显。qRT-PCR分析结果表明,在F-box家族成员中,共存在5个差异基因。其中、和在盐处理16 h的表达量均上调,和在盐处理16 h的表达量均下调。蚕豆F-box基因家族注释得到161个蚕豆F-box基因,分为11个亚族。其中5个重要的F-box基因在不同盐处理时间的表达量存在差异。
蚕豆;转录组测序;F-box家族;盐胁迫;表达模式
0 引言
【研究意义】蚕豆(L)是巢菜属一年生或越年生草本植物,有生物固氮之王的美誉,具有较高经济价值[1]。在蚕豆生长发育过程中,生物和非生物胁迫等因素对蚕豆产生影响,最终导致其产量和品质下降,造成经济损失。盐胁迫是其中一种主要的非生物胁迫,严重危害蚕豆的生长发育。F-box基因广泛参与植物生长发育及响应非生物胁迫途径,如细胞蛋白质降解、激素感知和信号传导等[2-4]。因此,研究蚕豆F-box基因功能和培育蚕豆耐盐品种具有重要意义。【前人研究进展】自然条件下,植物的生长会受到内部或外部的刺激,为了应对产生的刺激,植物已经发展出多种调节机制来协调它们的生命活动。其中,泛素26s蛋白酶体系统(ubiquitin-26s proteasome system,UPS)降解蛋白是一种重要的翻译后调控机制,并在真核生物中高度保守[5]。UPS催化多种泛素与靶底物的共价连接,使底物被26S蛋白酶体识别并进行蛋白水解。泛素化主要由泛素激活酶(ubiquitin-activating enzymes,E1s)、泛素偶联酶(ubiquitin-conjugating enzymes,E2s)和泛素蛋白连接酶(ubiquitin protein ligases,E3s)3种酶来完成。其中,E3s起到关键的底物识别功能[6]。F-box蛋白作为E3s的核心成分,即SCF(Skp1-Cul1-F-box)复合物,介导不同底物的特异性降解[7]。F-box基因广泛存在于各种植物,是植物中最大、进化最快的基因家族之一,越来越多的真核F-box基因被发现[8-11]。F-box基因在不同物种间的数量相差较大。动物体内的F-box基因数量相对较少,秀丽线虫是目前发现含有F-box基因最多的动物,具有326个,而人体内仅含有38个,果蝇只含有22个[12];植物中F-box基因家族成员个数远多于动物,模式植物苜蓿和拟南芥分别含有972和694个[13-14],农作物水稻、大豆、玉米和鹰嘴豆分别含有687、509、359和285个[15-16],经济作物苹果和梨分别含有517和226个[17-18]。F-box结构域一般位于蛋白的N端,含有40—50个氨基酸,与SKP1相互作用形成SCF复合物,而C端通常是介导底物识别特异性的二级结构[14]。在哺乳动物中,根据C末端二级结构的不同,F-box蛋白可分为3类。具有LRR结构域的FBXL,具有WD40重复结构域的FBXW和具有其他结构域的FBXO[19]。在植物中,通过对拟南芥和水稻F-box蛋白的全基因组分析,发现了大量结构域,如WD40(Tryptophan-aspartic acid 40)、kelch重复序列、LRR(Leucine-rich repeat)、FBD(F-Box and BRCT domain)、环指结构、PAS/PAC(Per Arnt Sim)、PPR(pentatricopeptide repeat)和TUB(tubby)等[15]。因此,F-box蛋白可以识别多种类型的底物,在植物生长发育、生物和非生物胁迫、激素信号转导等生物学过程中发挥重要作用。UFO是植物中发现的第一个F-box蛋白,在花分生组织鉴定和花器官发育中具有重要作用[20]。随着深入研究,越来越多的F-box蛋白被发现,并且这些蛋白的功能变得多样化。在拟南芥中,许多F-box蛋白参与植物激素反应。例如,含有F-box结构域的TIR1是拟南芥的生长素受体[21]。AFB 1-3是一种生长素信号转导的F-box蛋白,可以结合类似TIR1的Aux/IAA蛋白,促进生长素的应答[22]。F-box蛋白也可调控赤霉素信号通路,例如拟南芥的SLEEPY1(SLY1)和SNEEZY(SNE),水稻中的赤霉素不敏感矮杆2(GID2)[23]。在乙烯信号转导途径中,乙烯不敏感3(EIN3)通过结合F-box1/2(EBF1/2)进行降解[24]。COI1也属F-box蛋白,它是茉莉酸(JA)信号反应的一个重要因子,也是拟南芥JA依赖性反应必需的[25]。此外,F-box蛋白LKP1/ZTL、LKP2/FKL和FKF1参与光形态发生、昼夜节律和开花时间[26]。DOR是拟南芥中与ASK14和CUL1特异性相互作用的F-box蛋白,在耐旱条件下起负调控作用[27]。水稻F-box基因与葡萄糖延迟种子萌发有关[28]。此外,早期研究中也发现了一些植物的S位点F-box基因,证明其参与花粉管伸长,甚至参与自交不亲和[29]。【本研究切入点】目前,对于蚕豆的研究多集中在生理层面,在蚕豆中对F-box基因家族的鉴定和系统分析鲜见报道。【拟解决的关键问题】本研究利用生物信息学的方法对蚕豆F-box基因家族成员进行鉴定,并对其基因结构、亚细胞定位、蛋白保守结构域及系统进化等进行分析,同时利用qRT-PCR技术对F-box家族成员响应盐胁迫的差异表达模式进行分析,为后续研究蚕豆F-box基因的功能提供一定的理论依据。
1 材料与方法
1.1 材料
蚕豆(L)种子由湖北省农业科学院杂粮研究工程中心提供。于2018年9月,选取饱满、大小均等的2个不同品种(Y134耐盐和Y078不耐盐)的蚕豆种子进行发芽试验,用0.8% NaCl处理,分别于16和24 h收集具有胚胎1/4部分子叶的材料,每个处理设置3个生物重复,材料收集后液氮速冻,-80℃保存备用。
试验于2018年在长江大学农学院耐渍油料种质资源湖北省重点实验室进行。
1.2 蚕豆总RNA的提取、文库构建及转录组测序
植物总RNA提取采用TRNzol试剂法(TRNzol Universal Reagent,天根生化科技有限公司,北京,中国)分别提取蚕豆芽期胚的总RNA,并用TGem spectrophotometer Plus(天根生化科技有限公司,北京,中国)微量分光光度计检测RNA纯度(OD260/280比值)以及RNA浓度,最后用1%琼脂糖凝胶电泳检测。检测合格的样品送到广州赛哲生物科技股份有限公司进行转录组测序,并通过De Novo方法[30]组装得到116 093条Unigene,后续分析基于此测序结果进行。
1.3 蚕豆和拟南芥F-box基因的筛选
以盐胁迫转录组数据为基础,在NR、Swiss-prot和Pfam 3个数据库中进行注释,初步注释到451个F-box相关基因,并用TBtools软件[31]批量提取相应的蛋白序列。将这些蛋白序列于NCBI进行Blast搜索比对,同时利用CDD(conserved domain database)在线工具(https://www.ncbi.nlm.nih.gov/Structure/ bwrpsb/bwrpsb.cgi)对序列的保守结构域进行预测,去除重复序列及冗余转录本后,最终得到161个具有F-box结构域的蛋白序列。根据Wang等[32]对梨F-box基因家族成员保守结构域不同的方法进行分类,拟南芥蛋白序列从Ensembl Plants(http://plants.ensembl. org/index.html)数据库中下载获得。
1.4 蚕豆F-box基因家族保守域及其进化关系分析
利用序列分析软件Clustal W对161个蚕豆F-box蛋白进行多序列比对,去除保守域之外的区域,通过在线软件Web Logo 3(https://weblogo.Threeplusone. com/)对蚕豆F-box蛋白进行保守基序分析;利用MEGA-X软件的Clustal W程序,对161个蚕豆F-box蛋白和部分功能明确的拟南芥F-box蛋白(共15个)进行多序列比对,并用最大似然法(maximum likelihood)构建系统发育树,采用Jones-Thornton- Taylor(JTT)模型算法进行估计,并进行Bootstrap分析,重复值设置为1 000。通过在线程序iTOL(https:// itol.embl.de/)可视化系统发育树。
1.5 蚕豆F-box基因家族亚细胞定位预测、基因结构及Motif分析
利用在线软件Prot Comp 9.0(http://linux.softberry. com)对161个候选F-box基因家族各成员的氨基酸序列进行亚细胞定位预测分析;根据基因组注释信息,利用TBtools软件对F-box基因的基因结构进行可视化分析;利用在线软件MEME(http://meme-suite.org/)分析蚕豆F-box基因家族成员Motif类型和排列顺序,获得基因家族Motif特点。
1.6 蚕豆F-box基因的表达模式分析与验证
蚕豆转录组测序文库中各基因片段表达量的计算采用FPKM(fragments per kilobase million)法[33]。将数据标准化后,利用TBtools软件对F-box基因在2个不同处理时间和2个不同品种间的表达绘制热图。以蚕豆2个不同处理时间的2个品种为材料,选取了5个在处理16 h上、下调明显的F-box基因,进行实时荧光定量PCR。各样品RNA采用第一链cDNA合成试剂盒(Promega,Madison,USA)反转录cDNA,选用蚕豆作内参[34],采用Primer Premier 5.0设计荧光定量引物(表1),反应体系为模板cDNA 1 μL、正/反向引物各0.5 μL(10 μmol·L-1)、2×RealUniversal PreMix(SYBR Green, blue,FP201,TIANGEN,北京,中国)10 μL和ddH2O 8 μL。PCR反应程序为95℃ 15 min;95℃ 10 s,57℃ 20 s,72℃ 30 s,40个循环。每个样品设置3次技术重复。所用仪器为美国伯乐(BIO-RAD,CA,USA)CFX Connect实时荧光定量PCR仪,采用2-△△CT方法计算相对表达量的[35]。
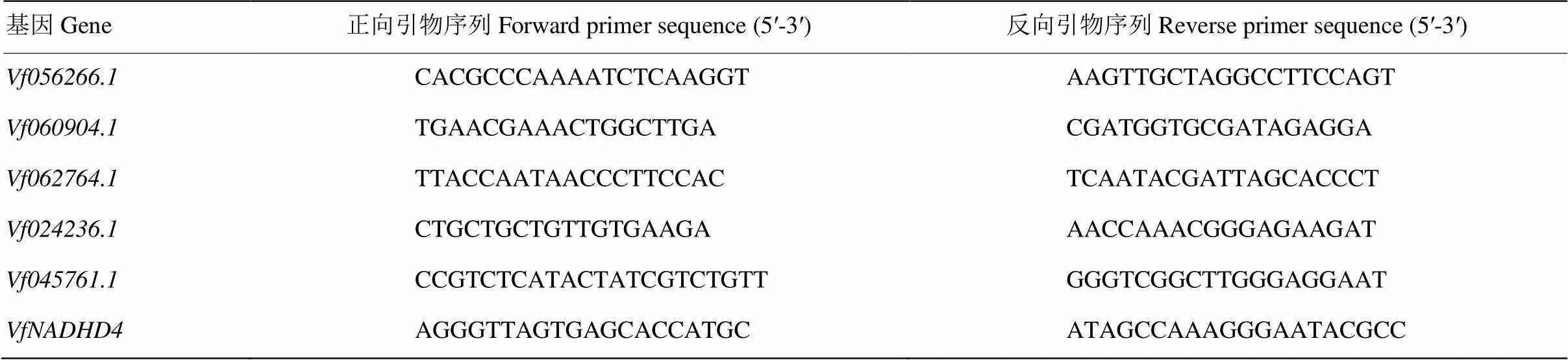
表1 实时荧光定量PCR所用的引物
2 结果
2.1 蚕豆F-box基因家族的鉴定及分类
基于盐胁迫转录组测序数据,在NR、Swiss-prot和Pfam 3个数据库中进行注释,初步注释到451个F-box相关基因,通过NCBI Blast预测,去除重复序列和冗余转录本后,最终得到161个蚕豆F-box基因。
利用SMART和CDD在线工具对所得到的161条F-box蛋白序列进行预测,并根据Wang等[18]对梨F-box基因家族的分类方法,按照每个F-box基因所包含的C端保守结构域类型,将其分为FBX、FBXFBA、FBXLRR、FBXPP2、FBXKelch、FBXTUB、FBXFBD、FBXDUF、FBXACTIN、FBXWD40和FBO 11类。其中FBX类基因64个,FBXFBA类基因27个,FBXLRR类基因19个,FBXPP2类基因8个,FBXKelch类基因16个,FBXTUB类基因7个,FBXFBD类基因10个,FBXDUF类基因2个,FBXACTIN类基因2个,FBXWD40类基因1个,FBO类基因5个。
2.2 蚕豆F-box蛋白的保守基序分析
利用序列分析软件Clustal W对161个蚕豆F-box蛋白进行多序列比对,去除保守域之外的不保守区域,对其保守结构域进行基序分析。通常F-box蛋白在N端含有40—50个氨基酸的F-box基序[18]。由图1蚕豆F-box蛋白的保守基序图可知,其约含49个氨基酸残基,包括1个极度保守的色氨酸残基(W)。此外,在F-box蛋白的保守基序中还存在其他保守氨基酸残基,如第4位的亮氨酸残基(L)、第5位的脯氨酸残基(P)、第12位的苏氨酸残基(T)、第30位的缬氨酸残基(V)。这些保守的氨基酸残基可能与色氨酸残基共同维持蚕豆F-box基序的α螺旋结构[36],进而发挥其特定的功能。
2.3 蚕豆与拟南芥F-box基因的进化关系与功能预测分析
利用MEGA-X软件构建蚕豆与拟南芥F-box蛋白家族的系统进化树,根据进化树(图2)的拓扑结构将2个物种的F-box家族蛋白进行聚类分析。研究表明,对植物的F-box蛋白进行系统发育分析时,具有相同C端结构域的F-box蛋白倾向于聚集在系统发育树的同一类群中[11]。因此,根据不同的特异性区域将F-box基因家族成员分为11个不同的亚群。FBX亚族(64)是由只含有F-box结构域的基因组成,不包含其他任何特定结构域,其成员数量多于其他子群。其他10个亚族由包含一个不可缺少的F-box结构域和其他可变结构域的基因组成,如LRR(命名为FBXLRR)、Tub(FBXTUB)、Kelch(FBXKelch)、FBA (FBXFBA)、FBD(FBXFBD)等。例如,FBXFBA和FBXFBD亚群分别表示含有FBA结构域或FBD结构域而没有其他可变域的基因。为了验证上述分类,利用蚕豆F-box全长序列与部分功能明确的拟南芥全长序列共同构建了一个系统发育树,事实上,观察到一些分组成员聚集在一起,例如FBXFBA、FBXFBD、FBXTUB和FBXPP2,这在一定程度上支持了基于C端结构域分类的方法。
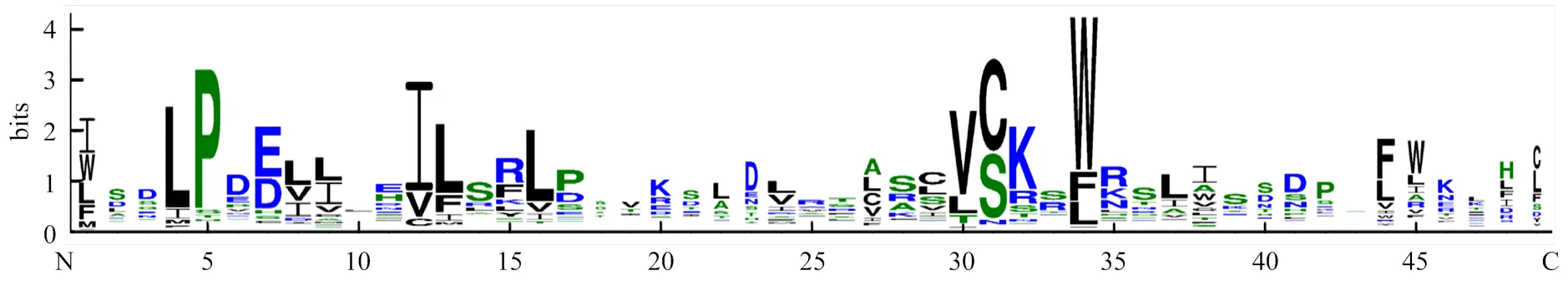
图1 蚕豆F-box蛋白保守基序
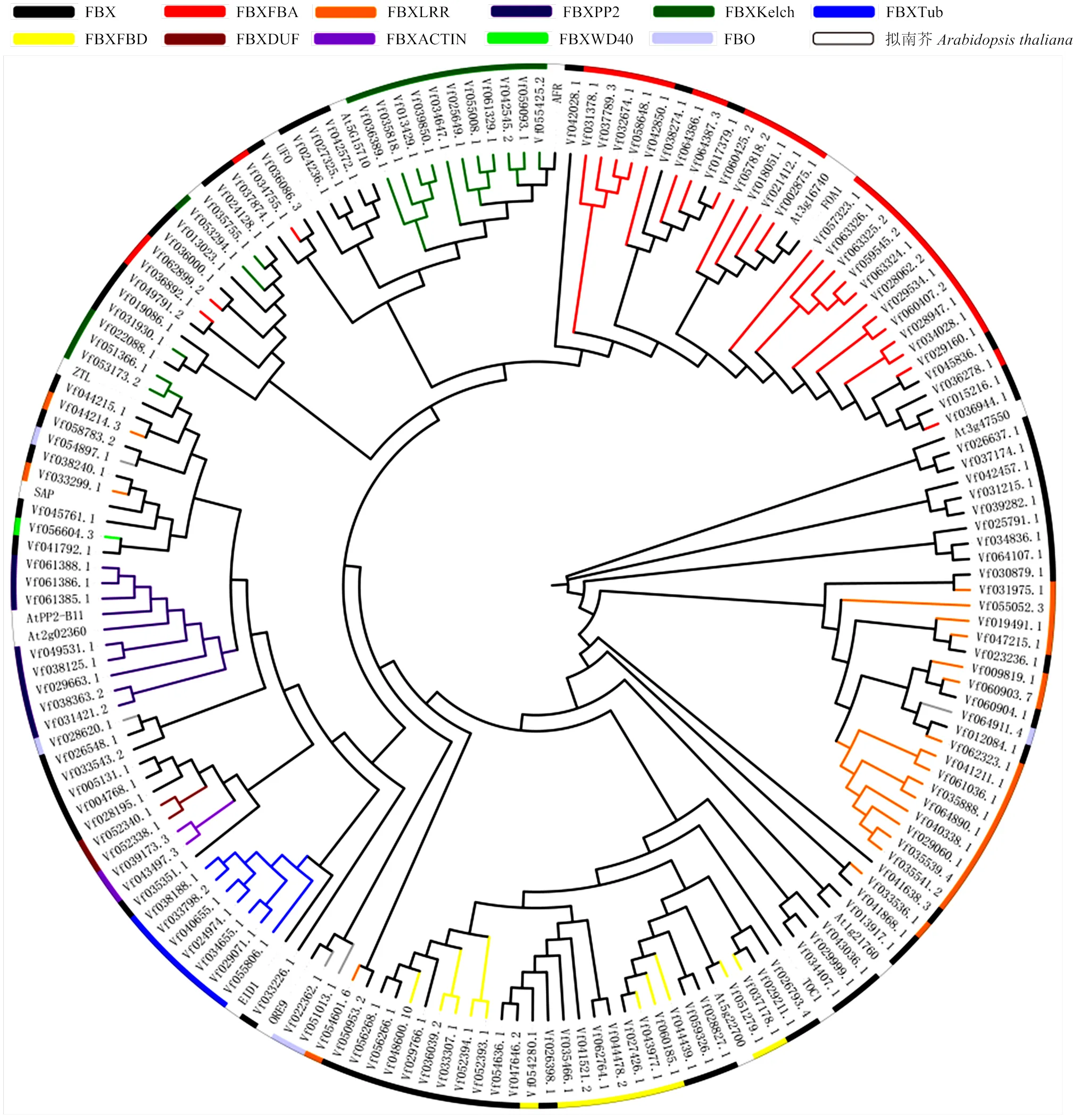
图2 蚕豆与拟南芥F-box蛋白家族的进化分析
由图2可知,一些F-box亚族与拟南芥F-box蛋白进化关系较近。例如,FBXFBA亚族中Vf057818.2、Vf018051.2、Vf021412.1、Vf002875.1与拟南芥At3g16740、FOA1位于同一进化亚枝上;FBX亚族中Vf026637.1、Vf037174.1与拟南芥At3g47550w位于同一进化亚枝上,Vf026793.4、Vf029211.1与拟南芥TOC1进化关系较近;FBXFBD亚族中Vf051279.1与拟南芥At5g22700位于同一进化亚枝上;FBXTub亚族与拟南芥EID1位于同一进化亚枝上;FBXPP2亚族与拟南芥AtPP2-B11、At2g02360位于同一进化亚枝上;FBXKelch亚族与拟南芥AFR位于同一进化亚枝,表明它们可能具有相似的功能。
2.4 蚕豆F-box基因的亚细胞定位预测
通过在线软件Prot Comp 9.0(http://linux.softberry. com)对161个蚕豆F-box个成员的氨基酸序列进行亚细胞定位预测(图3),结果显示,共有124个候选F-box基因位于细胞外,其中FBX亚族多达59个,占候选基因总数的47.58%,说明这些F-box蛋白可能参与胞外某些特定的生物学过程;37个位于细胞核中,其中FBXPP2亚族多达8个,占候选基因总数的21.62%,表明其可能通过调控细胞核基因的转录和翻译来发挥特定功能。
2.5 蚕豆F-box基因的基因结构及Motif分析
根据基因组注释信息,利用TBtools软件对F-box基因的基因结构进行可视化分析(图4),发现F-box基因家族成员无内含子结构,且每一个成员都含有UTR区和CDS区。根据F-box家族成员的进化关系可知,同一亚族的编码区长度大致相同。因此,基因结构分析在一定程度上也支持了基于C端结构域分类的方法。
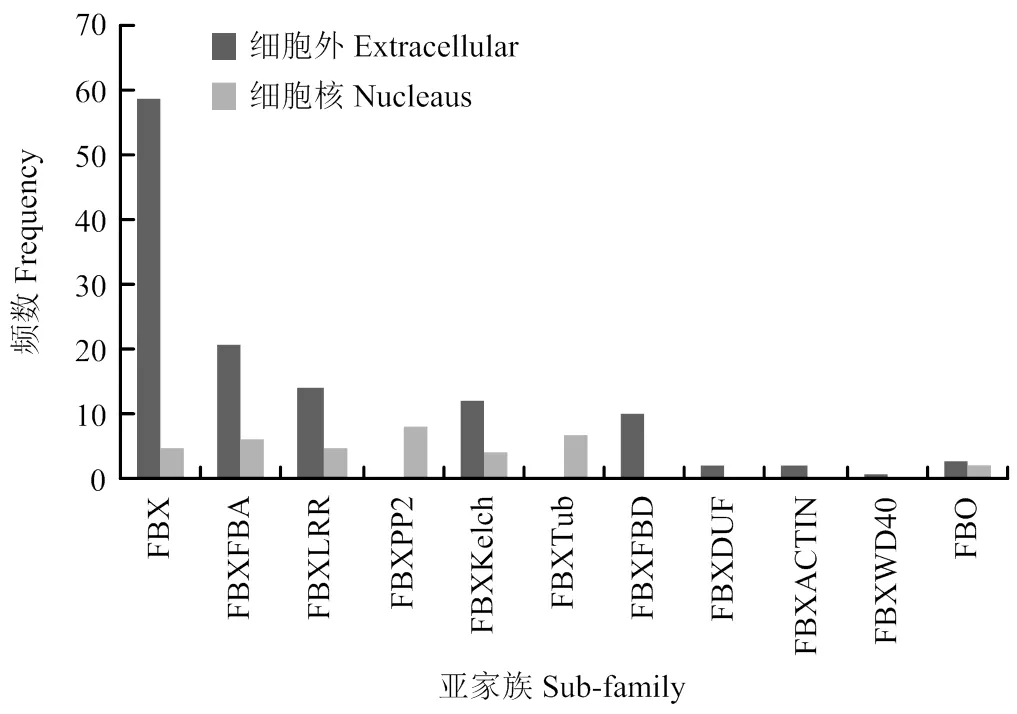
图3 蚕豆F-box基因家族的亚细胞定位预测
利用在线软件MEME(http://meme-suite.org/)分析蚕豆F-box基因家族成员基序类型和排列顺序。如图4所示,F-box基因家族预测含有20个Motif,F-box基因家族成员之间所包含的Motif数目及种类存在一定的差异。除了Vf036806.3和Vf033536.1不含Motif1外,其他均含有Motif1,相比Motif1,Motif2在蚕豆F-box基因家族中有60个成员不含该基序,Motif3在蚕豆F-box基因家族中有131个成员不含该基序。Motif4出现29次,Motif5出现17次,Motif6出现21次,Motif7出现35次,Motif8出现5次,Motif9出现11次,Motif10出现7次,Motif11出现51次,Motif12出现40次,Motif13出现39次,Motif14出现40次,Motif15出现42次,Motif16出现6次,Motif17出现6次,Motif18出现22次,Motif19出现25次,Motif20出现7次。结果表明,出现频率较高的Motif为F-box基因家族中非常重要的保守基序。对于出现频率较低的Motif,通过Smart在线分析,其功能的相关信息未知,有待日后进一步深入研究。
2.6 蚕豆F-box基因在盐处理不同时间的表达模式分析
基因的表达模式可以为基因功能提供重要的依据,从蚕豆盐胁迫转录组测序结果的基因表达数据库中,分别提取161个F-box家族基因在不同品种(Y134 T2-T1为以Y078(不耐盐)品种16 h盐处理表达量为参照,Y134(耐盐)品种16 h盐处理与之相比较的差异表达量;CK2-CK1为以Y078(不耐盐)品种16 h对照的表达量为参照,Y134(耐盐)品种16h对照与之相比较的差异表达量,同理T4-T3为以Y078(不耐盐)品种24 h盐处理表达量为参照,Y134(耐盐)品种24 h盐处理与之相比较的差异表达量;CK4-CK3为以Y078(不耐盐)品种24 h盐处理表达量为参照,Y134(耐盐)品种24 h盐处理与之相比较的差异表达量。由图5可知,随着盐处理时间的增加,蚕豆F-box基因的表达模式出现差异化。通过横向比对每个家族成员的表达差异,发现大部分成员在不同的时间节点均有高表达和低表达,一部分成员在16 h处理表达量明显升高,还有一些成员在24 h处理表达量明显降低。说明这些家族成员可能在盐胁迫前16 h中参与主要作用。为了进一步验证上述结果,并确定F-box基因在蚕豆盐胁迫下的参与情况,采用荧光定量PCR,在蚕豆2个品种和2个处理时间中进行差异表达分析(图6)。选取5个差异表达基因(在盐胁迫的2个时间点,以至少以一个时间节点的FDR<0.05,且差异倍数2倍及2倍以上的基因为差异表达基因),其中3个上调基因,2个下调基因。结果表明,5个蚕豆F-box基因的表达变化各不相同,其中在盐处理16和24 h的表达量上调;在盐处理16 h的表达量下调非常明显,盐处理24 h表达量无显著差异;在盐处理16 h上调非常明显,在盐处理24 h不明显,后者与转录组数据不符;在盐处理16 h表达量上调,盐处理24 h表达量上调不明显;在盐处理16 h下调非常明显,盐处理24 h表达量上调,这与转录组数据不符。盐处理16 h,qRT-PCR检测到的5个基因,有4个基因相对表达量与转录组数据基本一致。
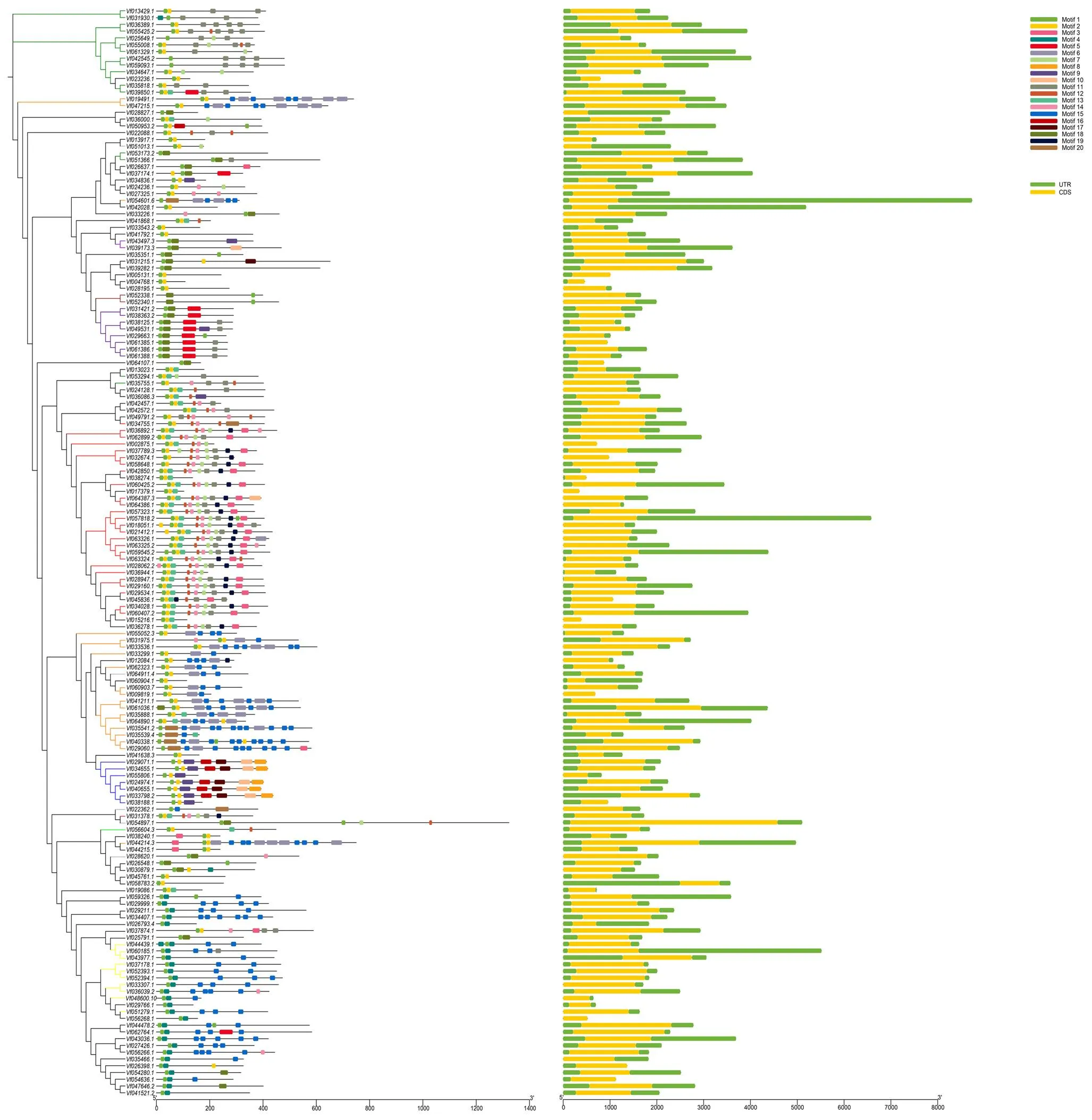
图4 蚕豆F-box基因家族结构和Motif分析
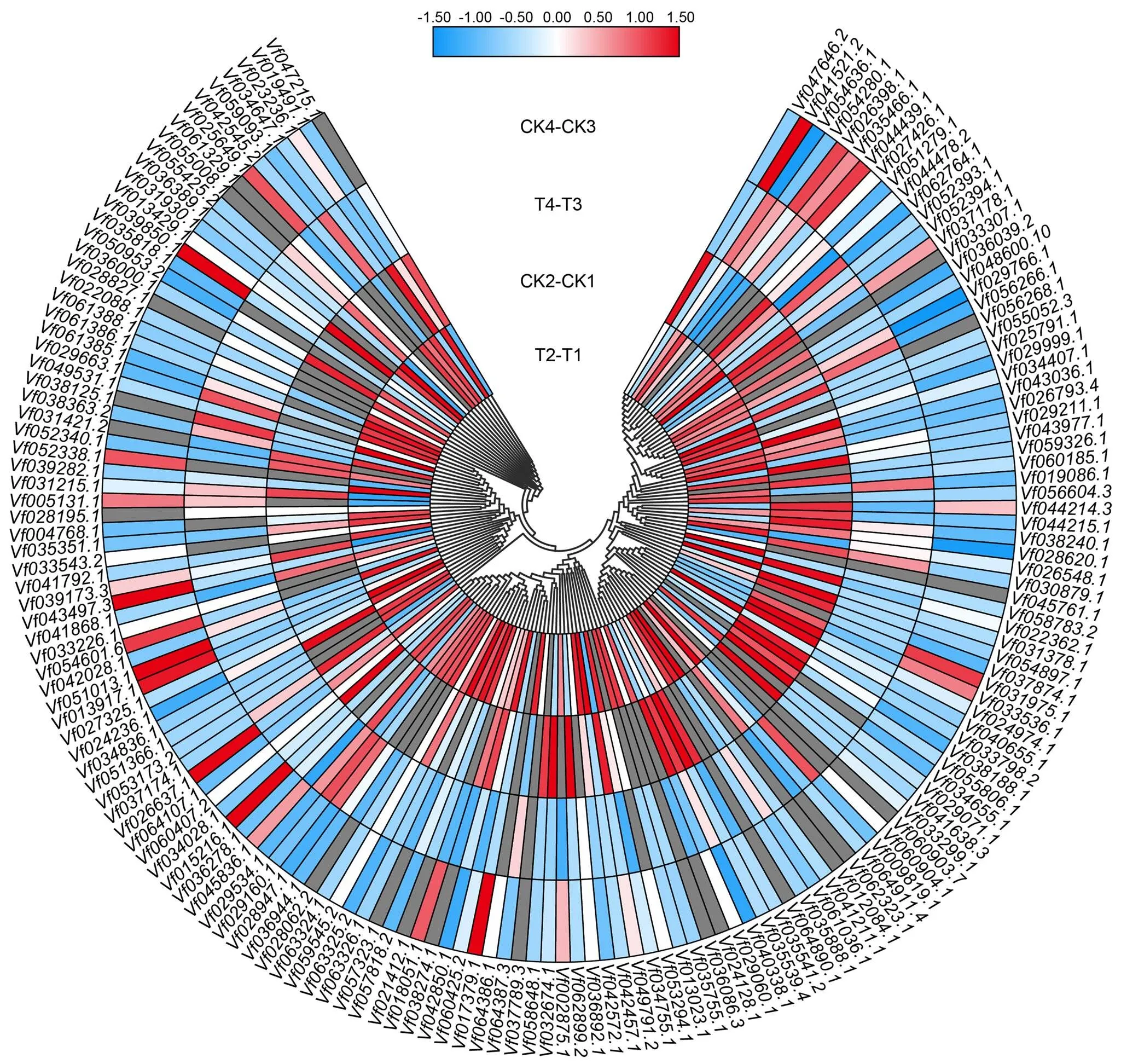
此图基于蚕豆盐胁迫转录组测序数据的计算得出,每种颜色代表相应表达量的数值,数值越大,表达量越高
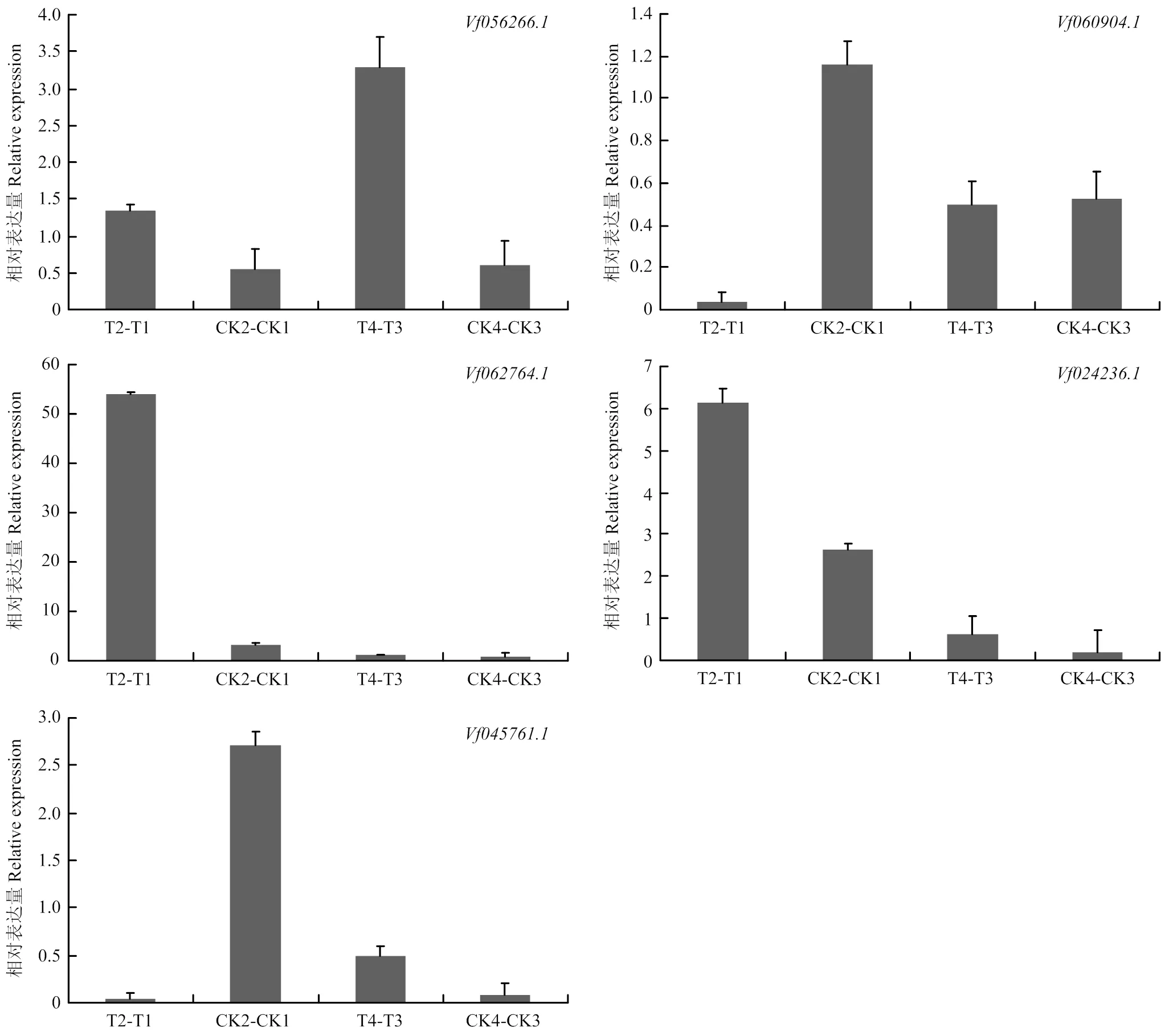
图6 蚕豆F-box基因的实时荧光定量PCR
3 讨论
在植物中,F-box作为一个较大的基因家族,它们参与了多种生物途径,在整个植物生命周期的一系列生物学过程中发挥重要作用。然而,大多数植物F-box基因的功能仍然未知,特别是在蚕豆中。由于蚕豆的全基因组序列还未公布,在很大程度上限制了其重要性状基因的研究。本研究基于蚕豆盐胁迫转录组数据,对蚕豆F-box基因家族进行系统分析。根据基因结构、保守基序、系统发育关系将其划分为不同亚族。在不同品种的不同盐胁迫下,对其表达模式进行了分析,并通过qRT-PCR进行了验证。
通过对蚕豆F-box蛋白的保守结构域进行基序分析,得出F-box基序中包含一个极度保守的色氨酸残基,这与其他植物相类似。因此,蚕豆也有可能普遍形成SCF复合物。此外,大多数已知的蚕豆C端结构域也存在于其他植物中,表明进化基因的保守性以及F-box蛋白在不同植物中的相似性。根据C端结构域的不同,蚕豆F-box蛋白可分为11个亚家族,其中最丰富的F-box蛋白属于FBX亚家族,含有未知的C端结构域。Motif分析结果表明,大多数基序也存在于其他植物中,而少数基序是豆科或蚕豆特有的。与其他已知的C端保守结构域一样,这些推测的新基序也可能在F-box蛋白与其底物的相互作用中发挥作用。那些含有豆科或蚕豆特异性基序的蛋白可能具有与豆科特异性细胞过程相关的功能,但具体功能有待深入研究。
本文使用基于C端底物相互作用域的F-box基因分类标准,再次肯定了之前基于N端F-box基序的分类,说明N端F-box基序可能与C端基序共同进化,这一点与其他植物相似[13]。此外,F-box基序生成的系统发育树与基因结构分析的结果是一致的,说明系统发育树准确地代表了蚕豆F-box基因家族的系统发育关系。然而,蚕豆F-box家族基因结构中无内含子,无内含子F-box基因在拟南芥、水稻、玉米、鹰嘴豆和胡杨中所占比例较高[16]。由亚细胞定位预测结果可知,蚕豆F-box蛋白主要存在于细胞外和细胞核中,少数在细胞质、细胞质膜和液泡中。大量的豆类F-box蛋白被预测有多个亚细胞定位。据报道,在拟南芥中检测到17个F-box蛋白大部分定位于胞腔内,单个F-box蛋白可以形成多个SCF复合物[37]。这些都说明了F-box蛋白在调控植物的生物学过程中具有多种作用。
基因表达模式与基因功能关系十分密切,通过研究蚕豆盐胁迫过程中基因表达的变化,可发现参与盐胁迫的相关功能基因。F-box基因被证实参与非生物胁迫反应,Zhao等[3]发现过表达的小麦的转基因烟草各项生理指标均高于野生型。Jia等[38]发现拟南芥F-box基因通过抑制ROS积累和影响体内Na+平衡来提高植株的耐盐性;An等[39]研究发现超表达能够提高植株的耐盐和耐旱性。本文基于蚕豆盐胁迫转录组数据,分析F-box基因在蚕豆盐胁迫下的差异表达模式,筛选出5个表达差异显著的基因,用于进一步qRT-PCR分析。qRT-PCR验证发现,有3个表达上调的差异基因和2个表达下调的差异基因,说明它们可能在蚕豆遭受盐胁迫过程中起着正向或负向调控作用。为研究蚕豆中参与盐胁迫过程中的相关F-box基因的功能和机制提供基础。
拟南芥F-box家族蛋白功能的研究报道很多,UFO参与调节花器官发育[40],AtPP2-B11对耐旱性起负调控作用,提高盐胁迫抗性,并参与ABA信号转导过程[38],AtTR1参与盐胁迫反应[41]。可根据进化树中位于同一进化亚枝中的拟南芥蛋白功能,预测研究物种蛋白的功能,严莉等[42]通过构建拟南芥与黑果枸杞的MYB转录因子的系统进化树,将位于相邻位置的黑果枸杞和拟南芥MYB转录因子列为具有相同或相似结构的转录因子,从而推测其功能的一致性。本研究所得的161个蚕豆F-box蛋白与15个功能明确的拟南芥F-box蛋白作系统进化树,根据15个拟南芥F-box蛋白进化关系的远近推测蚕豆F-box蛋白的功能。如Vf002875.1与At3g16740相邻,根据拟南芥At3g16740的功能推测Vf002875.1可能参与影响种子萌发的过程[43];推测Vf061385.1可能与拟南芥AtPP2-B11功能一致,参与盐胁迫反应[38];Vf026793.4与拟南芥TOC1进化关系较近,可能参与生物钟调节[44]。通过对拟南芥F-box蛋白保守结构域的搜索发现,含有同一C端结构域的蛋白进化到同一亚枝或进化关系较近,证明相邻或较近进化关系上的蛋白有可能具有相同或相似的功能,为研究F-box蛋白的功能提供了一种思路。此外,在后续研究中,将对F-box基因家族成员进行转基因试验,分析其在盐胁迫及其他非生物胁迫中的具体表型影响。
4 结论
基于蚕豆盐胁迫转录组测序数据,筛选注释了161个蚕豆F-box基因,且均含有F-box保守结构域。根据C末端氨基酸序列不同使家族成员多样化,分为11个亚家族,家族成员均无内含子结构,且家族成员在胞外和细胞核中均存在。、和正调控盐胁迫,和负调控盐胁迫,且盐胁迫下的F-box家族成员在不同处理时间下呈现出不同的响应。
[1] 王海飞, 关建平, 孙雪莲, 马钰, 宗绪晓. 世界蚕豆种质资源遗传多样性和相似性的ISSR分析. 中国农业科学, 2011, 44(5): 1056-1062.
Wang H F, Guan J P, Sun X L, Ma Y, Zong X X. Genetic diversity and similarity of global faba bean (L.) germplasm revealed by ISSR markers., 2011, 44(5): 1056-1062. (in Chinese)
[2] Baute J, Polyn S, De Block J, Blomme J, Van Lijsebettens M, Inzé D. F-Box protein FBX92 affects leaf size in., 2017, 58(5): 962-975.
[3] Zhao Z, Zhang G, Zhou S, Ren Y, Wang W. The improvement of salt tolerance in transgenic tobacco by overexpression of wheat F-box gene TaFBA1., 2017, 259: 71-85.
[4] Stefanowicz K, Lannoo N, Zhao Y, Eggermont L, Van Hove J, Al Atalah B, Van Damme E J. Glycan-binding F-box protein fromprotects plants frominfection., 2016, 16(1): 213-226.
[5] Smalle J, Vierstra R D. The ubiquitin 26S proteasome proteolytic pathway., 2004, 55: 555-590.
[6] Sadanandom A, Bailey M, Ewan R, Lee J, Nelis S. The ubiquitin-proteasome system: Central modifier of plant signalling., 2012, 196(1): 13-28.
[7] Ho M S, Ou C, Chan Y R, Chien C T, Pi H. The utility Fbox for protein destruction., 2008, 65(13): 1977-2000.
[8] Somers D E, Fujiwara S. Thinking outside the F-box: Novel ligands for novel receptors., 2009, 14(4): 206-213.
[9] Hua Z, Zou C, Shiu S H, Vierstra R D. Phylogenetic comparison of F-Box (FBX) gene superfamily within the plant kingdom reveals divergent evolutionary histories indicative of genomic drift., 2011, 6(1): e16219.
[10] Navarro-Quezada A, Schumann N, Quint M. Plant F-box protein evolution is determined by lineage-specific timing of major gene family expansion waves., 2013, 8(7): e68672.
[11] Xu G, Ma H, Nei M, Kong H. Evolution of F-box genes in plants: Different modes of sequence divergence and their relationships with functional diversification., 2009, 106(3): 835-840.
[12] Kipreos E T, Pagano M. The F-box protein family., 2000, 1(5): 1-7.
[13] Song J B, Wang Y X, Li H B, Li B W, Zhou Z S, Gao S, Yang Z M. The F-box family genes as key elements in response to salt, heavy mental, and drought stresses in., 2015, 15(4): 495-507.
[14] Jia Q, Xiao Z X, Wong F L, Sun S, Liang K J, Lam H M. Genome-wide analyses of the soybean F-box gene family in response to salt stress., 2017, 18(4): 818-835.
[15] JaIN M, NIJHAWAN A, ARORA R, AGARWAL P, RAY S, SHARMA P, KAPOOR S, TYAGI A K, KHURANA J P. F-Box proteins in rice. genome-wide analysis, classification, temporal and spatial gene expression during panicle and seed development, and regulation by light and abiotic stress., 2007, 143(4): 1467-1483.
[16] Cui H R, Zhang Z R, Lü W, Xu J N, Wang X Y. Genome-wide characterization and analysis of F-box protein-encoding genes in the Malus domestica genome., 2015, 290(4): 1435-1446.
[17] Gupta S, Garg V, Kant C, Bhatia S. Genome-wide survey and expression analysis of F-box genes in chickpea., 2015, 16(1): 1-15.
[18] Wang G M, Yin H, Qiao X, Tan X, Gu C, Wang B H, Cheng R, Wang Y Z, Zhang S L. F-box genes: Genome-wide expansion, evolution and their contribution to pollen growth in pear ()., 2016, 253: 164-175.
[19] 王秀燕, 孙莉萍, 张建锋, 李辉, 吕文清, 张其清. F-box蛋白家族及其功能. 生命科学, 2008, 20(5): 807-811.
Wang X Y, Sun L P, Zhang J F, Li H, Lü W Q, Zhang Q Q. F-box proteins and their functions., 2008, 20(5): 807-811. (in Chinese)
[20] Hepworth S R, Klenz J E, Haughn G W. UFO in theinflorescence apex is required for floral-meristem identity and bract suppression., 2006, 223(4): 769-778.
[21] Kepinski S, Leyser O. TheF-box protein TIR1 is an auxin receptor., 2005, 435(7041): 446-451.
[22] Walsh T A, Neal R, Merlo A O, Honma M, Hicks G R, Wolff K, Matsumura W, Davies J P. Mutations in an auxin receptor homolog AFB5 and in SGT1b confer resistance to synthetic picolinate auxins and not to 2,4-dichlorophenoxyacetic acid or indole-3-acetic acid in., 2006, 142: 542-552.
[23] Gomi K, Sasaki A, Itoh H, Ueguchi-Tanaka M, Ashikari M, Kitano H, Matsuoka M. GID2, an F-box subunit of the SCF E3 complex, specifically interacts with phosphorylated SLR1 protein and regulates the gibberellin-dependent degradation of SLR1 in rice., 2004, 37(4): 626-634.
[24] Gagne J M, Smalle J, Gingerich D J, Walker J M, Yoo S D, Yanagisawa S, Vierstra R D.EIN3-binding F-box 1 and 2 form ubiquitin-protein ligases that repress ethylene action and promote growth by directing EIN3 degradation., 2004, 101(17): 6803-6808.
[25] Sheard L B, Tan X, Mao H, Withers J, Ben-Nissan G, Hinds T R, Kobayashi Y, Hsu F F, Sharon M, Browse J, He S Y, Rizo J, Howe G A, Zheng N. Jasmonate perception by inositolphosphate-potentiated COI1-JAZ co-receptor., 2010, 468(7322): 400-405.
[26] Imaizumi T, Schultz T F, Harmon F G, Ho L A, Kay S A. FKF1 F-box protein mediates cyclic degradation of a repressor of CONSTANS in., 2005, 309(5732): 293-297.
[27] Zhang Y, Xu W Y, Li Z H, Deng X W, Wu W H, Xue Y B. F-Box protein DOR functions as a novel inhibitory factor for abscisic acid-induced stomatal closure under drought stress in., 2008, 148(4): 2121-2133.
[28] Song S, Dai X, Zhang W H. A rice F-box gene, OsFbx352, is involved in glucose-delayed seed germination in rice., 2012, 63(15): 5559-5568.
[29] Sonneveld T, Tobutt K R, Vaughan S P, Robbins T P. Loss of pollen-S function in two self-compatible selections of Prunus avium is associated with deletion/mutation of an S haplotype-specific F-Box gene., 2005, 17(1): 37-51.
[30] Grabherr M G, Haas B J, Yassour M, Levin J Z, Thompson D A, Amit I, Adiconis X, Fan L, Raychowdhury R, Zeng Q, Chen Z, Mauceli E, Hacohen N, Gnirke A, Rhind N, di Palma F, Birren B W, Nusbaum C, Lindblad- Toh K, Friedman N, Regev A. Full-length transcriptome assembly from RNA-Seq data without a reference genome., 2011, 29(7): 644-652.
[31] Chen C J, Xia R, Chen H, He Y H. TBtools, a Toolkit for biologists integrating various biological data handing tools with a user friendly interface., 2018, 3(27): 1020-1027.
[32] Wang G M, Yin H, Qiao X, Tan X, Gu C, Wang B H, Cheng R, Wang Y Z, Zhang S L. F-box genes: Genome-wide expansion, evolution and their contribution to pollen growth in pear ()., 2016, 253: 164-175.
[33] Kou Y, Qiao L, Wang Q. RETRACTED ARTICLE: Identification of core miRNA based on small RNA-seq and RNA-seq for colorectal cancer by bioinformatics., 2015, 36(4): 2249-2255.
[34] Zhang H M, Wheeler S L, Xia X, Colyvas K, Offler C E, Patrick J W. Transcript profiling identifies gene cohorts controlled by each signal regulating trans-differentiation of epidermal cells ofcotyledons to a transfer cell phenotype., 2017, 8: 2021.
[35] Livak K J, Schmittgen T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCT-method., 2001, 25(4): 402-408.
[36] Bai C, Sen P, Hofmann K, Ma L, Goebl M, Harper J W, Elledge S J. SKP1 connects cell cycle regulators to the ubiquitin proteolysis machinery through a novel motif, the F-box., 1996, 86(2): 263-274.
[37] Kuroda H, Yanagawa Y, Takahashi N, Horii Y, Matsui M. A comprehensive analysis of interaction and localization ofSKP1-like (ASK) and F-box (FBX) proteins., 2012, 7(11): e50009.
[38] Jia F Y, Wang C Y, Huang J G, Yang G D, Wu C G, Zheng C C. SCF E3 ligase PP2-B11 plays a positive role in response to salt stress in., 2015, 66(15): 4683-4697.
[39] An J P, Li R, Qu F J, You C X, Wang X F, Hao Y J. Apple F-Box protein MdMAX2 regulates plant photomorphogenesis and stress response., 2016, 7: 01685.
[40] Hepworth S R, Klenz J E, Haughn G W. UFO in theinflorescence apex is required for floral-meristem identity and bract suppression., 2006, 223(4): 769-778.
[41] 刘巧红, 杨亮, 刘志斌, 李旭锋, 杨毅. 拟南芥AtTR1在盐胁迫应答中的功能初探. 四川大学学报(自然科学版), 2016, 53(4): 895-901.
Liu Q H, Yang L, Liu Z B, Li X F, Yang Y. First exploration on protein function of Arabidopsis AtTR1 in response to salt stress., 2016, 53(4): 895-901. (in Chinese)
[42] 严莉, 王翠平, 陈建伟, 乔改霞, 李健. 基于转录组信息的黑果枸杞MYB转录因子家族分析. 中国农业科学, 2017, 50(20): 3991-4002.
Yan L, Wang C P, Chen J W, Qiao G X, Li J. Analysis of MYB Transcription Factor Family Based on Transcriptome Sequencing inMurr., 2017, 50(20): 3991-4002. (in Chinese)
[43] 段桂芳, 王立群, 李新梅, 赵福, 罗俊, 赵小英, 刘选明. 拟南芥F-box基因At3g16740的表达分析. 生命科学研究, 2013, 17(6): 486-492.
Duan G F, Wang L Q, Li X M, Zhao F, Luo J, Zhao X Y, Liu X M. Expression Analysis of F-box Gene At3g16740 in., 2013, 17(6): 486-492. (in Chinese)
[44] Más P, Kim W Y, Somers D E, Kay S A. Targeted degradation of TOC1 by ZTL modulates circadian function in., 2003, 426(6966): 567-570.
Analysis of F-Box gene family based on salt-stressed transcriptome sequencing inL
HAO ShuLin1, CHEN HongWei2, LIAO FangLi3, LI Li2, LIU ChangYan2, LIU LiangJun2, WAN ZhengHuang2, SHA Aihua1
(1College of Agriculture, Yangtze University/Hubei Collaborative Innovation Center for Grain Industry, Jingzhou 434025, Hubei;2Institute of Food Crops, Hubei Academy of Agricultural Sciences/Hubei Key Laboratory of Food Crop Germplasm and Genetic Improvement, Wuhan 430064;3Seed Authority of Jingzhou, Jingzhou 434020, Hubei)
【】The distribution structure and evolution of F-box gene family members inwere analyzed by bioinformatics method to study the expression patterns of family members and their responses to salt stress under different treatment times. It can provide a reference for the study of the biological function and the mechanism of F-box genes.【】Based on the salt-stressed transcriptome sequencing (RNA-seq) data, the NR, Swiss-Prot, PFAM and NCBI websites were used at the same time to screen and annotate the F-box genes of. The softwares including Web Logo 3, Prot Comp 9.0, MEGA-X and MEME were also applied to analyze the bioinformatics of conserved domain, subcellular localization, phylogenetic tree and Motif. Based on salt stress transcriptome data, the differential expression patterns of F-box gene family in(yz17134 salt tolerance and yz17078 salt intolerance) under salt stress were analyzed, and real-time fluorescence quantitative PCR (qrt-pcr) was used to verify the specific expression of part family members at 16 h and 24 h.】Based on salt-stressed transcriptome sequencing (RNA-seq) data, 161F-box genes were annotated and all contain F-box conserved domain. According to different C-terminal domains, they were divided into 11 subfamilies (FBX, FBXFBA, FBXLRR, FBXPP2, FBXKelch, FBXTUB, FBXFBD, FBXDUF, FBXACTIN, FBXWD40, and FBO). The analysis of the conserved domain showed that the F-box conserved Motif contained an extremely conserved tryptophan residue. By comparing and analyzing the evolutionary tree constructed by the F-box family ofand the F-box family of, it was found that most of the genes in the same C-terminal domain were clustered together. The results of subcellular localization prediction showed that 124 F-box genes were located outside the cell, and 37 F-box genes were located in the nucleus. The analysis of gene structure showed that there were no introns in the DNA sequences of the F-box family genes of, and all of them were composed of UTR zone and CDS zone. Analysis of F-box differential expression patterns based on salt-stressed transcriptome data showed that F-box gene expression inwas diverse from each other at two different processing time points, the expression was more obvious at 16 hours after salt treatment. The results of qRT-PCR analysis showed that there were five different genes in the F-box family. The expressions of,andwere all up-regulated at 16 hours after salt treatment,andwere both down-regulated at 16 hours after salt treatment.【】161F-box genes were identified by family annotation, and they were identified by family annotation, which were Evolutionarily divided into 11 subfamilies. 5 important genes were obtained through a series of bioinformatics analysis. What’s more, there exist difference among the expressions in diverse salt treatment time.
L; RNA-seq; F-box gene family; salt stress; expression pattern

10.3864/j.issn.0578-1752.2020.17.003
2019-12-19;
2020-03-05
国家重点研发计划(2019YFD1001303/2019YFD1001300)、国家现代农业产业技术体系建设项目(CARS-09)、国家食用豆产业技术体系建设专项(CARS-08-G13)、湖北省技术创新重大专项(2016ABA087,2018ABA090)
郝树琳,E-mail:shulinlin6@163.com。通信作者沙爱华,E-mail:aihuasha@163.com。通信作者万正煌,E-mail:zhwan168@163.com
(责任编辑 李莉)
