两种转基因抗虫棉L280 和L282 外源基因插入位点的分析
2020-08-29王鹏葛晓阳李付广
王鹏,葛晓阳,李付广
(中国农业科学院棉花研究所/棉花生物学国家重点实验室,河南安阳455000)
1983 年转基因烟草首次获得成功, 标志着植物转基因技术的诞生。 1997 年我国开始种植转基因抗虫棉花,并取得良好的抗虫效果,创造了巨大的经济效益。 随着转基因棉花种植面积的持续扩大,国家越来越重视转基因生物安全评价。 转基因材料的种植和审批必须要明确其分子特征,明确转基因植株T-DNA 的插入位点及侧翼序列是对转基因知识产权的保护, 可以作为监管和保护的依据,为进一步研究其分子机理提供理论基础。
基因组学技术的出现给生物学研究带来了革命性的变化,侧翼序列的测定仍然是确定基因结构的重要步骤。现阶段对侧翼序列的测定都是基于以下2 种方法,1 种是基于基因组文库连续区域或大片段克隆, 另1 种是基于聚合酶链式反应(Polymerase chain reaction,PCR)的近距离克隆。 根据这些方法的原理, 主要分为基于限制性内切酶的PCR、 基于随机引物的PCR 和基于接头的PCR[1]。使用常规的技术测定侧翼序列存在一些缺点,当扩增靶区域时,有些未知/ 已知区域中限制性内切酶位点的可用性成为限制因素[2],有些非特异性扩增产物太多[3],都会导致侧翼序列的鉴定效率与准确率下降。 由Liu 等[4]首次提出的热不对称交错PCR技术(Thermal asymmetric interlaced PCR,Tail-PCR),具有简单易行、灵敏度高、特异性好、重复性高、不涉及酶切连接等多种优势。 2007 年,Liu 等[5]对Tail-PCR 改进为了高效热不对称交错PCR(High-efficiencythermal asymmetric interlaced PCR,hiTail-PCR),在兼并引物的5’端增加了23 bp 的已知序列, 提高了Tail-PCR 的特异性与扩增效率。Wang 等[6]提出了融合引物与巢式聚合酶链式反应(Fusion primer and nested integrated PCR,FPNI-PCR),与Tail-PCR 和hiTail-PCR 原理类似,但效率更高、特异性更好、更加灵敏和准确。随着技术的改进提高, 对于已经创建了大型T-DNA 插入文库的生物,如水稻和拟南芥[7],以及转基因棉花不断发展的今天,1 种准确高效的方法具有重要的意义。
本研究通过FPNI-PCR 对cry2Ab4 转基因棉花材料L280 及cry2Ab4、vip3Aa11 双价转基因棉花材料L282 的T-DNA 在基因组上的插入位置进行测定分析。cry2Ab4 与vip3Aa11 两个外源杀虫基因均来自于苏云金芽胞杆菌(Bacillus thuringiensis,Bt),分别编码杀虫晶体蛋白和营养期杀虫蛋白,转基因植株对鳞翅目害虫如小地老虎、棉铃虫等具有较好的杀虫效果,对2 种转基因抗虫棉侧翼序列进行测定,为后续科研试验、专利保护以及安全评价等提供了重要的分子理论基础。
1 材料与方法
1.1 试验材料与试剂
转cry2Ab4 基因抗虫棉L280 与转cry2Ab4、vip3Aa11 双价抗虫棉L282 由本实验室通过农杆菌介导转化棉花下胚轴的方法获得,通过检测为稳定遗传的T5转基因材料。 Ex-Taq DNA 聚合酶、DNA marker、pMD18-T 载体等购自宝生物工程(大连)有限公司;PCR 产物回收试剂盒、琼脂糖凝胶回收试剂盒、DH5α 感受态菌株等购自全式金生物技术(北京)有限公司,PCR 引物的合成和克隆片段的测序由生工生物工程(上海)有限公司完成。
1.2 DNA 的提取
本研究对棉花基因组DNA 的完整性要求较高,参考李忠旺等[8]的CTAB 法提取DNA,注意在提取的过程中不要剧烈晃动,将提取的DNA 于4 ℃保存待用。
1.3 FPNI-PCR 引物及验证引物
参考Wang 等[7]在棉花基因组上设计9 个融合引物FP1-FP9、及2 个特定引物FSP1、FSP2。 根据转化时用到的植物表达载体pCAMBIA2300 的左边界(Left border,LB)和右边界(Right border,RB)的序列,由Primer Premier 5.0 软件设计, 分别设计3 对引物为:LSP1/RSP1、LSP2/RSP2、LSP3/RSP3。此外还分别设计验证L280 与L282 转基因植株T-DNA 完整性的引物各3 对:L280-a1/L280-b1、L280-a2/L280-b2、L280-a3/L280-b3;L282-a1/L282-b1、L282-a2/L282-b2、L282-a3/L282-b3 引物具体序列见表1。

表1 FPNI-PCR 的引物序列

表1 (续)
1.4 FPNI-PCR 扩增体系与反应条件
FPNI-PCR 扩增体系和扩增程序参考文献[7],具体体系与程序见表2。 第一轮PCR 以基因组DNA 为模板,分别将左边界引物LSP1 与9 个融合引物FP、 右边界引物RSP1 与9 个融合引物FP 配对,共18 对引物,按照第一轮PCR 程序进行扩增;第二轮PCR 分别以第一轮PCR 的18 个产 物 (1 μL) 作 为 模 板, 左 边 界 引 物 对 使 用LSP2/FSP1,右边界引物对使用RSP2/FSP1,按照第二轮PCR 程序进行扩增; 将第二轮PCR 的产物分别稀释50 倍,各取1 μL 作为模板,左边界引物对使用LSP3/FSP2, 右边界引物对使用RSP3/FSP2, 按照第三轮PCR 程序进行扩增,共计18 个产物,三轮PCR 结束后将有条带的送去测序,将测序结果分别与载体和棉花基因组进行比对分析。
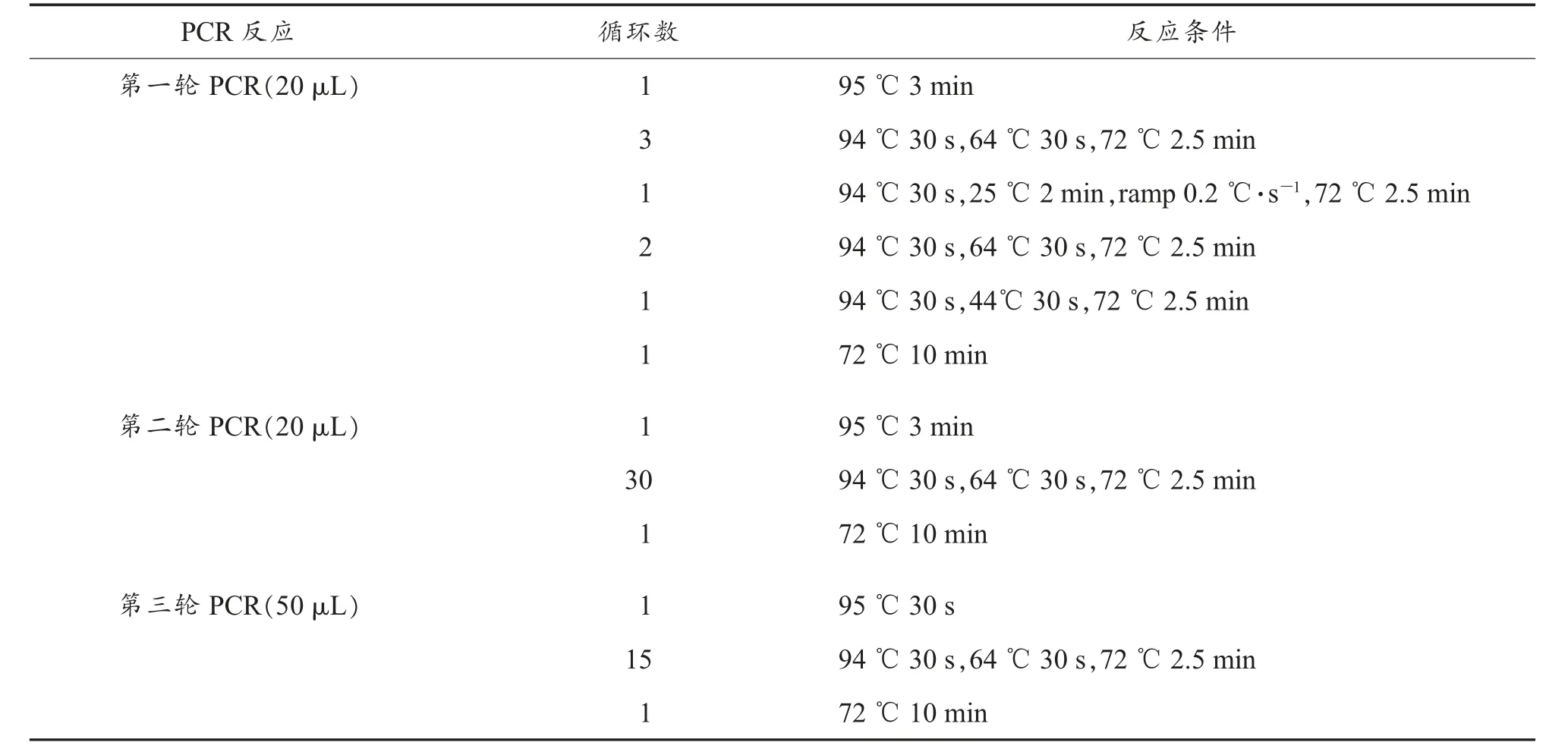
表2 三轮PCR 反应程序
1.5 侧翼序列的获取及比对分析
将第三轮扩增产物经琼脂糖电泳分离后,特异条带用胶回收试剂盒回收纯化, 将回收的PCR 片段连接到pGEM-T easy 载体,转化大肠杆菌,每个转化随机挑5 个单克隆菌落, 使用通用引物M13进行测序验证。 根据测序的结果,分别与载体骨架pCAMBIA2300 左右边界序列以及Cotton FGD 的棉花基因组数据库 (https://cottonfgd.org/) 进行比对, 通过比对结果确定T-DNA 左右边界在基因组上的插入位置。
1.6 侧翼序列的验证与T-DNA 区域的分析
根据插入的位置, 在T-DNA 两侧的棉花基因组上设计引物(图2A、2C),L280-F1 或L280-F2 结合载体的左边界序列LSP3 配对扩增,L280-R1 或L280-R2 结合载体的右边界序列RSP3 配对扩增;L282-F1 或L282-F2 结合载体的左边界序列LSP3配对扩增,L282-R1 或L282-R2 结合载体的右边界序列RSP3 配对扩增, 以此对T-DNA 插入位点进行验证。 同时分别在T-DNA 区域内设计引物进行扩增验证(图2A、2C),将扩增的结果与预测的条带大小比较验证, 根据验证的结果将T-DNA 的完整序列在棉花基因组中的插入位置标识出来。
2 结果与分析
2.1 FPNI-PCR 扩增结果
分别选取转cry2Ab4 抗虫棉(田间编号L280)与转cry2Ab4、vip3Aa11 双价抗虫棉 (田间编号L282)的DNA 作为模板,通过FPNI-PCR 进行三轮扩增,扩增产物通过琼脂糖凝胶电泳(图1),L280左边界有8 个条带,大于500 bp 的有6 条,主要集中在500~2 000 bp;右边界没有条带,可能是载体右侧边缘存在缺失等原因造成的。 L282 左边界扩增的4 个有条带, 右边界扩增的2 个有条带,6 个条带均大于500 bp。
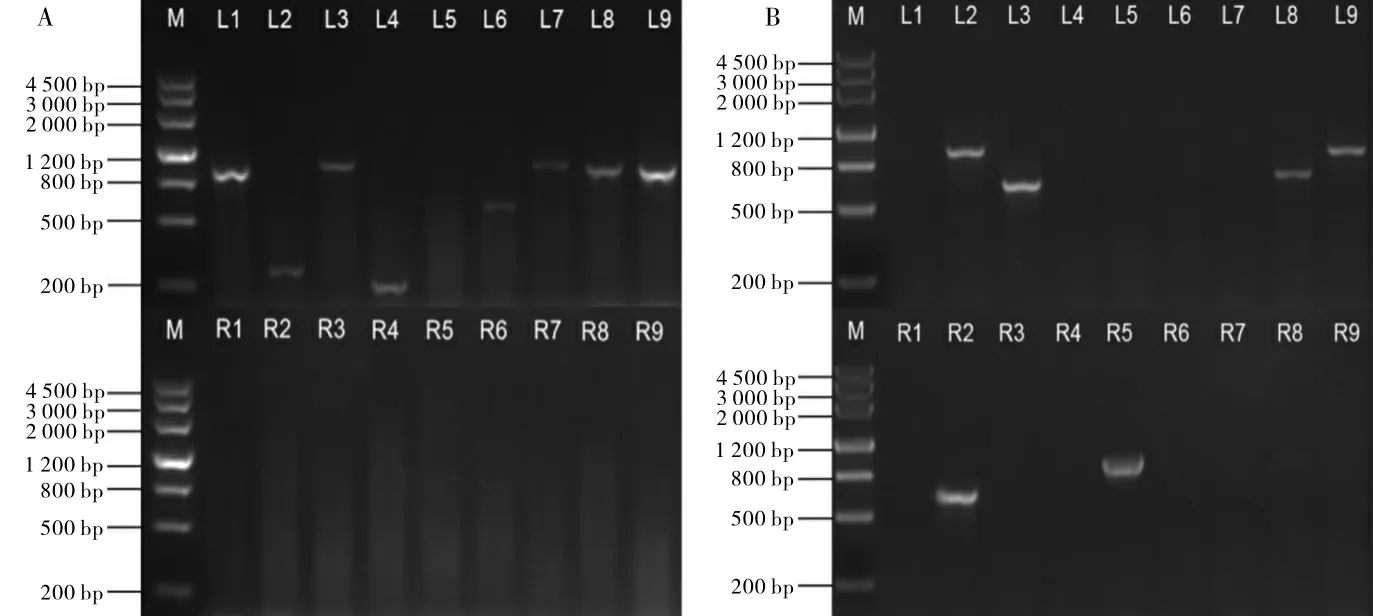
图1 两个转基因抗虫棉FPNI-PCR 的电泳图
2.2 侧翼序列的克隆分析及验证
根据电泳结果,将扩增条带切胶回收,纯化后的产物测序验证。 将测序结果分别与Cotton FGD的棉花基因组数据库和载体骨架pCAMBIA2300左右边界序列进行比对,结果表明,L280 的扩增条带L1、L3、L6、L7、L8、L9 的序列均能与载体骨架pCAMBIA2300 和棉花基因组比对上,T-DNA 插入位点位于棉花基因组D13 染色体上61 309 969-61 309 997 bp 区间 (图2A) 基因组缺失27 bp,T-DNA 左右两端分别缺失15 bp 与28 bp。 小于500 bp 的2 个条带与T-DNA 和棉花基因组都没有相似序列,推测这2 个条带可能是非特异性扩增产物。L282 扩增的6 个条带核苷酸比对结果表明,这6 个条带与载体骨架pCAMBIA2300 和棉花基因组都有部分相同的核苷酸序列,T-DNA 插入位点位于D11 染色体上11 069 019-11 069 039 bp 区间(图2C),基因组缺失19 bp,T-DNA 左右两端分别缺失21 bp 与17 bp。 根据基因组比对的结果,分别选取L280 和L282 两个材料LB 与RB 的侧翼序列各1 000 bp ,通过计算分析,侧翼序列的AT 碱基含量较高。 材料L280 的LB 与RB 侧翼序列AT碱基含量分别为72%、69%; 材料L282 的LB 与RB 侧翼序列AT 碱基含量分别为64%、67%。 为进一步验证T-DNA 插入的准确性, 分别在T-DNA插入位点两侧的棉花基因组DNA 上设计特异性引物与载体边界序列进行PCR 扩增(图2A、2C),产物测序结果表明分离得到的侧翼序列是正确的。
2.3 T-DNA 完整性分析
根据在图2A 和2C 上标识的T-DNA 特异性引物,经PCR 扩增、琼脂糖凝胶电泳,从图2B 和图2D 中可以看出, 条带大小与预测一致。 说明T-DNA 插入的各个元件都保持了较好地完整性。
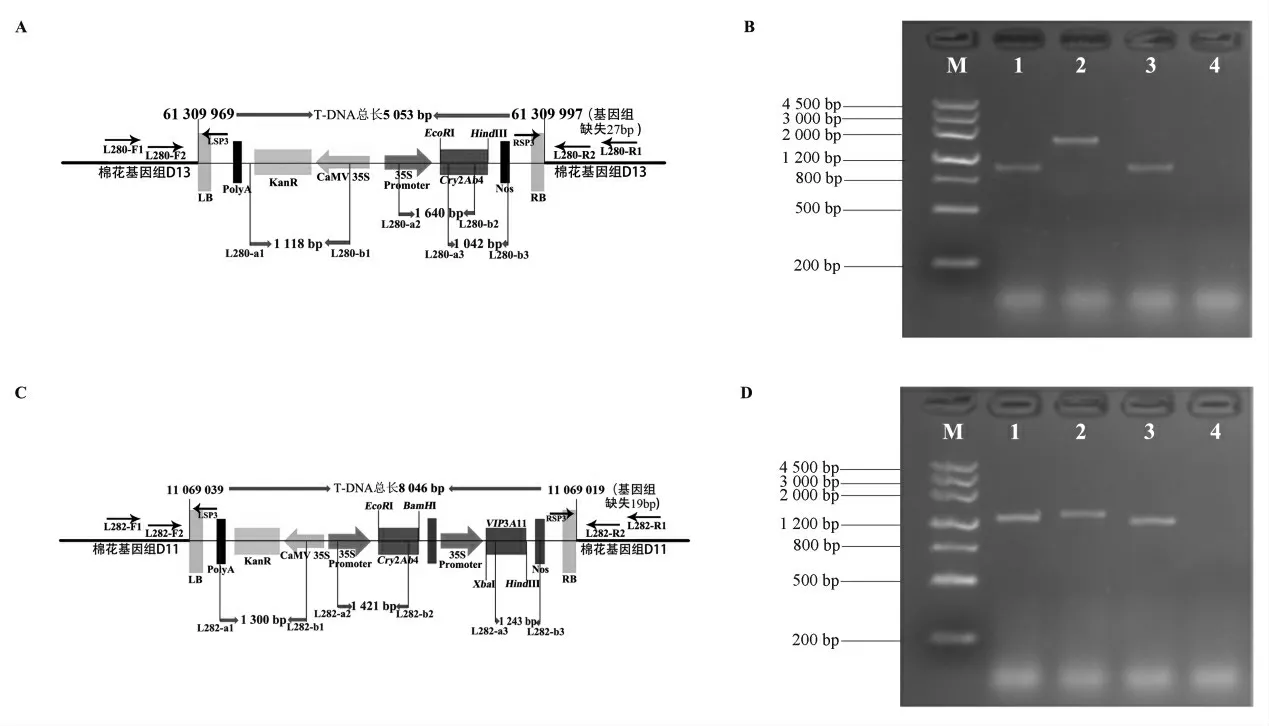
图2 两个转基因抗虫棉T-DNA 插入位点及完整性示意图
3 讨论
本研究为了分析cry2Ab4 基因抗虫棉(L280)与转cry2Ab4+vip3Aa11 基因双价抗虫棉(L282)的插入位点,探究了FPNI-PCR 在棉花中高效的侧翼序列克隆方法。 研究结果表明,18 个泳道只要有1 个扩增的条带可以与载体骨架与基因组分别比对上,就可以确定插入位点,然后通过在基因组上设计特异性的引物即可进行验证。随着基因编辑技术的发展,功能基因的不断挖掘以及未来棉花突变体库的建立,1 种高效的侧翼序列测定的方法是必不可少的。 FPNI-PCR 在本实验中能够高效对侧翼序列进行测定,因此在载体边界序列信息清楚的情况下,使用该方法省时省力,特异性高。转基因抗虫棉的种植不仅能够减少棉花生产的成本和棉农的劳动强度, 同时也对生态环境起到良好的保护作用。 转基因的侧翼序列就是该品种的标签,明确转基因抗虫棉的插入位点对申请安全证书有一定帮助,对改善转基因棉花市场的监管也能起到积极的作用。 因此,测定两种抗虫棉的侧翼序列是对其进行后续研究的必要步骤。
4 结论
本研究通过FPNI-PCR 成功扩增出cry2Ab4基因抗虫棉(L280)与转cry2Ab4+vip3Aa11 基因双价抗虫棉 (L282) 的侧翼序列。 确定了它们的T-DNA 插入位置, 分别位于棉花基因组染色体D13 上61 309 969-61 309 997 bp 区 间 和D11 上11 069 019-11 069 039 bp 区间。 通过设计特异性引物, 对插入位点的准确性与T-DNA 区域的完整性分别进行了验证分析。检测两种抗虫棉的侧翼序列不仅能够为品种的保护在申请专利时提供理论依据, 也在后续研究以及安全证书的申请提供数据支撑。
