小麦PIN基因家族的鉴定及表达分析
2020-08-04刘培勋万洪深郑建敏罗江陶蒲宗君
刘培勋,万洪深,郑建敏,罗江陶,蒲宗君
(四川省农业科学院作物研究所/农业农村部西南地区小麦生物学与遗传育种重点实验室,成都 610066)
0 引言
【研究意义】小麦是全球重要的粮食作物,中国作为世界上最大的小麦生产国和消费国,其生产对保障国家粮食安全具有重要意义。小麦籽粒硬度是国内外小麦分类和定价的重要因素之一,是当前专用小麦育种的重要指标。不同硬度小麦面粉用途不同,硬麦适用于制作面包和面条等,软麦适用于制作饼干、糕点和酿酒等。明确小麦籽粒硬度遗传基础,对于改良小麦品质,培育具有不同硬度的小麦品种具有重大意义。【前人研究进展】前人研究发现,小麦籽粒硬度受多个遗传因子控制并受环境影响。GREENWELL等[1]首次用SDS方法从软质小麦水洗淀粉中分离获得了与籽粒硬度相关的Friabilin蛋白,其分子量为15 kD,该蛋白位于小麦5DS染色体臂的Ha位点,在软粒小麦中大量表达,在硬质小麦中含量极少或者缺失[2]。小麦籽粒硬度受PIN(puroindoline)基因控制[3],包括Pina和Pinb,2个基因的氨基酸序列相似性达60%,共同作用影响籽粒柔软度。当二者同时存在时,小麦籽粒表现为软质,当Pina缺失或Pinb发生变异时,小麦籽粒表现为硬质。HEINZE等[4]将PIN基因转入硬粒小麦(Triticum turgidumL.var.durum),导致其籽粒品质发生改变并显著降低了磨粉所需能量。GASPARIS等[5]运用RNA介导的基因沉默技术使PIN基因沉默,发现PIN蛋白显著减少,小麦籽粒硬度增加。WILEY等[6]运用组织印记等技术,发现小麦PIN蛋白的合成和积累仅发生在籽粒胚乳细胞中。世界各地学者对不同地区小麦资源PIN基因的变异类型开展了研究并发现了大量变异类型[7-10]。随着对PIN基因研究的深入,与硬度相关的grain softness protein-1(gsp-1)也被分离出来,该基因与PIN基因高度同源,且共同存在于5DS染色体上,3个基因的物理顺序为PinB-PinA-Gsp1[11]。此外,WILKINSON等[12]在普通小麦cDNA中发现一种与puroindoline b核苷酸序列相似性高达70%的类PIN基因,称为puroindoline b-2。陈锋等[13]发现该基因有4种变异类型,分别为Pinb-v1、Pinb-v2、Pinb-v3和Pinb-v4。利用缺体材料将其分别定位为7DL、7BL、7B和7AL上。研究表明,小麦籽粒硬度不仅由基因决定,还受环境及栽培措施等的影响。王月福等[14]研究了氮肥用量对不同小麦品种籽粒蛋白质组分含量变化及加工品质的影响,结果表明,增施氮肥能够显著提高籽粒蛋白质各组分的含量,提高湿面筋含量和沉降值,从而增加籽粒硬度。李友军等[15]研究表明,氮钾配合施用可降低小麦品种豫麦50和郑麦9023的籽粒硬度。然而,其改变小麦籽粒硬度的分子遗传基础尚不明确。【本研究切入点】目前,对于小麦籽粒硬度的研究主要集中于PIN基因(Pina和Pinb),对其所属基因家族的同源基因的研究尚不全面。【拟解决的关键问题】本研究以小麦基因组参考序列(iwgsc_refseqv1.0 2018)为基础,鉴定PIN基因家族成员,分析基因分布、基因结构、保守结构域、顺式作用元件和系统进化关系等,明确小麦PIN家族基因在不同组织不同发育阶段的表达特异性和不同硬度小麦样本中的表达模式,为解析该基因功能奠定基础,为小麦遗传改良提供理论参考。
1 材料与方法
1.1 试验材料和处理方法
小麦品种蜀麦969和川麦601来自于四川省。蜀麦969是筋力最强、硬度最高的四川主推品种之一,川麦601是筋力最弱、硬度最低的品种之一。于2018年10月至2019年5月种植于四川省农业科学院德阳试验基地(31°14′N,104°24′E)。播种时各施75 kg·hm-2的磷肥(过磷酸钙)和钾肥(硫酸钾)作为基肥,常规组施150 kg·hm-2的尿素,低氮组施75 kg·hm-2的尿素,尿素的施用分为2次,一半作为基肥施用;另一半作为拔节肥施用。在开花30 d时,收取饱满的种子,液氮速冻后,-80℃保存备用,用于提取RNA。使用单粒谷物测定仪KSCS4100检测成熟小麦种子籽粒硬度指数,使用近红外谷物品质分析仪Infratec TM 1241检测粗蛋白含量。
1.2 小麦PIN基因家族的鉴定和系统发育分析
从UniProt数据库[16]下载小麦已知PIN蛋白序列[17]和大麦hordoindoline序列[18],作为搜索小麦同源基因的探针序列(电子附表1)。小麦全基因组序列、蛋白序列和注释文件来源于URGI[19]。根据小麦PIN蛋白序列和大麦hordoindoline序列,使用HMMER3.0软件[20-21]建立HMM模型,在小麦全基因组蛋白序列中搜索同源序列。同时利用本地BLASTP程序[22],同源比对小麦蛋白序列,进行鉴定,同时满足2种鉴定方法的被认为是小麦PIN基因家族成员。对鉴定出的小麦PIN基因,利用Clustal X 2.0进行序列比对,利用MEGA 7.0软件[23]最大似然法构建进化树。
1.3 小麦PIN基因的序列分析
使用PFAM和CDD和数据库[24-25]对鉴定出的小麦PIN基因进行保守结构域鉴定。利用ExPASy进行预测小麦PIN基因家族蛋白序列的分子量和等电点[26]。使用MEME在线程序[27]鉴定蛋白保守motif,通过GSDS在线软件[28]分析基因结构。此外,使用PlantCARE软件[29]对每个基因编码序列上游2 000 bp的DNA序列分析顺式元件,并运用TBtools工具[30]进行可视化作图。
1.4 小麦PIN基因的表达分析
从Wheat Expression Browser(powered by expVIP)[31-32]下载小麦品种中国春在不同时期不同组织的转录组表达数据,用于分析小麦PIN基因的表达模式,采用实时荧光定量PCR对小麦PIN基因的表达进行鉴定。使用Omega biotek公司植物RNA提取试剂盒(R6827)提取开花后30 d的小麦籽粒RNA,并反转录成cDNA。根据小麦PIN基因序列,利用Oligo 7软件设计定量PCR引物(表1),由上海生工生物工程有限公司合成。实时荧光定量PCR反应体系(20 μL)包括10 μL SYBR®Green Realtime PCR Master Mix、1 μL primer#1 (10 μmol·L-1)、1 μL primer #2 (10 μmol·L-1)、1 μL Template DNA和4 μL PCR grade water。反应程序为95℃ 2 min;95℃ 15 s,54℃ 30 s(同时收集信号值),40个循环,溶解曲线分析为54℃—95℃。使用β-Actin作为相对定量的内参基因,采用2-ΔΔCt方法进行数据计算,基因的相对表达量使用(平均值±标准差)表示。

表1 TaPINs实时荧光定量PCR分析所用引物Table 1 Primer sequences used in quantitative real-time PCR analysis of TaPINs
2 结果
2.1 小麦PIN基因的鉴定和序列分析
通过BLAST和Hidden Markov Model方法从小麦基因组数据库中共鉴定到19个小麦PIN家族基因,并随机命名为TaPIN1—TaPIN19,这些基因集中成簇分布于1B/1D、5A/5B/5D、7A/7B/7D染色体(电子附表2),编码148—327个氨基酸,相对分子量为16.39—37.19 kD,等电点为6.35—9.34(电子附表3)。Conserved Domains search分析结果表明,所有TaPINs基因都包含AAI_SS结构域(cl07890),该结构域是AAI_LTSS超级家族的特征,因此,PINs基因属于AAI_LTSS超级家族成员。AAI_LTSS超级家族为高等植物特有,包括谷物型阿尔法淀粉酶抑制剂(alpha amylase inhibitor)、脂质转移蛋白(lipid transfer protein)、种子储存蛋白(seed storage protein)和其他类似蛋白。Pfam数据库进一步分析表明,19个PIN蛋白结构域的Top Hit不同,其中,9个蛋白(TaPIN6—TaPIN10、TaPIN14—TaPIN16和TaPIN19)为Tryp_alpha_amyl(PF00234.22),剩下的10个(TaPIN1—TaPIN5、TaPIN11—TaPIN13、TaPIN17—TaPIN18)为Gliadin(PF13016.6)(电子附表4)。
2.2 小麦PIN基因的系统发育分析、蛋白保守结构域及基因结构分析
19个TaPINs蛋白序列的系统发育分析显示该基因家族可以分为A和B两大类,其中,作为参考的小麦PIN蛋白序列和大麦hordoindoline序列与A类聚为同一类。A类又进一步分为A1、A2和A3亚类,A1包括TaPIN9和TaPIN10,A2包括TaPIN14、TaPIN15、TaPIN16和TaPIN19,A3包括TaPIN6、TaPIN7和TaPIN8;B类可进一步分为B1和B2亚类,B1包括TaPIN11、TaPIN12、TaPIN13、TaPIN17和TaPIN18,B2包括TaPIN1、TaPIN2、TaPIN3、TaPIN4和TaPIN5(图1)。为了研究小麦PIN基因的结构特征,根据其进化关系进行保守motif分析,通过MEME数据库共鉴定12个保守motif(图2),所有序列均包含motif1、motif3、motif4和motif5,其中,属于PIN基因特征之一的富色氨酸区域位于motif3。A类中独有motif6和motif7,而B类中特有motif 8—motif12。如图2所示,除TaPIN15有1个长的内含子和TaPIN1有2个短内含子外,大部分TaPINs基因仅有1个外显子,没有内含子。19个TaPINs基因上游序列含多个顺式作用元件(图3),包括与抗逆相关的脱落酸作用元件(ABRE)、茉莉酸甲酯作用元件(CGTCA-motif和TGACG- motif)、干旱胁迫反应元件(MBS)、水杨酸作用元件(TCA-element)、抗逆元件(TC-rich repeats)、赤霉素反应元件(P-box和GARE-motif)和低温反应元件(LTR);与种子发育相关的胚乳发育元件(GCN4_motif)、蛋白代谢调控元件(O2-site)和种子特异调控元件(RY-element)。丰度最大的3种元件为ABRE、CGTCA和TGACG,均与抗逆性有关。
2.3 小麦PIN基因在不同组织、不同时期的表达分析
利用小麦转录组数据分析了小麦品种中国春在不同组织和发育时期的表达(图4和电子附表5),19个TaPINs基因在小麦籽粒中的相对表达量显著高于其他组织部位,而在根茎叶等组织中表达量低,几乎不表达。在小麦拔节期和开花期几乎不表达,而在籽粒形成期在籽粒中大量表达表明TaPINs主要在籽粒发育过程中起作用。不同基因成员间表达差异显著,TaPIN14、TaPIN15和TaPIN12等相对表达量较低。
2.4 小麦PIN基因在不同硬度小麦中的相对表达分析
选取了3个不同籽粒硬度的小麦样品(SM969MN籽粒硬度指数为40.2,粗蛋白含量为13.4%;SM969LN硬度指数为35.3,粗蛋白含量为11.4%;CM601LN籽粒硬度指数为25.4,粗蛋白含量为10.7%),分析了TaPINs基因在不同硬度小麦中的表达模式(图6),在3个样品中,TaPIN9和TaPIN10均表达量较高,但随着施氮量降低或是在弱筋小麦品种中,其他几个基因表达量相对较低,而TaPIN9和TaPIN10表达比例非常高。比较几个表达量相对较低的基因的表达模式发现,仅有TaPIN9和TaPIN10的表达量随籽粒硬度的减低,相对表达量上调。TaPIN16和TaPIN6则呈现出相反的趋势,随籽粒硬度的降低,表达量下调。其他几个基因(TaPIN14、TaPIN15和TaPIN19)相对表达量很低。
3 讨论
根据系统发育树,可将本研究鉴定出的19个小麦PIN基因分为A和B两大类,通过同源比对和查阅小麦基因组蛋白注释可知,TaPIN9为Pina,TaPIN10为Pinb,属于A类。Pfam数据库分析表明,19个PIN蛋白结构域的Top Hit不同,A类的9个基因为Tryp_alpha_amyl(protease inhibitor/seed storage/LTPfamily),B类的10个基因为Gliadin(Cys-rich Gliadin N-terminal),推测至少A类的9个基因在影响小麦籽粒硬度中发挥相似功能,而B类则与另一类与PIN基因高度同源的基因类别Avenin-like b(AVNLB)高度相关[33]。A类又分为3个亚类,A1亚类包含了Pina和Pinb。A2亚类包括TaPIN16、TaPIN19、TaPIN14和TaPIN15,该亚类与Pinb亲缘关系较近,且与之前报道的Pinb的同源基因puroindoline b-2的变异类型数量吻合,均为4个,推测其为已报道的puroindolineb-2。前人利用小麦中国春缺体材料对该类基因进行了定位,认为Pinb-2v1位于7DL,Pinb-2v2位于7BL,Pinb-2v3位于7B,Pinb-2v4位于7AL[34]。本研究利用生物信息学方法,在小麦中国春的基因组中进行鉴定,得出TaPIN14和TaPIN15均位于7A,TaPIN16位于7B,TaPIN19位于7D染色体,与前人研究略有差异。A3亚类包括TaPIN6、TaPIN7和TaPIN8,分别位于5A、5B和5D染色体,蛋白序列比对发现,TaPIN8为已报道的gsp-1,但位于5A和5B的同源序列TaPIN6和TaPIN7之前未见报道。A2和A3亚类均在小麦的3套染色体中存在,而A1则只存在于5D染色体,这可能是由于小麦漫长的进化过程中发生了大量基因重组(转座因子插入、基因组缺失、重复和逆序)造成[35]。
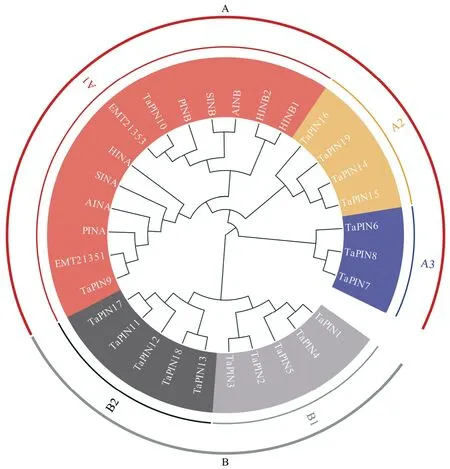
图1 TaPINs蛋白序列构建的系统发育树Fig.1 Phylogenetic tree of TaPINs using the complete protein sequences

图2 19个TaPINs蛋白的保守序列和基因结构图Fig.2 The conserved motifs and gene structure analysis of 19 TaPINs

图3 19个TaPINs基因顺式作用元件分析Fig.3 Cis-acting element analysis of 19 TaPINs

图4 TaPINs基因在小麦品种中国春不同发育时期不同组织中的表达情况Fig.4 Expression profiles of TaPINs in different growth stages and different tissues of Chinese Spring

图5 TaPINs基因家族中A类部分基因相对表达分析Fig.5 Relative expression of TaPINs belong to class A
鉴定出的19个小麦PIN基因与近缘种大麦、燕麦[36]、黑麦[37]中已报道的同源蛋白共同构建的进化树见图1中A1亚类。可见,4个麦类作物均含有该基因,其他麦类未发布完整基因组,因此,其含有PIN同源基因的数量未知。PINA与粗山羊草中的EMT21351聚在一起,PINB与EMT21353聚在一起,且蛋白序列几乎一致,再次验证了小麦D基因组来源于粗山羊草。从该基因的进化分析来看,粗山羊草与小麦亲缘关系最近,燕麦和黑麦次之,大麦与小麦亲缘最远。
通过对该基因家族编码区上游2 000 bp的DNA序列进行顺式作用元件分析,发现启动子区除了种子发育相关元件外,还存在大量抗逆相关元件,推测该类基因除影响籽粒硬度外,还参与小麦植株后期的抗性调控。DHATWALIA等[38]认为puroindoline在植物种子中具有抗菌活性,可能是一种membranotoxin膜毒素,在植物抵御微生物病原体的防御机制中发挥作用。KIM等[39]研究表明,puroindoline具有体外抗菌活性,通过转基因手段进行过表达,证明其是一种有效的抗真菌蛋白。HANEY等[40]对puroindoline抗菌活性进行了检测,表明puroindoline衍生的多肽均与DNA结合,阻断体内大分子的合成。虽然序列信息和前人研究均表明puroindoline参与抗菌活动,但基于该基因家族仅限于在种子内表达,其具体对哪些病害起作用,其调控方式等尚需进一步试验来明确。
目前,对Pina和Pinb的研究尚不能完全解释小麦籽粒硬度变异的机制。AYALA等[41]研究了西班牙南部安达鲁西亚小麦地方品种puroindoline基因的分子特征,发现13个硬质小麦样本,仅有7个可以用Pina-D1或Pinb-D1突变来解释。NIRMAL等[42]对硬籽粒小麦的基因型进行了鉴定,表明除了Pina和Pinb变异,还存在其他基因影响小麦硬度。CHEN等[43]利用电子显微镜分析的硬质和软质节节麦,不能用PIN等位基因的序列变异来解释。DARLINGTON等[44]对3个硬质大麦和3个软质大麦中的puroindoline的同源基因hordoindoline进行了序列比对,大麦中硬质与软质品种中该基因并没有差异。这些研究表明硬度是受多基因控制的数量性状。除Pina和Pinb表达量较高外,其他几个同源基因表达量非常低,对籽粒硬度的影响有限。除Pina和Pinb之外,该基因家族其他成员同样具有AAI_SS功能结构域,其在籽粒硬度调控中也可能具有共同作用,如果这些基因的表达量提升到相近水平后,能否改良籽粒硬度性状,尚需进一步试验来明确。
4 结论
在小麦全基因组范围中共鉴定出19个TaPINs基因家族成员,可分为两大类。与已报到的其他麦类作物同源基因构建的系统发育表明,从该基因来看,小麦和粗山羊草亲缘关系最近,其次是燕麦、黑麦和大麦。该基因家族均只在籽粒中表达,但部分基因表达量很低。小麦籽粒硬度的调节以Pina和Pinb为主,推测其他基因家族成员也具有相似功能,但受表达量低的限制,对籽粒硬度影响较小。
