燕麦 AsSnRK2.7编码蛋白的分子特征及基因表达特异性研究
2020-07-31向殿军满丽莉王晓东张巍巍李志刚
向殿军,满丽莉,王晓东,张巍巍,刘 鹏,李志刚
(1.内蒙古民族大学农学院,内蒙古通辽 028042;2.内蒙古民族大学生命科学学院,内蒙古通辽 028042)
植物蔗糖非发酵-1相关蛋白激酶(sucrose non-fermenting-1-related protein kinase,SNRK)家族属于CDPK-SNRK超家族,与酵母的蔗糖非发酵蛋白激酶1(SNF1)和哺乳动物的AMP激活的蛋白激酶(AMPK)同源[1]。在酵母和哺乳动物中,SNF1和AMPKs在碳水化合物代谢中扮演重要角色;而在植物中,SnRKs在连接胁迫信号和新陈代谢通路方面发挥关键作用[2-4]。根据氨基酸序列和结构域的相似性,植物SnRKs又可分为SnRK1、SnRK2和SnRK3三个亚家族[4]。
SnRK2蛋白激酶是主要的信号转导因子,在调节真核生物适应环境方面起核心作用。水稻中的SAPK4和小麦中的TaSRK2C1均属于SnRK2蛋白激酶家族基因。在盐胁迫下,SAPK4基因的过表达减少了水稻中Na+和Cl-的积累,并使水稻光合作用和生长状况得到明显改善[5]。与野生型烟草相比,转TaSRK2C1基因烟草中具有较高水平的游离脯氨酸和可溶性碳水化合物,说明TaSRK2C1的异源表达提高了烟草对高盐、脱水和低温胁迫的耐受性[6]。TaSnRK2.8基因的异源过表达可改善拟南芥的主根长、相对含水量、细胞膜稳定性、渗透势和叶绿素含量等生理特性,从而使拟南芥对干旱、盐和冷胁迫的耐受性得到增强[7]。SnRK2蛋白激酶可通过控制镉离子引发的活性氧积累,调节植物对重金属胁迫的耐受性[8]。在干旱胁迫下,脱落酸(ABA)使植物气孔关闭,从而减少水分损失,而拟南芥snrk2e(snrk2.6/ost1)突变体则破坏了ABA诱导的气孔关闭机制,抑制气孔开放,表明SnRK2E参与ABA信号传导[9]。
SnRK2蛋白激酶主要通过ABA依赖或非ABA依赖途径在应答逆境胁迫的防御机制中起着至关重要的作用[10]。其中,ABA依赖型的SnRK2蛋白激酶(如拟南芥SnRK2D、SnRK2I和SnRK2E)在ABA信号传递中起关键作用[11],其调控通路模型主要由可溶性ABA受体(PYR/PYL/RCAR)、蛋白磷酸酶2C(PP2C)和SnRK2丝氨酸/苏氨酸蛋白激酶家族组成[12-14]。在没有ABA的情况下,PP2Cs发生去磷酸化作用,钝化了SnRK2s活性,进而抑制SnRK2s磷酸化调控的反应通路。
目前,一些植物体内的蛋白质已被鉴定为SnRK2蛋白激酶的底物[15-17]。此外,在一些异源表达SnRK2基因的植物中,也发现一些SnRK2蛋白激酶作用的靶标基因[6-7]。如小麦TaSRK2C1蛋白激酶能显著上调烟草DREB2、DREB1A和DREB2三个中枢调节因子的表达水平[6],而小麦TaSnRK2.8蛋白激酶则能调控拟南芥中CBF3、CBF2、CBF1、RD29B、RD20A、ABI5、ABI4、ABI3、ABA2和ABA1基因的mRNA水平[7]。
目前,SnRK2基因家族在小麦[6-7]、波兰小麦[18]和二穗短柄草[19]等一些燕麦(AvenasativaL.)近源种中已得到研究,但有关SnRK2基因在燕麦中的功能尚未见报道。因此,本研究以皮燕麦品种美达为材料,借助前人获得的干旱胁迫下燕麦RNA序列信息,克隆SnRK2蛋白激酶家族基因AsSnRK2.7,利用生物信息学手段和烟草表皮细胞瞬时表达试验分别对AsSnRK2.7蛋白的分子特征和亚细胞定位进行分析,同时借助qRT-PCR技术对AsSnRK2.7基因的非生物胁迫反应和组织表达特异性进行研究,以期为揭示AsSnRK2.7基因应答逆境胁迫的分子机制及其功能奠定基础。
1 材料与方法
1.1 试验材料
供试材料为美国皮燕麦品种美达(Monida),购于北京正道生态科技有限公司,由内蒙古民族大学作物遗传育种实验室扩繁并保存。
1.2 材料处理
将发育良好、饱满的燕麦种子用0.1% HgCl2溶液表面消毒30 min,用无菌水洗涤数次后,将种子放在铺有两层滤纸的培养皿上,置于发芽箱(23±1 °C,光照12 h,黑暗12 h)中,每日添加10 mL无菌水,至种子发芽。将发芽整齐一致的种子分成两份,一份转移至盛有1/2 Hoagland培养液的水培盘(32.5 cm×26 cm×5.5 cm)中,一份播种到土壤中进行培养。培养条件为相对湿度70%,光照强度400 μmol·m-2·s-1,20 ℃暗培养9 h,25 ℃光照15 h。待水培燕麦幼苗长至三叶一心期时,分别进行ABA、干旱、高盐和低温胁迫处理。ABA胁迫处理:用ABA溶液(100 μmol·L-1)喷洒燕麦;干旱胁迫处理:水培液中添加PEG6000(25 mmol·L-1);高盐胁迫处理:水培液中添加NaCl(300 mmol·L-1);低温胁迫处理:将水培燕麦幼苗置于低温(4 ℃)光照培养箱中。水培燕麦在胁迫处理0、1、2、4、8、16、24、48和72 h后,剪取叶片,液氮迅速冷冻后-70 ℃保存,用于胁迫表达模式分析,其中胁迫处理 24 h的叶片用于基因克隆;待盆栽燕麦正常生长至苗期、分蘖期、抽穗期、灌浆期和成熟期时,分别收集叶、根、茎和穗组织(抽穗期、灌浆期和成熟期),液氮迅速冷冻后-70 ℃保存,用于组织表达模式分析。
1.3 燕麦 AsSnRK2.7基因的克隆

表1 本研究所用的引物
1.4 燕麦 AsSnRK2.7基因生物信息学分析
运用ORF finder(https://www.ncbi.nlm.nih.gov/orffinder/)预测基因的开放阅读框。采用Translate(https://web.expasy.org/translate/)推导目的基因编码的氨基酸序列。使用BLASTP(https://blast.ncbi.nlm.nih.gov/Blast.cgi)对编码蛋白进行同源搜索。利用NCBI中的CD-Search(https://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)预测编码蛋白的保守结构域。采用ClustalX软件进行多序列比对分析,基于输出的序列比对结果,利用GeneDoc和MEGA 5.1软件分别进行多重序列比对图和系统进化树的绘制。运用SMART(http://smart.embl-heidelberg.de/)工具预测蛋白的功能区和激活位点。利用ProtParam(https://web.expasy.org/protparam/)预测编码蛋白的理化性质;利用TMpred(http://www.ch.embnet.org/software/TMPRED_form.html)预测编码蛋白的跨膜结构;利用ProtScale(https://web.expasy.org/protscale/)编码蛋白的疏水性/亲水性。用DictyOGlyc 1.1(http://www.cbs.dtu.dk/services/dictyoglyc/)预测编码蛋白的O-糖基化修饰位点;利用Motif Scan(https://myhits.isb-sib.ch/cgi-bin/motif_scan)预测编码蛋白的生物活性位点;利用NetPhos 3.1(http://www.cbs.dtu.dk/services/NetPhos/)预测编码蛋白的磷酸化修饰位点。利用PONDR(http://www.pondr.com/)分析编码蛋白的无序化;利用TargetP 1.1(http://www.cbs.dtu.dk/services/TargetP-1.1/)预测氨基酸序列导肽;利用STRING(https://string-db.org/cgi/input.pl)预测编码蛋白的互作蛋白。利用SOPMA(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)分析编码蛋白的二级结构,利用Phyre 2(http://www.sbg.bio.ic.ac.uk/phyre2/html/page.cgi?id=index)分析编码蛋白的三级结构,利用PDBsum(http://www.ebi.ac.uk/thornton-srv/databases/pdbsum/Generate.html)检测模型质量。
1.5 燕麦 AsSnRK2.7基因的亚细胞定位
1.6 燕麦 AsSnRK2.7基因的表达特性分析
2 结果与分析
2.1 燕麦 AsSnRK2.7全长cDNA的克隆结果
以干旱胁迫24 h的燕麦叶片cDNA第一链为模板,通过RT-PCR扩增,得到长度约250 bp和1 000 bp的2条cDNA片段(图1),其中,1 000 bp的条带与预期大小一致。回收长度约 1 000 bp的片段,进行T-A克隆后并测序,发现该片段长度为1 074 bp。ORF预测结果表明,该基因包含一个1 074 bp的开放阅读框,编码357个氨基酸。BLAST结果显示,该基因编码的氨基酸序列与二倍体长穗偃麦草(Thinopyrumelongatum,QDA34118.1)、粗山羊草(Aegilopstauschii,XP_020174011.1)、波兰小麦(Triticumpolonicum,ALL27274.1)、二穗短柄草(Brachypodiumdistachyon,NP_001304805.1)、水稻(Oryzasativa,XP_004975639.1)、谷子(Setariaitalica,XP_004975639.1)、高粱(Sorghumbicolor,XP_021318906.1)和拟南芥(Arabidopsisthaliana,NP_195711.1)SnRK2蛋白激酶氨基酸序列的一致性分别为99.75%、96.92%、96.64%、96.36%、91.64%、90.50%、89.97%和 62.01%。利用NCBI CD-Search在线工具对该基因编码的蛋白进行功能结构域预测,发现该蛋白含有一个STKc_SnRK2结构域和一个PKc_like superfamily结构域,属于SnRK2蛋白激酶家族成员(图2)。将获得的cDNA片段命名为AsSnRK2.7,提交至Genbank,获得登录号为MN729577。
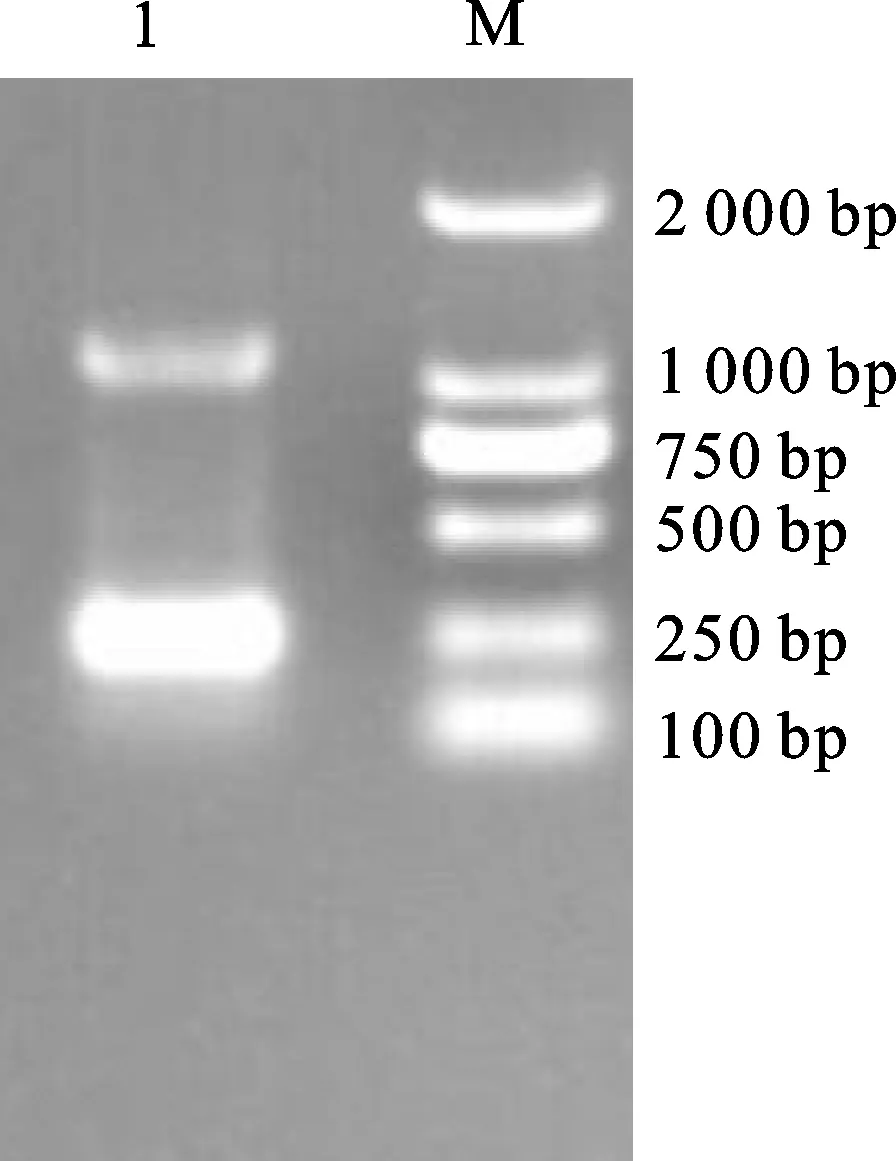
1:cDNA扩增产物;M:Marker DL2000。
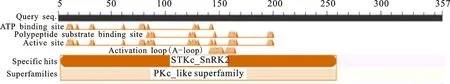
图2 AsSnRK2.7蛋白功能结构域预测结果
2.2 燕麦AsSnRK2.7蛋白多序列比对和系统进化分析
多序列比对结果表明,AsSnRK2.7蛋白与玉米(Zeamays,ACG50009.1)、粳稻(OryzasativaJaponica,BAD17998.1)、波兰小麦(Triticumpolonicum,ALL27272.1)、拟南芥(Arabidopsisthaliana,NP_567945.1)和二穗短柄草(BrachySnRK2家族成员在C端呈现多变特异性,而在N端表现出高度的保守性(图3)。

Ⅰ:蛋白激酶ATP结合信号区;Ⅱ:N-肉豆蔻酰化作用位点;Ⅲ:丝氨酸/苏氨酸蛋白激酶活性位点;Ⅳ:激活环;Ⅴ:跨膜螺旋位点;Ⅵ:响应渗透胁迫所需的结构域Ⅰ;Ⅶ:响应ABA所需的结构域Ⅱ。
进化树分析表明,来自10个物种的22个SnRK2蛋白被分成3个类群(图4)。其中,AsSnRK2.7蛋白与普通小麦(Triticumaestivum,ACU65228.1)、波兰小麦(Triticumpolonicum,AHC00229.1)、玉米(Zeamays,ACG50009.1)、水稻(Oryzasativa,BAD18002.1)、拟南芥(Arabidopsisthaliana,NP_176290.1)、烟草(Nicotianatabacum,AAL89456.1)和马铃薯(Solanumtuberosum,AFR68941.1)的7个SnRK2成员亲缘关系最近,被聚为Group Ⅰ(SnRK2b)。
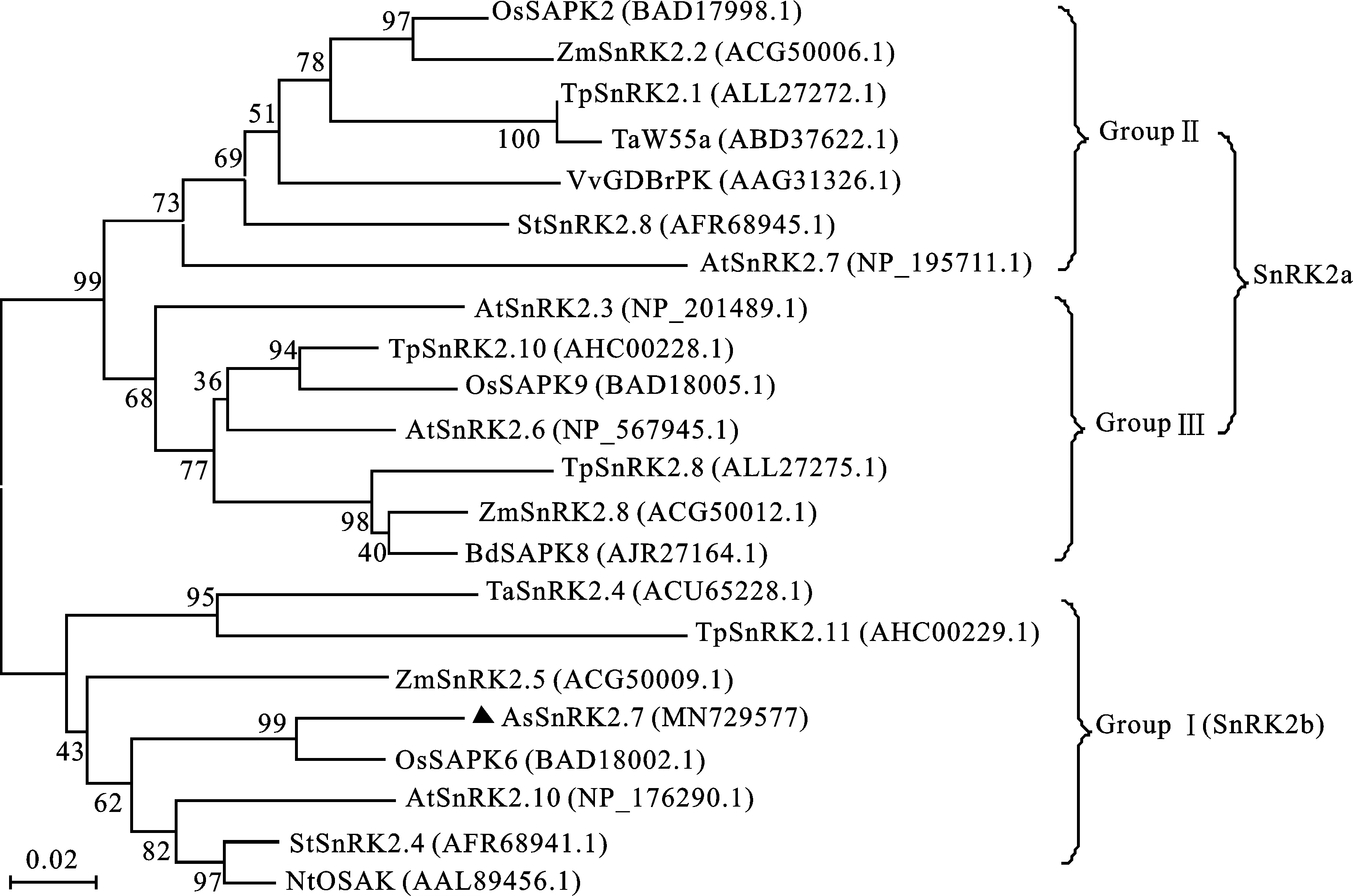
图4 不同物种SnRK2蛋白的聚类分析
2.3 AsSnRK2.7蛋白功能区与生物活性位点的预测
O-糖基化修饰位点预测结果表明,AsSnRK2.7蛋白中第133和307位的氨基酸残基可能成为O-糖基化修饰的位点。NetPhos预测结果显示,该蛋白中有12个丝氨酸、9个苏氨酸和7个络氨酸残基可能成为磷酸化修饰的位点。利用Motif Scan在线工具预测AsSnRK2.7生物活性位点,结果表明,该蛋白包含1个酰胺化位点(324~327 aa)、1个cAMP和cGMP依赖性蛋白激酶磷酸化位点(247~250 aa)、5个酪蛋白激酶Ⅱ磷酸化位点(96~99 aa、129~132 aa、172~175 aa、250~253 aa和290~293 aa)、2个N-肉豆蔻酰化作用位点(110~115 aa和239~244 aa)、3个蛋白激酶C磷酸化位点(54~56 aa、172~174 aa和250~252 aa)、2个酪氨酸激酶磷酸化位点(94~102 aa和140~146 aa)、1个蛋白激酶ATP结合信号区(10~33 aa)、1个丝氨酸/苏氨酸蛋白激酶活性位点(119~131 aa)、富含谷氨酸的区域(318~347 aa)、1个蛋白激酶结构域(4~60 aa)和1个酪氨酸蛋白激酶位点(4~85 aa)。
2.4 燕麦AsSnRK2.7蛋白的理化性质、跨膜结构和疏水性/亲水性分析
基于ProtParam工具对AsSnRK2.7蛋白理化性质分析,结果显示,该蛋白含有20种氨基酸,带正电氨基酸(Arg+Lys)和负电氨基酸(Asp+Glu)比例分别为13.16%和15.69%,含量较低的2种氨基酸分别为色氨酸(0.8%)和半胱氨酸(2.2%),含量较高的4种氨基酸分别为谷氨酸(10.4%)、亮氨酸(7.0%)、赖氨酸(7.0%)和丙氨酸(6.4%);理论等电点为5.63,分子量为 40 914.63。该蛋白总平均疏水性(GRAVY)、脂肪系数和不稳定性系数分别为-0.540、75.94和43.08,是一种亲水且不稳定蛋白(总平均疏水性负值是亲水蛋白,正值为疏水蛋白;稳定蛋白系数小于40,不稳定蛋白系数大于40)。
利用TMPred在线分析软件对燕麦AsSnRK2.7蛋白进行跨膜结构分析,结果表明,该蛋白仅存在一个分值大于500的跨膜区域(180~200 aa),该跨膜区域既有从内到外的跨膜能力(分值991),又有从外到内的跨膜能力(分值734),推断AsSnRK2.7为跨膜蛋白。利用ProtScale在线工具对燕麦AsSnRK2.7蛋白进行疏水性/亲水性分析,结果显示,该蛋白包括明显的疏水区(分值大于0.5)和亲水区(分值小于-0.5),且疏水区域少于亲水区域。此外,组成AsSnRK2.7蛋白的357个氨基酸中,第339和340位氨基酸亲水性最强,分值为-3.300;第191和192位氨基酸疏水性最强,分值为2.189。这些结果表明,燕麦AsSnRK2.7可能是一种亲水蛋白,与ProtParam在线工具预测结果一致。
2.5 AsSnRK2.7蛋白的固有无序化分析以及导肽和互作蛋白的预测
利用PONDR在线程序对AsSnRK2.7蛋白进行折叠无序化分析,发现AsSnRK2.7是一个部分无序蛋白。AsSnRK2.7蛋白包含6个无序化区域,分别位于3~4 aa、23~36 aa、38~47 aa、159~172 aa、291~304 aa和309~356 aa,最长的无序区域包含48个氨基酸残基,无序化氨基酸总数为103个,无序化程度为28.85%。
利用TargetP 1.1在线工具对AsSnRk2.7蛋白进行导肽预测,结果表明,燕麦AsSnRK2.7蛋白的分泌途径信号肽、线粒体目标肽、叶绿体转运肽和其他位置导肽的预测分值分别为0.026、0.168、0.036和0.891,预测可靠性为2级,不存在导肽酶切位点,不具有导肽性。
利用STRING在线程序预测显示,在小麦中发现10个与AsSnRK2.7同源蛋白(丝氨酸/苏氨酸蛋白激酶)的互作蛋白(图5)。其中,包含6个碱性亮氨酸拉链(bZIP)转录因子、3个胱硫醚-β-合成酶和1个环核苷酸-单磷酸依赖蛋白激酶。
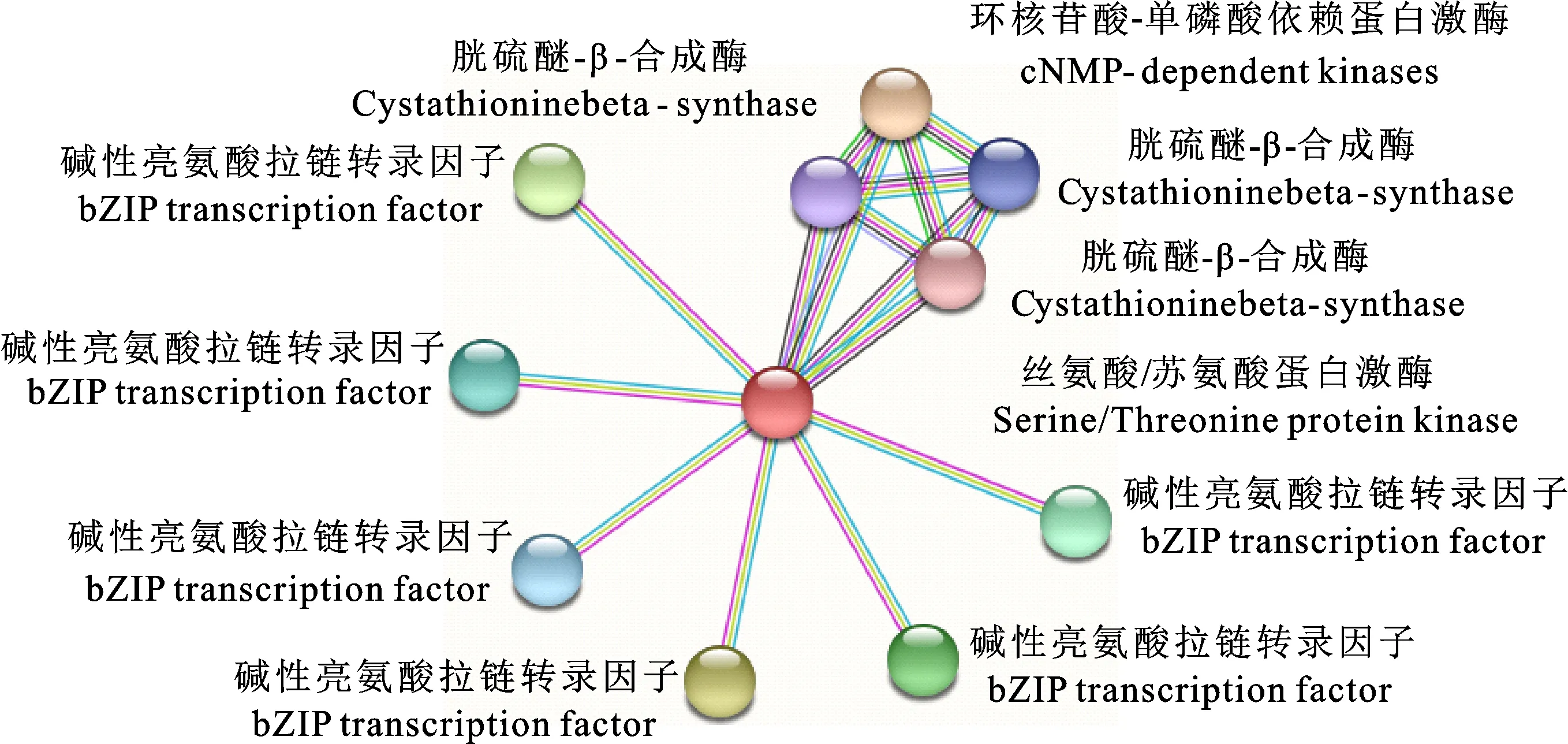
图5 小麦中AsSnRK2.7同源蛋白的互作蛋白
2.6 AsSnRK2.7蛋白的二级结构和三级结构 分析
AsSnRK2.7蛋白是一种混合型结构的蛋白质,其二级结构主要为α-螺旋和无规则卷曲,分别占38.94%和40.06%,而β-转角和延伸链占比较少,分别为5.60%和15.41%。
由图6A可以看出,AsSnRK2.7蛋白三级结构主要为无规则卷曲和α-螺旋,与其二级结构预测结果相一致。利用PDBsum,对AsSnRK2.7蛋白三级结构建模结果可靠性进行检测,结果(图6B)可以看出,AsSnRK2.7模型氨基酸在最佳允许区(A、B和L区域)、次允许区(a、b、l和p区域)、一般允许区(-a、-b、-l和-p区域)和不允许区的比列分别为64.1%、29.8%、4.8%和 1.3%。在最佳允许区和次允许区的氨基酸的比例为93.9%,说明所构建的AsSnRK20.7蛋白三级结构模型符合立体化学的规则。

A:AsSnRK2.7蛋白的三级结构;B:AsSnRK2.7蛋白模型的拉氏构象。
2.7 AsSnRK2.7蛋白质亚细胞定位分析
从图7可以看出,在pCAMBIA2300-CaMV35S-GFP转化的烟草表皮细胞中,绿色荧光信号分布在细胞质、细胞核和细胞膜等区域。而35S-AsSnRK2.7-GFP转化的烟草表皮细胞中,绿色荧光信号主要分布在细胞核。将GFP、CHI、DAPI以及DIC通道融合后,35S-AsSnRK2.7-GFP融合蛋白所表达的绿色荧光与蓝色的DAPI相重合,表明AsSnRK2.7蛋白在烟草表皮细胞中定位于细胞核。
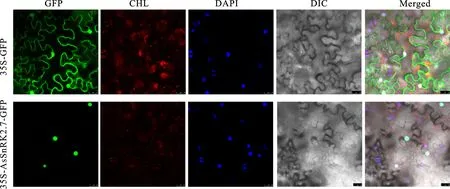
GFP:绿色荧光;CHL:叶绿体自发荧光;DAPI:细胞核特异标记;DIC:微分干涉对比;Merge:叠加场。35S-GFP为空载体,35S-AsSnRK2.7-GFP为包含AsSnRK2.7的融合蛋白载体。标尺=25 μm。
2.8 AsSnRK2.7基因的qPCR表达分析
实时定量PCR结果(表2)显示,AsSnRK2.7基因在燕麦被检测组织中为组成型表达。在根组织中,AsSnRK2.7基因在苗期表达量最高,灌浆期次之,成熟期最低。在叶片组织中,AsSnRK2.7基因在分蘖期的表达量最高,分别是苗期、抽穗期、灌浆期和成熟期的5.57、2.61、2.33和20.19倍。在茎和穗组织中,AsSnRK2.7基因表达量分别在抽穗期和灌浆期最高,在成熟期最低。
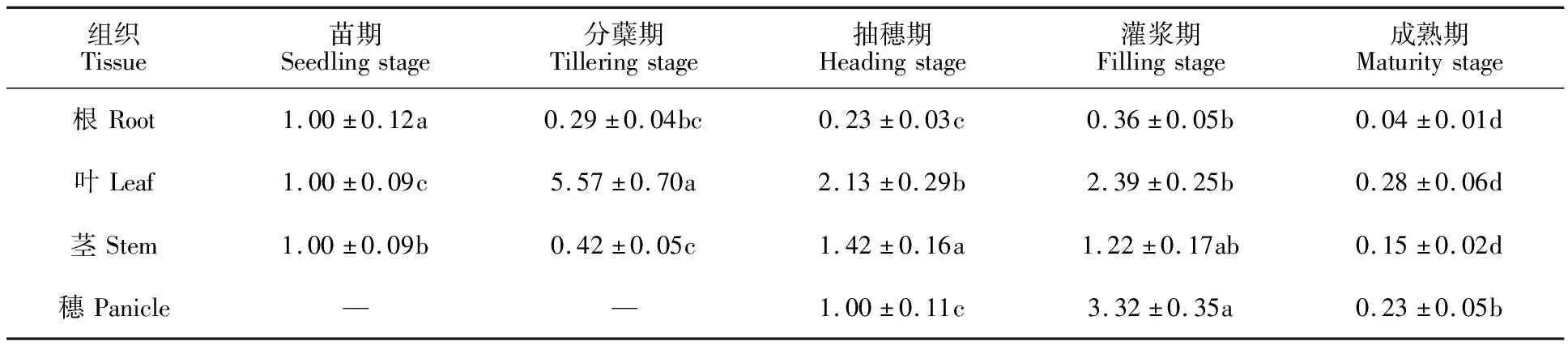
表2 不同组织中 AsSnRK2.7基因的相对表达量
从表3可以看出,ABA不能激活AsSnRK2.7基因的表达,但其表达积极响应PEG、盐和低温(4 ℃)的胁迫。在PEG胁迫下,AsSnRK2.7基因出现了双峰表达模式,峰值分别出现在2 h和16 h,且在2 h表达量最高。在盐胁迫下,AsSnRK2.7基因的表达量随处理时间的延长呈现先上升后下降的趋势,在4 h达到最高。在低温胁迫下,AsSnRK2.7基因的表达量在0~4 h持续上调,在16 h达到最高。
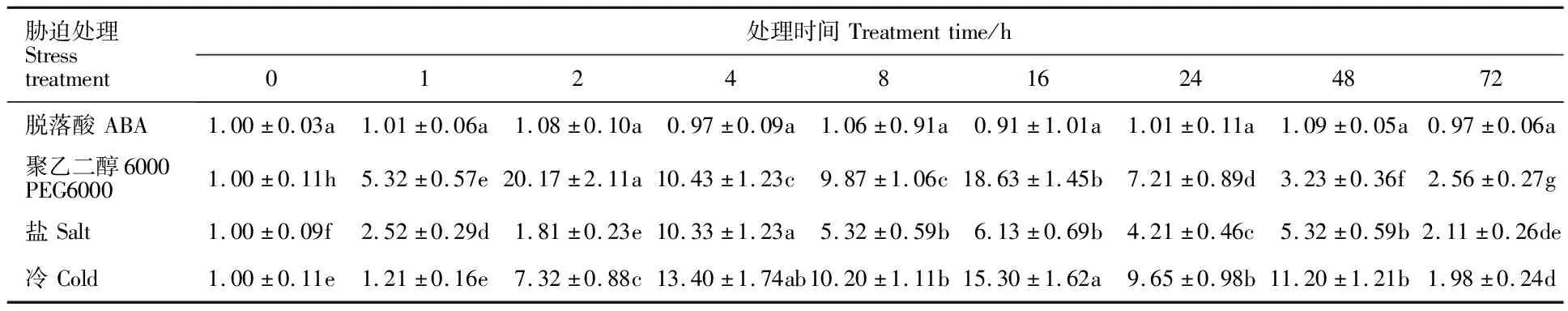
表3 不同胁迫下 AsSnRK2.7基因的转录水平
3 讨 论
SnRK2蛋白激酶基因属于多基因家族,在水稻[22]、玉米[23]和拟南芥基因组中分别存在10、11和10个AsSnRK2基因[24]。本研究依据干旱胁迫下燕麦转录组中的序列信息,利用RT-PCR技术,首次从燕麦中分离出一个SnRK2蛋白激酶基因AsSnRK2.7。AsSnRK2.7基因cDNA长度为1 074 bp,与波兰小麦[18]和二穗短柄草[19]SnRK2.7基因的开放阅读框序列长度完全一致。生物信息学分析表明,AsSnRK2.7蛋白含有SnRK2蛋白激酶家族特有的功能域,包括蛋白激酶ATP结合信号区、N-肉豆蔻酰化作用位点、丝氨酸/苏氨酸蛋白激酶活性位点、激活环、跨膜螺旋位点和响应渗透胁迫所需的结构域Ⅰ等重要功能区,表明不同植物中SnRK2蛋白激酶成员结构十分保守,这与前人在小麦[6-7]、波兰小麦[18]和二穗短柄草[19]中的研究结果一致。
N-肉豆蔻酰化作用位点和跨膜区是蛋白质在植物应答环境胁迫反应中调节膜靶向和信号转导的关键区域[25-26]。TaSnRK2.4、TaSnRK2.7和TaSnRK2.8蛋白均定位在细胞膜、细胞质和细胞核中,且过表达这3个基因均能增强拟南芥对干旱、盐和冷胁迫的耐受性[7,27-28]。TaSnRK2.4、TaSnRK2.7和 TaSnRK2.8蛋白中均存在一个潜在的N-肉豆蔻酰化作用位点和一个潜在的跨膜区,表明SnRk2s可能与细胞膜或核受体结合而相互作用[7,27-28]。本研究发现,AsSnRK2.7蛋白中也存在一个潜在的N-肉豆蔻酰化作用位点和一个潜在的跨膜区,但仅定位在细胞核中。
尽管在5个发育阶段的根、叶、茎和穗(抽穗期、灌浆期和成熟期)组织中均能检测到AsSnRK2.7基因的表达,但在不同发育阶段的不同组织中,AsSnRK2.7基因的相对表达量不同,这与小麦中SnRK2成员研究的报道一致。如小麦TaSnRK2.7基因的表达量在苗期的根组织中较高,而在苗期的叶片、孕穗期的穗和抽穗期的剑叶组织中较低[28]。TaSnRK2.4基因在孕穗期的穗中表达量较高,在苗期的根和抽穗期的剑叶中次之,而在苗期的叶片中最低[27]。这些结果表明,SnRK2基因存在着明显的空间和时间上的表达差异,在正常生长条件下调节不同生理代谢过程。
根据C末端富集的酸性氨基酸的类型,SnRK2蛋白激酶可分为两个亚组:富含天冬氨酸的SnRK2a(为ABA依赖型SnRK2s)和富含谷氨酸的SnRK2b(为非ABA依赖型SnRK2s)[2,29]。根据序列相似性、功能域结构和细胞功能,植物SnRK2亚家族可分为三个亚组:Group Ⅰ、Group Ⅱ和Group Ⅲ,其中,Group Ⅱ和Group Ⅲ两个亚组属于SnRK2a,而Group Ⅰ属于SnRK2b[1]。Group III中的SnRK2成员对ABA具有强烈的响应,Group II成员不受或微弱受ABA激活,Group I成员不被ABA胁迫诱导,而Group Ⅰ、Group Ⅱ和Group Ⅲ成员均受渗透胁迫的激活[22,24]。本研究发现,AsSnRK2.7属于Group I(SnRK2b)中成员,对外源ABA的诱导不敏感,说明AsSnRK2.7蛋白激酶可能是一个非ABA依赖型SnRK2成员,这与Group I成员不被ABA胁迫诱导的研究结果一致。然而,在玉米中ZmSnRK2.4、ZmSnRK2.5、ZmSnRK2.7和小麦中的TaSnRK2.4也属于Group I成员,但却能被ABA胁迫诱导[23,27]。此外,在本研究中,AsSnRK2.7基因能够积极应答低温、盐和PEG胁迫,表明AsSnRK2.7基因同其他SnRK2成员一样在植物信号转导和胁迫耐受性中起关键作用。然而,前人研究表明,ZmSnRK2.7基因不含低温应答元件(LTRE),但仍可被冷胁迫所诱导[23]。所以,非ABA依赖型SnRK2基因结构与其受逆境胁迫应答的关系还需进一步深入研究。
已有研究表明,SnRK2蛋白激酶N端激活环上丝氨酸残基的可逆磷酸化可调控SnRK2活性[31-33]。目前对于非ABA依赖型SnRK2的可逆磷酸化报道较少,但有关ABA依赖型SnRK2的可逆磷酸化的研究较多。SnRK2蛋白激酶在响应渗透胁迫与ABA过程中自身磷酸化机制不同,渗透胁迫诱导的磷酸化水平普遍高于ABA诱导的磷酸化水平,另外在渗透胁迫下不同的SnRK2蛋白激酶的磷酸化水平也不同[31]。烟草中的NtOSAK和拟南芥中的SnRK2.10均属于GroupI,为响应渗透胁迫的非ABA依赖型SnRK2成员,这2个蛋白激酶激活环中均存在2个蛋白磷酸化位点,且这2个位点的可逆磷酸化均可调节激酶活性,并存在顺序性,其中一个丝氨酸的磷酸化是另一个丝氨酸磷酸化所必需的[29-30]。然而,Group III中拟南芥ABA依赖型激酶则是通过2个位点单独磷酸化来调控激酶活性[30]。本研究基于NetPhos在线分析,发现AsSnRK2.7蛋白质的激活环(ICDFGYSKSSLLHSKPKSTVGTPAYIAPE)上存在2个可逆的蛋白质磷酸化残基(带下划线的氨基酸残基),分别位于Ser-154和Ser-158,但其激活环上的2个磷酸化位点磷酸化是否对AsSnRK2.7蛋白激酶活性调控具有协同作用,还需进一步的研究。
SnRK2蛋白激酶可通过磷酸化作用来影响下游底物的活性。SnRK2蛋白激酶如SnRK2.2/SRK2D、SnRK2.6/SRK2E和SnRK2.3/SRK2I可以在体外磷酸化拟南芥AREB1蛋白[11]。目前,植物中仅有少数蛋白被被证实为SnRK2蛋白激酶的底物,包括转录bZIP型转录因子ABF/AREB和AP2/ERF-型转录因子DREB[15,17]。此外,Wang等[16]通过定量磷酸化蛋白组学鉴定了一些SnRK2s底物,这些SnRK2s底物包括参与开花时间调控、RNA和DNA结合、miRNA和表观遗传调控、信号转导(ABF2、AREB3、TAF5、EEL和FBH3等)、叶绿体功能及许多其他细胞过程的蛋白质。本研究使用STRING在线程序在小麦中找到与AsSnRK2.7同源蛋白可能存在互作关系的蛋白,包括bZIP转录因子、胱硫醚-β-合成酶和环核苷酸-单磷酸依赖蛋白激酶。这些蛋白对植物抗逆性有重要影响,因此,这些蛋白的发现将为利用燕麦AsSnRK2.7基因来改善植物的抗逆性提供理论基础。
