蝴蝶兰PhNAC1基因序列分析及对低温胁迫的响应
2020-07-30梁芳张燕牛苏燕袁秀云崔波
梁芳 张燕 牛苏燕 袁秀云 崔波

摘 要: 為探讨NAC转录因子在蝴蝶兰低温胁迫响应中的分子调控机理,该研究以蝴蝶兰的叶片为材料,运用RT-PCR及RACE技术克隆得到一条蝴蝶兰的NAC转录因子基因完整的cDNA序列,命名为PhNAC1(GenBank登录号MF797909),并分析了其在两种低温条件下的表达模式。结果表明:PhNAC1基因cDNA序列全长1 442 bp,ORF全长942 bp,编码313个氨基酸。预测其蛋白分子量为35.22 kDa,等电点为6.95,属于稳定亲水性蛋白。二级结构预测表明,无规则卷曲和延伸链为该蛋白的主要结构元件,与三级结构预测结果基本相符。PhNAC1编码的氨基酸序列与其他已登录的兰科植物NAC蛋白进行同源序列比对,表明与小兰屿蝴蝶兰(XP_0205763790)亲缘关系较近,序列一致性达97%,其次为铁皮石斛(XP_020695081),一致性为84%。实时荧光定量PCR分析表明,PhNAC1基因在营养器官和生殖器官中均有表达,在蕊柱中的表达量最高。在11 ℃/6 ℃低温条件下,PhNAC1基因的转录表达水平在前5天随着处理时间逐渐升高,到第7天开始下降;在4 ℃低温条件下,PhNAC1基因的表达水平在处理0.5 h时表达量有所下降,1 h后表达量上升至对照水平,之后无明显变化,在处理24至48 h又逐渐升高,推测PhNAC1基因参与蝴蝶兰低温胁迫响应。
关键词: 蝴蝶兰, NAC转录因子, 表达特性, 低温胁迫, 序列分析
中图分类号: Q785;Q786 文献标识码: A
文章编号: 1000-3142(2020)06-0845-09
开放科学(资源服务)标识码(OSID) :
Abstract: NAC transcription factors are involved in many processes of plant development, which play an important role in stress response. The NAC transcription factor named PhNAC1 (GenBank accession No. MF797909) was cloned from the leaves of Phalaenopsis using RT-PCR and RACE method. The full-length of PhNAC1 was 1 442 bp, which contained a 942 bp ORF that encoding a protein with 313 amino acids residues. The molecular weight of the putative protein was 35.22 kDa and the theoretical pI was 6.95, a hydrophilic and unstable protein. Prediction of secondary structure showed that the random coil and extended strand were the main structural elements of the protein, which conformed to the prediction of tertiary structure. Amino acid sequence alignment and phylogenetic tree analysis between the protein of PhNAC1 and NACs from other Orchids showed that PhNAC1 was close to Phalaenopsis equestris (XP_020576379) with the sequence identity of 97%, followed by the Dendrobium catenatum (XP_020695081) with 84%. The qRT-PCR analysis indicated that the PhNAC1 gene was expressed in both of the vegetative and reproductive organs, and the expression level was the highest in the column. Under the cold stress of 11 ℃/6 ℃, the expression level of PhNAC1 gene was increased significantly with treatment time in leaves during the first five days, and then decreased at the 7th day. Under the cold stress of 4 ℃, the expression level was decreased slightly at 0.5 h, then recovered and remained to the initial level at 1 h after treatment, whereas the expression level was increased obviously after treating for 24、48 h, indicating that PhNAC1 may be involved in cold stress response of Phalaenopsis. This research will be useful for study of molecular mechanism of NAC transcription factor in cold stress response in Phalaenopsis species.
Key words: Phalaenopsis, NAC transcription factor, expression pattern, cold stress, sequence analysis
基因的转录调控控制着植物生长发育的许多重要生理过程,如逆境应答、信号转导、形态建成等。转录因子通过与特异的目的基因启动子区相结合,激活或抑制目的基因的转录效率,从而使植物对外界刺激做出响应。近年来,NAC、MYB、WRKY、AP2/EREBP等转录因子在植物逆境中的响应机制被广泛研究(Scarpecie et al., 2013;Xue et al., 2014;Guan et al., 2014;Butt et al., 2017)。其中,NAC转录因子是目前发现数量最大的植物特有的转录因子家族之一(Olsen et al., 2005)。最早在矮牵牛NAM、拟南芥ATAF1/2和CUC2基因编码蛋白的N端发现一段高度保守的氨基酸序列,以此3个基因的首字母命名為NAC结构域,并将包含NAC结构域的蛋白称为NAC转录因子。NAC转录因子的研究多集中在被子植物。通过对拟南芥和水稻的NAC转录因子进化分析,NAC结构域可分为两组18个亚组,NAC结构域高度保守,仅有少数氨基酸差异,在NAC蛋白C端转录激活区有13个保守基序(Ooka et al., 2003)。
NAC转录因子参与植物生长发育诸多过程,包括胚的发育(Larsson et al., 2011;Zhao et al., 2011)、侧根形成(Hao et al., 2011)、叶片衰老(Jia et al., 2018)、果实成熟(Kou et al., 2012)、激素信号传递和调控(Wang et al., 2014)等。此外,NAC转录因子在植物逆境应答过程中起着关键作用(Shao et al., 2015)。现已在许多植物中分离到NAC基因证明其能提高植物对低温胁迫的抗性,如水稻SNAC2(Hu et al., 2008)、小麦TaNAC8(Xia et al., 2010)、TaNAC2(Mao et al., 2012)和TaNAC47(Zhang et al., 2015)、芒草MlNAC5(Yang et al., 2015)和MlNAC9(Zhao et al., 2016)、苜蓿MfNAC3(Qu et al., 2016)等。虽然与抗逆相关的NAC转录因子在拟南芥和水稻中研究较为深入,但在兰花中的研究还见之甚少,仅见Mita et al.(2006)报道蕙兰(Cymbidium faberi)CyNAC1转录因子参与高温(25~30 ℃)导致幼嫩花芽的坏死过程,发现在变异的抗高温植株中,该基因表达量明显比野生型低。关于兰科植物在低温胁迫过程中NAC转录因子的相关研究目前尚未见报道。
蝴蝶兰(Phalaenopsis amabilis)是世界著名的高档花卉,在我国各地广泛栽培。原产于热带亚热带地区,性喜暖畏寒,生长适温为18~28 ℃,温度过高或过低均会限制蝴蝶兰的生长。因此,低温是影响蝴蝶兰生长的重要环境因子之一。我国北方地区蝴蝶兰均在现代化温室内种植,冬春季节温度较低时需对温室进行加温,因此导致生产成本升高,耗能巨大制约了蝴蝶兰产业的健康发展。因此,研究蝴蝶兰抗冷的生理生化机制,培育抗冷性新品种,对于蝴蝶兰产业的健康可持续发展具有重要意义。
1 材料与方法
1.1 材料与处理
所用材料为蝴蝶兰栽培品种“大辣椒”(Phalaenopsis hybrid ‘Big Chili),由郑州师范学院生物工程研究所提供。两种低温处理条件:将5叶期的蝴蝶兰植株置于植物人工光照培养箱(美国,PERCIVAL E-41HO2)内27 ℃/22 ℃预培养15 d,使所有的实验苗Fv/Fm≥0.79。实验采用模拟自然状态逐步降温法,分为两个阶段,第一阶段低温驯化:昼夜温度20 ℃/16 ℃处理3 d,16 ℃/11 ℃处理3 d;第二阶段11 ℃/6 ℃低温处理7 d。其他培养条件:光暗比12 h/12 h,光强为60 μmol·m-2·s-1,相对湿度70%~90%。取样方法:以27 ℃/22 ℃预培养结束取样为对照,昼夜温度11 ℃/6 ℃处理第1、2、3、5、7天取样。另外4 ℃低温处理,用蝴蝶兰3叶期瓶苗,置于4 ℃冰箱内分别于0、0.5、1、2、4、8、12、24和48 h取样。
1.2 方法
1.2.1 总RNA的提取和cDNA第一链的合成 各样品总RNA的提取采用多糖多酚植物总RNA提取试剂盒。对提取的总RNA经检测合格后,用M-MLV反转录酶对其进行反转录合成单链cDNA第一链,用于PhNAC1基因的克隆;用PrimeScript RT reagent Kit with gDNA Eraser试剂盒反转录成cDNA第一链,用于实时荧光定量PCR(qRT-PCR)检测目的基因表达量。
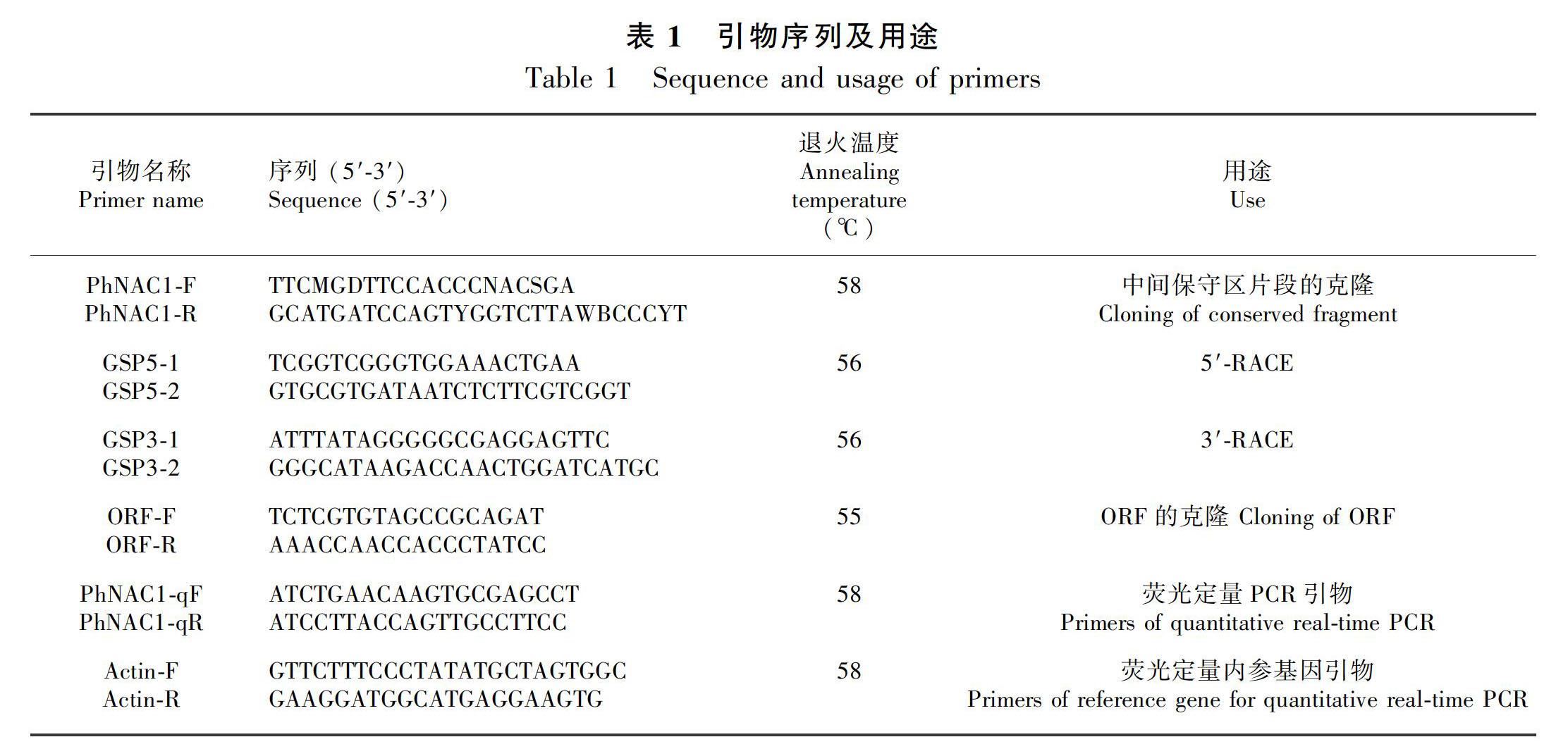
1.2.2 PhNAC1基因全长的扩增及ORF的预测与验证 利用DNAMAN和Primer 5.0软件,以GenBank中已登录的JF831198(铁皮石斛, Dendrobium candidum)、KC954544(铁皮石斛, Dendrobium officinale)和AB257312(蕙兰, Cymbidium hybrid)为模板,设计1对简并引物PhNAC1-F和PhNAC1-R(表1),用于扩增蝴蝶兰NAC基因中间保守片段。将得到的中间保守序列经BLAST比对正确后,分别设计1对5′端特异引物(GSP5-1、GSP5-2)和3′端特异引物(GSP3-1、GSP3-2)。对中间保守区片段、5′-RACE扩增片段和3′-RACE扩增片段进行分析比对并拼接, 通过BLAST对得到的基因全长进行同源性比对,确定得到的基因为NAC基因。在ORF区两端设计特异引物ORF-F和ORF-R对ORF序列进行验证。
1.2.3 PhNAC1基因的生物信息学分析 蝴蝶兰PhNAC1基因蛋白结构域、理化性质、亲疏水性、磷酸化位点、以及蛋白质的二级和三级结构等采用在线软件进行分析。此外,核苷酸与氨基端序列的同源性用DNAMAN软件进行比对分析,并用Clustal X和MEGA构建系统进化树。
1.2.4 PhNAC1基因的表达特性分析 取蝴蝶兰的根、叶(第2片)、花葶、花芽、萼片、翼瓣、唇瓣及蕊柱,迅速投入液氮中速冻后-80 ℃冰箱保存,用于PhNAC1基因在不同组织中的表达分析。低温胁迫处理均取第2片成熟叶,每个取样点有三个生物学重复,每个重复由三株不同植株混合而成。
根据得到的ORF序列,设计qRT-PCR引物PhNAC1-qF和PhNAC1-qR,以蝴蝶兰Actin基因作为内参基因。采用SYBR Premix Ex Taq II试剂盒进行qRT-PCR,反应体系为25 μL,反应条件:95 ℃ 15 s,58 ℃ 15 s,72 ℃ 15 s(40個循环),反应在Eppendorf Mastercycler荧光定量PCR仪上进行,每个样品重复3次,同时做阴性对照。基因相对表达量用公式2-ΔΔCt来计算(Vandesompele et al., 2002)。
2 结果与分析
2.1 PhNAC1基因全长的扩增
以蝴蝶兰叶片cDNA为模板,以引物PhNAC1-F和PhNAC1-R进行中间片段的扩增,得到约350 bp的保守片段(图1:A),经BLASTn比对分析表明克隆的片段为NAC基因片段。根据该片段序列,利用引物GSP3-1和GSP3-2进行3′-RACE扩增,得到大小约780 bp目的片段(图1:B),利用引物GSP5-1和GSP5-2进行5′-RACE扩增,得到大小约320 bp目的片段(图1:C)。将测序得到的中间保守区片段、5′-RACE扩增片段及3′-RACE扩增片段进行分析拼接,得到全长为1 442 bp的基因序列,其中包括一个完整的长为942 bp ORF,编码313个氨基酸。对ORF区进行克隆验证,得到997 bp的片段(图1:D), 与全长部分序列完全相M. DL2000 DNA 标记; 1. 中间保守片段; 2. 3′-RACE片段; 3. 5′-RACE片段; 4. 开放阅读框。
同。经BLAST分析, 所得的核苷酸序列与小兰屿蝴蝶兰(Phalaenopsis equestris, XP_020576379)一致性为98%,与小野芭蕉(Musa acuminata)NAC79基因序列一致性为79%,通过比对分析表明克隆的基因为NAC家族基因。将该基因命名为PhNAC1,GenBank登录号MF797909。
2.2 PhNAC1基因的生物信息学分析
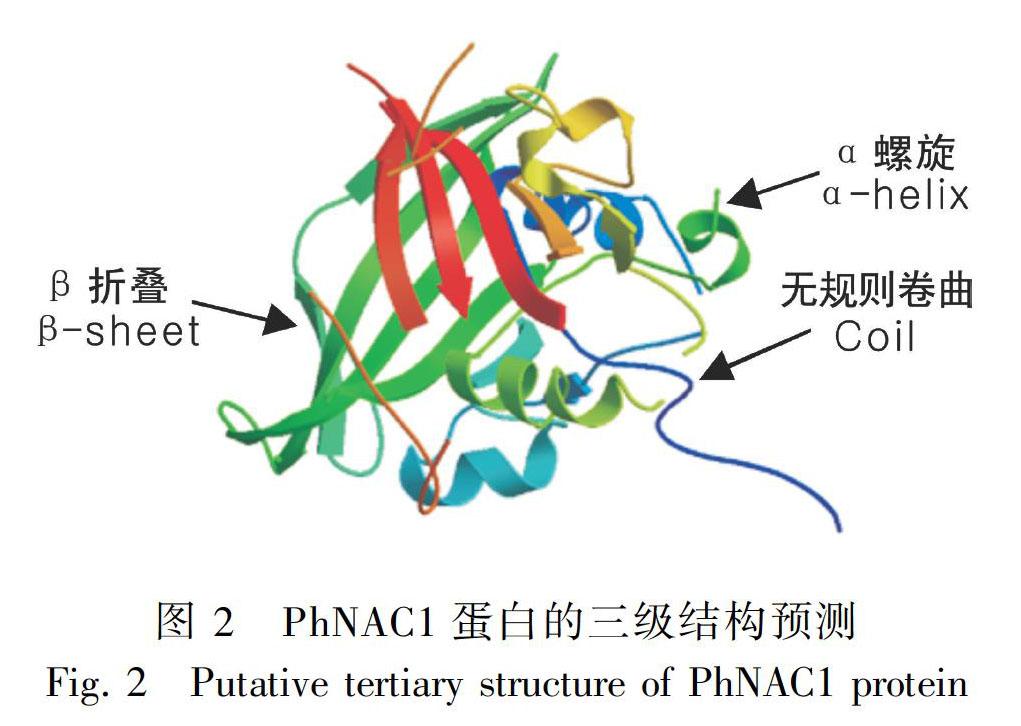
2.2.1 PhNAC1蛋白的基本性质及空间结构分析 通过对PhNAC1基因推导的氨基酸序列进行分析,表明该蛋白属于植物特有的NAC转录因子家族。CDD数据库分析结果表明,该蛋白第8至第134个氨基酸为NAC结构域。生物信息分析结果表明PhNAC1蛋白属于亲水性蛋白,该蛋白分子质量约为35.22 kDa、理论等电点pI为6.95,分子式为C1563H2375N417O477S18;正负电荷残基总数均为35。在组成PhNAC1蛋白的20种氨基酸中,Ser所占的比例最高,为10.2%,His和Cys所占比例最低,仅为1.6%。该蛋白脂肪指数56.10,不稳定指数为33.53,属于稳定蛋白。PhNAC1蛋白序列中存在41个潜在的磷酸化位点,包括21个Ser位点、14个Thr位点和6个Tyr位点。对PhNAC1蛋白进行二级结构预测,结果表明该蛋白二级结构由α螺旋(Hh)、伸展链(Ee)和无规则卷曲(Cc)组成,其中无规则卷曲占70.6%,伸展链占27.48%,α螺旋仅占1.92%。
采用SWISS-MODEL同源建模的方法,以4dul.1 (拟南芥ANAC019)为模型对PhNAC1进行蛋白质三级结构建模(图2),结果表明PhNAC1与4dul.1相似性为56.33%,推测PhNAC1是与胁迫响应相关的NAC转录因子。
2.2.2 PhNAC1基因同源性与系统进化分析 将蝴蝶兰PhNAC1基因编码的氨基酸序列(AXF50252)与已登录到NCBI上的其他4种兰科植物的 NAC蛋白进行序列比对分析(图3),PhNAC1与小兰屿蝴蝶兰(Phalaenopsis equestris, XP_020576379)的氨基酸序列一致性最高为97%,均为313个氨基酸,二者仅有8个氨基酸的差异;其次为铁皮石斛(Dendrobium catenatum, XP_020695081),二者序列一致性为84%;与深圳拟兰(Apostasia shenzhenica, PKA66962)和杂交兰(Cymbidium hybrid, BAF36563)的一致性较低,分别为51%和32%。将PhNAC1氨基酸序列经BLAST比对,选择序列一致性较高的15种植物,利用NJ法构建无根系统进化树(图4),显示蝴蝶兰与小兰屿蝴蝶兰、铁皮石斛、深圳拟兰亲缘关系最近,其次为番红花(Crocus sativus, ABU40776),与其他植物的亲缘关系较远。
2.3 PhNAC1基因的表达特性分析
由图5可知,PhNAC1基因在蝴蝶兰的营养器官和生殖器官中均有表达,在蕊柱中的表达水平最高,明显高于其他组织,在根、叶和翼瓣中表达水平最低。在11 ℃/6 ℃低温条件下,PhNAC1基因的转录表达水平在处理1、2、3、5 d逐渐升高,处理第7天明显下降(图6:A);在4 ℃低温条件下,PhNAC1基因的表达水平在处理0.5 h表达量下降,处理1 h后表达量上升至对照水平,之后无明显变化,在处理24 h和48 h后又明显升高(图6:B)。
3 讨论与结论
NAC转录因子是一类数量巨大的植物转录因子,在植物的生长发育调控及抗逆响应中发挥重要的作用(Sun et al., 2018;Singh et al., 2016)。Liu et al.(2019)报道NAC转录因子参与高温诱导的荔枝花序败育。Wang et al.(2014)首次报道西瓜NAC转录因子CcNAC1和CcNAC2参与光信号途径和生长激素信号途径。通过抑制ABA信号通路相关基因,过表达ONAC066可以提高水稻对稻瘟病和白叶枯病的抗性(Liu et al., 2018)。本文从蝴蝶兰中通过同源克隆的方法得到一个NAC转录因子基因PhNAC1,对该基因编码的氨基酸序列进行分析,发现在N端具有NAC结构域,该结构域是NAC转录因子特有的结构域。运用生物信息学方法对PhNAC1蛋白进行分析预测,PhNAC1是一种稳定的亲水性蛋白,具有多个潜在的磷酸化位点。将PhNAC1蛋白与其他已登录的兰科植物NAC蛋白序列进行比较,发现都具有高度保守的N端和多变的C端,N端含有A、B、C、D、E 5个典型亚结构域(Puranik et al., 2012)。进化分析表明,PhNAC1与小兰屿蝴蝶兰的亲缘关系最近,仅有8个氨基酸的差异,序列一致性达到97%。三级空间结构预测表明,PhNAC1与拟南芥ANAC019的相似度最高,推测PhNAC1是与胁迫响应相关的NAC转录因子(Jenson et al., 2010)。
用实时荧光定量PCR对PhNAC1基因在蝴蝶兰各个组织中的表达特性进行了研究,发现PhNAC1基因在蝴蝶兰的根、叶及各个花器官组织A-E下划线部分分别代表NAC结构域中5个保守的亚结构域。
中均有表达,其中在蕊柱中的表达量明显高于其他部位和组织。蝴蝶兰的蕊柱(Column)是雄蕊和花柱、柱头完全愈合而成的一种柱状体,顶端着生一个花粉块。PhNAC1基因在蕊柱中的高表达机理还有待进一步研究。PhNAC1基因在低温胁迫条件下的表达分析表明,在11 ℃/6 ℃的昼夜温度条件下,该基因的表达水平随着处理时间逐渐升高,到第5天表达量是对照的34倍,第7 天表达量开始下降。在4 ℃低温条件下,处理0.5 h表达量有所下降,之后开始上升恢复到对照水平维持至12 h,到24 h后表达量又开始上升,由此推测PhNAC1基因在蝴蝶兰低温胁迫响应中发挥作用。拟南芥和水稻全基因组表达谱研究表明,一些NAC基因受到高温、干旱、低温或病害中的一个或多个因素诱导表达。如拟南芥中的ANAC019、ANAC055、ANAC072(RD26)均对不同非生物胁迫有应答反应(Fujita et al., 2004;Delessert et al., 2005;Fang et al., 2008);在水稻中,OsNAC6和SNAC2超量表达可以提高幼苗发育阶段对干旱、高盐和低温的耐受性(Lu et al., 2007;Nuruzzaman et al., 2010)。
本研究首次克隆了蝴蝶兰的NAC基因PhNAC1,分析了其在两种低温条件下的表达模式,结果显示其在低温胁迫24 h后即开始上调表达,表明蝴蝶兰PhNAC1转录因子能够对低温做出积极响应,以及时调节下游冷相關基因的表达,减轻逆境对植物可能造成的伤害。
参考文献:
R2R3 MYB gene, in transgenic Arabidopsis plays an important role in drought tolerance [J]. BMC Plant Biol, 17(1): 142. Doi: 10.1186/s12870-017-1078-3.
DELESSERT C, KAZAN K, WILSON IW, et al., 2005. The transcription factor ATAF2 represses the expression of pathogenesis-related genes in Arabidopsis [J]. Plant J, 43(5): 745-757.
FANG Y, YOU J, XIE K, et al., 2008. Systematic sequence analysis and identification of tissue-specific or stress-responsive genes of NAC transcription factor family in rice [J]. Mol Genet Genom, 280(6): 547-563.
FUJITA M, FUGITA Y, MARUYAMA K, et al., 2004. A dehydration-induced NAC protein, RD26, is involved in a novel ABA-dependent stress-signaling pathway [J]. Plant J, 39(6):863-876.
GUAN QM, YUE XL, ZENG HT, et al., 2014. The protein phosphatase RCF2 and its interacting partner NAC019 are critical for heat stress-responsive gene regulation and thermotolerance in Arabidopsis [J]. Plant Cell, 26: 438-453.
HAO YJ, WEI W, SONG QX, et al., 2011. Soybean NAC transcription factors promote abiotic stress tolerance and lateral root formation in transgenic plants [J]. Plant J, 68: 302-313.
HU HH, YOU J, FANG Y, et al., 2008. Characterization of transcription factor gene SNAC2 conferring cold and salt tolerance in rice [J]. Plant Mol Biol, 67(1-2): 169-181.
JENSEN MK, KJAERSGAAD T, NIELSEN MM, et al., 2010. The Arabidopsis thaliana NAC transcription factor family: Structure-function relationships and determinants of ANAC019 stress signaling [J]. Biochem J, 426(2): 183-196.
JIA D, GONG X, LI M, et al., 2018. Overexpression of a novel apple NAC transcription factor gene, MdNAC1, Confers the dwarf phenotype in transgenic apple (Malus domestica) [J]. Genes, 9(5): 229. Doi:10.3390/genes9050229.
KOU X, WATKINS CB, GAN S, 2012. Arabidopsis AtNAP regulates fruit senescence [J]. J Exp Bot, 63: 6139-6147.
LARSSON E, SITBON F, SUNDSTROM J, et al., 2011. NAC regulation of embryo development in conifers [J]. BMC Proceed, 5(7): 67. Doi: 10.1186/1753-6561-5-S7-P67.
LIU H, WANG CC, CHEN H, et al., 2019. Genome-wide transcriptome analysis reveals the molecular mechanism of high temperature-induced floral abortion in Litchi chinensis [J]. BMC Genomics, 20(1):127. Doi: 10.1186/s12864-019-5493-8
LIU Q, YAN SJ, HUANG WJ, et al., 2018. NAC transcription factor ONAC066 positively regulates disease resistance by suppressing the ABA signaling pathway in rice [J]. Plant Mol Biol, 98(4-5):289-302.
LU PL, CHEN NZ, AN R, et al., 2007. A novel drought-inducible gene, ATAF1, encodes a NAC family protein that negatively regulates the expression of stress-responsive gene in Arabidopsis [J]. Plant Mol Biol, 63(2): 289-305.
MAO X, ZHANG H, QIAN X, et al., 2012. TaNAC2, a NAC-type wheat transcription factor conferring enhanced multiple abiotic stress tolerances in Arabidopsis [J]. J Exp Bot, 63(8): 2933-2946.
MITA S, HENMI R, OHNO H, 2006. Enhanced expression of genes for ACC synthase, ACC oxidase, and NAC protein during high-temperature-induced necrosis of young inflorescences of Cymbidium [J]. Physiol Plant, 128: 476-486.
NURUZZAMAN M, MANIMEKALAI R, SHARONI MA, et al., 2010. Genome-wide analysis of NAC transcription factor family in rice [J]. Gene, 465: 30-44.
OLSEN NA, ERNST AH, LEGGIO LL, et al., 2005. NAC transcription factors: Structurally distinct, functionally diverse [J]. Trends Plant Sci, 10(2): 80-88.
OOKAH, SATOH K, DOI K, et al., 2003. Comprehensive analysis of NAC family genes in Oryza sativa and Arabidopsis thaliana [J]. DNA Res, 10(6): 239-247.
PURANIK S, SAHU PP, SRIVASTAVA PS, et al., 2012. NAC proteins: Regulation and role in stress tolerance [J]. Trends Plant Sci, 17(6): 369-381.
QU YT, DUAN M, ZHANG ZQ, et al., 2016. Overexpression of the Medicago falcata NAC transcription factor MfNAC3 enhances cold tolerance in Medicago truncatula [J]. Environ Exp Bot, 129: 67-76.
SCARPECI ET, ZANOR IM, MUELLER-ROEBER B, et al., 2013. Overexpression of AtWRKY30 enhances abiotic stress tolerance during early growth stages in Arabidopsis thaliana [J]. Plant Mol Biol, 83(3): 265-277.
SHAO H, WANG H, TANG X, 2015. NAC transcription factors in plant multiple abiotic stress responses: Progress and prospects [J]. Front Plant Sci, 6: 902. Doi: 10.3389/fpls.2015.00902.
SINGH S, GROVER A, NASIM M, 2016. Biofuel potential of plants transformed genetically with NAC family genes [J]. Front Plant Sci, 7: 22. Doi: 10.3389/fpls.2016.00022.
SUN H, HU ML, LI JY, et al., 2018. Comprehensive analysis of NAC transcription factors uncovers their roles during fiber development and stress response in cotton [J]. BMC Plant Biol, 18(1): 150. Doi: 10.1186/s12870-018-1367-5.
VANDESOMPELEJ, PRETER DK, PATTYN F, et al., 2002. Accurate normalization of real-time quantitative RT-PCR data by geometric averaging of multiple internal control genes [J]. Genom Biol, 3(7):1-11.
WANG Z, RASHOTTE AM, DANE F, 2014. Citrullus colocynthis NAC transcription factors CcNAC1 and CcNAC2 are involved in light and auxin signaling [J]. Plant Cell Rep, 33(10): 1673-1686.
XIA N, ZHANG G, SUN YF, et al., 2010. TaNAC8, a novel NAC transcription factor gene in wheat, responds to stripe rust pathogen infection and abiotic stresses [J]. Physiol Mol Plant Pathol, 74(5-6): 394-402.
XUE Y, WANG YY, PENG HR, et al., 2014. Transcription factor MdCBF1 gene increases freezing stress tolerance in transgenic Arabidopsis thaliana [J]. Biol Plantarum, 58(3): 499-506.
YANG XW, WANG XY, JI L, et al., 2015. Overexpression of a Miscanthus lutarioriparius NAC gene MlNAC5 confers enhanced drought and cold tolerance in Arabidopsis [J]. Plant Cell Rep, 34(6): 943-958.
ZHAO P, WANG WJ, SUN MX, 2011. Characterization and expression pattern analysis of DcNAC gene in somatic embryos of Dendrobium candidum Wall Ex Lindl [J]. Plant Cell Tissue Organ Cult, 107(1): 151-159.
ZHAO X, YANG XW, PEI SQ, et al., 2016. The Miscanthus NAC transcription factor MlNAC9 enhances abiotic stress tolerance in transgenic Arabidopsis [J]. Gene, 586: 158-169.
ZHANG LN, ZHANG LC, XIA C, et al., 2015. The novel wheat transcription factor TaNAC47 enhances multiple abiotic stress tolerances in transgenic plants [J]. Front Plant Sci, 6: 1174. Doi: 10.3389/fpls.2015.01174.
ZHOU Y, HUANG WF, LIU L, et al., 2013. Identification and functional characterization of a rice NAC gene involved in the regulation of leaf senescence [J]. BMC Plant Biol, 13: 132. Doi: 10.1186/1471-2229-13-132.
(責任编辑 何永艳)
