CRISPR/Cas13系统敲减HEK293T细胞ku70和lig4基因表达
2020-07-29王豪强李国玲黄广燕李紫聪郑恩琴徐铮杨化强吴珍芳张献伟刘德武
王豪强,李国玲,黄广燕,李紫聪,郑恩琴,徐铮,杨化强,2,吴珍芳,2,张献伟,2,刘德武
·生物技术与方法·
CRISPR/Cas13系统敲减HEK293T细胞和基因表达
王豪强1,李国玲1,黄广燕1,李紫聪1,郑恩琴1,徐铮1,杨化强1,2,吴珍芳1,2,张献伟1,2,刘德武1
1 华南农业大学 动物科学学院 国家生猪种业工程技术研究中心,广东 广州 510642 2 温氏食品集团股份有限公司,广东 云浮 527400
成簇的规律间隔的短回文重复序列 (Clustered regularly interspaced short palindromic repeats, CRISPR)/CRISPR相关蛋白 (CRISPR-associated proteins,Cas) 系统是目前基因编辑、基因表达研究的热点,其中靶向RNA的CRISPR/Cas13系统的开发为RNA的干扰和编辑提供了新的方向。文中针对HEK293T细胞非同源末端连接 (Nonhomologous end joining,NHEJ) 通路修复关键因子Ku70和Lig4的编码序列,设计并合成CRISPR/Cas13a、b系统相应的gRNA,检测其对和基因表达的影响。结果显示,Cas13a对和的RNA敲减效果可以达到50%以上,Cas13b对和的RNA敲减效果分别达到92%和76%;同时Cas13a、b系统能将Ku70和Lig4蛋白水平分别下调至68%和53%。CRISPR/Cas13系统可有效敲减HEK293T细胞RNA和蛋白质表达,为基因功能和调控研究提供一种新的策略。
CRISPR/Cas13,,,基因敲减
CRISPR/Cas系统是来源于细菌与古菌的一种后天免疫防御系统,主要用于防止外来质粒或噬菌体的侵入[1],其中ⅡCRISPR/Cas9系统能对基因组DNA实现精确编辑,目前已被广泛应用于农业生产及医学领域[2]。最近研究发现一种新型的靶向RNA的CRISPR蛋白Cas13,它的发现及开发为人类干扰和编辑RNA提供更便捷的方法。Cas13属于2类Ⅵ型CIRSPR关联蛋白家族,该蛋白目前共发现4种亚型,分别是Cas13a (也称作C2c2)、Cas13b、Cas13c和Cas13d[3]。与CRISPR/Cas9类似,Ⅵ型CRISPR系统含有一个效应蛋白即Cas13蛋白,它与CRISPR RNA (crRNA) 结合后形成一个crRNA引导的RNA靶向复合物[4-7]。截至目前,所发现的Cas13蛋白均有两个截然不同的核糖核酸酶活性。其一,主要用于pre-crRNA的加工,有利于Ⅵ型干扰复合物的形成;其二,由两个高等真核和原核核酸结合结构域 (Higher eukarytoes and prokaryotes nucleotide-binding domain,HEPN) 提供,是病毒干扰过程中降解外来RNA的关键。另外,除了Cas13蛋白外,部分Ⅵ型系统拥有额外的附属蛋白,这些蛋白是非必需的,但具有调节干扰效果的活性[5-6]。Ⅳ-B和Ⅵ-C系统缺乏适应性基因和,说明它们可能利用同一基因组内的其他Cas位点获取适应模块,或者丢失适应模块,不再需要获取新的原始间隔序列。特别地,Cas13b存在两种亚型Ⅵ-B1和Ⅵ-B2,其中Ⅵ-B1系统拥有一个额外的开放阅读框Csx28,它编码一种小蛋白,该小蛋白带着一个假定的跨膜结构域(或信号肽) 和一个发散的HEPN结构域;Ⅵ-B2系统拥有一个不同的开放阅读框Csx27,它编码一种包含3–4个可预测的跨膜结构域。Ⅵ-D系统具有更小的Cas13蛋白,并且携带一个与原核防御系统有关的WYL结构域附属蛋白。张锋实验室根据Cas13蛋白靶向RNA的性质,设计并开发了靶向RNA的CRISPR/Cas13a、b系统[8],通过设计28/30 nt的RNA互补序列,可以实现特异性切割靶RNA和非特异性切割周围环境的RNA。研究表明,来源于纤毛菌属的LwaCas13a具有50%左右的RNA敲减水平[8],来源于普氏菌属 (sp. P5-125) 的PspCas13b在萤火虫素酶报告载体上具有90%–95%的RNA敲减水平[9]。相对传统的RNA干扰 (RNA interference,RNAi) 技术,CRISPR/Cas13系统具有更好的专一性和更低的脱靶效应[8-11];相对于CRISPR/Cas9等DNA编辑工具,RNA水平的编辑避免了任何由于对遗传基因组DNA操作所引发的脱靶效应的担忧,为基因编辑器的改造和疾病的治疗提供了多种可选择的高效手段。
本课题组前期研究发现RNA干扰NHEJ关键因子Ku70、Ku80、PNKP、Lig4等可显著提高猪原代成纤维细胞的同源定向修复 (Homology- directed repair,HDR) 效率[12-13],同时CRISPR干扰Ku70和Ku80同样能够显著提高HDR编辑效率[14-15]。因此,本研究尝试利用CRISPR/Cas13a、b系统特异性敲减HEK293T细胞和基因表达,为提高HDR效率提供一种新的策略,为CRISPR/Cas13蛋白的研究提供经验。
1 材料与方法
1.1 材料
Cas13a系统 (#91906和#91924)、Cas13b系统(#103854和#103862) 和mCherry2-C1质粒 (# 54563) 均来源于美国Addgene。HEK293T购自美国标准生物品收藏中心 (#ATCC ACS-4500)。
1.2 Cas13a、b系统构建
针对人(GeneID: 2547) 和(GeneID: 3981) 2个基因的CDS序列分别设计对应的gRNA (表1和表2),并由北京六合华大基因科技有限公司合成gRNA oligo引物。参考Thermo Fisher Scientific的Fast DigestⅠ说明书线性化Cas13a、b的U6质粒。将上述合成的引物退火后与线性化的U6质粒16 ℃过夜连接,详细体系见TaKaRa T4 DNA Ligase (#2011A) 说明书。将连接好的环状DNA转化到大肠杆菌DH5α感受态细胞中,并划线涂板。第2天将扩大培养的单克隆菌落(AmpR) 进行Sanger测序,测序引物为U6 (human): CCGTAACTTGAAAGT ATTTCG,将测序正确的菌液在氨苄青霉素终浓度为100 μg/mL的LA培养基中扩大培养,并用Omega bio-tek公司的Endo-Free Plasmid Mini Kit II (#D6950) 进行质粒抽提备用,为后续HEK293T细胞转染做准备。

表1 Cas13a和Cas13b靶向ku70的gRNA序列

表2 Cas13a和Cas13b靶向lig4的gRNA序列
1.3 细胞培养与传代
将液氮保存的HEK293T在37 ℃水浴锅中快速解冻,解冻的细胞经600×、5 min离心后,使用含10% FBS (Gibco) 和1% Anti-Anti (Invitrogen) 的DMEM (Invitrogen) 生长培养基重悬细胞并进行培养,培养条件为37 ℃、5% CO2。细胞生长至90%时,用PBS清洗2遍,随后加入适量的Trypsin/EDTA (0.05%,Gibco) 消化1–2 min,待细胞完全脱落后用生长培养基终止反应,600×离心5 min后,使用生长培养基重悬细胞并接种至新的细胞培养皿中继续培养。
1.4 实时荧光定量PCR检测Cas13a、b系统对mRNA的干扰效果
当HEK293T细胞汇合度达到70%–90%时,参考Thermo Fisher Scientific公司的Lipofectamine 3000 Reagent说明书转染细胞。以24孔板为例,每孔将500 ng Cas13a、b质粒分别和500 ng 相应的U6-gRNA共转染至细胞中。转染48 h后,抽提细胞RNA,经反转录后对细胞cDNA进行实时荧光定量PCR,检测和的RNA表达水平,荧光定量PCR引物见表3。
1.5 Cas13a、b系统的转染效率
为了鉴定Cas13a、b系统的转染效率,将不同的Cas13a、b系统(包括Cas13质粒和U6-gRNA质粒) 分别同mCherry2-C1质粒共转HEK293T细胞,质粒用量均为500 ng/孔,步骤与上述保持一致。待细胞转染48 h后,使用流式细胞术 (BD accuri C6 plus) 检测红光细胞比例,通道为FL3。红光细胞比例间接反映Cas13系统的转染效率。
1.6 Western blotting检测目的基因的蛋白水平
将上述转染48 h的细胞用PBS清洗2遍后,使用碧云天的Western及IP细胞裂解液 (#P0013) 裂解细胞。将5×蛋白缓冲液加入裂解后的样品中并在100 ℃水中煮沸5–10 min。使用BCA法测定蛋白浓度便于SDS-PAGE上样。将蛋白总量为20 μg的蛋白样品上样至12%的SDS-PAGE凝胶孔中,70 V电泳30 min,90 V电泳1–2 h。使用iBlot 2 Gel Transfer Device系统 (Invitrogen) 将蛋白条带转移至PVDF膜上,5%脱脂奶粉封闭2 h后,4 ℃过夜孵育1︰2 000 KU70 (Proteintech, #10723-1- AP) 兔多克隆抗体或1︰2 000 LIG4 (Abcam,#ab193353) 兔单克隆抗体和1︰2 000 β-Actin (Cell signaling,#4970S) 兔单克隆抗体,TBST洗膜3次,TBS洗膜1次后,室温孵育山羊抗兔IgG 1–2 h,并按上述方法洗膜。将清洗好的膜浸湿并显色后置于UVP显色装置曝光拍照,随后用ImageJ 1.52a (National Institutes of Health,USA) 软件进行灰度扫描分析[16-17],将目的蛋白灰度值参照内参灰度值进行归一化处理3次。使用GraphPad Prism 8.0软件将归一化处理的数据 作图。
1.7 统计分析


表3 实时荧光定量PCR引物信息
2 结果与分析
2.1 Cas13a、b系统构建
将合成的gRNA退火后与Ⅰ酶切的线性化U6质粒进行连接。单克隆菌扩大培养后进行Sanger测序,测序结果(图1) 符合预期。
2.2 Cas13a、b系统对ku70和lig4基因mRNA水平的影响
针对和基因,我们分别为Cas13a系统设计3对gRNA,Cas13b系统设计6对gRNA。将Cas13a、b系统共转染HEK293T细胞,48 h后荧光定量PCR显示,所有Cas13a、b系统的不同gRNA对mRNA的表达水平均呈现出显著下调的现象 (<0.01),其中Cas13a-g2对的敲减效果可以达到56%,Cas13b-g5对的敲减效果高达92% (图2A)。另外,在对基因的干扰上,仅有Cas13b-g2和g5展现出显著的敲减效果,分别达到70%和76%的敲减水平 (图2B),其余如Cas13a-g3和Cas13b-g4则具有较大的RNA敲减趋势 (<0.1)。
2.3 Cas13a、b系统转染效率评估
将共转Cas13系统和红光质粒mCherry2-C1的HEK293T细胞用PBS清洗2次,经胰酶消化并离心,PBS重悬后,使用流式细胞术检测红光细胞比例。如图3所示,红色荧光细胞数约占总细胞数的65.7%–71.0%,即3对Cas13a的gRNA和6对Cas13b的gRNA转染效率无明显差异。
2.4 Cas13a、b系统对Ku70和Lig4蛋白水平的影响
将Cas13a、b系统转染HEK293T细胞,48 h后裂解并变性,用于Western blotting实验。使用ImageJ软件分析,结果显示,Cas13a-g2、g3和Cas13b-g2、g4–g6均能将Ku70蛋白表达水平下调20%以上,其中Cas13a-g3和Cas13b-g2在各自系统中具有最大的下调能力,分别能够将Ku70蛋白表达下调至74%和68% (图4A)。此外,除Cas13a-g2外其余gRNA均能使Lig4蛋白水平下调20%以上,其中Cas13a-g3和Cas13b-g4在各自系统中具有最大的下调能力,分别能够将Lig4蛋白水平下调至73%和53% (图4B)。

图1 Cas13a、b系统重构质粒测序
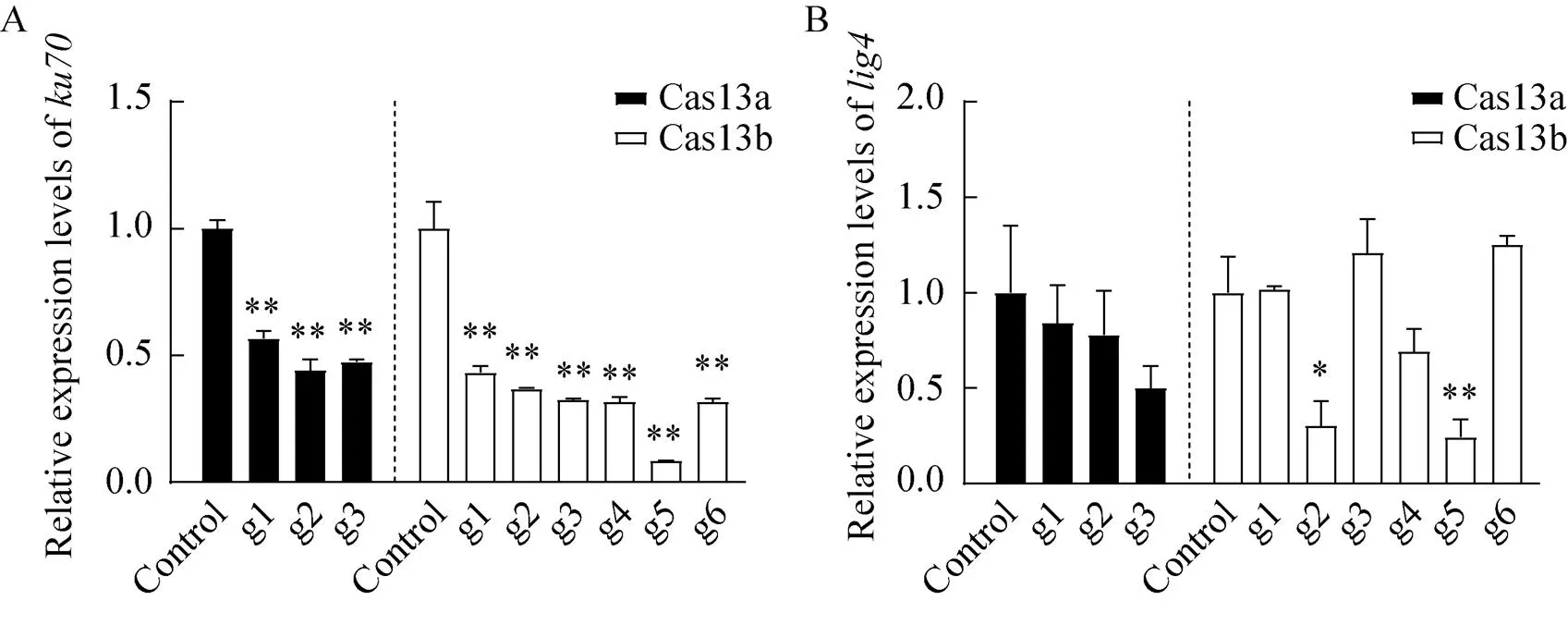
图2 Cas13a、b对ku70和lig4基因mRNA水平的影响(A: Cas13a、b系统对ku70基因mRNA水平的影响;B: Cas13a、b系统对lig4基因mRNA水平的影响)

图3 Cas13a、b系统转染效率评估(A: mCherry红光校正Cas13a、b系统的流式细胞图;B: Cas13a、b系统干扰ku70的转染效率;C: Cas13a、b系统干扰lig4的转染效率)

图4 Cas13a、b对Ku70和Lig4蛋白水平的影响(A: Cas13a、b对Ku70蛋白水平的影响;B: Cas13a、b对Lig4蛋白水平的影响)
3 讨论
CRIPSPR/Cas13系统是一种新兴的有望替代RNA干扰的RNA编辑技术[8-9]。本研究使用CRISPR/Cas13a、b系统能够有效降低和的转录和蛋白表达水平,但不同的gRNA拥有不同的敲减效果。最近研究表明,哺乳动物细胞中,靶向RNA的低二级结构能够大大提高转录本的敲减效果[8]。由于Cas13系统不具备解旋酶活性,RNA的高级结构影响gRNA靶向能力,故而在gRNA设计时需要考虑RNA的可靶向性以及周围二级结构对RNA切割能力的影响[18]。人的Ku70和Lig4蛋白分别由和基因编码,根据Ensembl数据库显示,基因存在8个转录本,其中6个能够翻译形成蛋白,基因具有5个能够翻译成蛋白的转录本,这些差异的转录本主要是mRNA的前体或由于可变剪切造成的。基于Cas13蛋白的切割原理,本研究将荧光定量PCR引物设计在靶位点两端,以便检测RNA水平的变化,初步了解敲减事件的发生。由于多转录本具有相同的靶位点,可能影响目的蛋白的下调水平,因此我们建议设计gRNA时尽可能避免可变剪切带来的影响。另外,部分gRNA使得目的基因的转录水平和蛋白水平表达不一致。原因可能如下:1) 基因的表达一般分为转录和翻译两个层面,尤其是真核生物的基因表达存在时间和位置的时空间隔。蛋白表达存在一定的滞后性,48 h可能不足以使得转录水平和翻译水平保持一致。而过久的转染时间会导致一定的细胞毒性问题。2) 受可变剪切的影响[19],可靶向RNA的种类大于1,使得靶向能力分散。另外,多转录本的存在影响基因表达调控,进而影响蛋白表达水平。3) 蛋白调控过程是复杂的,DNA修复因子如Ku70、Lig4等存在交互网络,而这个调控网络尚未完全被人类了解。4) Cas13质粒大小约13 kb,基因的干扰表达受转染效率的影响较大。5) gRNA靶向特异性影响Cas13系统对mRNA的调控效率,特异性高、脱靶效率低的gRNA更容易富集在靶位点区域,具有更快、更强地干扰基因表达的能力。另外,和敲减效果的不同除了gRNA的影响外,Cas13系统不同亚型的切割活性也是影响敲减效果的因素之一。Konermann等发现在哺乳动物细胞中Cas13d具有比Cas13a、b更强烈、更可观的敲减效果[7],这个现象可能是由于Cas13d拥有更小的蛋白分子量,能够更容易地转染或包装到细胞内,并对细胞的转录和翻译有着较低的负担所造成的。
与RNAi相比,尽管Cas13系统未能将转录水平敲减至1%以下,但其对蛋白的敲减已达到RNAi的水平[12]。另外,CRISPRi和NgAgoi也仅能敲减Ku70和Lig4约40%–60%的蛋白表达 量[14-15]。这些结果证明Cas13a、b系统成功达到我们的预期,并具备巨大的潜在价值。目前,CRISPR/Cas13a、b系统对蛋白敲减效果有限,主要在于Cas蛋白分子量巨大,转染或病毒包装以及细胞内蛋白运转困难。因此,Rauch等基于人类蛋白特性设计并合成了CIRTS系统 (CRISPR/Cas inspired RNA targeting system)[20],整个系统仅有2.7 kb,克服了Cas13系统分子量大和免疫原性的限制。此外,Cas13系统除了靶向RNA,敲减目的RNA的作用外,还可用于RNA编辑,纠正由于基因突变所导致的疾病[9,21]。它不改变基因组序列的特性让其具有可逆性,似乎在基因治疗等方面比CRISPR/Cas9系统更为安全。本研究使用CRISPR/Cas13a、b系统主要针对Ku70和Lig4蛋白,筛选出了具有显著敲减效果的gRNA。在不编辑基因组的条件下,为基因功能和调控研究提供一种可行方案。
[1] Deveau H, Garneau JE, Moineau S. CRISPR/Cas system and its role in phage-bacteria interactions. Annu Rev Microbiol, 2010, 64: 475–493.
[2] Hsu PD, Lander ES, Zhang F. Development and applications of CRISPR-Cas9 for genome engineering. Cell, 2014, 157(6): 1262–1278.
[3] O’Connell MR. Molecular mechanisms of RNA targeting by Cas13-containing type VI CRISPR-Cas systems. J Mol Biol, 2019, 431(1): 66–87.
[4] East-Seletsky A, O’Connell MR, Knight SC, et al. Two distinct RNase activities of CRISPR-C2c2 enable guide-RNA processing and RNA detection. Nature, 2016, 538(7624): 270–273.
[5] Smargon AA, Cox DBT, Pyzocha NK, et al. Cas13b is a type VI-B CRISPR-associated RNA-guided RNase differentially regulated by accessory proteins Csx27 and Csx28. Mol Cell, 2017, 65(4): 618–630.e7.
[6] Yan WX, Chong SR, Zhang HB, et al. Cas13d is a compact RNA-targeting type VI CRISPR effector positively modulated by a WYL-domain-containing accessory protein. Mol Cell, 2018, 70(2): 327–339.e5.
[7] Konermann S, Lotfy P, Brideau NJ, et al. Transcriptome engineering with RNA-targeting type VI-D CRISPR effectors. Cell, 2018, 173(3): 665–676. e14.
[8] Abudayyeh OO, Gootenberg JS, Essletzbichler P, et al. RNA targeting with CRISPR-Cas13. Nature, 2017, 550(7675): 280–284.
[9] Cox DBT, Gootenberg JS, Abudayyeh OO, et al. RNA editing with CRISPR-Cas13. Science, 2017, 358(6366): 1019–1027.
[10] Elbashir SM, Harborth J, Lendeckel W, et al. Duplexes of 21-nucleotide RNAs mediate RNA interference in cultured mammalian cells. Nature, 2001, 411(6836): 494–498.
[11] Jackson AL, Bartz SR, Schelter J, et al. Expression profiling reveals off-target gene regulation by RNAi. Nat Biotechnol, 2003, 21(6): 635–637.
[12] Li GL, Liu DW, Zhang XW, et al. Suppressing Ku70/Ku80 expression elevates homology-directed repair efficiency in primary fibroblasts. Int J Biochem Cell Biol, 2018, 99: 154–160.
[13] Quan R, Li GL, Mo JX, et al. Effects of RNA interference on the porcine NHEJ pathway repair factors on HR efficiency. Hereditas (Beijing), 2018, 40(9): 749–757 (in Chinese). 全绒, 李国玲, 莫健新, 等. RNA干扰猪NHEJ通路修复因子对HR效率的影响. 遗传, 2018, 40(9): 749–757.
[14] Ye LP, Wang CK, Hong LJ, et al. Programmable DNA repair with CRISPRa/i enhanced homology-directed repair efficiency with a single Cas9. Cell Discov, 2018, 4: 46.
[15] Li GL, Quan R, Wang HQ, et al. Inhibition of Ku70 and Ku80 by CRISPR interference, not NgAgo interference, increases the efficiency of homologous recombination in pig fetal fibroblasts. J Integr Agric, 2019, 18(2): 438–448.
[16] Carvajal-Vergara X, Sevilla A, D’Souza SL, et al. Patient-specific induced pluripotent stem-cell-derived models of LEOPARD syndrome. Nature, 2010, 465 (7299): 808–812.
[17] 用ImageJ对条带进行定量分析[EB/OL]. [2020- 01-05]. https://www.plob.org/article/14865.html.
[18] Abudayyeh OO, Gootenberg JS, Konermann S, et al. C2c2 is a single-component programmable RNA- guided RNA-targeting CRISPR effector. Science, 2016, 353(6299): aaf5573.
[19] Wang ET, Sandberg R, Luo SJ, et al. Alternative isoform regulation in human tissue transcriptomes. Nature, 2008, 456(7221): 470–476.
[20] Rauch S, He E, Srienc M, et al. Programmable RNA-guided RNA effector proteins built from human parts. Cell, 2019, 178(1): 122–134.e12.
[21] Abudayyeh OO, Gootenberg JS, Franklin B, et al. A cytosine deaminase for programmable single-base RNA editing. Science, 2019, 365(6451): 382–386.
Knockdown the expression ofandin HEK293T cells by CRISPR/Cas13 system
Haoqiang Wang1, Guoling Li1, Guangyan Huang1, Zicong Li1, Enqin Zheng1, Zheng Xu1, Huaqiang Yang1, 2, Zhenfang Wu1, 2, Xianwei Zhang1, 2, and Dewu Liu1
1 National Engineering Research Center for Breeding Swine Industry, College of Animal Science, South China Agricultural University, Guangzhou 510642, Guangdong, China 2 Wens Foodstuff Group Co., Ltd, Yunfu 527400,Guangdong,China
Clustered regularly interspaced short palindromic repeats (CRISPR)/CRISPR-associated proteins (Cas) system is a hotspot of gene editing and gene expression research, in which CRISPR/Cas13 system provides a new direction for RNA interference and editing. In this study, we designed and synthesized the corresponding gRNAs of CRISPR/Cas13a and CRISPR/Cas13b systems in non-homologous end joining (NHEJ) pathway, such as Ku70 and Lig4, and then detected the expression ofandin HEK293T cells. The CRISPR/Cas13a system could efficiently knockdown the mRNA expression ofandmore than 50%, and CRISPR/Cas13b system also suppressedandabout 92% and 76%, respectively. Also, CRISPR/Cas13a, b systems could down-regulate Ku70 and Lig4 proteins level to 68% and 53%, respectively. The study demonstrates that the CRISPR/Cas13 system could effectively knockdown the expression of RNA and protein in HEK293T cells, providing a new strategy for gene function and regulation research.
CRISPR/Cas13,,, gene knockdown
10.13345/j.cjb.190494
November 1, 2019;
February 19, 2020
Supported by: National Transgenic Major Projects (No. 2016ZX08006002), Project of R & D Plan in Key Areas of Guangdong, China (No. 2018B020203002).
Dewu Liu. Tel: +86-20-85284816; E-mail: dwliu@scau.edu.cn
Xianwei Zhang. Tel: +86-766-2986345; E-mail: zxianw@163.com
国家转基因重大专项 (No. 2016ZX08006002),广东省科技创新战略专项 (No. 2018B020203002) 资助。
王豪强, 李国玲, 黄广燕, 等. CRISPR/Cas13系统敲减HEK293T细胞和基因表达. 生物工程学报, 2020, 36(7): 1414–1421.
WangHQ, LiGL, HuangGY, et al. Knockdown the expression ofandin HEK293T cells by CRISPR/Cas13 system. Chin J Biotech, 2020, 36(7): 1414–1421.
(本文责编 郝丽芳)
