鱼类遗传连锁图谱构建及QTL定位的研究进展
2020-07-28陈军平胡玉洁李学军
陈军平,胡玉洁,王 磊,田 雪,李学军
( 河南师范大学 水产学院,河南省水产动物养殖工程技术研究中心,河南 新乡 453007 )
随着水产养殖业的快速发展,其已成为我国农业的重要组成部分,全国鱼类养殖产量呈不断上升态势。但是,近些年随着鱼类野生资源的减少和长期人工繁殖,养殖品种出现了种质资源退化、生长速度慢、饲料转化率低、抗逆性差等现象[1]。针对这些问题,通过遗传育种手段,选育出优良品种,提高品质是解决此问题的重要手段之一[2]。随着分子生物学和基因组学的快速发展,构建高密度的遗传连锁图谱,有效利用与数量性状相关的分子标记,并结合标记辅助育种从而选育出生长速度快、品质优、抗逆性好的新品种可以有效地促进鱼类养殖产业的健康可持续发展。
由于传统选育方法在选择生长、抗病、耐盐碱等经济性状时较为缓慢或不稳定,因此需要借助分子标记的方法进行辅助选择。其中,遗传图谱是伴随着分子标记的发展而演化过来的,它主要就是通过遗传重组或交换从而定位不同标记或者基因在图谱上的相对位置。数量性状位点(QTL)定位是将数量性状位点定位在连锁图谱的某一位点,从而能够在一定程度上辅助育种研究。高密度和高分辨率的遗传连锁图谱为鱼类重要经济性状的QTL精细定位和基因克隆提供了理论依据。鉴于连锁图谱在基因组和遗传研究中具有重要作用,笔者拟通过对鱼类遗传图谱构建和QTL定位研究进行综述,从而为后续的相关研究提供参考和依据。
1 遗传连锁图谱构建
遗传连锁图谱是基因标记的有序列表,连锁图谱的构建主要包括4个组成部分:多态性标记、合适的作图家系群体、对作图家系进行基因分型和用于构建图谱的软件及分析。
1.1 DNA分子标记
以DNA分子标记为基础构建遗传连锁图谱是实现数量性状基因定位和分子标记辅助育种的重要基础,与传统的遗传标记相比,DNA分子标记具有标记位点多、多态性高、标记数量多、重复性好、不受环境影响、不受组织类别和发育阶段等影响。目前,用于水产动物遗传图谱构建的分子标记主要有限制性片段长度多态性(RFLP)、随机扩增多态性DNA (RAPD)、扩增片段长度多态性(AFLP)、简单重复序列(SSR)和单核苷酸多态性(SNP)等。RFLP是发展和应用最早的DNA分子标记技术,它通过基因组酶切位点的缺失、重排或突变使RFLP谱带表现出不同程度的多态性,可重复性好,但RFLP对DNA需求量较大,其分析一次只能检测一个酶切位点、操作过于复杂、提供的信息较少、成本较高[3]。RAPD技术是以PCR技术为基础的一种DNA多态性标记,相对于RFLP来说,RAPD操作简单,检测位点多,要求DNA量少,成本较低,但其稳定性不高且呈显性,不能区分纯合子和杂合子,会丢失一些相关的遗传信息[4]。AFLP同时具备RFLP和RAPD的优点,具有检测位点多,分布均匀,覆盖度高等特点,在早期这种分子标记被广泛用于遗传图谱的构建。但它也是显性标记,在一定程度上会降低标记的遗传信息量,因此一些研究者认为AFLP标记并非构建高密度遗传连锁图谱的理想分子标记[5]。目前,SSR标记和SNP标记这两种类型的DNA标记在遗传图谱中应用最为广泛。SSR标记是近些年发展起来的第二代分子标记,其呈共显性、稳定性强、信息含量比较丰富,而且这种标记也可用于不同群体和作图家系遗传图谱的整合。2000年,孙效文等[6]利用RAPD技术与SSR技术建立了鲤鱼(Cyprinuscarpio)的遗传连锁图谱; Liu等[7]利用905个SSR标记构建了鳙鱼(Aristichthysnobilis)的高密度遗传连锁图谱。SNP标记主要是指在基因组水平上由单个核苷酸的变异所引起的DNA序列多态性,它具有密度高、稳定性强、呈共显性等特点,是一种比较理想的作图标记。彭文竹等[8]基于鲤鱼250 K SNP位点分型芯片及所测得的黄河鲤(C.carpiohaematoperus)家系 106 个个体的位点分型数据,利用拟测交原理,构建了雌雄两个分别包含 50 个连锁群的高密度连锁图谱;Wan等[9]利用SNP标记构建了团头鲂(Megalobramaamblycephala)的高密度遗传连锁图谱。
1.2 连锁图谱的作图群体
遗传连锁图谱构建过程中对亲本和作图群体的选择尤其重要,主要条件包括:(1)应保证亲本之间遗传背景差异大,即亲本之间多态性高,以便在后期提供大量的遗传信息;(2)尽量选择亲缘关系较远的个体进行杂交而获得子代。所以,在遗传图谱构建过程中,常用的作图群体包括重组自交系(RIL)、双单倍体(DH)、F2群体和回交(BC)群体。在鱼类中构建重组自交系群体是一个巨大的挑战,通常需要5~6年的时间来构建符合要求的群体,侯宁等[10]以大头鲤(C.pellegrini)、荷包红鲤(C.carpiovar.wuyuanensis)抗寒品系的重组自交系群体为材料构建了遗传图谱,并成功定位了与体长性状相关的主效QTL区间;刘奕等[11]以165个双单倍体褐牙鲆(Paralichthysolivaceus)群体作为试验材料构建了遗传图谱;由于F2群体可以产生较多的分离位点,具有亲本之间等位基因所有的遗传信息,所以该群体常用于多种水产动物构建高密度遗传图谱,如黄河鲤[12]、尼罗罗非鱼(Oreochromisniloticus)[13]、日本花鲈(Lateolabraxjaponicus)[14]等。高国强等[15]以荷包红鲤抗寒品系(♂)和云南大头鲤(♀)为祖父母本培育了110个F2个体作为作图群体构建了鲤鱼的遗传连锁图谱;王宣朋[16]以饲料转化率有明显差异的镜鲤(C.carpiovar.specularis)选育F2代作为作图群体构建了遗传连锁图谱。另外,在构建遗传图谱过程中,作图群体的大小对遗传图谱的精度有着重大影响,作图群体越大,图谱分辨率和精度越高;反之,则越低。另外,作图群体特点不一,研究者们可以根据研究需求选择不同的群体来开展研究。在试验过程中多使用几个参考群体,可以定位更多的标记,这样标记可以在至少一个作图群体中提供信息[17]。
1.3 鱼类遗传图谱研究进展
遗传连锁图谱是基因组学和遗传学研究的重要工具,也是开展选育工作的重要手段之一。虽然水产动物的相关研究相对于陆生动植物起步较晚,但发展迅速。目前,国内外水产科技人员已在多种鱼类中成功构建了遗传连锁图谱,如草鱼(Ctenopharyngodonidella)[18]、鲢鱼(Hypophthalmichthysmolitrix)[19]、罗非鱼[20]、黄河鲤、褐石斑鱼(Epinephelusbruneus)[22]、点带石斑鱼(E.coioides)[23]、虹鳟(Oncorhynchusmykiss)[24]、太平洋蓝鳍金枪鱼(Thunnusorientalis)[25]、褐牙鲆[26]、黄颡鱼(Pelteobagrusfulvidraco)[27]、南方鲇(Silurusmeridionalis)[28]、大黄鱼(Pesudosciaenacrocea)[29]、鲫鱼(Carassiusauratus)[30]、大菱鲆(Scophthalmusmaximus)[31]、大口黑鲈(Micropterussalmoides)[32]、乌苏拟鲿(Pseudobagrusussuriensis)[33]、斑点叉尾(Ictaluruspunctatus)[34]等。
1.3.1 褐牙鲆
2011年,宋文涛等[35]基于微卫星标记构建了褐牙鲆遗传连锁图谱,这也是我国首次利用微卫星标记构建褐牙鲆遗传连锁图谱,图谱总长为1320.4 cM,由24个连锁群组成; 2013年,刘奕[36]首次基于褐牙鲆双单倍体群体并利用446个微卫星标记、128个EST-SSR标记构建了褐牙鲆遗传连锁图谱,图谱总长为1214.7 cM,标记之间的平均间隔为2.2 cM,图谱覆盖率为94%。2015年,Shao等[37]首次利用Lep-MAP构建了基于RAD测序的褐牙鲆高分辨率遗传图谱,该遗传图谱全长3497.29 cM,标记的平均间隔为0.47 cM,共分布于24个连锁群,其中有12 712个SNPs并对应7430个有效位点,这也是目前国内所报道的关于褐牙鲆分辨率最高的遗传图谱。
1.3.2 虹鳟
虹鳟属鲑形目,鲑科,太平洋鲑属,是最早开展基因组研究的鱼类之一。
2015年,Palti等[38]基于虹鳟F2和回交群体分别构建了遗传图谱,在对F2群体构建图谱时共利用5612个SNP标记和267个微卫星标记相结合进行了连锁分析,最终有5851个标记定位到29个染色体上,图谱总长为3588 cM,平均标记间隔为3.2 cM;Liu等[39]基于两个半同胞家系构建了遗传图谱,共有683个SNP标记用于构建遗传图谱,图谱总长为5483 cM,标记平均间隔为8.6 cM;共有438个SNP标记用于构建雄性遗传图谱,图谱总长为2964 cM,标记平均间隔为6.8 cM。2016年,Gonzalez-pena等[40]利用SNP芯片构建了虹鳟全同胞家系遗传图谱,共有47 839个SNP标记分布于29个连锁群,每个连锁群平均有1650个SNP标记,雌性图谱总长为4248 cM,雄性图谱总长为2214 cM。
1.3.3 鲤鱼
2000年,孙效文等[6]采用56个RAPD标记、26个鲤鱼的SSLP标记、19个鲫鱼的SSLP标记、70个斑马鱼的SSLP标记、91个鲤鱼基因标记分析了黑龙江鲤(C.carpiohaematopterus)和柏氏鲤(C.pellegrini)杂交得到的46个F2个体的基因型并构建了鲤鱼的第一代遗传图谱,这也是国内首次报道关于鲤鱼的遗传图谱; 2016年,Peng等[21]基于鲤鱼250 K高通量SNP基因分型构建了超高密度、高分辨率遗传图谱,其中共筛选出28 194个SNP标记分别坐落于14 146个不同的位置上,遗传图谱总长度为10 595.94 cM,标记位置的平均间距为0.75 cM,标记的平均间距为0.38 cM,这张图谱也是迄今为止鲤鱼中密度最大的一张连锁图谱。2018年,Wang等[12]以2b-RAD技术基于鲤鱼F2家系构建高密度遗传连锁图谱,将6230个SNPs和65个微卫星标记定位到50个连锁群中,覆盖黄河鲤基因组的98.2%。
1.3.4 其他重要鱼类
除了以上几种鱼类外,还有很多鱼类基因组作图研究工作发展迅速。
Tsai等[41]利用96 396个SNP标记构建了大西洋鲑(Salmosalar)高密度遗传连锁图谱,其中,雄性和雌性图谱中平均每0.05 cM和0.07 cM就有一个SNP标记,这也是鲑鱼中密度最高的遗传图谱;Uchino等[42]利用470个微卫星标记对太平洋蓝鳍金枪鱼构建了遗传连锁图谱,该图谱包含24个连锁群,其中雄性图谱为992.9 cM,雌性图谱为1332.3 cM,该图谱覆盖了其基因组的86.5%;Nunes等[43]使用7192个SNP标记构建了大盖巨脂鲤(Piaractusbrachypomus)遗传连锁图谱,这也是第一次对大盖巨脂鲤进行遗传图谱的构建,图谱全长为2811 cM,包含27个连锁群,平均标记间隔为0.39 cM,在与斑马鱼的基因组比较分析后发现,大多数连锁群与斑马鱼的染色体同源,此外,还对大量标记进行了注释,并鉴定了一些潜在的性状功能基因;Pang等[44]利用8460个SNP标记构建了鲫鱼的高密度遗传连锁图谱,图谱全长为4047 cM,标记平均间隔为0.478 cM,该图谱覆盖了鲫鱼基因组的98.76%;Wang等[45]基于F1群体并利用2974个SNP标记构建了乌鳢(Channaargue)高密度性别平均图谱;2017年,Lin等[46]对杂交罗非鱼F2群体构建了遗传连锁图谱,该图谱有151个SSR标记和633个SNP标记,标记平均间隔为2.73 cM,图谱全长2076.7 cM,共由22个连锁群组成。
1.4 简化基因组技术在鱼类基因组学中的应用
随着高通量测序技术及鱼类基因组学的快速发展,限制性酶切位点相关 DNA 测序(RAD-seq)[47]、特异性位点扩增片段测序(SLAF-seq)[48]和基于测序的基因分型(GBS)[49]等是近些年发展较快的简化基因组测序技术,因其通量高、操作简单、成本低且不受参考基因组的限制被广泛用于水生生物分子标记的开发、高密度遗传图谱的构建、QTL定位及群体遗传学分析等领域。其中,2b-RAD[50]和dd-RAD[51]是在传统RAD-seq技术的基础上发展起来的,在一定程度上改善了传统RAD-seq技术的缺陷,简化了建库流程,节约了成本,分型准确性也得到了提高(表1)。

表1 不同简化基因组测序技术特点对比
目前,广泛用于遗传连锁图谱的软件有HighMap[52]、Joinmap 4.0[53]及IciMapping[54]等。其中,HighMap通过采用多次排序和纠错策略,提高了测序数据的使用效率及运算速度,减少了人工操作时间,而且能够提供更准确的标记排序及高质量图谱;Joinmap 4.0凭借Windows系统可视化操作界面可分析大量的作图群体,使用简单,作图速度快,可同时在多个LOD值下进行多个连锁群的构建,内置标记数据处理等功能;IciMapping是一款集遗传连锁图谱构建与QTL定位于一体的软件,该软件运行速度快,可用于双亲衍生群体的QTL与环境的互作分析,经过不断更新,软件的运行效率、稳定性及遗传连锁图谱的质量得到较大地改善,但IciMapping与其他软件相比适用的群体类型较少。在构建遗传连锁图谱的过程中,选择合适的作图软件也尤为重要,以确保图谱的准确性。
2 鱼类QTL定位研究进展
QTL是控制数量性状的基因在基因组中的位置,它以遗传连锁图谱为基础,检测作图群体中基因型和数量性状的数值,通过连锁分析确定QTL在遗传连锁图谱上的相对位置[55]。由于鱼类的生长速度、肉质、抗病、饲料转化率等重要经济性状受许多基因、环境因素相互作用的影响,所以,这就需要通过QTL定位来准确估计每个基因位点对性状的贡献率,从而减少环境方差的影响,提高选育的精确度,加快选育进程。高质量的遗传连锁图为QTL的定位提供了框架,并促进了许多水产养殖品种的遗传辅助选择和育种。如褐牙鲆的淋巴囊肿病抗性性状已成功定位并应用于标记辅助育种,罗非鱼[56]、褐牙鲆[57]和半滑舌鳎(Cynoglossussemilaevis)[58]等通过QTL定位已将性别决定位点定位于遗传图谱中,这些遗传连锁图谱已被广泛用于重要性状的QTL定位,并为这些性状的定位克隆打下了基础。
2.1 鱼类QTL定位的主要性状
目前,国内外已对多种鱼类的重要经济性状进行了QTL定位,尤其是以罗非鱼、褐牙鲆、鲤鱼及其他重要养殖鱼类为主,这些鱼类的数量性状已成功定位在图谱中(表2)。
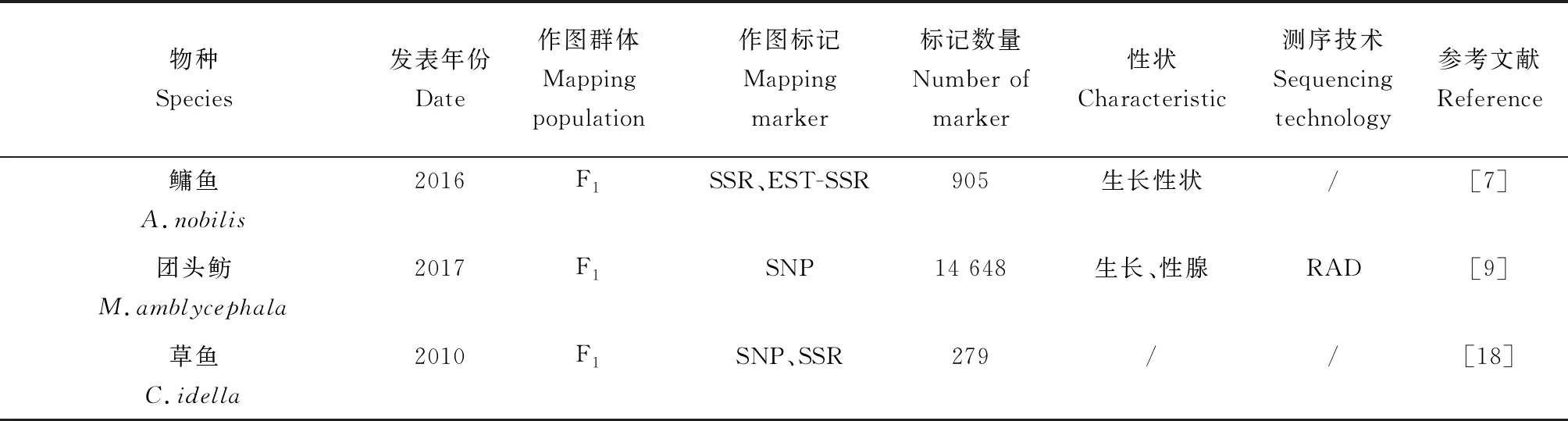
表2 重要经济鱼类遗传连锁图谱构建及QTL定位情况

(续表2)
2.1.1 罗非鱼
罗非鱼作为开展QTL定位研究较多的物种之一,关于其研究的性状主要集中在生长速度、抗病、耐盐碱、性别决定和耐低温等方面。最早报道关于罗非鱼抗寒性状相关的QTL已成功定位到23号连锁群上[59]。与性别决定性状相关的几个QTL位点也成功定位在不同的连锁群上。2015年,Palaiokostas等[56]利用dd-RAD测序法构建了遗传连锁图谱,在LG1区域内定位两个与性别决定相关的QTL位点,在LG20区域内发现一个与性别决定相关的新位点; 2017年,Wessels等[61]基于dd-RAD测序法揭示了在LG23中,温度变化是性反转的决定因素;2018年,Gu等[62]首次采用QTL-seq和连锁分析方法鉴定出两个与耐盐性状相关的全基因组显著水平的QTL,分别位于chrLG4 和chrLG18,其中,对chrLG18进行了精细定位,最终在chrLG18的23 Mb处发现一个主效QTL,可解释表型变异高达79%,该主效QTL区域内发现至少有10个基因随高盐胁迫有显著反应;Lin等[46]对杂交罗非鱼F2群体的脂肪酸ω-3进行了定位,与ω-3相关QTL位点分别定位在LG11、LG18、LG19和LG20上,可解释表型变异为5%~7.5%,表明ω-3脂肪酸含量是由多个基因决定的,这是首次对罗非鱼的总脂肪和ω-3进行QTL定位。
2.1.2 褐牙鲆
目前,对褐牙鲆QTL定位的研究主要集中在抗病和生长性状方面,其他性状方面研究较少。在生长方面,2015年,Cui等[63]采用有丝分裂雌核发育方法构建160个双单倍体个体,最终获得42个主效QTL,在第6和第9染色体之间存在一个相互作用的QTL,可解释表型变异达到25.19%,其中第9染色体上的QTL同时控制着7个生长性状;2017年,李宁等[57]在偏分离条件下对褐牙鲆生长性状进行了主成分定位,在4个图谱中检测到3个加性QTL,分别位于6号、9号和22号连锁群上。在抗病方面,2014年,Wang等[64]定位了关于抗鳗弧菌病QTL位点,通过基因组扫描鉴定出4个多态性SSR标记,其中位于LG7中的两个标记在感病和抗病群体之间有显著差异;2016年,Wang等[65]对褐牙鲆迟钝爱德华氏菌(Edwardsiellatarda)抗病性进行了QTL定位,利用两种不同的分析模型发现6个与抗病性相关的QTL,其中有2个QTL是主效QTL,可解释表型变异为16.0%~89.5%;Shao等[37]采用RAD-seq技术定位9个与抗鳗弧菌(Vibrioanguillarum)病相关的QTL并定位了4个与抗鳗弧菌病相关的基因;Fuji等[66]对淋巴囊肿病抗病位点进行了定位,发现主效QTL附近的微卫星标记Poli-8TUF与抗病性QTL紧密连锁,该标记的一个等位基因与LD抗病性呈显著相关,后来利用含有该等位基因的雌性个体作为亲本选育了一个新群体,养殖试验结果表明,该抗病基因由母本完全遗传给子代,所有的子代均对LD具有抗性,大大提高了子代的成活率,而另外两个对照组试验由于不具有该等位基因,子代的成活率仅为4.5%和6.1%。
2.1.3 鲤鱼
目前关于鲤鱼QTL定位的研究主要集中在生长和形态特征、饲料转化率(FCR)、生理生化和性别决定等方面。关于鲤鱼的遗传图谱构建和QTL定位也取得了较快发展。2011年,肖同乾[67]利用415个微卫星标记筛选与鳞被相关标记,最终获得7个与鳞被极显著相关的微卫星标记并定位到LG3上;2013年,Laghari等[68]选用109个SSR标记、31个EST-SSR标记和167个SNP标记得到了影响体质量和生长速率的7个QTL; 2016年,Peng等[21]选用28 194个SNPs标记检测到22个与生长相关的QTL和7个与性别二态性相关的QTL;2018年,Chen等[69]对黄河鲤头型性状进行了QTL定位和全基因组关联分析(GWAS),最终定位到12个与头型显著相关的位点;Feng等[70]对F2群体的生长性状进行了定位,得到了21个与生长相关的QTL,可解释表型变异介于16.3%~38.6%。
在大多数水产养殖品种中,饲料约占总生产成本的70%。因此,饲料转化率的大小对水产养殖品种和行业的发展都极其重要,由于饲料摄取量不平衡,每个个体的饲料摄入量难以在水产养殖种类中测量。2012年,Wang等[71]基于镜鲤F2群体利用560个多态性标记检测到15个与饲料转化率相关的QTL区间,并分布在9个连锁群上,其中可解释表型变异最大的QTL为52.20%。
在生理生化方面,2012年,于彬彬等[72]对德国镜鲤F1群体DNA进行基因组检测,最终检测到9个与酸性磷酸酶相关的QTL,最大的可解释表型变异为13.8%;徐玉兰[73]对镜鲤F1代群体的肝脏、前肠和后肠3个不同组织中的超氧化物歧化酶活量进行了QTL定位,在3个不同组织中分别发现7个、5个和6个影响超氧化物歧化酶性状的显著性QTL区间。
2.1.4 其他重要养殖鱼类
Boison等[74]分别对3个大西洋鲑种群的心肌病综合征(CMS)抗病性进行了定位,鉴定了2个与心肌病综合征抗性高度相关的QTL,分别位于第12和第17连锁群上,可解释表型变异为7.6%~31.7%,其中有4个影响病毒抗性的功能基因与鉴定到的QTL密切相关,通过试验发现,大西洋鲑对心肌病综合征的抗性是遗传的,可以通过选择育种群体以提高心肌病综合征抗性,减少疾病的暴发。Pedersen等[75]在之前的研究基础上,通过欧洲和北美大西洋鲑第一代和第二代回交群体证实了色素沉着和生长性状是由基因组的多个区域决定。Wang等[76]首次利用日本花鲈对虹彩病毒(SGIV)抗病性进行了定位,最终定位了1个全基因组显著水平的QTL和3个可能性QTL,可解释表型编辑为7.5%~15.6%,表明虹彩病毒抗病性是由多个基因或因素控制的;另外还在21号染色上检测到一个候选基因MECOM,发现MECOM对虹彩病毒感染的反应有差异表达,这也为后续了解虹彩病毒抗病性提供了重要信息。Sawayama等[77]对真鲷虹彩病毒(RSIVD)抗病性进行了定位,最终在一尾雄鱼中检测到一个与真鲷虹彩病毒抗病性相关的主效QTL,利用这尾雄鱼繁殖了两个家系(Fam-2014和Fam-2015)并进行了两次养殖试验,经过两次养殖试验均发现一个QTL与真鲷虹彩病毒抗病性显著相关,其中在Fam-2014家系中,该位点可解释表型变异高达31.1%。Pang等[44]首次对鲫鱼饲料转化率进行了定位,其可解释表型变异介于15.2%~20.9%,这也是世界上第一次对鲫鱼饲料转化率进行定位。最终从5个QTL区域中筛选出7个潜在的候选基因,并将其定位在鲫鱼的LG25和LG49上,在这些基因中,有3个候选基因cse1l、mapk11和myh14与GTPase或ATP相关,分别参与了能量代谢。Sun等[78]利用鳜鱼全同胞家系进行了生长性状和性别决定的QTL定位,在LG23(标记r1_73194附近)上发现一个与性别决定显著相关的QTL,可解释表型变异为53.3%;在LG4上检测到了11个与生长性状显著相关的QTL,可解释表型变异为12.4%~17.2%。Zhang等[79]对布氏鲳鲹生长形状进行了QTL定位,检测23个与生长性状相关显著性位点,且这些位点均位于LG15上,可解释表型变异为16.6%~21.3%。Liu等[7]对鳙鱼用于生长的性状进行了定位,发现一个位于LG9全基因组水平的位点,其中TP53BP2基因与鳙鱼生长性状显著相关。
2.2 QTL作图方法
一方面,随着研究者对数量性状定位研究的深入,虽然对很多鱼类进行了QTL定位,但只有少数鱼类数量性状在不同的遗传背景中得到了证实和精细定位。另一方面,由于受到QTL检测技术的限制和大量低密度遗传图谱的出现,无法有效检测出一些重要性状的位点。目前在鱼类QTL定位检测中常用的方法分别有单标记回归分析法、区间作图法、复合区间作图法[80]。单标记分析是检测与单个标记相关的QTL的最简单方法[81]。王宣朋[16]对与饲料转化率性状相关的标记进行了单标记回归分析。单标记分析的主要优点在于它不需要参照组或完整的连锁图谱,并且可以应用于在相同条件下培育的任何群体,当标记数量较少时,是一种非常有效的方法。然而,该方法的缺点主要是是其检测QTL的能力低,仅允许与要鉴定的标记紧密相关的QTL,并不能识别是同一个还是多个QTL连锁。区间作图是基于回归模型和最大似然分析原理分析QTL的,其需使用大量DNA标记并均匀覆盖整个基因组,并分析每个连锁群相邻标记所处的位置及遗传效应。尹森[82]利用MAPOTL 6.0对与镜鲤肝组织中碱性磷酸酶相关的标记进行区间作图分析。Lin等[13]采用区间作图法对雄、雌罗非鱼的体质量进行了QTL分析。该方法与单标记分析法相比增加了QTL定位和检测能力[83]。但是,该方法每次检测只用到两个标记并没有充分利用其他标记所提供的信息,导致效率低下。在复合区间作图中,区间作图与线性回归相结合,除了用于区间作图的相邻连锁标记之外,还包括统计模型中的其他遗传标记。与单标记回归分析和区间作图相比,复合区间作图的主要优势是其在定位QTL方面具有更高的精度和检测能力[84]。但该方法也有一定的缺陷,并不能分析与环境互相作用等复杂的遗传学问题,还需要在大量分子标记中筛选一些有效的标记。在水产养殖物种中,因为复合区间作图具有更好的定位和检测能力,经常被用于重要性状的QTL定位。目前,研究者正在开发更多新的QTL定位方法以提高QTL定位的准确性。
3 展 望
随着生物技术和分子标记技术的不断发展,鱼类遗传图谱的构建也迅速发展,许多鱼类都建立了遗传连锁图谱,甚至对一些鱼类建立了超高密度和分辨率的遗传图谱,并对一些重要经济性状进行了QTL定位,这对比较基因组学和分子标记辅助育种的研究提供了较好理论基础。但是,鱼类很多重要经济性状都是通过多基因控制的,它们遗传背景及发生过程都比较复杂,导致分子育种过程较为缓慢。主要原因在于:(1)不是所有的群体都适合构建遗传图谱,一些作图群体也会受到分子标记的限制,使用的标记较少,导致遗传图谱质量不高且覆盖率低;(2)遗传图谱的构建和QTL定位的分析软件发展得还不够完善;(3)对一些鱼类重要经济性状的定位研究还不够深入,无法实现QTL的精准定位[85]。针对以上问题,建议在未来研究过程中增加样本量,开发更多的分子标记,选择合适的作图群体并结合相应的分子标记以构建高密度连锁图谱,加强鱼类重要经济性状的鉴定能力。近些年,以RAD-seq技术为代表的简化基因组技术发展迅猛,由于RAD-seq能开发出更多的分子标记且密度更高,所以,该技术应用更为广泛。另外,RAD-seq技术在简化基因组构建过程中能够克服参考基因组质量不高的问题[86]。目前,该技术已广泛应用于遗传图谱的构建、群体遗传学研究和性状关联研究等并取得了较大的突破。随着简化基因组技术应用愈发广泛及对基因组学研究的不断深入,人们可以在此基础上对鱼类构建更多超高密度、高覆盖率的遗传图谱,定位更多与重要经济性状相关的QTL,加快水产养殖业的健康、可持续发展。
