野生和养殖的异齿裂腹鱼肠道菌群结构分析
2020-07-28王金林王且鲁王万良曾本和潘瑛子周建设
王金林,王且鲁,王万良,曾本和,潘瑛子,周建设
( 西藏自治区农牧科学院 水产科学研究所,西藏 拉萨 850002 )
鱼类肠道是消化吸收的重要器官,也是鱼类与外界环境接触面积最大的器官,而肠道菌群是肠道的重要组成部分,且对动物的免疫调节、代谢吸收也有重要影响[1-3]。脊椎动物肠道菌群以厚壁菌门、拟杆菌门、变形菌门、梭杆菌门为主[4]。肠道菌群通过长期自然选择,与宿主间形成了一个相对稳定的微生态系统,从而增强宿主对有害环境的自我调节能力。目前已有研究表明,鱼类肠道菌群的定殖主要与养殖水环境、饲喂饵料及鱼卵表面中的细菌有关,其中养殖水环境和饲喂的饵料为主要因素[5]。肠道菌群分泌的酶可以帮助宿主分解食物增强营养的消化吸收,其分泌的抗生素类物质则可阻止病原微生物的侵入。通过对肠道菌群结构特点的研究,可为减少鱼类病害的发生提供一定的理论参考数据。
传统肠道菌群结构的分析研究方法包括传统分离培养法、变性梯度凝胶电泳(DGGE)、随机扩增引物多态性分析(RAPD)[6],这些方法的局限性在于无法鉴定丰度极小的微生物,从而导致研究结果与样品中真正的菌群结构出现较大偏差。近年来,MiSeq高通量测序技术应运而生,该技术基于细菌16S rRNA基因在功能上的高度保守型及对应序列不同位点的高变性,在保守区设计通用引物PCR扩增然后对高变区进行测序分析和菌种鉴定[7-8]。这种方法克服了传统方法的局限性,对生物物种之间的亲缘关系和差异有了较为准确的分析。该测序技术可同时定量分析多个样本中的群落结构,从而真实全面地反映微生物群落结构的基本特征[9]。
异齿裂腹鱼(Schizothoraxo′connori)隶属鲤形目、鲤科、裂腹鱼亚科、裂腹鱼属,别名异齿弓鱼。其腹部肛门附近具有特化形成裂隙的成排鳞片,即“裂腹”由来[10]。异齿裂腹鱼分布于雅鲁藏布江上、中游的干支流及附属水体,营底栖生活,在干支流水质清澈、砾石底质的河道处活动,为产区主要经济鱼类之一。异齿裂腹鱼生长缓慢、性成熟晚且繁殖力低,因此对过度捕捞和环境破坏较为敏感[10]。笔者以野生和人工养殖的异齿裂腹鱼为试验对象,研究两种异齿裂腹鱼肠道菌群结构的异同,以期为减少异齿裂腹鱼养殖过程中病害的发生提供一定的理论参考依据。
1 材料与方法
1.1 样品来源
试验用异齿裂腹鱼野生组采自雅鲁藏布江日喀则江段,体质量(1.00±0.13) kg。人工养殖组为水泥池驯化养殖3年的异齿裂腹鱼,体质量(1.00±0.20) kg。两组鱼体质健壮、体表完好,可用于试验。
1.2 样品采集方法
野生组和人工养殖组各取5尾异齿裂腹鱼,于无菌环境下,用解剖剪剪开异齿裂腹鱼体腔,取出肠道,用无菌的生理盐水冲洗肠道外壁及内容物。样品采集后进行分类编号保存,寄至上海美吉生物公司进行DNA提取和MiSeq高通量测序分析。两组样品DNA提取完成后,分别将两组DNA样品进行混合,然后分类编号,其中野生组异齿裂腹鱼为Y1、Y2、Y3,人工养殖组异齿裂腹鱼为C1、C2、C3。
1.3 数据分析
数据的统计分析以每个样品中的运算分类单元作为分类和计算的依据。根据不同的相似度水平,对所有的序列进行运算分类单元划分,在97%的相似水平下的运算分类单元进行生物信息统计分析。16S细菌和古菌核糖体数据库使用Silva数据库(Release128 http:∥www.arb-silva.de)。设置差异显著水平为0.05,当P<0.05时表示差异显著,P≥0.05表示差异不显著。
2 结 果
2.1 样品序列数目
人工养殖组C1、C2、C3和野生组Y1、Y2、Y3测得的序列条数分别为47 068、46 869、40 417、43 194、39 942和46 791(表1),序列平均长度约为435 bp,符合高通量测序要求。
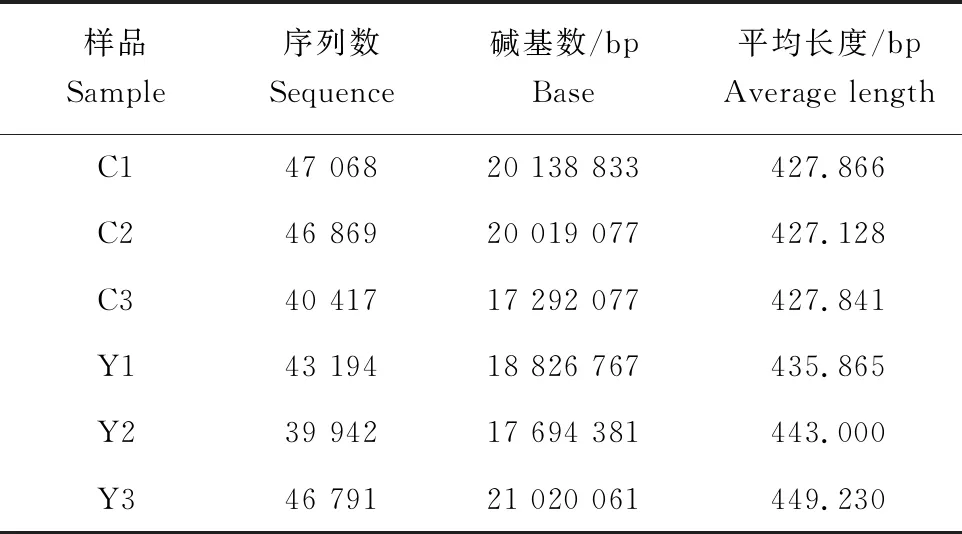
表1 样品序列数统计表
2.2 菌群多样性分析
2.2.1 稀释性曲线
以97%相似性水平为标准划分可操作分类单元运算分类单元,其中人工养殖组C1、 C2、C3和野生组Y1、Y2、Y3样品运算分类单元数据分别可划分为302、259、263、751、469、313。基于随机选取一定数量的测序序列及其对应的运算分类单元种类得到的稀释性曲线,当序列数达到10 000~40 000时曲线趋于平坦(图1),通过对样品数据进行抽平分析,6组样品的取样条数为44 046,表明C1、C2、C3和Y1、Y2、Y3 6组样品数据可靠,测序数量接近饱和,能够真实有效地反映样品中优势细菌的数量关系。
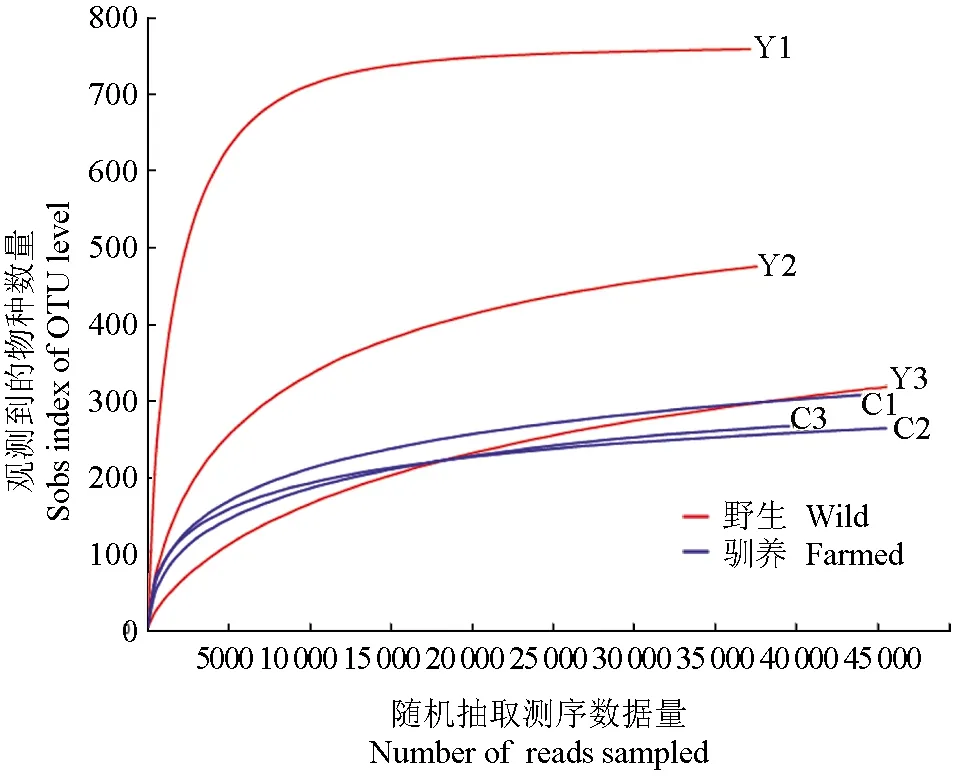
图1 稀释性曲线
2.2.2 菌群结构多样性指数分析
通过测序序列计算出6个异齿裂腹鱼中细菌多样性指数(表2),6组样品的覆盖率均超99%,表明数据测序完整;其中Y1样品的香农指数最大为5.46,辛普森指数最小为0.01,说明在野生组异齿裂腹鱼Y1样品鱼中肠道的细菌多样性最高。对野生组和人工养殖组组间多样性指数差异进行t检验(表3),得到丰富度指数Chao(用来估计样品中所含运算分类单元数目)和Ace(用来估计群落中运算分类单元数目),多样性指数香农指数(用来估算样品中微生物多样性)和辛普森指数(用来定量描述一个区域的生物多样性)的P>0.05,即野生和人工养殖两种模式下异齿裂腹鱼肠道菌群的丰富度和多样性差异不显著。

表3 野生组和人工养殖组组间差异检验数据
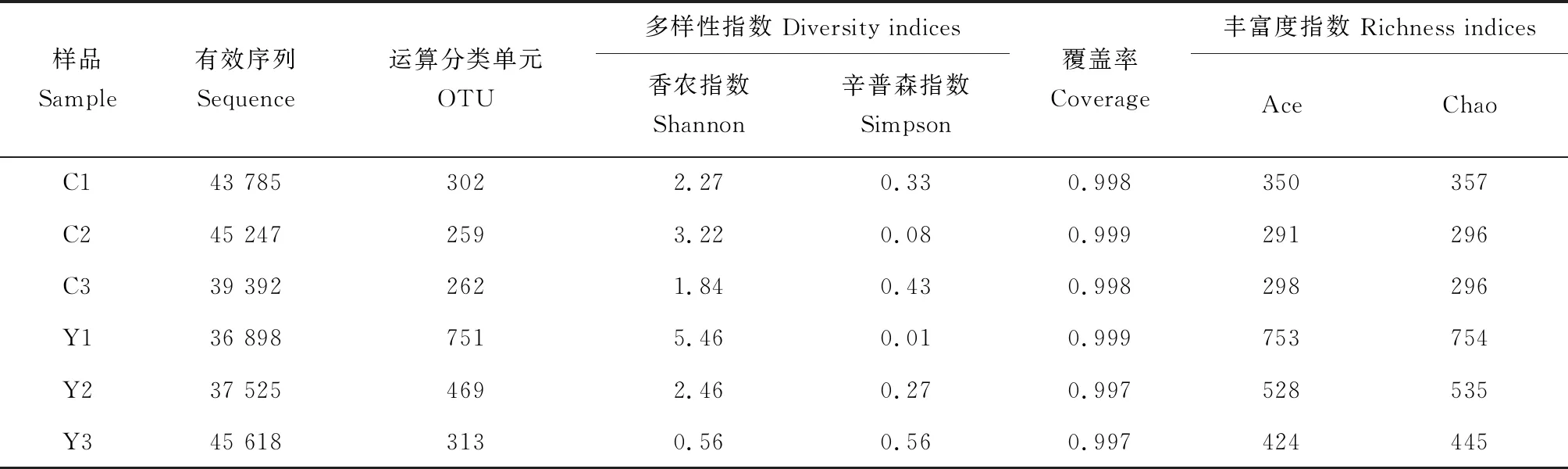
表2 基于16S rRNA基因序列的细菌多样性指数
2.3 菌群结构组成
根据分类结果,在6个异齿裂腹鱼肠道样品中检测到的细菌归属于25个门、54个纲、111个目、219个科、393个属,主要门类有变形菌门、厚壁菌门、放线菌门、蓝细菌门、梭杆菌门、拟杆菌门、绿弯菌门、疣微菌门、衣原体门和软壁菌门。异齿裂腹鱼野生组中Y1样品中含有10个门类细菌,其中优势菌群为放线菌门(53.89%),次优势菌群为厚壁菌门(14.87%);Y2样品中含有8个门类细菌,其中优势菌群为变形菌门(70%),次优势菌群为梭杆菌门(15.61%),未发现拟杆菌门和软壁菌门;Y3样品中含有6个门类细菌,其中优势菌群为变形菌门(94.82%),次优势菌群为厚壁菌门(1.93%),未发现绿弯菌门、疣微菌门、衣原体门和软壁菌门;C1样品中含有9个门类细菌,其中优势菌群为蓝细菌门(61.19%),次优势菌群为变形菌门(28.57%);C2样品中含有9个门类细菌,其中优势菌群为变形菌门(52.78%),次优势菌群为蓝细菌门(37.26%);C3样品中含有9个门类细菌,其中优势菌群为蓝细菌门(69.97%),次优势菌群为变形菌门(24.31%)(表4),在人工养殖组的3个样品中均未发现软壁菌门。

表4 样品优势细菌门类及相对丰度 %
通过对不同样品中所含菌群丰度最高的10个运算分类单元进行分析,有助于了解样品中所含的主要细菌类型,因此依据每个样品中相对丰度最高的前10种运算分类单元在属水平上对每个样品的菌群结构及分布进行统计分析(图2)。其中,Y1中可分类的细菌主要是微球菌属(Micrococcaceae)(4.9%)、暗黄类诺卡氏菌(Nocardioidesfulvus)(3.7%)、微杆菌属(Microbacterium)(3.7%)、蓝细菌(2.1%)、鲸杆菌属(Cetobacterum)(1.3%)、气单胞菌属(Aeromonas)(1.1%)、红杆菌科(0.6%)和希瓦氏菌属(Shewatiella)(0.6%);Y2中可分类的细菌主要是气单胞菌属(49%)、鲸杆菌属(14.8%)、希瓦氏菌属(9%)、红杆菌科(0.8%)、FukuN57(0.8%)、α-变形细菌纲(0.15%)、微球菌属(0.6%)和暗黄类诺卡氏菌(0.3%);Y3中可分类的细菌主要是气单胞菌属(92%)、希瓦氏菌属(0.3%)和微球菌属(0.1%);C1中可分类的细菌主要是蓝细菌(59%)、红杆菌科(12.2%)、FukuN57(8%)、瘦鞘丝藻属(Leptolyngbya)(1.2%)、α-变形细菌纲(1.2%)、鲸杆菌属(0.7%)、玫瑰球菌属(Roseococcus)(0.7%)和Subsection Ⅲ(0.5%);C2中可分类的细菌主要是FukuN57(19%)、蓝细菌(16%)、瘦鞘丝藻属(15%)、红杆菌科(12.1%)、Subsection Ⅲ(5.2%)、玫瑰球菌属(4.2%)、α-变形细菌纲(4.2%)和希瓦氏菌属(0.8%);C3中可分类的细菌主要是蓝细菌(70%)、红杆菌科(10%)、FukuN57(5.9%)、玫瑰球菌属(1.1%)、α-变形细菌纲(1%)、单胞菌属(0.8%)、瘦鞘丝藻属(0.5%)、鲸杆菌属(0.4%)和Subsection Ⅲ(0.1%)。
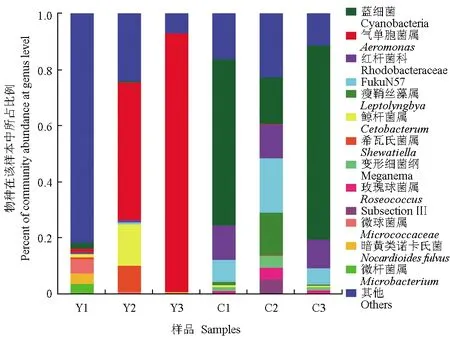
图2 样品种属水平细菌种群结构分布
2.4 基于运算分类单元数量的肠道菌群结构组成和系统进化树
通过在门、属的分类水平上对异齿裂腹鱼肠道内菌群结构进行分析,结果表明,在野生和人工养殖条件下,异齿裂腹鱼的优势菌种有所不同。基于野生组和人工养殖组中全部运算分类单元作韦恩图(图3),比较两种养殖条件下样品间的差异。异齿裂腹鱼肠道样品有效运算分类单元总数为930,两种养殖模式下异齿裂腹鱼肠道共有运算分类单元数为258个,人工养殖组特有的运算分类单元数为98个,野生组特有的运算分类单元数为574个。可见在野生和人工养殖两种条件下,异齿裂腹鱼的肠道内均具有一定比例的特有菌群。

图3 两种养殖模式下异齿裂腹鱼肠道共有运算分类单元分析
由样品聚类树(图4)可以看出,野生组和人工养殖组的异齿裂腹鱼的肠道菌群进化关系较远,说明野生和人工养殖条件下异齿裂腹鱼肠道菌群结构相似度较低,存在一定的差异。

图4 样品聚类树
3 讨 论
3.1 野生与养殖异齿裂腹鱼肠道菌群的丰富度和多样性差异
肠道是营养物质消化吸收的场所,鱼类不同的生理状态会形成不同的菌群结构,而不同的菌群结构会影响异齿裂腹鱼的生理状态。本次研究的6个样品基于Illumina MiSeq平台高通量测序分析,比较了异齿裂腹鱼在野生和人工养殖两种模式下的肠道菌群结构。通过对样品序列数目分析,可得6个样品的有效序列数为39 942~47 068,可归为259~751个运算分类单元;通过对样品菌群多样性分析,可知本次试验数据可靠,能够真实有效地反映出样品中优势细菌的数量关系,野生和人工养殖异齿裂腹鱼肠道菌群的丰富度和多样性差异不显著。关于淡水鱼类肠道菌群结构已有诸多报道[11-13],李学梅等[14]发现,在相同饲料和水环境条件下,银鲫(Carassiusauratusgibelio)和异育银鲫的肠道微生物群落结构无显著差异,二者肠道微生物群落结构与斑点叉尾(Ictaluruspunctaus)差别较大;王琴等[15]发现,鲢鱼(Hypophthalmichthysmolitrix)和鳙鱼(Aristichthysnobilis)肠道菌群结构相似性较高,匙吻鲟(Polyodonspathala)与鲢鱼、鳙鱼的肠道菌群相似性低。
3.2 野生与养殖异齿裂腹鱼肠道菌群的组成结构分析
本研究结果显示,MiSeq高通量测序技术准确分析了异齿裂腹鱼在两种养殖模式下肠道菌群结构的不同,克服了常规分离细菌培养的限制。对高通量测序结果进行分析,本研究结果显示,野生组异齿裂腹鱼肠道的优势菌群是气单胞菌属、微球菌属和微杆菌属;人工养殖组肠道优势菌群是蓝细菌和红杆菌。有研究表明鱼类肠道细菌群落的形成受水中细菌、饵料等影响[16],而有关鱼类肠道细菌与养殖环境中细菌之间的关系尚未明确。Cahill[17]认为,肠道菌群是环境或饵料中存在并且能在肠道内繁殖的细菌群落。Munro等[18]认为,鱼类肠道菌群的主要来源是所摄取的活饵料而不是养殖水体。相反,部分研究结果表明,鱼类肠道菌群与饲料和水中的细菌并不相同[19]。异齿裂腹鱼为杂食性偏植食性鱼类[20],人工驯化后异齿裂腹鱼能够形成一定的摄食习性。陈孝煊等[21]指出,水体的盐度、水温、微生物种类、鱼类摄食的饵料及鱼类不同的生理状态和不同发育阶段都会影响鱼类消化道菌群结构的形成,水体中的菌群也会对鱼类的消化道菌群结构造成直接影响[22-23];王建建等[24]在对野生和养殖银鲳(Pampusargenteus)消化道菌群结构分析发现,两者菌群结构存在明显差异;Holben等[25]也发现,由于不同的生长环境,养殖型和野生型鲑鱼肠道菌群结构存在明显差异;李可俊等[26]对长江河口8种野生鱼类的肠道内菌群多样性分析比较后发现,生活在不同水层及同水层但食性不同的鱼类其肠道菌群结构存在较大差异。由此可推测,异齿裂腹鱼的肠道菌群结构随养殖环境的差异而发生改变。
本研究通过对两种养殖模式下异齿裂腹鱼肠道菌群结构的深入分析,对异齿裂腹鱼肠道菌群的结构、优势菌群及多样性有了一定认识,研究结果可为异齿裂腹鱼的健康养殖及人工养殖环境的微生态调控技术构建提供理论依据。
