南美白对虾4种病原体可视化快速检测试剂盒(生物芯片法)的研发
2020-07-23宋居易刘昱辰张苏楠江高伟杨华卫
宋居易 刘昱辰 张苏楠 江高伟 杨华卫


摘要:利用反向点杂交技术,研制灵敏度较高的南美白对虾4种病原体检测试剂盒,其检测指标包括致对虾急性肝胰腺坏死病副溶血弧菌(VpAHPND)、对虾白斑综合征病毒(WSSV)、对虾传染性皮下及造血组织坏死病毒(IHHNV)、对虾肝肠胞虫(EHP),用重组质粒分别对其进行了最低检测限测试,并与现有推荐方法进行比较。本研究研发的检测试剂盒对4种病原体的最低检测限为1 μl 10~100 拷贝。特异性检测结果表明,4种病原体彼此不产生交叉反应,特异性良好,与各病原体推荐检测方法的结果相比,阳性符合率完全一致。本研究研发的检测试剂盒具有操作方便,灵敏度高,特异性好,检测时间短,设备要求简单等特点,适合应用于南美白对虾VpAHPND、WSSV、IHHNV、EHP的检测、筛查工作中。
关键词:南美白对虾;病原体;生物芯片法;反向点杂交技术
中图分类号:S945.4文献标识码:A文章编号:1000-4440(2020)03-0656-10
Development of a rapid visualization detection kit (biochip method) for four pathogens in Penaeus vannamei
SONG Ju-yi1,LIU Yu-chen2,ZHANG Su-nan2,JIANG Gao-wei2,YANG Hua-wei2
(1.Jiangsu Institute of Agriculture Sciences for Region along the Yangtze River, Rugao 226541, China;2.Jiangsu Huntarray Biological Technology Co., Ltd., Nantong 226400, China)
Abstract:The reverse hybridization technique was used to develop the rapid visualization detection kit (biochip method) for four pathogens in Penaeus vannamei. The detection indices included acute hepatopancreatic necrosis disease-causing Vibrio parahaemolyticus (VpAHPND), white spot syndrome virus (WSSV), infectious hypodermal and hematopoietic necrosis virus (IHHNV), Enterocytozoon hepatopenaei (EHP). The recombinant plasmid was used to test the minimum detection limit, and the results based on kits were compared with those based on the common methods. The minimum detection limit of four pathogens based on this detection kit was 10-100 copies per microliter. In addition, the results of specificity tests showed that there was no cross-reaction among four pathogens. Compared with the results of the recommended detection methods, the positive coincidence rate was completely consistent. The detection kit introduced in this study has the characteristics of easy operation, high sensitivity, good specificity, short detection time and simple equipment. It is suitable for the screening and detection of VpAHPND, WSSV, IHHNV and EHP in Penaeus vannamei.
Key words:Penaeus vannamei;pathogen;biochip method;reverse hybridization technique
南美白對虾是重要的经济虾品种,具有较大的经济效益和社会效益[1-3]。有研究发现,病害对于渔业产值的影响仅次于台风、洪涝等自然灾害[4]。致对虾急性肝胰腺坏死病副溶血弧菌(VpAHPND)、对虾白斑综合征病毒(WSSV)、对虾传染性皮下及造血组织坏死病毒(IHHNV)、对虾肝肠胞虫(EHP)是导致许多地区南美白对虾大批量死亡的主要原因之一[5-6],对虾一旦被这些病原体侵入,便无法治愈,造成大量损失。所以从源头防止携带这些病原体的虾苗进入养殖环境极其重要,这就需要采用准确、高效、灵敏的检测手段进行检测,及时处理携带病原体的虾苗。苗种检测往往要求快速、高效、全面、准确,但是目前使用的核酸检测手段无法满足这些要求,各种分子检测手段还有很大的局限性。各病原体检测的国家标准方法及行业标准多采用PCR或者巢式PCR法,但是PCR法的最低检测限较低[7],巢式PCR法的步骤较为繁琐,检测指标单一,并且容易产生核酸污染[8-10]。荧光定量PCR法使用染料法对病原体进行检测,无法对提取过程进行质量控制,并且在指标较多时采用熔解曲线法容易造成误读。探针法的检测指标少,对于大多数四通道的荧光定量PCR仪而言,无法实现单管检测4个以上病原体指标[11-12],并且成本较高,不利于大规模推广。本研究拟在前人研究基础上,针对VpAHPND、WSSV、IHHNV、EHP的基因序列分别设计特异性引物、探针,构建生物芯片检测体系,以期研发出一种能够高效、快速检测这4种对虾病原体的理想试剂盒产品。
1材料与方法
1.1试驗材料
1.1.1主要试剂及仪器动物组织基因组提取试剂盒、核酸杂交液、前处理液、后处理液、杂交洗涤液1、杂交洗涤液2、显色液、酸杂交晶格均购自江苏猎阵生物科技有限公司,PCR扩增试剂(10×Buffer、Taq DNA 聚合酶、尿嘧啶-N-糖基化酶、脱氧尿嘧啶、脱氧核糖核苷三磷酸)、硝酸纤维素膜购自赛多利斯集团,微孔板恒温振荡器购自杭州奥盛仪器有限公司。
1.1.2供试样本含有WSSV、EHP病原体的对虾组织样本及VpAHPND样本来源于中国水产科学研究院黄海水产研究所,含有IHHNV病原体的对虾组织样本以及无病原体的健康对虾组织样本从江苏省南通市当地采样获得。其中,含有IHHNV病原体的对虾组织经过国家标准方法检测为IHHNV阳性,无病原体的健康对虾组织样本经过4种病原体的国家标准方法或世界动物卫生组织(OIE)和亚太水产养殖中心网(NACA)推荐方法检测为阴性。
1.1.3供试质粒标准品阳性供试质粒由江苏猎阵生物科技有限公司前期获得,分别取VpAHPND、WSSV、EHP、IHHNV的特异性基因片段进行扩增,再将片段插入到pUCm-T载体中,将连接产物转化至DH5α感受态细胞中,进行阳性菌落筛选后提取质粒,进行质粒质量浓度测定并将其稀释至1 ng/μl,构建质粒标准品,-20 ℃保存,使用PCR验证并测序[由生工生物工程(上海)股份有限公司完成]的方法检测是否正确插入了目的片段。采用超微量分光光度计测定质粒DNA浓度和纯度,根据摩尔定律计算单位体积质粒所含的DNA拷贝数。质粒拷贝数=[质粒质量浓度×质粒体积×6.02×1 023.00]/[(载体长度+片段长度)×660.00]
1.2试验方法
1.2.1供试引物、探针的设计根据4种对虾病原体(VpAHPND、WSSV、EHP、IHHNV)及对虾18S rDNA特异性基因序列,设计该基因的扩增引物序列和检测探针序列,利用在线数据库Blast进行比对,确保引物、探针的特异性,并将设计好的序列交由生工生物工程(上海)股份有限公司合成,具体信息见表1、表2。
1.2.2多重PCR扩增体系的建立 利用单因素控制法将合成的引物依据不同的上下游配比进行混合,对Mg2+浓度、脱氧核糖核苷三磷酸用量、上下游引物浓度配比以及一轮、二轮PCR的2种退火温度和扩增循环数进行探索,利用琼脂糖凝胶电泳观察条带的扩增情况,得到最优的扩增条件。
1.2.3芯片制备工艺的建立利用江苏猎阵生物科技有限公司独有的反向点杂交技术研发平台建立芯片制备工艺技术。使用15×柠檬酸钠缓冲液(SSC)浸泡硝酸纤维素膜,60 ℃烘干,用稀释液稀释探针后滴加到芯片上,在0.4 mol/L NaOH溶液中变性,15×SSC浸泡10 min,80 ℃烘干,其探针排布如图1显示。
1.2.4芯片检测体系的建立以对虾肝肠胞虫特异性基因质粒3 fg/μl为PCR扩增模板,利用已经成熟的扩增体系和芯片制备生产工艺研究合适的杂交条件(杂交时间、杂交温度、洗涤时间、洗涤次数、显色温度、显色时间等),最终获得合适的芯片检测体系。
1.2.5对虾组织样本提取根据江苏猎阵生物科技有限公司提供的海洋动物组织基因组提取试剂盒(磁珠法)的使用说明书进行操作,取20 mg对虾组织加至混有50 μl磁珠、200 μl裂解液1、200 μl裂解液2、50 μl蛋白酶K的试管中,60 ℃ 1 500 r/min恒温振荡20 min,取出后放入磁力架中,静置数秒后用移液器吸出液体并弃置,加入70 μl洗脱液,置于72 ℃恒温振荡器中5 min,取出放入磁力架中,取上清液即可。
1.2.6最低检测限试验用三羟甲基氨基甲烷(Tris)-乙二胺四乙酸(EDTA)缓冲液(TE)对含有4种病原体靶标基因碱基序列的重组质粒进行10倍梯度稀释,稀释至10.000 pg/μl(约1 μl 3.3×106拷贝)、0.300 fg/μl(约1 μl 100.0拷贝)、0.030 fg/μl(约1 μl 10.0拷贝)、0.003 fg/μl(约1 μl 1.0拷贝),然后进行生物芯片检测,共进行3次平行试验。根据探针排布情况观察是否出现对应的信号点,确定其最低检测限及特异性。确定各指标最低检测限后,采用提取的阴性虾基因组,将含有4种病原体(VpAHPND、WSSV、EHP、IHHNV)靶标基因碱基序列的标准质粒10倍梯度稀释至其最低检测限质量浓度进行检测,以模拟正常染病的虾基因组检测情况,观察试验结果。
1.2.7特异性试验用TE对含有本研究4种病原体靶标基因碱基序列的重组质粒进行10倍梯度稀释,稀释至10.000 pg/μl,与阴性虾基因组共同进行生物芯片检测,共进行3次平行试验。
1.2.8生物芯片法与推荐方法的对比验证以含有VpAHPND、WSSV、EHP、IHHNV的对虾组织样本为试验材料,比较其推荐方法[9,13-15]与本研究所述生物芯片法的检测结果。分别利用现有的推荐方法(所使用的引物见表3)和本研究所述生物芯片法检测供试样本,比较阳性符合率,并将巢式PCR或PCR扩增产物送至生工生物工程(上海)股份有限公司进行测序确认,从而对试剂盒性能进行评价。
2结果与分析
2.1多重PCR扩增体系的建立
通过一系列优化(包括Mg2+浓度的梯度优化和正反向引物的比例优化等)确定18 μl五重PCR扩增反应液的组分,每个组分加入3 μl含4种病原体靶标基因碱基序列的重组质粒,质粒质量浓度为10 pg/μl,并且根据退火温度的梯度试验结果确定合适的扩增条件。通过双链扩增退火温度梯度试验结果,确定一轮PCR退火温度为55 ℃。PCR反应条件为:50 ℃ 2 min;95 ℃ 4 min;95 ℃ 30 s,55 ℃ 30 s,72 ℃ 20 s,40个循环;95 ℃ 30 s;68 ℃ 30 s,72 ℃ 5 s,3个循环。图2显示,当一轮PCR退火温度为55 ℃时,分别扩增含有本研究4种病原体靶标基因碱基序列的质粒标准品,均可见明显的扩增条带。
2.2生物芯片检测体系的建立
對杂交时间、杂交温度、显色时间、显色温度等进行研究,确立合适的杂交检测条件:杂交时间9 min,杂交温度45 ℃,显色时间3 min,显色温度45 ℃。图3显示,生物芯片检测条件为:杂交时间分别为3 min、6 min、9 min、12 min;杂交条件为45 ℃,1 500 r/min;洗涤条件为:45 ℃,2 min,采用洗涤液洗涤2次;显色条件为:45 ℃,1 500 r/min,3 min。模板为3 fg/μl EHP质粒标准品。杂交时间为9 min和12 min时,信号点较为明显且信号相差不大,所以选择9 min作为最终的杂交时间。
2.3最低检测限分析
最低检测限试验结果(图4)显示,生物芯片法对VpAHPND的最低检测限为1 μl 1拷贝,对IHHNV、WSSV的最低检测限为1 μl 10拷贝,对EHP的最低检测限为1 μl 100拷贝。
图5显示,将最低检测限浓度的质粒与阴性虾基因组混合,VpAHPND、WSSV、IHHNV的最低检测限可达到1 μl 10拷贝,EHP的最低检测限为1 μl 100拷贝,VpAHPND、IHHNV、WSSV、EHP在最低检测限下的信号均明显增强。
A:对虾急性肝胰腺坏死病副溶血弧菌(VpAHPND);B:对虾肝肠胞虫(EHP);C:对虾白斑综合征病毒(WSSV);D:对虾传染性皮下及造血组织坏死病毒(IHHNV)。M:Marker DL2000;1~8:退火温度分别为51 ℃、52 ℃、53 ℃、54 ℃、55 ℃、56 ℃、57 ℃、58 ℃。
1st:第一次试验结果;2nd:第二次试验结果;3rd:第三次试验结果。
2.4特异性分析
对本研究4个病原体较高浓度的质粒和阴性虾基因组用生物芯片法进行检测,结果(图6)显示,较高浓度的质粒及阴性虾基因组均未产生非特异性信号,证明特异性良好。
2.5生物芯片法与推荐方法结果的对比验证
VpAHPND检测结果(图7)显示,利用AP4巢式PCR方法进行检测时,阴性对照组无明显条带,阳性对照组有明显条带,表明检测结果是有效的。1号、4号、6号样本在巢式PCR一轮扩增即出现目的条带,为强阳性或中阳性样本,2号样本在二轮扩增出现目的条带,为弱阳性样本,对AP4巢式PCR二轮产物进行测序,确认阳性样本为1号、2号、4号、6号样本。利用生物芯片法检测时,杂交质控信号点出现说明生物芯片检测成功,结果显示,1号、2号、4号、6号样本出现明显的阳性信号,说明2种方法的检测结果一致。
A:1 μl 1 000拷贝质粒模板;B:1 μl 100拷贝质粒模板;C:1 μl 10拷贝质粒模板;D:1 μl 1拷贝质粒模板。VpAHPND、WSSV、IHHNV、EHP见图2注。1st:第一次试验结果;2nd:第二次试验结果;3rd:第三次试验结果。
VpAHPND、WSSV、IHHNV、EHP见图2注。1st:第一次试验结果;2nd:第二次试验结果;3rd:第三次试验结果。
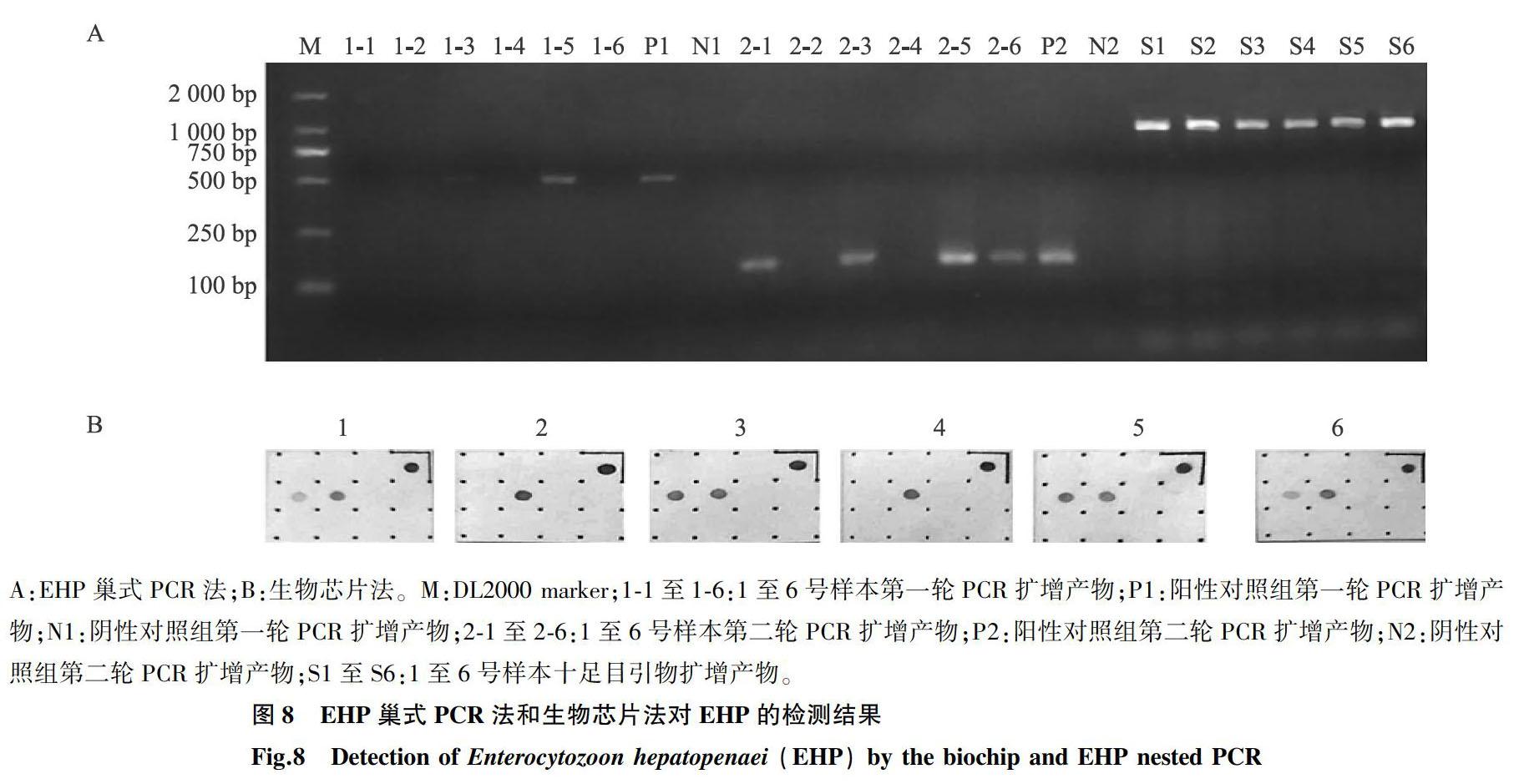
EHP检测结果(图8)显示,利用EHP巢式PCR法进行检测时,阴性对照组无条带,阳性对照组有明显条带。使用十足目引物扩增提取的虾基因组出现明显条带,说明基因组提取成功,检测结果有效。5号样本在PCR一轮扩增即出现目的条带,为强阳性或中阳性样本,1号、3号、6号样本在PCR二轮扩增出现目的条带,为弱阳性样本,对PCR二轮扩增产物进行测序,确认EHP阳性样本为1号、3号、5号、6号样本。利用生物芯片法检测时,杂交质控、全程质控均出现信号点说明检测成功,结果显示,1号、3号、5号、6号样本出现明显的阳性信号,说明2种方法检测结果一致。
VpAHPND、WSSV、IHHNV、EHP见图2注。N:阴性虾基因组。1st:第一次试验结果;2nd:第二次试验结果;3rd:第三次试验结果。
A:AP4巢式PCR法;B:生物芯片法。M:DL2000 marker;1-1至1-6:1至6号样本第一轮PCR扩增产物;P1:阳性对照组第一轮PCR扩增产物;N1:阴性对照组第一轮PCR扩增产物;2-1至2-6:1至6号样本第二轮PCR扩增产物;P2:阳性对照组第二轮PCR扩增产物;N2:阴性对照组第二轮PCR扩增产物。
A:EHP巢式PCR法;B:生物芯片法。M:DL2000 marker;1-1至1-6:1至6号样本第一轮PCR扩增产物;P1:阳性对照组第一轮PCR扩增产物;N1:阴性对照组第一轮PCR扩增产物;2-1至2-6:1至6号样本第二轮PCR扩增产物;P2:阳性对照组第二轮PCR扩增产物;N2:阴性对照组第二轮PCR扩增产物;S1至S6:1至6号样本十足目引物扩增产物。
WSSV检测结果(图9)显示,利用文献[13]的套式PCR检测法进行检测时,阴性对照组无条带,阳性对照组有明显条带。使用十足目引物扩增提取的虾基因组出现明显条带,说明基因组提取成功,检测结果有效。1号、3号样本在PCR一轮扩增出现目的条带,为强阳性或中阳性样本,对PCR二轮扩增产物进行测序,确认WSSV阳性样本为1号、3号样本。利用生物芯片法检测时,杂交质控、全程质控均出现信号点表明检测成功,结果显示,1号、3号样本出现明显的阳性信号,说明2种方法检测结果一致。
IHHNV检测结果(图10)显示,利用文献[14]的普通PCR方法进行检测时,阴性对照组无条带。由于所取的样本为先前标准方法检测的阳性样本,故复检的样本1~8均有目的条带,为强阳性或中阳性样本,对PCR扩增产物进行测序,确认IHHNV阳性样本为1~8号样本。利用生物芯片法检测时,杂交质控、全程质控均出现信号点表明检测成功,1~8号样本出现非常明显的阳性信号,说明2种方法检测结果一致。
综上所述,本研究生物芯片法分别与4种病原体的推荐检测方法的检测结果一致,阳性符合率均为100%。
A:巢式PCR法;B:生物芯片法。M:DL2000 marker;1-1至1-4:1至4号样本第一轮PCR扩增产物;P1:阳性对照组第一轮PCR扩增产物;N1:阴性对照组第一轮PCR扩增产物;2-1至2-4:1至4号样本第二轮PCR扩增产物;P2:阳性对照组第二轮PCR扩增产物;N2:阴性对照组第二轮PCR扩增产物;S1至S4:1至4号样本十足目引物扩增产物。
A:普通PCR法;B:生物芯片法。M:DL2000 marker;1~8:1~8号样本;N:阴性对照。
3讨论
生物芯片检测方法在各领域均发挥一定作用,特别是在人体的体外诊断中应用较为广泛,如新生儿耳聋遗传基因诊断、人乳头瘤病毒基因分型诊断、结核分枝杆菌Spoligotyping(48型)分型诊断等[16-18],其优势在于检测指标多,区分度高(可以区分单碱基突变)[19],但由于其灵敏度相对较低,手动操作较繁琐,自动化仪器设备成本高,极大制约了该方法在其他领域的发展[20]。
本研究将多重PCR单链扩增技术和独特的生物芯片制备技术相结合,具有成本低,检测指标多,速度快,灵敏度高等特点,不同于传统生物芯片通过热变性产生单链的方法,而是利用引物量和退火温度的双重不对称性,产生大量单链[21-22],提高检测效率的同时,简化了操作步骤。由于引物标有生物素,所以单链中同样存在生物素标记,在杂交过程中,目标单链可以与探针结合,而一端的生物素与标记有磷酸脂酶的链酶亲和素结合,在底物显色液的作用下显色,从而达到可视化检测的目的。
利用不对称扩增产生单链,相较于常用的热变性方法,效率更好,操作更加方便[23-24]。在研发过程中发现,在两轮退火温度不对称扩增体系下,单链扩增情况与双链扩增情况呈一定程度的正相关,故可以通过双链扩增条带的检测结果来判断单链扩增情况。这可能是因为在二轮PCR中,单链的产生同样来源于双链。正是由于良好的单链产生效率才使得杂交系统仅需要很短的杂交、洗涤、显色时间即可完成多个指标的检测,省去了热变性的步骤,结合配套的基因组提取试剂盒,仅需要3.5 h即可完成对虾急性肝胰腺坏死病副溶血弧菌、对虾白斑综合征病毒、对虾传染性皮下及造血组织坏死病毒、对虾肝肠胞虫4个指标的检测,在目前同类的反向点杂交检测技术中,处于领先水平[25-26]。前期大量的试验摸索,使该生物芯片法实现较高的检测灵敏度,在目前已发表的核酸分子检测技术相关的文章中,其最低检测限处于较高水平[27-28],可以与目前流行的巢式PCR[29]、荧光定量PCR[30]、等温聚合酶扩增技术(RPA)和数字PCR等相媲美[31-32],并且具有其他方法所不具备的优势,包括试剂成本低,仪器要求简单等。不仅如此,该方法所有原材料、仪器、试剂均可以实现国内自主化生产,可以有效抵御由于国际贸易大环境所导致的贸易风险。所以,具有较好的实用性和市场前景。
在试验结果稳定性方面,生物芯片法同样具有一定程度的优势。在试验过程中,发现传统的PCR方法,特别是多重PCR或者巢式PCR,即使是国家标准方法或者OIE/NACA推荐方法也可能会产生非特异性扩增,影响最终结果的判读。生物芯片法是通过探针杂交来判断靶标基因碱基序列的,相较于传统的PCR或巢式PCR法,出现非特异性信号点的可能性较低,即使产生非特异性扩增,由于探针与非特异性单链的不互补也不会产生非特异性信号点,是较为稳定的检测系统。在产品设计时,为了使产品更加完善,在研发之初就加入了全程质量控制体系,即在引物-探针体系中加入十足目特异性引物和探针,在对虾组织提取检测过程中,根据该质控信号点来进一步判断是否存在假阴性结果。同时,为了防止扩增产物的污染,加入了dUTP-UNG防污染系統,一定程度上减少了扩增产物产生污染的可能性。
参考文献:
[1]VALDERRAMA D, ENGLE C R. Economic optimization of shrimp farming in Honduras[J]. Journal of the World Aquaculture Society, 2002, 33(4):398-409.
[2]LIAO D S, LIAO I C. An economic evaluation of shrimp farming industry in Taiwan[M]. New Jersey: Wiley-blackwell, 2007:187-196.
[3]BARBIER E B, COX M. An economic analysis of shrimp farm expansion and mangrove conversion in Thailand[J].Land Economics, 2004, 80(3):389-407.
[4]ZHONG G P. Fisheries in China: progress, problems, and prospects[J]. Canadian Journal of Fisheries and Aquatic Sciences, 1997, 54(54):224-238.
[5]VIOLETA M B, ANTONIO L G, JESS A, et al. Echinacea purpurea and Uncaria tomentosa reduce the prevalence of WSSV in whiteleg shrimp (Litopenaeus vannamei) cultured under laboratory conditions[J]. Aquaculture, 2012(358):164-169.
[6]PIYACHAT S, NATTHINEE J, ANEJIT K, et al. A cohort study of 196 Thai shrimp ponds reveals a complex etiology for early mortality syndrome(EMS)[J]. Aquaculture, 2018, 493:26-36.
[7]TANG K, DURAND S V, WHITE B L, et al. Postlarvae and juveniles of a selected line of Penaeus stylirostris are resistant to infectious hypodermal and hematopoietic necrosis virus infection[J]. Aquaculture, 2000, 190(3/4):203-210.
[8]JAROENLAK P, SANGUANRUT P, WILLIAMS B A, et al. A nested PCR assay to avoid false positive detection of the microsporidian Enterocytozoon hepatopenaei (EHP) in environmental samples in shrimp farms[J]. PLoS One, 2016, 11(11): 1-15.
[9]DANGTIP S, SIRIKHARIN R, SANGUANRUT P, et al. AP4 method for two-tube nested PCR detection of AHPND isolates of Vibrio parahaemolyticus[J]. Aquaculture Reports, 2015, 2(2): 158-162.
[10]NATIVIDAD K D, NOMURA N, MATSUMURA M, et al. Detection of white spot syndrome virus DNA in pond soil using a 2-step nested PCR[J]. Journal of Virological Methods, 2008, 149(1): 28-34.
[11]HAN J E, TANG K F, PANTOJA C R, et al. qPCR assay for detecting and quantifying a virulence plasmid in acute hepatopancreatic necrosis disease (AHPND) due to pathogenic Vibrio parahaemolyticus[J]. Aquaculture, 2015,442: 12-15.
[12]QIU L, CHEN M, WAN X, et al. Detection and quantification of shrimp hemocyte iridescent virus by TaqMan probe based real-time PCR.[J]. Journal of Invertebrate Pathology, 2018,154: 95-101.
[13]國家质量监督检验检疫总局国家标准化管理委员会.白斑综合征(WSD)诊断规程:GB/T 28630.2-2012[S]. 北京:中国标准出版社,2012:1-14.
[14]国家质量监督检验检疫总局国家标准化管理委员会.对虾传染性皮下及造血组织坏死病毒(IHHNV)检测PCR 法:GB/T 25878-2010[S]. 北京:中国标准出版社,2011:1-6.
[15]NACA. A New and improved PCR detection method for Enterocytozoon hepatopenaei (EHP) based on a gene encoding a spore wall protein[R]. Bangkok: The Network of Aquaculture Centres, 2018.
[16]YAO Y C, LI N , HU L S, et al. Comparison of the Tellgenplex HPV DNA test with the PCR-reverse dot blot assay for human papillomavirus genotyping[J]. Asian Pacific Journal of Tropical Medicine, 2018, 11(2):57-62.
[17]HIJIKATA M, KEICHO N, VAN DUC L, et al. Spoligotyping and whole-genome sequencing analysis of lineage 1 strains of Mycobacterium tuberculosis in Da Nang, Vietnam[J]. PLoS One, 2017, 12(10):1-13 .
[18]LI S, PENG Q, LIAO S, et al. A reverse dot blot assay for the screening of twenty mutations in four genes associated with NSHL in a Chinese population[J]. PLoS One, 2017, 12(5): 45-49.
[19]ALQASSAB T, SHAYAN P, KAMKAR A, et al. Reverse line blot hybridization assay as a suitable method for the determination of food adulteration in example of sausage samples[J]. European Food Research and Technology, 2019, 245(8): 1677-1683.
[20]ARVINDHAN N, RADOSLAV J, NARENDRA W. Dot blot analysis for measuring global N6-methyladenosine modification of RNA: methods and protocols[M].Totowa: Humana Press, 2019:263-271.
[21]CHAN V, YAM I, CHEN F E, et al. A reverse dot-blot method for rapid detection of non-deletion α thalassaemia[J]. British Journal of Haematology, 1999, 104(3): 513-515.
[22]BUMM T, ELSEA C R, CORBIN A S, et al. Characterization of murine JAK2V617F-positive myeloproliferative disease[J]. Cancer Research, 2006, 66(23): 11156-11165.
[23]BOBY N, ALI S A, PREENA P, et al. Detection of multiple organisms based on the distance-dependent optical properties of gold nanoparticle and dark-field microscopy[J]. Talanta, 2018,188: 325-331.
[24]WANG J, LI H, LI T, et al. Determination of bacterial DNA based on catalytic oxidation of cysteine by G-quadruplex DNAzyme generated from asymmetric PCR: application to the colorimetric detection of Staphylococcus aureus[J]. Microchimica Acta, 2018, 185(9):410.
[25]SABALZA M, YASMIN R, BARBER C A, et al. Detection of Zika virus using reverse-transcription LAMP coupled with reverse dot blot analysis in saliva[J]. PLoS One, 2018, 13(2):1-17.
[26]XU R, HU B, TIAN Y, et al. Development of padlock probe combined with dot-blot hybridization based methods for detection of bacterial spot of melon leaves[J]. Scientia Agricultura Sinica, 2017, 50(4): 679-688.
[27]WANG J, WANG J, LI R, et al. Rapid and sensitive detection of canine distemper virus by real-time reverse transcription recombinase polymerase amplification[J]. BMC Veterinary Research, 2017, 13(1):241-247.
[28]KRISTINE Y, RICHARD F. Real-time quantitative PCR and fast QPCR have similar sensitivity and accuracy with HIV cDNA late reverse transcripts and 2-LTR circles[J]. Journal of Virological Methods, 2008,153(2):253-256.
[29]石蓮琴,翟国莲,吴杰,等.云南地区猪毕氏微孢子虫的分子检测及其基因型鉴定[J].南方农业学报,2018,49(8):1654-1659.
[30]温书香,安利民,赵协,等.猪伪狂犬病毒实时荧光定量PCR检测方法的建立及初步应用[J].江苏农业科学,2019,47(7):50-53.
[31]OLSEN J B, LEWIS C J, MASSENGILL R L, et al. An evaluation of target specificity and sensitivity of three qPCR assay for detecting environmental DNA from Northern Pike (Esox lucius)[J].Conservation Genetics Resources,2015,7(3):615-617.
[32]TADMOR A D, OTTESEN E A, LEADBETTER J R, et al. Probing individual environmental bacteria for viruses by using microfluidic digital PCR[J]. Science, 2011, 333(6038):58-62.
(责任编辑:王妮)
