SLFN14抗LINE-1分子机制研究
2020-07-22毛洋丁寄葳陈淑敏岑山李晓宇
毛洋,丁寄葳,陈淑敏,岑山,李晓宇
研究报告
SLFN14抗LINE-1分子机制研究
毛洋,丁寄葳,陈淑敏,岑山,李晓宇
中国医学科学院&北京协和医学院医药生物技术研究所免疫生物学室,北京 100050
长散在核重复序列1 (long interspersed nuclear element-1, LINE-1)是迄今为止发现的人体基因组中唯一具有自主转座活性的逆转录转座子,其转座常引起宿主基因组不稳定,从而导致包括癌症在内的各种严重基因疾病的发生。宿主因子在宿主抗LINE-1转座中发挥着重要作用。宿主因子SLFN14作为免疫系统重要组成员,具有抗病毒活性。本实验室研究发现SLFN14对于LINE-1的转座具有抑制作用。为进一步探究其具体的作用机制,通过对LINE-1复制周期中的转录、翻译、逆转录、整合环节进行实验分析,证实SLFN14能够通过影响LINE-1 mRNA转录过程及其半衰期,降低LINE-1 mRNA的水平,从而影响LINE-1蛋白及cDNA表达水平,最终导致LINE-1复制受阻。同时,通过对SLFN14活性中心的定位,本研究还发现SLFN14的抗LINE-1活性与其核糖核酸内切酶结构域和核糖体结合结构域密切相关。上述研究结果展示了SLFN14调控LINE-1复制的机制,进一步完善了宿主因子调控网络,为控制因LINE-1复制引起的基因组不稳定提供了新思路。
转座;逆转录转座子;LINE-1;SLFN14;5ʹ-UTR内部启动子区
转座子,又被称为转座元件(transposable elements, TEs),是一类能在基因组移动的DNA重复序列,约占基因组总量的45%[1]。LINE-1是目前发现的人类基因组中唯一具有自主转座活性的转座子,占据人类基因组的17%,其全长约为6000 bp[2],包括5'端和3'端的非翻译区(untranslated regions, UTR)和两个开放式读码框架ORF1及ORF2 (open reading frame 1 protein, open reading frame 2 protein)。5'-UTR是其内部启动子区域[3];ORF1p是RNA结合蛋白,具有分子伴侣活性[4];ORF2p具有逆转录酶活性和核酸内切酶活性[5~8]。LINE-1的转座方式为“复制–粘贴”型转座,即在LINE-1的逆转录转座过程中,首先RNA聚合酶II介导LINE-1启动子启动转录,转录生成的LINE-1 RNA既能作为LINE-1的基因组RNA,又能行使mRNA的功能。随后,LINE-1 RNA出核,进入细胞质,翻译出ORF1p和ORF2p,两者能与LINE-1 RNA结合形成核糖核蛋白复合物(ribonucleoprotein complexes, RNPs)。LINE-1 RNPs进入细胞核,识别宿主细胞基因组DNA上的目的序列,并在双链DNA的其中一条链上切开一个切口,逆转录酶引导LINE-1 RNA逆转录生成LINE-1 cDNA,该过程被称为靶位点引导反转录(target-site primed reverse transcription, TPRT)。最后,DNA的另一条链也被切开,继续进行另一条DNA链的合成,LINE-1 cDNA被整合入到基因组中新的位点,完成逆转录转座[9~11]。
LINE-1转座对基因组的结构和功能都会产生重要影响。在正常分化的细胞中,LINE-1启动子为高甲基化状态,不进行转座[12~15];而在生殖细胞发育、胚胎发育的早期则为激活状态,一旦对其进行抑制,小鼠胚胎无法正常发育[16]。由此可见,宿主细胞对LINE-1的活跃与抑制具有严格的调控。同时,各项研究表明LINE-1在多数肿瘤细胞中高表达[17,18],这表明在病理状态下,活跃的LINE-1会对基因组结构和功能造成不利影响进而引起基因疾病和癌症,这可能是由于LINE-1可以通过插入、缺失、重组和转导等方式影响基因组的完整性和不稳定性。因此,通常情况下,细胞会通过表观遗传学修饰、RNA干扰和宿主限制因子调控等手段对LINE-1的转座进行严格控制[19,20]。表观遗传学修饰主要包括DNA甲基化和组蛋白修饰,它能在转录层面抑制LINE-1转座;RNA干扰途径主要在转录后层面抑制LINE-1转座;宿主限制因子也是近年来研究的热点之一,目前已发现多种宿主限制因子能够抑制LINE-1转座活性[21],其中包括APOBEC家族蛋白、SAMHD1蛋白以及本课题组正在研究的Schlafen (SLFN)家族蛋白。
SLFN家族作为胸腺细胞成熟和激活的调节剂被首次发现[22],随后多篇研究报道了它在其他细胞中的多种功能,如调控细胞增殖、调节T细胞和巨噬细胞分化、抑制肿瘤细胞的移动和侵袭及增强肿瘤细胞对化疗药物的敏感性等[23]。这些研究证实SLFN蛋白是免疫系统的重要组成部分,在抗病毒复制和抗肿瘤方面发挥着重要作用[23]。有研究证实,部分SLFN家族成员具有抗逆转录病毒活性,并且SLFN14作为SLFN家族成员之一已被证实是一种新型抗病毒因子[24,25],考虑到LINE-1在复制周期上与逆转录病毒具有相似性,分析认为SLFN14可能对LINE-1具有抑制作用。因此本研究对SLFN14是否具有抑制LINE-1转座的活性进行研究,并对其抗LINE-1活性位点进行分析,同时对SLFN14抗LINE-1机制进行了初步探讨。
1 材料与方法
1.1 细胞培养
HEK293T、HeLa细胞系(购于美国ATCC细胞库)培养于含有10%胎牛血清(购自美国Gibco公司)的DMEM (购自美国Life Technologies公司)。HEK293T细胞与HeLa细胞均以1∶6的比例进行传代,传代后的细胞置于37℃、5% CO2培养箱静置培养,接种后2~3 d即可长成致密单层。
1.2 脂质体介导的细胞转染
HEK293T与HeLa细胞系以合适的密度(转染过表达质粒时细胞密度应为70%~90%)接种于相应的孔板中。接种24 h后,HEK293T细胞使用Lipofectamine 2000 (购自美国Life Technologies公司)、HeLa使用TransEasy (购自北京博雅创新科技发展有限公司)按说明书进行质粒转染。转染后,将培养皿放置于培养箱中静置培养4~6 h,之后更换细胞对应的完全培养液正常培养。转染48 h后收样。
1.3 逆转录转座实验(retrotransposition assay)
对转染48 h后的HeLa细胞进行计数,在六孔板中接种2.0×105个细胞/孔的HeLa细胞,用含有适宜浓度的G418培养基进行培养。10~12 d后,用PBS润洗六孔板,每孔加入1 mL甲醇固定10 min,随后结晶紫染色10 min,最后用蒸馏水冲洗干净。对形成的细胞集落进行计数。
1.4 总RNA、cDNA提取和Real-time PCR
按广州美基生物科技有限公司的RNA提取试剂盒使用说明书提取细胞总RNA,测定RNA浓度。取2 µg所提取RNA,使用随机引物通过M-MLV反转录酶反转录为cDNA,以反转录的cDNA为模板进行相对定量PCR扩增检测。PCR扩增引物见表1,PCR反应体系见表2。反应条件:98℃ 2 min;98℃ 10 s,55℃ 15 s,72℃ 2 min 15 s,40个循环。
按照QIAamp DNA Mini Kit基因组提取试剂盒说明书要求提取cDNA,取2 µL cDNA进行绝对定量PCR实验。PCR扩增引物见表1,PCR反应体系见表2。反应条件:98℃ 2 min;98℃ 10 s,72℃2 min 40 s,40个循环。

表1 PCR扩增所需引物序列
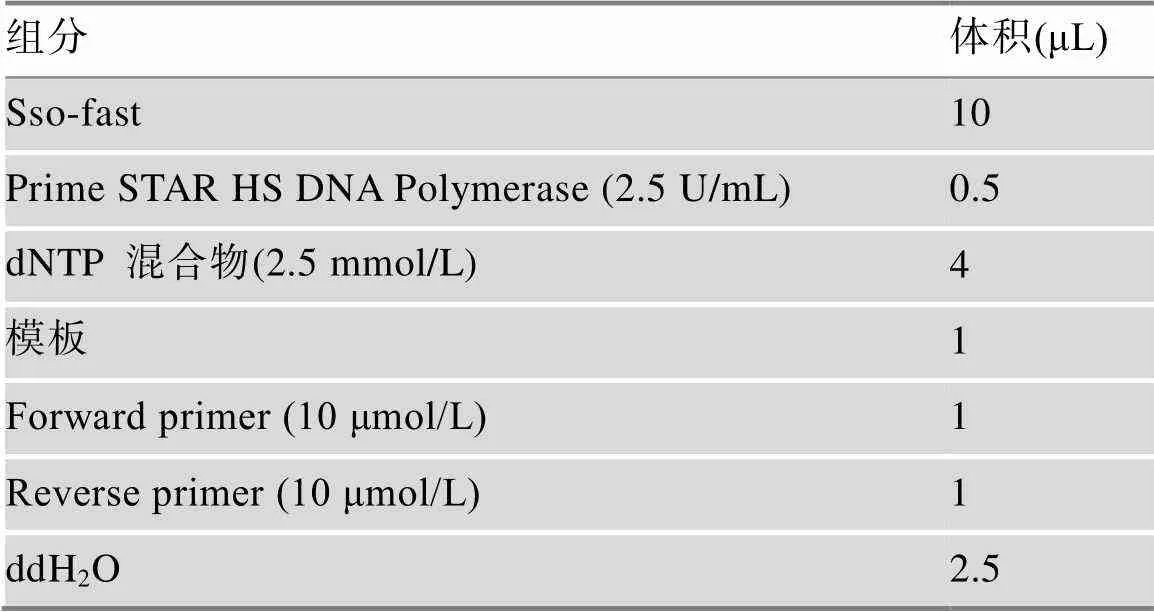
表2 PCR扩增体系
1.5 免疫印迹(Western blot)分析
提取收集细胞总蛋白后,通过Western blot 法检测细胞系中SLFN14-flag或ORF1蛋白表达量。SDS-PAGE进行蛋白分离,恒压电流浓缩胶70 V,分离胶120 V。湿转法进行蛋白转膜,冰浴中以75 V恒压电转75 min。质量分数5%的脱脂奶粉室温封闭1 h。按照说明书标示比例加入一抗稀释液(购自苏州新赛美生物科技有限公司)稀释的β-actin抗体(购自美国Abcam公司,SC-47778)、flag抗体(购自美国Sigma-Aldrich公司,#14793)、ORF1p抗体(由中国医学科学院病原微生物研究所郭斐研究员实验室惠赠),4℃轻摇过夜。次日,洗膜后按照体积比1∶5000比例加入PBST稀释的二抗,室温轻摇1 h。最后辣根过氧化物酶化学发光法显色。
1.6 免疫荧光实验
HeLa细胞铺Ф35 mm共聚焦专用皿,每皿接种5×104个细胞。细胞置于37℃、5% CO2培养箱培养24 h后转染,继续培养48 h,收细胞处理。PBS润洗细胞3次,4%细胞固定液室温固定10 min;吸弃细胞固定液,加入0.02% Triton X-100溶液,室温打孔10 min;PBS洗涤细胞3次,2% BSA溶液封闭1 h后,一抗孵育1 h;回收一抗,PBS洗涤细胞3次,荧光二抗孵育1 h;回收二抗,PBS洗涤细胞3次,加入1~2滴含DAPI的封片液。激光共聚焦显微镜观察。
1.7 Luciferase报告系统实验
HEK293T铺六孔板,转染48 h后收取细胞。细胞吹打混匀,吸取50 µL混匀的细胞液至白色底板中,加入50 µL 2×裂解液,混匀后置于37℃孵箱中放置25 min;将孵育好的样品混匀后吸取10 µL至白色底板中,再加入40 µL luciferase荧光底物,混匀后酶标仪检测。
1.8 LEAP (LINE-1 element amplification protocol)实验
HeLa细胞铺10 cm细胞培养皿,24 h后转染。继续培养48 h后收取细胞。加入细胞裂解液裂解1 h后离心20 min,收集上清液。配置50 mL 8%和16%的蔗糖溶液(1 mol/L NaCl 1.6 mL,0.1 mol/L MgCl22.5 mL,1 mol/L Tris-HCl (pH 7.5) 1 mL,1 mol/L DTT 50 µL,蛋白酶抑制剂1片,无RNA酶水45 mL及4 g或8 g蔗糖)。随后将配制好的蔗糖溶液加入到超速离心管中,上层加入裂解液,4℃超速离心41,000 r/min,3 h。超速离心后用100 µL无RNA酶水溶解离心管底部样品,1∶1加入甘油,–20℃保存。取一部分样品定量后,取适量样品加入到49 µL LEAP反应体系中,反应体系组分见表3。37℃孵育1 h,将反转录后的cDNA进行Real-time PCR扩增,测定各组cDNA量。LEAP实验所需引物序列见表4。
1.9 RNA稳定性实验
HEK293T铺六孔板,转染24 h后用放线菌素D (5 µg/mL)进行处理,分别于0、0.5、1、2、4、8 h收集细胞,提取细胞总RNA。利用Real-time PCR对LINE-1 RNA进行检测。
1.10 统计学分析
实验数据采用GraphPad Prism 5 进行处理,以3次实验的平均值和标准误(mean±SEM)表示。组间比较用检验。
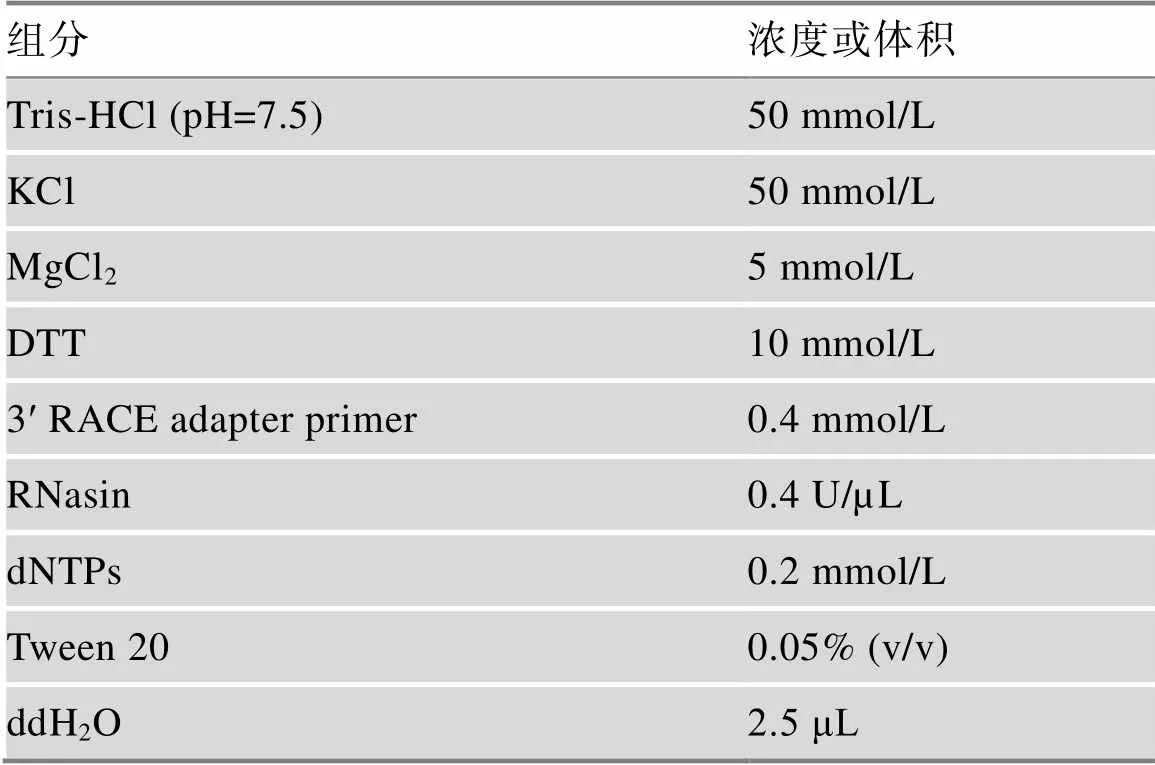
表3 LEAP孵育体系

表4 LEAP实验所需引物序列
2 结果与分析
2.1 SLFN14抑制LINE-1转座
为确定SLFN14能否抑制LINE-1转座活性,本研究进行了逆转录转座实验。该实验是利用Thierry Heidmann实验室的报告质粒CMV-LINE-1-NEORT(以下简称LINE-1质粒),该质粒在LINE-1的3ʹ-UTR上游设计了一个含有内含子的新霉素()抗性基因,此基因只有在LINE-1质粒成功进行逆转录转座去除内含子后才能表达新霉素抗性(图1A)。在含有G418抗性的培养基中,含有成功进行逆转录转座LINE-1的HeLa细胞才能存活,并形成细胞集落,细胞集落数目的多少便能反映LINE-1转座效率的高低[26]。
在HeLa细胞中,共同转染LINE-1与不同浓度的SLFN14-flag质粒,通过逆转录转座实验观察发现,不同转染量SLFN14-flag对含LINE-1细胞的集落形成产生影响。结果显示,随着SLFN14表达的增加,LINE-1形成的细胞集落数量有明显的下降(图1B)。此结果表明,外源表达的SLFN14能明显抑制LINE-1转座且这种抑制活性与SLFN14的表达呈剂量依赖性关系。同时还发现,随着SLFN14表达量的增加,LINE-1 ORF1p的表达也在下降,而且下降的幅度与LINE-1转座水平下降的趋势一致(图1C),这表明SLFN14有可能直接抑制LINE-1 ORF1p蛋白的表达,抑或是通过抑制LINE-1 RNA表达水平来降低ORF1p蛋白的表达。基于此分析,后续的实验中将直接通过检测ORF1p的表达量来反映SLFN14对LINE-1转座的影响。
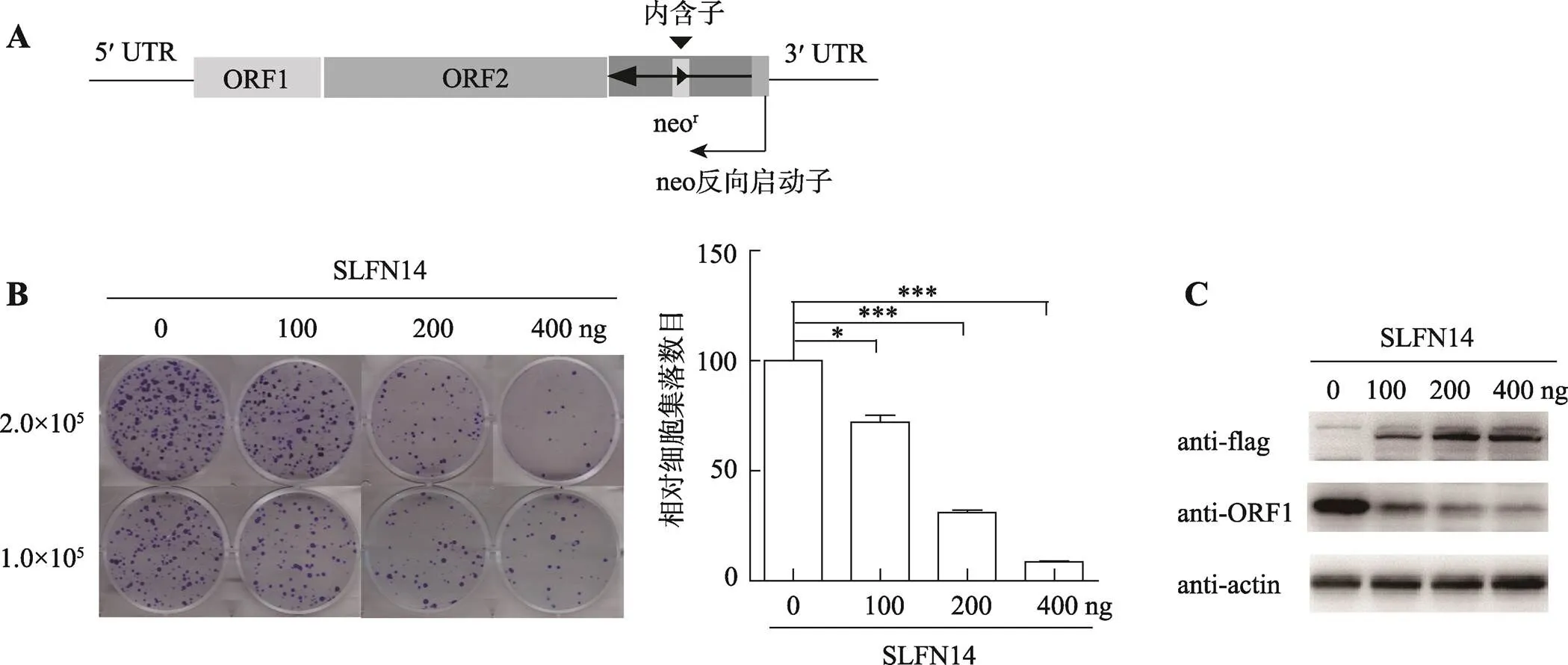
图1 SLFN14抑制LINE-1逆转录转座
A:逆转录转座实验原理示意图。抗性基因中含有一段反向的内含子,此基因只有在LINE-1质粒成功进行逆转录转座去除内含子后才能表达新霉素抗性。B:逆转录转座实验检测分别转染0、100、200、400 ng SLFN14-flag及1000 ng LINE-1质粒后LINE-1逆转录转座形成的细胞集落数量变化。将未转染SLFN14-flag的对照组细胞集落数目看作100;*:< 0.05,***:< 0.001。C:免疫印迹法检测SLFN14对LINE-1 ORF1p表达的影响。
2.2 SLFN14抑制LINE-1的结构域分析
SLFN14包括3个分别具有内切核糖核酸酶活性、核糖体结合功能和解旋酶活性的关键结构域,因此本研究分别构建了缺失以上3个关键结构域的SLFN14 dC1、dC2、dC3、dN1、dN2、dN3六个截短体(图2A)以对不同结构域进行分析。
从2.1实验可知,SLFN14对LINE-1转座的影响可以通过LINE-1 ORF1p的表达体现。因此,为确定SLFN14 dC1、dC2、dC3、dN1、dN2、dN3六个截短体是否仍然具有抗LINE-1活性,本研究共同转染LINE-1与SLFN14的六个截短体质粒,并以SLFN14野生型作为对照。通过检测ORF1p蛋白表达的变化,发现与野生型SLFN14相比,dC1和dC2依然能够一定程度抑制ORF1p的表达活性,证明SLFN14的C端的解旋酶结构域不是SLFN14抑制LINE-1转座活性的关键结构域;另一方面dN3的表达对LINE-1 ORF1p的影响与未转染SLFN14-flag质粒的空白对照组相比没有显著性差异,这提示dN3完全丧失了抗LINE-1活性(图2B)。此结果表明SLFN14解旋酶结构域在其抗LINE-1活性中可能作用有限,而内切核糖核酸酶结构域与核糖体结合结构域在其中发挥重要作用。
利用激光共聚焦显微镜对SLFN14及其截短体在细胞中的分布情况进行了观察。结果显示,保留主要抗LINE-1活性的dC1的细胞定位情况与野生型完全一致,主要定位于细胞质中;而完全丧失了抗LINE-1活性的SLFN14 dN3则有明显弥散现象,在细胞质、细胞核均有分布(图2C)。
上述实验结果表明,SLFN14 dN3不抑制LINE-1 ORF1p表达。基于此,在后续实验中以SLFN14 dN3为阴性对照进行SLFN14抗LINE-1活性的机制研究。
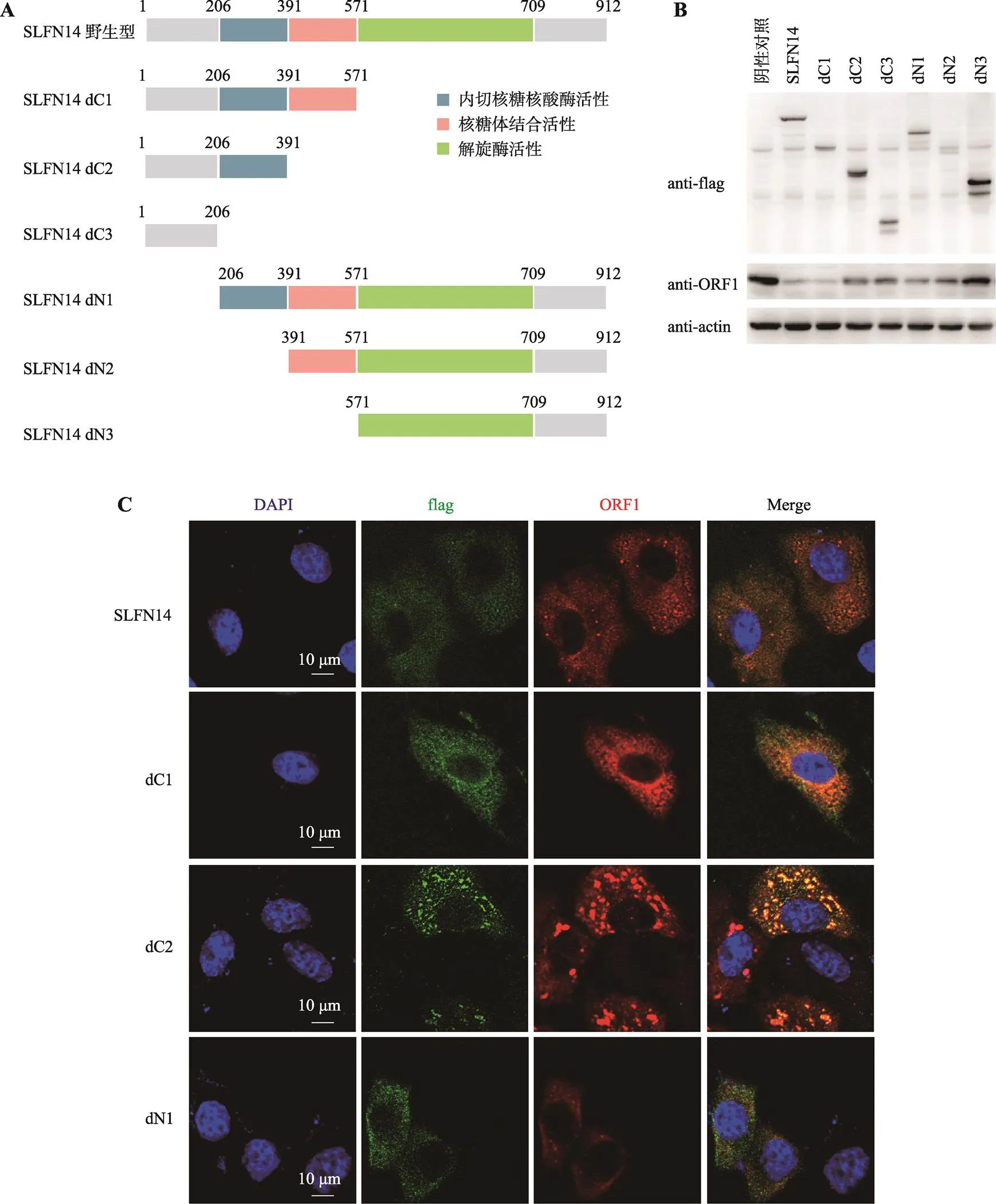
图2 SLFN14抑制LINE-1转座的活性结构域定位
A:SLFN14野生型及各个截短体结构域示意图。B:Western blot检测HEK293T细胞中转染SLFN14-flag野生型及各个截短体质粒时对ORF1p表达情况的影响。将转染LINE-1及pcDNA4.0质粒组作为阴性对照,将转染LINE-1及SLFN14-flag野生型质粒组作为阳性对照。C:免疫荧光实验检测HeLa细胞中转染SLFN14-flag野生型及各个截短体质粒时SLFN14蛋白在细胞内的定位情况。
2.3 SLFN14并非通过影响ORF2p逆转录酶活性影响LINE-1 cDNA水平
以上实验证实SLFN14可以抑制LINE-1逆转录转座,这意味着SLFN14可能会减少插入宿主基因组的LINE-1拷贝数。为验证SLFN14是否会减少插入到细胞基因组中的LINE-1 cDNA,本研究设计了一对引物,其中正向引物只与拼接的基因结合,这样只有从拼接的RNA中逆转录的LINE-1 cDNA才能被扩增(图3A)。与仅转染LINE-1质粒的对照组相比,SLFN14的表达呈剂量依赖性地降低LINE-1 cDNA水平(图3B),而SLFN14 dN3的表达不降低LINE-1 cDNA水平(图3C)。
SLFN14减少LINE-1 cDNA拷贝数的原因有多种,最可能的是影响了LINE-1 ORF2p逆转录酶的活性或直接减少了其RNA的拷贝数,为此首先采用LEAP实验来给予验证。利用细胞内各组分沉降系数的差异进行差速离心,分离各个组分,通过超速区带离心分离得到LINE-1 RNP。对其定量后,利用RNP中自带的ORF2p以及LINE-1 RNA模板模拟TPRT过程,反转录出LINE-1 cDNA并用实时定量PCR进行定量[27]。在等量RNP的情况下,检测SLFN14对逆转录过程的影响,结果显示SLFN14不影响LINE-1的逆转录过程(图3D),提示SLFN14对LINE-1 cDNA的影响并非通过影响LINE-1 ORF2p逆转录酶活性而实现的。由于LINE-1 RNA同时作为编码蛋白质的mRNA和RNA中间体产生新的cDNA拷贝,因此SLFN14更可能是影响了LINE-1 RNA 的转录,SLFN14影响LINE-1 ORF1p表达同样证明了这一观点。
2.4 SLFN14降低LINE-1 RNA水平
将LINE-1和不同浓度的SLFN14质粒共转染HEK293T细胞,然后Real-time PCR测定总LINE-1 RNA,结果显示,随着SLFN14的增加,LINE-1 RNA明显下降且呈剂量依赖性(图4A),SLFN14 dN3则与对照组相比无显著差异(图4B),这表明SLFN14影响了LINE-1 RNA水平。
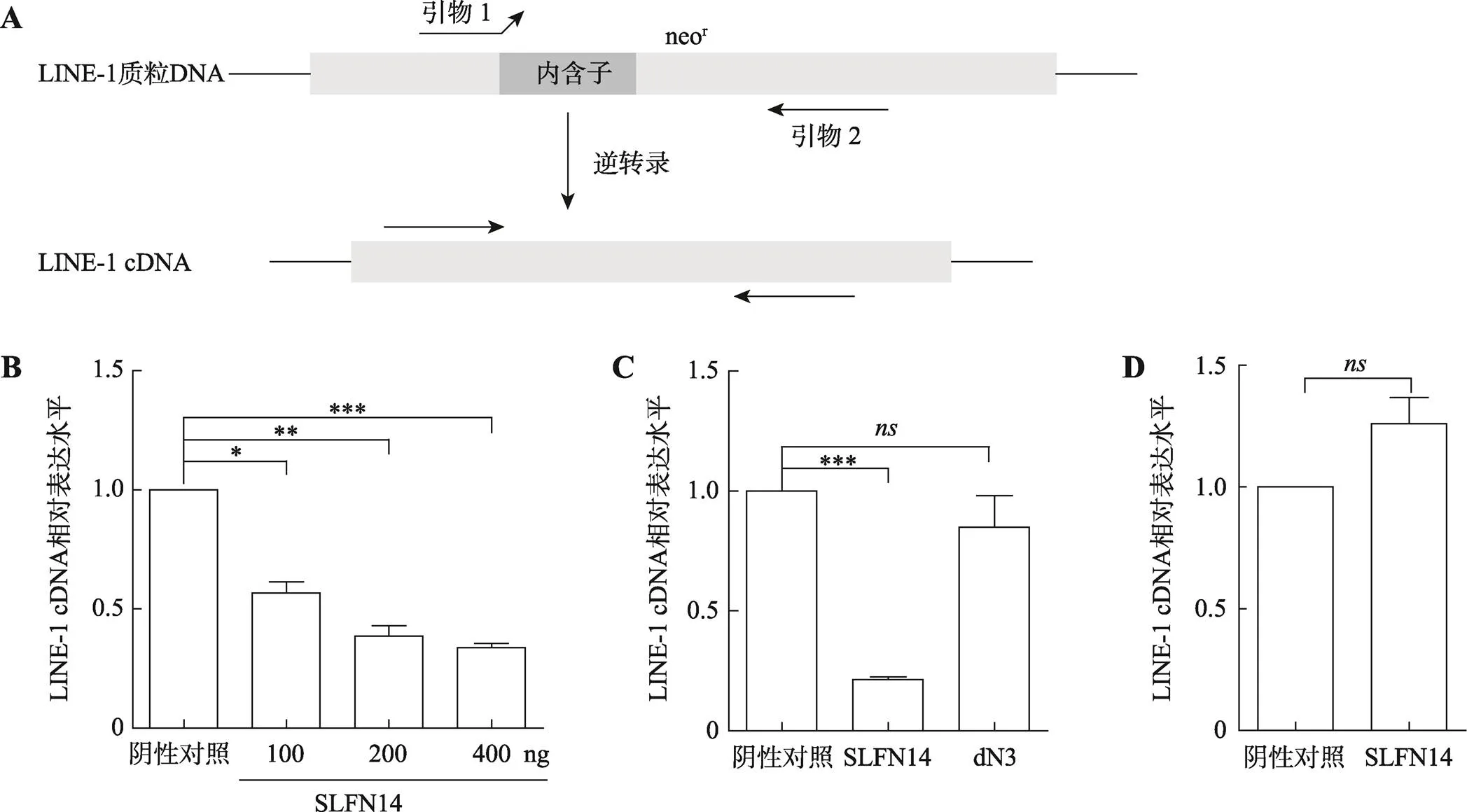
图3 SLFN14并非通过影响ORF2p逆转录酶活性而影响LINE-1 cDNA水平
A:扩增LINE-1 cDNA的引物示意图。此正向引物被设计成只与拼接的耐新霉素基因结合,这样只有从拼接的RNA中逆转录的LINE-1 cDNA才能被扩增。B:Real-time PCR检测梯度转染野生型SLFN14后LINE-1 cDNA的变化情况。C:Real-time PCR检测转染野生型SLFN14及截短体SLFN14 dN3后LINE-1 cDNA的变化情况。D:LEAP实验检测ORF2p逆转录活性对LINE-1 cDNA水平的影响。将转染LINE-1及pcDNA4.0质粒组作为阴性对照组;*:< 0.05;**:<0.01;***:< 0.001;:无显著性差异。

图4 SLFN14能够降低LINE-1 RNA水平
A:Real-time PCR检测梯度转染野生型SLFN14后LINE-1 RNA的变化情况。B:Real-time PCR检测转染野生型SLFN14及截短体SLFN14 dN3后LINE-1 RNA的变化情况。转染LINE-1及pcDNA4.0质粒组作为阴性对照组;***:< 0.001;:无显著性差异。C:Real-time PCR检测放线菌素D处理后不同时间段SLFN14对LINE-1 RNA降解情况的影响。
那么SLFN14是在转录前还是转录后水平发挥作用呢?有研究证实兔源SLFN14具有核酸内切酶活性,能够降解RNA[28]。因此本研究利用放线菌素D能够抑制RNA聚合酶II活性的特性,通过测量LINE-1 RNA的衰减变化进而验证SLFN14是否对LINE-1 RNA稳定性产生影响。将LINE-1和SLFN14质粒(1 mg)共转染HEK293T细胞24 h后放线菌素D(5 µg/mL)处理,于0、0.5、1、2、4、8 h后收集细胞,提取细胞总RNA,利用Real-time PCR对LINE-1 RNA进行检测。结果显示,SLFN14对于LINE-1 RNA降解有作用(图4C),这表明SLFN14可以在转录后水平影响LINE-1 RNA的降解。
2.5 SLFN14通过抑制LINE-1 5ʹ-UTR的内部启动子来抑制LINE-1 RNA的产生
SLFN14是否也会在LINE-1转录前水平发挥作用呢?LINE-1的5ʹ-UTR包含具有转录启动功能的启动子,这对于LINE-1的转录至关重要。为确定SLFN14是否影响LINE-1启动子活性,将LINE-1 5ʹ-UTR启动子序列克隆至pGL3-basic启动子活性报告质粒(图5A),该质粒的多克隆位点下游是一个缺少启动子序列的荧光素酶(luciferase)序列,只有在其上游插入具有启动子活性的序列,才能被激活进而表达出荧光素酶,而后加入相应底物,便可以通过酶标仪测定生物荧光变化,反映基因的表达情况。在排除SLFN14对CMV强启动子影响(图5B)后,结果显示,野生型SLFN14会极大程度地抑制LINE-1 5ʹ-UTR启动子活性(图5C),而SLFN14 dN3则无抑制作用(图5D),这表明SLFN14也可以在转录前水平抑制了LINE-1 5ʹ启动子活性从而抑制LINE-1 RNA表达,最终影响LINE-1逆转录转座。
3 讨论
近年来对SLFN家族成员,特别是SLFN14的研究较少,其功能还没有被了解完全。迄今为止对SLFN14唯一的功能描述是称其为一真正的哺乳动物内切核糖核酸酶,而目前对SLFN14病理性研究主要集中于血液病方向,SLFN14是最近发现的与遗传性血小板减少相关的基因之一,研究认为SLFN14可能参与了血小板形成和成熟过程及核糖体的降解[29~32]。此外,有研究表明在流感病毒感染时SLFN14表达于细胞核,并对流感病毒的核蛋白(nuclearprotein, NP)表达进行限制[25],这提示SLFN14可能与NP相互作用。对此,本课题组也对SLFN14与LINE-1核蛋白ORF1p的相互作用进行探究,但却没有发现两者存在相互作用(结果未显示)。这提示SLFN14抗LINE-1转座机制可能与其抗流感感染机制不同,SLFN14是通过间接作用影响LINE-1 ORF1p的表达。
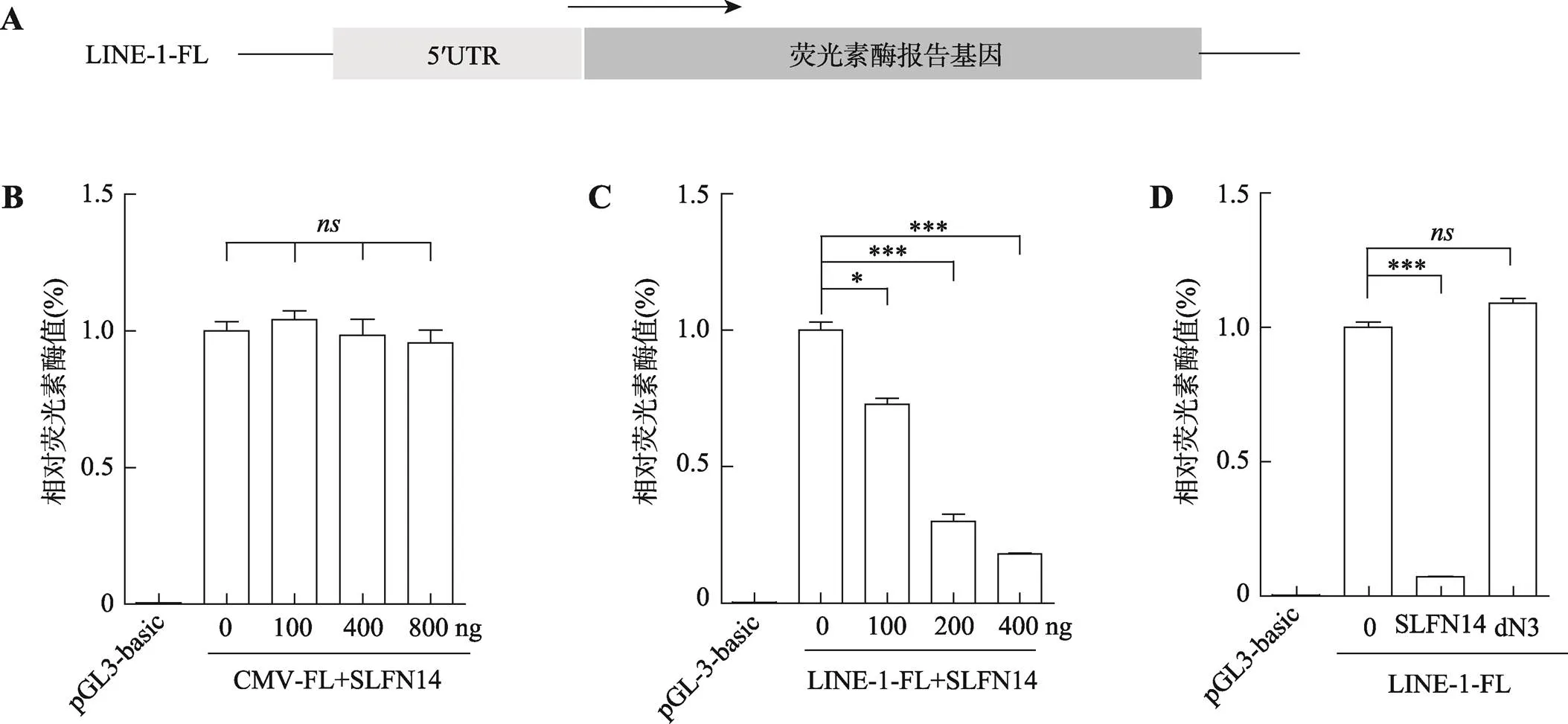
图5 SLFN14能够通过抑制LINE-1 5ʹ-UTR启动子活性来抑制LINE-1转座
A:用于检测LINE-1 5ʹ-UTR活性的LINE-1-FL质粒示意图。B:Luciferase启动子活性报告系统检测转染野生型SLFN14对CMV启动子活性的影响。C:Luciferase启动子活性报告系统检测转染野生型SLFN14后对LINE-1 5ʹ-UTR内部启动子活性的影响。D:Luciferase启动子活性报告系统检测转染SLFN14截短体dN3对LINE-1 5ʹ-UTR内部启动子活性的影响。*:< 0.05,***:< 0.001,:无显著性差异。
本实验室对SLFN14的序列进行了分析,经蛋白亚细胞定位预测工具网站(https://www.genscript. com/psort.html)比对,SLFN14具有核定位序列,预测其核定位序列为PRVKKLH。通过免疫荧光实验发现,野生型SLFN14主要定位于细胞质中。对此,Fletcher等[32]同样证实了这一点,即SLFN14主要定位于细胞质当中,在细胞核中仅有少量点状分布。然而,SLFN14截短体的细胞分布却发生了显著变化,丧失抗LINE-1活性的截短体dN3在细胞质、细胞核中弥散分布。因此推测一旦SLFN14大量停留在细胞核中,便会丧失抗LINE-1活性。这表明正确的细胞分布对于SLFN14的抗LINE-1活性至关重要,细胞分布的改变会对其活性产生负面影响。
通过The Human Protein Atlas数据库(https:// www.proteinatlas.org/)分析SLFN14在细胞中蛋白表达水平低,且经验证实验室常见细胞系无内源SLFN14蛋白表达,因此没有对内源SLFN14的抗LINE-1转座活性进行进一步验证,具有一定的局限性。同时,SLFN14更详细的抗LINE-1转座作用机制,特别是与SLFN14通过何种方式影响LINE-1 5ʹ-UTR的相关分子机制研究还有待进一步研究。
综上所述,本研究发现SLFN14可以抑制LINE-1的转座活性,并将其活性中心定位于N端的核糖核酸内切酶及核糖体结合结构域上,此部分的缺失会导致SLFN14细胞定位发生变化,从而使SLFN14丧失抗LINE-1活性;进一步实验证实SLFN14能够在转录前通过抑制LINE-1 5ʹ启动子活性从而降低LINE-1 RNA产生,也能在转录后加速LINE-1 RNA的降解,随后减少LINE-1 ORF1p及cDNA的表达,最终抑制了LINE-1的转座。
[1] Lander ES, Linton LM, Birren B, Nusbaum C, Zody MC, Baldwin J, Devon K, Dewar K, Doyle M, FitzHugh W, Funke R, Gage D, Harris K, Heaford A, Howland J, Kann L, Lehoczky J, LeVine R, McEwan P, McKernan K, Meldrim J, Mesirov JP, Miranda C, Morris W, Naylor J, Raymond C, Rosetti M, Santos R, Sheridan A, Sougnez C, Stange-Thomann Y, Stojanovic N, Subramanian A, Wyman D, Rogers J, Sulston J, Ainscough R, Beck S, Bentley D, Burton J, Clee C, Carter N, Coulson A, Deadman R, Deloukas P, Dunham A, Dunham I, Durbin R, French L, Grafham D, Gregory S, Hubbard T, Humphray S, Hunt A, Jones M, Lloyd C, McMurray A, Matthews L, Mercer S, Milne S, Mullikin JC, Mungall A, Plumb R, Ross M, Shownkeen R, Sims S, Waterston RH, Wilson RK, Hillier LW, McPherson JD, Marra MA, Mardis ER, Fulton LA, Chinwalla AT, Pepin KH, Gish WR, Chissoe SL, Wendl MC, Delehaunty KD, Miner TL, Delehaunty A, Kramer JB, Cook LL, Fulton RS, Johnson DL, Minx PJ, Clifton SW, Hawkins T, Branscomb E, Predki P, Richardson P, Wenning S, Slezak T, Doggett N, Cheng JF, Olsen A, Lucas S, Elkin C, Uberbacher E, Frazier M, Gibbs RA, Muzny DM, Scherer SE, Bouck JB, Sodergren EJ, Worley KC, Rives CM, Gorrell JH, Metzker ML, Naylor SL, Kucherlapati RS, Nelson DL, Weinstock GM, Sakaki Y, Fujiyama A, Hattori M, Yada T, Toyoda A, Itoh T, Kawagoe C, Watanabe H, Totoki Y, Taylor T, Weissenbach J, Heilig R, Saurin W, Artiguenave F, Brottier P, Bruls T, Pelletier E, Robert C, Wincker P, Smith DR, Doucette-Stamm L, Rubenfield M, Weinstock K, Lee HM, Dubois J, Rosenthal A, Platzer M, Nyakatura G, Taudien S, Rump A, Yang H, Yu J, Wang J, Huang G, Gu J, Hood L, Rowen L, Madan A, Qin S, Davis RW, Federspiel NA, Abola AP, Proctor MJ, Myers RM, Schmutz J, Dickson M, Grimwood J, Cox DR, Olson MV, Kaul R, Raymond C, Shimizu N, Kawasaki K, Minoshima S, Evans GA, Athanasiou M, Schultz R, Roe BA, Chen F, Pan H, Ramser J, Lehrach H, Reinhardt R, McCombie WR, de la Bastide M, Dedhia N, Blocker H, Hornischer K, Nordsiek G, Agarwala R, Aravind L, Bailey JA, Bateman A, Batzoglou S, Birney E, Bork P, Brown DG, Burge CB, Cerutti L, Chen HC, Church D, Clamp M, Copley RR, Doerks T, Eddy SR, Eichler EE, Furey TS, Galagan J, Gilbert JG, Harmon C, Hayashizaki Y, Haussler D, Hermjakob H, Hokamp K, Jang W, Johnson LS, Jones TA, Kasif S, Kaspryzk A, Kennedy S, Kent WJ, Kitts P, Koonin EV, Korf I, Kulp D, Lancet D, Lowe TM, McLysaght A, Mikkelsen T, Moran JV, Mulder N, Pollara VJ, Ponting CP, Schuler G, Schultz J, Slater G, Smit AF, Stupka E, Szustakowki J, Thierry-Mieg D, Thierry-Mieg J, Wagner L, Wallis J, Wheeler R, Williams A, Wolf YI, Wolfe KH, Yang SP, Yeh RF, Collins F, Guyer MS, Peterson J, Felsenfeld A, Wetterstrand KA, Patrinos A, Morgan MJ, de Jong P, Catanese JJ, Osoegawa K, Shizuya H, Choi S, Chen YJ, Szustakowki J, International Human Genome Sequencing Consortium. Initial sequencing and analysis of the human genome., 2001, 409(6822): 860–921.
[2] Rodić N, Burns KH. Long interspersed element-1 (LINE-1): passenger or driver in human neoplasms?, 2013, 9(3): e1003402.
[3] Leibold DM, Swergold GD, Singer MF, Thayer RE, Dombroski BA, Fanning TG. Translation of LINE-1 DNA elements in vitro and in human cells., 1990, 87(18): 6990–6994.
[4] Martin SL. Nucleic acid chaperone properties of ORF1p from the non-LTR retrotransposon, LINE-1., 2010, 7(6): 706–711.
[5] Piskareva O, Ernst C, Higgins N, Schmatchenko V. The carboxy-terminal segment of the human LINE-1 ORF2 protein is involved in RNA binding., 2013, 3: 433–437.
[6] Christian CM, deHaro D, Kines KJ, Sokolowski M, Belancio VP. Identification of L1 ORF2p sequence important to retrotransposition using Bipartile Alu retrotransposition (BAR)., 2016, 44(10): 4818–4834.
[7] Ye ZJ, Liu QP, Cen S, Li XY. The function of LINE-1-encoded reverse transcriptase in tumorigenesis., 2017, 39(5): 368–376.叶仲杰, 刘启鹏, 岑山, 李晓宇. LINE-1编码的逆转录酶在肿瘤形成过程中的作用. 遗传, 2017, 39(5): 368– 376.
[8] Duan XC, Jin X, Xie Y, Jiao N, Liu J, Wang XY, Lv ZJ. Different effects on reporter gene expression by distinct L1-ORF2 segements., 2009, 31(1): 50– 56.段肖翠, 靳霞, 谢英, 焦宁, 刘静, 王晓燕, 吕占军. L1-ORF2不同片段对报告基因表达产生不同影响. 遗传, 2009, 31(1): 50–56.
[9] Goodier JL, Kazazian HH Jr. Retrotransposons revisited: the restraint and rehabilitation of parasites., 2008, 135(1): 23–35.
[10] Babushok DV, Kazazian HH Jr. Progress in understanding the biology of the human mutagen LINE-1., 2007, 28(6): 527–539.
[11] Ostertag EM, Kazazian HH Jr. Biology of mammalian L1 retrotransposons., 2001, 35: 501–538.
[12] Crowther PJ, Doherty JP, Linsenmeyer ME, Williamson MR, Woodcock DM. Revised genomic consensus for the hypermethylated CpG island region of the human L1 transposon and integration sites of full length L1 elements from recombinant clones made using methylation-tolerant host strains., 1991, 19(9): 2395–2401.
[13] Thayer RE, Singer MF, Fanning TG. Undermethylation of specific LINE-1 sequences in human cells producing a LINE-1-encoded protein., 1993, 133(2): 273–277.
[14] Woodcock DM, Lawler CB, Linsenmeyer ME, Doherty JP, Warren WD. Asymmetric methylation in the hypermethylated CpG promoter region of the human L1 retrotransposon., 1997, 272(12): 7810–7816.
[15] Hata K, Sakaki Y. Identification of critical CpG sites for repression of L1 transcription by DNA methylation., 1997, 189(2): 227–234.
[16] Jachowicz JW, Bing X, Pontabry J, Bošković A, Rando OJ, Torres-Padilla ME. LINE-1 activation after fertilization regulates global chromatin accessibility in the early mouse embryo., 2017, 49(10): 1502–1510.
[17] Rodic N. LINE-1 activity and regulation in cancer., 2018, 23: 1680–1686.
[18] Liu Q, Wang JH, Li XY, Cen S. The connection between LINE-1 retrotransposition and human tumorigenesis., 2016, 38(2): 93–102.刘茜, 王瑾晖, 李晓宇, 岑山. 逆转录转座子LINE-1与肿瘤的发生和发展. 遗传, 2016, 38(2): 93–102.
[19] Bodak M, Yu J, Ciaudo C. Regulation of LINE-1 in mammals., 2014, 5(5): 409–428.
[20] Wang XY, Zhang Y, Yang N, Cheng H, Sun YJ. DNMT3a mediates paclitaxel-induced abnormal expression of LINE-1 by increasing the intragenic methylation., 2020, 42(1): 100–111.王昕源, 张雨, 杨楠, 程禾, 孙玉洁. DNMT3a通过提升基因内部甲基化介导紫杉醇诱导的LINE-1异常表达. 遗传, 2020, 42(1):100–111.
[21] Goodier JL. Restricting retrotransposons: a review., 2016, 7: 16.
[22] Schwarz DA, Katayama CD, Hedrick SM. Schlafen, a new family of growth regulatory genes that affect thymocyte development., 1998, 9(5): 657–668.
[23] Liu FR, Zhou PT, Wang Q, Zhang MC, Li D. The Schlafen family: complex roles in different cell types and virus replication., 2018, 42(1): 2–8.
[24] Li MQ, Kao E, Gao X, Sandig H, Limmer K, Pavon- Eternod M, Jones TE, Landry S, Pan T, Weitzman MD, David M. Codon-usage-based inhibition of HIV protein synthesis by human schlafen 11., 2012, 491(7422): 125–128.
[25] Seong RK, Seo SW, Kim JA, Fletcher SJ, Morgan NV, Kumar M, Choi YK, Shin OS. Schlafen 14 (SLFN14) is a novel antiviral factor involved in the control of viral replication., 2017, 222(11): 979–988.
[26] Esnault C, Maestre J, Heidmann T. Human LINE retrotransposons generate processed pseudogenes., 2000, 24(4): 363–367.
[27] Kopera HC, Flasch DA, Nakamura M, Miyoshi T, Doucet AJ, Moran JV. LEAP: L1 element amplification protocol., 2016, 1400: 339–355.
[28] Pisareva VP, Muslimov IA, Tcherepanov A, Pisarev AV. Characterization of novel ribosome-associated endoribonuclease SLFN14 from rabbit reticulocytes., 2015, 54(21): 3286–3301.
[29] Stapley RJ, Pisareva VP, Pisarev AV, Morgan NV. SLFN14 gene mutations associated with bleeding., 2020, 31(3): 407–410.
[30] Marconi C, Di Buduo CA, Barozzi S, Palombo F, Pardini S, Zaninetti C, Pippucci T, Noris P, Balduini A, Seri M, Pecci A. SLFN14-related thrombocytopenia: identification within a large series of patients with inherited thrombocytopenia., 2016, 115(5): 1076–1079.
[31] Fletcher SJ, Johnson B, Lowe GC, Bem D, Drake S, Lordkipanidzé M, Guiú IS, Dawood B, Rivera J, Simpson MA, Daly ME, Motwani J, Collins PW, Watson SP, Morgan NV. SLFN14 mutations underlie thrombocytopenia with excessive bleeding and platelet secretion defects., 2015, 125(9): 3600–3605.
[32] Fletcher SJ, Pisareva VP, Khan AO, Tcherepanov A, Morgan NV, Pisarev AV. Role of the novel endoribonuclease SLFN14 and its disease-causing mutations in ribosomal degradation., 2018, 24(7): 939–949.
SLFN14 inhibits LINE-1 transposition activity
Yang Mao, Jiwei Ding, Minshu Chen, Shan Cen, Xiaoyu Li
Long interspersed nuclear element-1 (LINE-1) is the only active autonomous transposon in the human genome. Its transposition frequently induces host genome instability, leading to a variety of genetic diseases, including cancers. The host factors play important roles in inhibiting LINE-1 retrotransposition. As an important component of the immune system, the host factor SLFN14 has antiviral activity. Our laboratory shows that SLFN14 possesses potent inhibitory activity against LINE-1 retrotransposition. To explore the potential mechanism of SLFN14 inhibition, we analyzed its effects on transcription, translation, reverse transcription and insertion in the LINE-1 replication cycle. We confirmed that SLFN14 could suppress the LINE-1 mRNA level by affecting its transcription and degradation, thereby diminishing the protein and cDNA levels of LINE-1, which eventually block the LINE-1 retrotransposition. Further, by mapping the active domains of SLFN14, we found its inhibitory activity on LINE-1 being closely related to its endoribonuclease and ribosome binding domains. These results demonstrate the mechanism of SLFN14 in regulating LINE-1 replication, which further provide new insights for improving the regulation network of host factors for controlling genomic instability caused by LINE-1 replication.
transposition; retrotransposon; LINE-1; SLFN14; 5 ʹ-UTR internal promotor region
2020-04-26;
2020-05-22
国家科技重大专项(编号:2018ZX10301408-004)和中国医学科学院医学与健康创新工程项目(编号:CIFMS 2016-I2M-2-002)资助[Supported by the National Science and Technology Major Project of the Ministry of Science and Technology of China (No. 2018ZX10301408-004)] and Chinese Academy of Medical Sciences Innovation Fund for Medical Sciences (No. CIFMS 2016-I2M- 2-002)]
毛洋,在读硕士研究生,专业方向:宿主因子对转座子调控机制研究。E-mail: yangmao-MY@outlook.com
李晓宇,博士,副研究员,研究方向:病毒学。E-mail: xiaoyulik@hotmail.com
岑山,博士,研究员,研究方向:病毒学。E-mail: shancen@imb.pumc.edu.cn
10.16288/j.yczz.20-081
2020/5/26 10:53:37
URI: http://kns.cnki.net/kcms/detail/11.1913.R.20200525.1400.002.html
(责任编委: 宋旭)
