小麦‘Fielder’ ω-醇溶蛋白基因及启动子的克隆与序列分析
2020-07-21郭丹丹李保云
郭丹丹 李保云
(中国农业大学 农学院/北京市作物遗传改良重点实验室/教育部作物杂种优势研究与利用重点实验室,北京 100193)
小麦是世界上重要的三大粮食作物之一。小麦具有面筋蛋白,所以能够被加工成面条、面包、馒头和饼干等各种面食品。面筋蛋白主要由单体形式的醇溶蛋白和多聚体形式的谷蛋白组成。根据在酸性聚丙烯酰胺凝胶电泳(Acid polyacrylamide-gel electrophoresis,A-PAGE)中迁移率的不同,醇溶蛋白可分为α-、β-、γ-和ω-醇溶蛋白4种类型,其中α-、β-、γ-醇溶蛋白中脯氨酸、苯丙氨酸和谷氨酰胺的含量较少,但是含硫氨基酸较多,因此被称为富硫醇溶蛋白,其中的半胱氨酸是维持单肽及肽链间二硫键的重要因素;而ω-醇溶蛋白通常不含半胱氨酸,无法参与链内或链间二硫键的形成,多数只含1个甲硫氨酸残基,因此被称为贫硫醇溶蛋白,有少数ω-醇溶蛋白含有奇数个半胱氨酸残基,能够参与谷蛋白聚合体的形成,导致面粉的品质变劣[1-3]。醇溶蛋白含有能够诱发麸质不耐受人群乳糜泻(Celiac disease,CD)疾病的肽段,主要类型是α-、β-和γ-醇溶蛋白,在ω-醇溶蛋白中也含有部分肽段[4]。通过RNAi介导使醇溶蛋白含量降低或沉默,能够在不影响总蛋白和淀粉含量的前提下达到降低CD毒性的目的[5]。因此克隆ω-醇溶蛋白基因对小麦品质的遗传改良具有重要意义。
ω-醇溶蛋白基因的基本结构,包括信号肽、N末端非重复区、占肽段90%~96%的中间重复区及C末端非重复区4部分[6]。六倍体小麦可能含有 15~18 个ω-醇溶蛋白基因[7],编码基因位于第1部分同源染色体短臂上[8],由1A、1D染色体基因编码的蛋白在A-PAGE上的迁移率较慢,称为ω1,2-醇溶蛋白(46~58 ku),推导的氨基酸序列N末端前3个通常为ARE/Q或KEL;由1B染色体基因编码的蛋白在A-PAGE上的迁移率较快,称为ω5-醇溶蛋白(55~65 ku),N末端前3个氨基酸为SRL[9-10]。ω1,2-醇溶蛋白在中间重复区的重复单元类型与ω5-醇溶蛋白有明显区别,这种差异也表现在氨基酸组成上,ω1,2-醇溶蛋白的谷氨酰胺与脯氨酸残基个数比为4∶3,而ω5-醇溶蛋白的比值为5∶2[11]。
ω-醇溶蛋白基因最早被克隆,是由于与γ-醇溶蛋白具有交叉杂交的性质,才会在对γ-醇溶蛋白基因进行克隆时被鉴定到[12]。利用电泳或RP-UPLC等方法对醇溶蛋白分离后回收,再进行N末端测序或质谱鉴定也鉴定到一些醇溶蛋白[13-14]。通过免疫化学方法制备特定醇溶蛋白的单克隆抗体,对ω-醇溶蛋白的分离和鉴定也发挥了重要作用[14]。通过基因组水平、转录水平和蛋白水平相结合的方法在二倍体祖先种乌拉尔图小麦中鉴定到2个ω-醇溶蛋白基因及其蛋白[4]。同样,利用三代全长转录组测序技术结合质谱技术,也鉴定到六倍体普通小麦品种‘小偃81’中有5个能够表达的ω-醇溶蛋白[15];通过将‘中国春’基因组进行De Novo BioNano组装,结合RNA-seq数据同样获得19条ω-醇溶蛋白基因的序列信息[16]。目前对小麦高分子量谷蛋白亚基(High molecular weight glutenin subunit, HMW-GS)和低分子量谷蛋白亚基(Low molecular weight glutenin subunit, LMW-GS)基因的调控研究已经较为深入[17-20]。但是,ω-醇溶蛋白基因家族庞大,相似性较高,假基因比例高,基因中间重复区含有较多的重复单元,GC含量较高,这些因素导致相关研究更加困难。已有研究多数集中在区分不同ω-醇溶蛋白及其基因的差异,对不同ω-醇溶蛋白基因启动子的差异比较研究报道较少,本研究以春小麦‘Fielder’为材料,通过PCR和克隆测序法分离ω-醇溶蛋白基因,分析编码区和启动子序列差异,旨在探明小麦中不同ω-醇溶蛋白基因的启动子序列差异,以期为ω-醇溶蛋白基因的转录调控机理研究奠定基础。
1 材料与方法
1.1 试验材料
本试验所用六倍体普通小麦(TriticumaestivumL.) 品种‘Fielder’和‘中国春’(CS)均由本实验室保存。
1.2 试验方法
1.2.1蛋白质提取
取1粒小麦种子充分碾碎,置于1.5 mL离心管中,加入800 μL 75%乙醇,室温震荡过夜,提取醇溶蛋白。
1.2.2A-PAGE的制备及电泳
采用FeSO4-Vc-H2O2催化系统制备A-PAGE[21],500 V稳压电泳3.5 h,对醇溶蛋白进行分离。
1.2.3DNA提取
将小麦种子播种在培养皿上,暗培养1周左右,取4叶1心期的叶片,用CTAB法提取基因组DNA。
1.2.4引物设计及扩增
根据已经公布的‘CS’ω-醇溶蛋白基因上下游序列[16],分别设计能够扩增不同基因组上的ω-醇溶蛋白基因及启动子的引物,详见表1。
其中上游引物位于ω-醇溶蛋白基因起始密码子上游1 kb左右;下游引物位于终止密码子下游100 bp左右。
采用大连宝生物(Takara)公司的Tks GflexTMDNA Polymerase高保真酶,按说明书进行PCR扩增。

表1 ω-醇溶蛋白基因编码区和启动子克隆引物及测序引物Table 1 The ω-gliadin genes cloning primers andsequencing primers
1.2.5目的片段回收与连接转化
将扩增产物在1%琼脂糖凝胶上进行分离,用琼脂糖凝胶回收试剂盒(北京天根生化科技有限公司)回收目的条带,与pEasy-Blunt Zero载体(北京全式金生物公司)连接,将连接产物转化大肠杆菌Trans-T1感受态细胞(北京全式金生物公司),涂布于含卡那霉素(Kan)的LB培养基上,37 ℃倒置培养过夜。
1.2.6重组克隆筛选
挑取一定数量的单克隆,用M13引物进行菌落PCR检测,将阳性克隆送北京擎科生物科技有限公司测序。首先用载体上的M13F和M13R引物进行测序,再根据ω-醇溶蛋白基因非重复区差异设计引物,对不同基因组的ω-醇溶蛋白基因进行测序,引物信息详见表1。
1.2.7序列分析
采用NCBI(https:∥www.ncbi.nlm.nih.gov/)和DNAMAN软件对测序结果进行比对、拼接及翻译。利用MegaX软件(https:∥www.megasoftware.net/)进行系统发生树的构建,启动子序列提交PlantCARE网站(http:∥bioinformatics.psb.ugent.be/webtools/plantcare/html/)进行motif分析。
2 结果与分析
2.1 小麦种子醇溶蛋白的A-PAGE
提取‘CS’和‘Fielder’种子的醇溶蛋白进行A-PAGE。从图1看出,2个材料的ω-醇溶蛋白谱带差异较大。‘CS’在ω1,2-醇溶蛋白区有2条带;在ω5-醇溶蛋白区有3条带,其中有2条带非常接近,共5条ω-醇溶蛋白条带。‘Fielder’在ω1,2-醇溶蛋白区有2条带,并且这2条带的迁移率与‘CS’非常接近;在ω5-醇溶蛋白区有5条带,其中有2条带非常接近,共7条ω-醇溶蛋白条带。
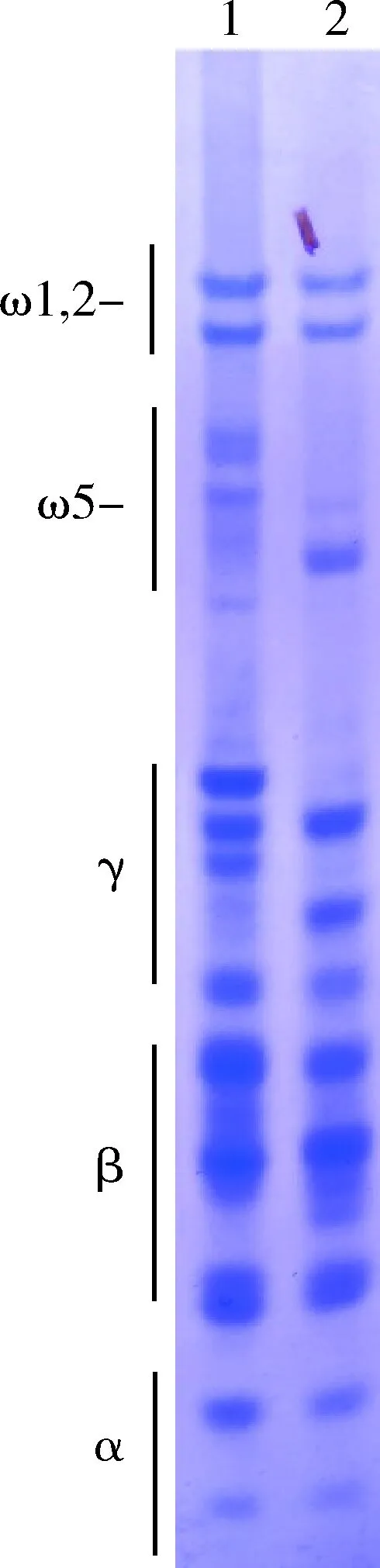
α-、β-、γ-和ω-分别表示按迁移率不同划分的4组醇溶蛋白类型;ω1,2-和ω5-分别为ω-醇溶蛋白的2种类型。1和2分别为‘Fielder’和‘CS’。α-, β-, γ- and ω- are the four types of wheat gliadin;ω1,2- and ω5- are the two types of ω-gliadin. Lane 1 and 2 are ‘Fielder’ and ‘CS’.图1 小麦种子醇溶蛋白的A-PAGEFig.1 The A-PAGE of wheat grain gliadin
2.2 小麦种子ω-醇溶蛋白基因及启动子克隆
利用3组ω-醇溶蛋白基因特异引物(表1),以‘Fielder’的基因组DNA为模板进行PCR扩增,得到约2 kb左右的目的条带(图2)。将目的条带回收后,连接T载体并转化至大肠杆菌中,挑选阳性单克隆进行序列测定,共获得11种序列。将这些序列在NCBI网站进行Blast,相似性较高的是ω-醇溶蛋白基因,进一步分析发现这些序列含有ω-醇溶蛋白基因的基本结构,即均为ω-醇溶蛋白基因及其启动子序列(GenBank登录号:MN441496~MN441506)。
这些基因序列可以分为2组,ARE/Q和SRL型ω-醇溶蛋白基因。编码区长度、推导的氨基酸数目以及重复单元种类和数量各不相同(表2)。
ARE/Q型ω-醇溶蛋白基因编码区长度范围在 972 ~1 158 bp,推导的氨基酸主要含有PQQPFP和PFPQQPQQ这2种类型的重复单元;SRL型ω-醇溶蛋白基因编码区的长度变异范围为1 303~1 419 bp,主要重复单元为FPQQQ和PQQQFP。

M,1 kb ladder,1、2和3分别为ωA、ωB和ωD引物的扩增产物。M is 1 kb ladder. Lane 1, 2 and 3 show the amplifications of primer pairs of ωA, ωB and ωD, respectively.图2 ω-醇溶蛋白基因扩增产物Fig.2 The amplification of ω-gliadin genes

表2 ω-醇溶蛋白基因及推导的氨基酸序列比较Table 2 The comparison of gene and deduced amino acid sequences of ω-gliadin
2.2.1ω-醇溶蛋白基因编码区分析
A和D基因组上的ω-醇溶蛋白基因相似性较高,从‘Fielder’中利用基因组特异性引物共扩增得到的ARE和ARQ型ω-醇溶蛋白基因序列分别有3种(表2),不同类型和数量的Indel是造成不同基因编码区长度变异的主要因素。其中MN441497和MN441505的编码区长度最长,为1 158 bp。在其余4种ARE/Q型基因序列中,共有5种类型的Indel,长度分别为15、18、24、48和123 bp(图3)。MN441503中含有123、48和15 bp 3种类型Indel;MN441504中含有18、48和15 bp 3种类型Indel;MN441496中只含有18 bp的Indel;MN441506中只含有24 bp的Indel。共有70处SNP位点分布于这6种ω-醇溶蛋白基因序列之间,其中22个SNP为同义突变,48个SNP为非同义突变。这些Indel碱基数均为3的倍数,SNP变异也没有引入额外的终止密码子,因此6种ARE/Q型ω-醇溶蛋白基因序列均含有完整编码框。不同长度和数量的Indel组合以及不同的SNP变异,使基因呈现多态性。其中MN441496和MN441497只有18 bp Indel的差异。
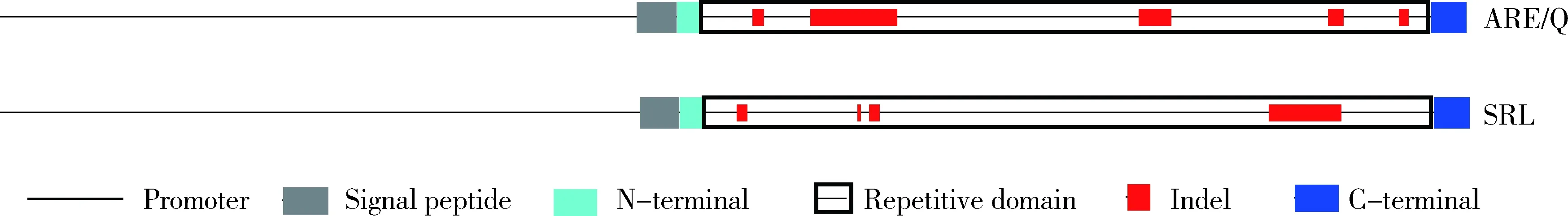
图3 ARE/Q型和SRL型ω-醇溶蛋白基因模式图Fig.3 The schematic diagram of ARE/Q and SRL type ω-gliadin genes
MN441499在135 bp处有1个18 bp的Indel,在151 bp处插入了1个碱基A,导致推导的氨基酸序列与醇溶蛋白相似性较低,推测其可能为移码突变的假基因。在另外4种SRL型ω-醇溶蛋白基因的中间重复区共发现了3种类型的Indel(图3)。MN441500和MN441502在303 bp处均有1个3 bp的Indel,MN441498、MN441499、MN441500和MN441501在相距12 bp后的位置有1个18 bp的Indel,MN441498、MN441499、MN441501和MN441502在相距552 bp后的位置有1个102 bp的Indel,这些Indel碱基数均是3的倍数,没有引起移码突变。在4种SRL型ω-醇溶蛋白基因中共发现27个SNP位点,其中有7个SNP为同义突变,17个为非同义突变。还有3个位点的SNP引入了提前终止密码子,造成推导的氨基酸序列变短,其中1个SNP位点位于MN441498序列ATG下游1 260 bp处,三联体密码子TAC突变为TAA;另外2个位点位于MN441501序列编码区274和 1 279 bp 处,编码脯氨酸的密码子CAG和CAA突变为终止密码子TAG和TAA。
2.2.2ω-醇溶蛋白基因的进化树分析
将克隆的ω-醇溶蛋白基因序列在NCBI的NR数据库中进行Blast,并与44条来源于不同小麦属具有完整编码框的ω-醇溶蛋白基因构建Neighbor-joining系统发生树。从图4可知,系统发生树被分成2个分支,6种ARE/Q型ω-醇溶蛋白基因主要与普通小麦(TriticumaestivumL.)、山羊草属(AegilopstauschiiL.)、乌拉尔图小麦(TriticumurartuL.)及圆锥小麦(TriticumturgidumL.)中的ω-醇溶蛋白基因聚类在1个大分支上,而4种SRL型ω-醇溶蛋白基因主要与普通小麦(TriticumaestivumL.)和二粒小麦(TriticumdicoccoidesL.)中的ω-醇溶蛋白基因聚在1个分支上。通过进化树分析可以看到ARE/Q型ω-醇溶蛋白基因在进化上与SRL型ω-醇溶蛋白基因相对独立。
2.2.3ω-醇溶蛋白基因启动子分析
ω-醇溶蛋白基因与小麦中其他储藏蛋白基因启动子区类似[17-18,22],ATG上游序列包含有许多保守的motif,其中ARE/Q型ω-醇溶蛋白基因启动子含有29种motif;SRL型ω-醇溶蛋白基因启动子含有27种motif(表3),相同的motif有19种,但是分布的位置不同,这些motif是SPA、MYB、DOF和B3等重要转录因子的识别序列。
在ARE/Q型ω-醇溶蛋白基因中,MN441496和MN441497、MN441503和MN441504以及MN441505和MN441506的启动子序列分别相同,SRL型ω-醇溶蛋白基因的5种启动子序列均不相同。ARE/Q和SRL型基因启动子序列差异较大(表3),但是存在保守的motif组合,比如P-box和N-motif组成的endosperm box只出现在ARE/Q型醇溶蛋白基因启动子序列的-300 bp处,但出现在SRL型醇溶蛋白基因启动子序列的-300 bp和 -600 bp 处。有些motif只特定出现在1种序列中,比如ARE/Q型ω-醇溶蛋白基因-137、-172、-389、-498、-499、-675、-946和-968 bp处的motif,SRL型-187、-367、-626和-933 bp处的motif。这些motif出现在不同基因启动子的不同位置,可能会对基因的表达产生不同的影响。
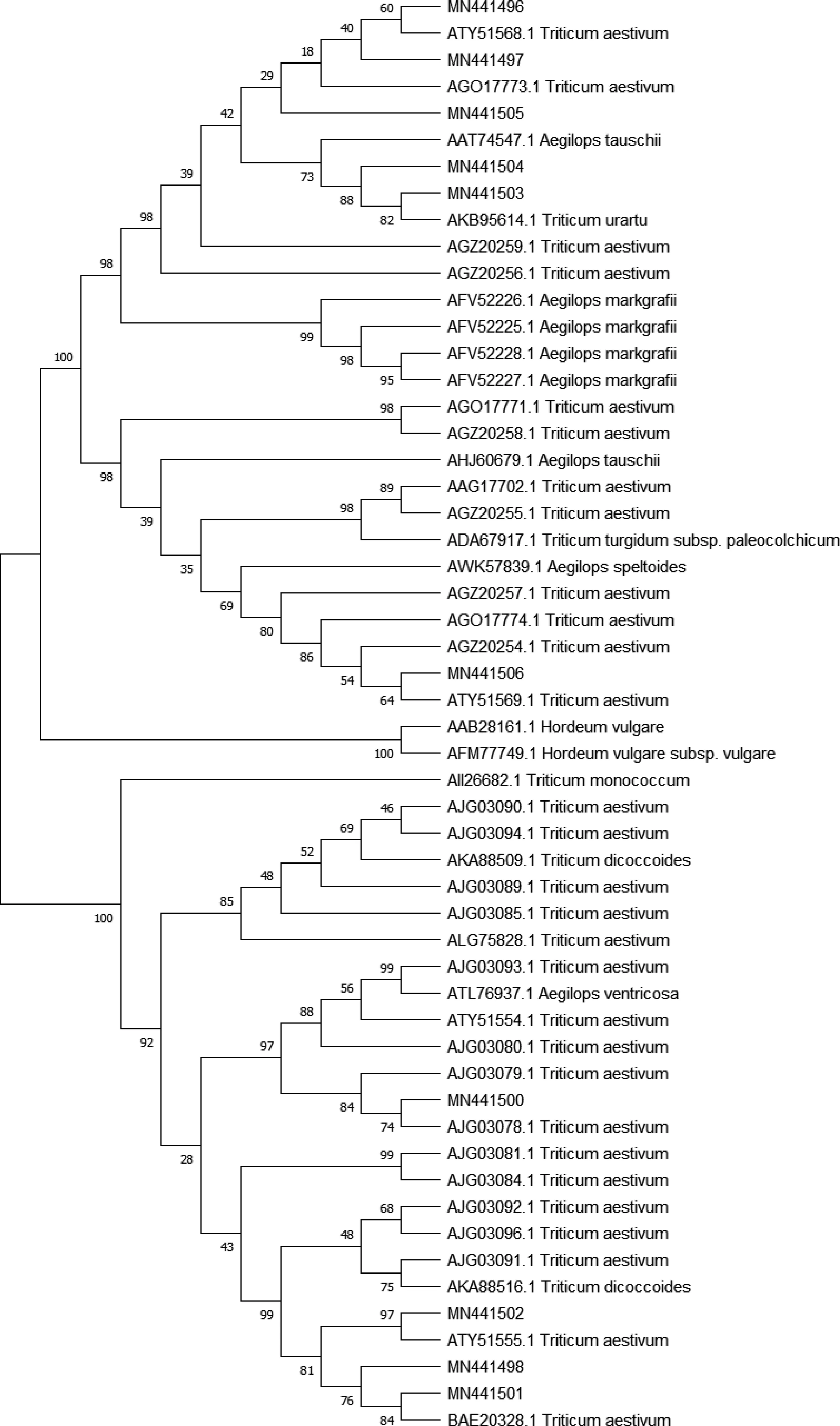
数字表示可信度。The numbers in the figure indicate the credibility.图4 克隆的ω-醇溶蛋白基因与非冗余ω-醇溶蛋白基因进化树分析Fig.4 Phylogenic tree analysis based on the cloned genes and non-redundant ω-gliadins

表3 ARE/Q和SRL型醇溶蛋白基因启动子区保守motif分布Table 3 The distribution of conserved motif in ARE/Q and SRL type ω-gliadin genes promoter region
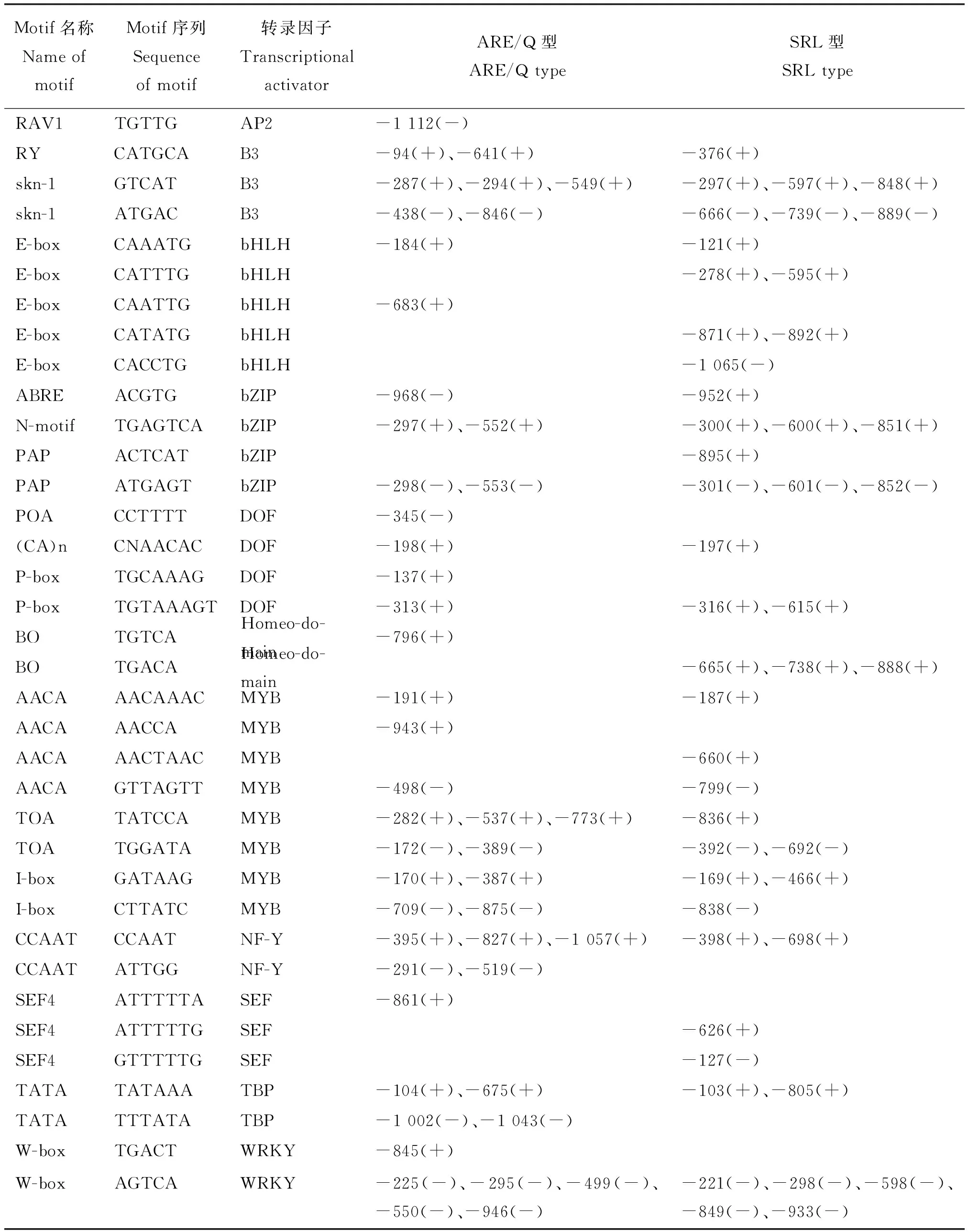
表3(续)
3 讨 论
不同小麦材料中的ω-醇溶蛋白基因拷贝数不同,研究表明,六倍体普通小麦中的ω-醇溶蛋白基因数量在15~18个[7]。根据重新组装的基因组信息、2-DE(双向蛋白电泳)和RNA-seq信息,‘CS’共有5种能够转录并且表达的ω-醇溶蛋白基因[16],因此在A-PAGE上可以看到5条清晰的ω-醇溶蛋白条带(图1)。‘Fielder’在A-PAGE上共有7条ω-醇溶蛋白条带,推测至少应有7种能够表达的ω-醇溶蛋白,因此根据‘CS’中ω-醇溶蛋白基因的上下游序列信息,对‘Fielder’中的ω-醇溶蛋白基因的编码区和启动子序列进行克隆,共得到了11条序列,其中1条为移码突变的假基因,2条含有提前终止密码子,8条具有完整编码框。由于ω-醇溶蛋白有限的水解位点,质谱分析相对困难[23],更为重要的是ω-醇溶蛋白基因等位变异广泛,但数据库中完整的基因数量较少,大部分预测的蛋白分子量较小,可能是丢失了一部分中间重复区[10]。本研究采用单克隆测序的方法,能够准确知道每条DNA序列的长度,并且通过拼接不同位置的测序结果,大大降低了缺失中间重复区的可能。这种通过简单的PCR进行克隆的方法,避免了2-DE和质谱等繁琐的试验过程,即可获得ω-醇溶蛋白基因序列,简单易行且成本较低。缺点是需要避开高GC含量的中间重复区设计出合适的引物进行测序,并且由于重复单元较多,需要设计多对引物对测序结果反复验证,避免拼接结果缺失部分重复单元,另外得到的基因序列并不能与A-PAGE上的条带一一对应。
含奇数个半胱氨酸的醇溶蛋白可能作为链内终止剂,参与谷蛋白大聚体的形成[3]。本研究克隆的ω-醇溶蛋白基因中,MN441506推导的氨基酸序列含有1个半胱氨酸和甲硫氨酸,MN441502在C末端含有1个半胱氨酸,这2个ω-醇溶蛋白可能通过分子间二硫键对谷蛋白大聚体的形成及稳定性产生影响,进而影响小麦品质性状。
Glu-1基因启动子中的顺式作用元件可以组成保守的顺式作用调控模块(conserved cis-regulatory modules,CCRM)对基因表达进行精确调控,并且表达量较高的x-型HMW-GS除了保守的CCRM外,还含有能够被R2R3-MYB转录因子识别的motif[16,22]。LMW-GS基因启动子也含有保守的非编码调控元件,大部分的s-和i-型LMW-GS基因呈现逐渐增加的表达模式,而m-型LMW-GS基因则呈先升高再降低的表达模式,不同基因表达模式的差异与启动子区motif的数量和分布密切相关[17]。本研究克隆的ω-醇溶蛋白基因启动子区也含有大量保守的motif,不同种类基因之间motif的数量和分布不同(表3),相同种类基因之间也在一些motif上有差异。其中由GCN4和P-box这2种motif组成的endosperm box对储藏蛋白基因的表达起到非常重要的作用,LMW-GS基因启动子区GCN4和P-box motif的数量和组合多态性使同一类型的基因呈现不同的表达模式[17]。在本研究克隆的ω-醇溶蛋白基因中也发现了类似的现象,ω1,2-醇溶蛋白基因启动子区含有这2种motif组成的3种组合类型,每种类型所含的motif种类和数量不同。ω1,2-醇溶蛋白基因启动子只含有1个典型的endosperm box,而ω5-醇溶蛋白基因则含有2个。这些motif的组合形式也可能会造成ω-醇溶蛋白基因之间的差异表达,有待进一步研究。
