玉米耐深播主效QTL qMES20-10 的精细定位及差异表达基因分析
2020-07-02任蒙蒙张红伟王建华王国英
任蒙蒙 张红伟 王建华 王国英 郑 军,*
1 中国农业科学院作物科学研究所, 北京100081; 2 中国农业大学农学院, 北京100193
干旱胁迫严重影响玉米生长发育, 是造成玉米减产的最重要非生物胁迫因素[1]。我国有大量玉米种植在干旱半干旱地区, 通过改善栽培措施如增加播种深度, 能够减轻干旱胁迫对玉米生长发育的影响[2]。对于普通的玉米品种, 增加播种深度会导致出苗率下降, 造成苗期缺苗而影响产量[3]。具有耐深播特性的玉米材料相对于普通玉米材料, 在深播条件下具有更长的中胚轴和更强的顶土能力, 能适应较深的播种条件, 其根系能从更深层的土壤吸收水分,有助于玉米应对干旱胁迫。因此, 研究玉米耐深播特性的遗传机制对于改良普通杂交种的耐深播特性具有重要的指导意义[4]。
目前对作物耐深播的研究主要集中在大麦、小麦和水稻等作物[5-7]。在深播条件下, 不同的作物通过延伸不同的组织将胚芽顶出土层。水稻、燕麦等作物主要延伸中胚轴和第一节间[8], 大麦、小麦等作物延伸其胚芽鞘和第一节间[9-10], 而玉米和高粱则主要延伸其中胚轴[11-12]。在水稻直播技术的推广中,需要培育耐深播的水稻品种以提高出苗率。Lu 等[13]用469 份水稻自交系进行全基因组关联分析, 以中胚轴和根的长度作为表型指标, 挖掘了23 个与耐深播相关的位点。Wu 等[14]分别在水培和5 cm 沙土中对270 份水稻自交系进行了深播表型鉴定, 并用重测序得到的基因型数据进行了关联分析, 共挖掘出16 个与耐深播相关的位点。Zhao 等[15]对621 个水稻品种进行深播表型分析, 发现中胚轴伸长受深层土壤覆盖诱导, 对田间耐深播能力具有重要意义, 同时利用关联分析的方法鉴定出了13 个耐深播相关的QTL, 并对其中两个主效基因OsML1和OsML2进行了验证。在玉米耐深播的研究中, 赵光武等[16]对12 个玉米自交系耐深播相关性状的分析表明, 中胚轴伸长是玉米种子顶土出苗的主要动力, 深播条件下的出苗率与玉米的中胚轴长度呈正相关, 因此也可以用深播条件下的中胚轴长度来评价玉米的耐深播能力。Liu 等[17]用玉米优良自交系B73 和Mo17组配的280 份DH 系(IBM Syn10)进行了深播发芽能力的QTL 定位, 共发现了32 个相关的QTL, 并在这些QTL 的定位区间内鉴别出6 个与深播发芽能力相关的候选基因。上述研究表明, 作物耐深播能力是由多个位点控制的数量性状, 需要通过QTL 定位的方法挖掘相关基因。
目前, 借助分子标记辅助选择, 利用高代回交群体构建近等基因系, 是精细定位QTL 并最终获得候选基因的常用方法[18]。近等基因系遗传背景相似,只在包含QTL 区段的染色体片段上存在差异, 在控制环境的情况下, 可认为表型的差异只是由于该染色体片段的不同引起, 从而能精确定位在该区段上的QTL。另外, 利用RNA-Seq 的方法, 分析近等基因系中基因差异表达的情况, 结合精细定位的结果,也是预测候选基因并对目标性状进行遗传解析的有效方法[19]。
本研究所用材料为3681-4 和X178, 其中3681-4为本实验室鉴定的一个具有耐深播特性的玉米自交系[20], X178 为玉米优良杂交种农大108 的母本。实验室前期已通过构建F2:3家系进行了耐深播性状的初定位, 在玉米10 号染色体长臂上发现了一个主效QTLqMES20-10, 并将其定位到了 SSR 标记umc1506 与bnlg1028 之间大约5.24 Mb 的区间[21]。在此基础上, 构建了高代回交群体, 对qMES20-10进一步精细定位, 并分析近等基因系中差异表达基因的情况, 为该位点候选基因的挖掘提供了材料和参考依据。
1 材料与方法
1.1 实验材料
亲本材料为玉米自交系3681-4 和X178 在20 cm播深下的中胚轴长度(简称为MES20)有极显著差异(图1-C)。实验室前期工作用X178 作为轮回亲本, 通过 umc1506 与 bnlg1028 两个分子标记筛选保留qMES20-10的杂合区段, 并对其他非目标QTL 进行背景控制, 构建了170 份BC3F3:4家系。
在此基础上利用分子标记辅助选择, 2015年冬季在海南三亚种植了目标区段为杂合基因型的BC3F3群体2600 株, 通过初定位区间的两端标记umc1506 和bnlg1028, 筛选出243 个交换单株, 将这些交换单株与X178 回交获得BC4F1群体。2016年夏季在北京顺义和2016年冬季在海南将BC4F1群体自交获得大约200 份BC4F2:3家系。
1.2 表型鉴定方法
采用20 cm 播种深度鉴定表型。首先在一个塑料盒(长60 cm, 宽40 cm, 高16 cm)中装上6 根PVC管(直径20 cm, 高24 cm), 在PVC 管的底部平铺4 cm 厚混匀的蛭石(蛭石与水的质量比为1∶1), 均匀点上种子后, 用蛭石填满PVC 管。然后将盒子密封, 黑暗处理。环境温度25℃, 相对湿度60% (图1-A)。播种14 d 后, 将幼苗取出, 用直尺测量幼苗的中胚轴长度(图1-B)。从BC3F3:4每个家系中随机选择30粒种子种到一个PVC 管中鉴定表型, 用表型的平均值代表该家系的表型值。对于从BC4F2:3家系中选取的用作后代测验的交换单株上的穗子, 每个单穗按照上述方法种植180 粒种子(6 个PVC 管)作为分离群体, 进行基因型和表型鉴定。
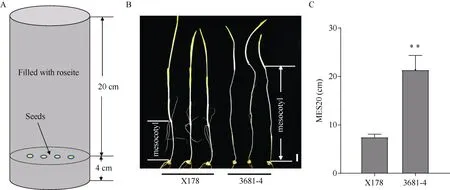
图1 表型鉴定方法和亲本材料表型Fig. 1 Phenotyping method and phenotype of the two parents
1.3 基因型鉴定方法
取玉米苗期幼嫩叶片, 用CTAB 法提取基因组DNA[23], 用于基因型鉴定。BC3F3:4家系全基因组的基因型鉴定采用了北京康普森生物技术有限公司定制的Maize iSlect6K 芯片, 共5179 个SNP 标记, 均匀覆盖了玉米全基因组[22]。精细定位群体的基因型鉴定采用SSR 分子标记或InDel 标记, 所用的SSR分子标记, 来源于MaizeGDB (http://www.maizegdb.org/), InDel 标记根据B73 和Mo17 基因组序列比对得到的InDel 位点进行设计[24]。引物由上海生工生物工程有限公司合成, 引物信息如表1。
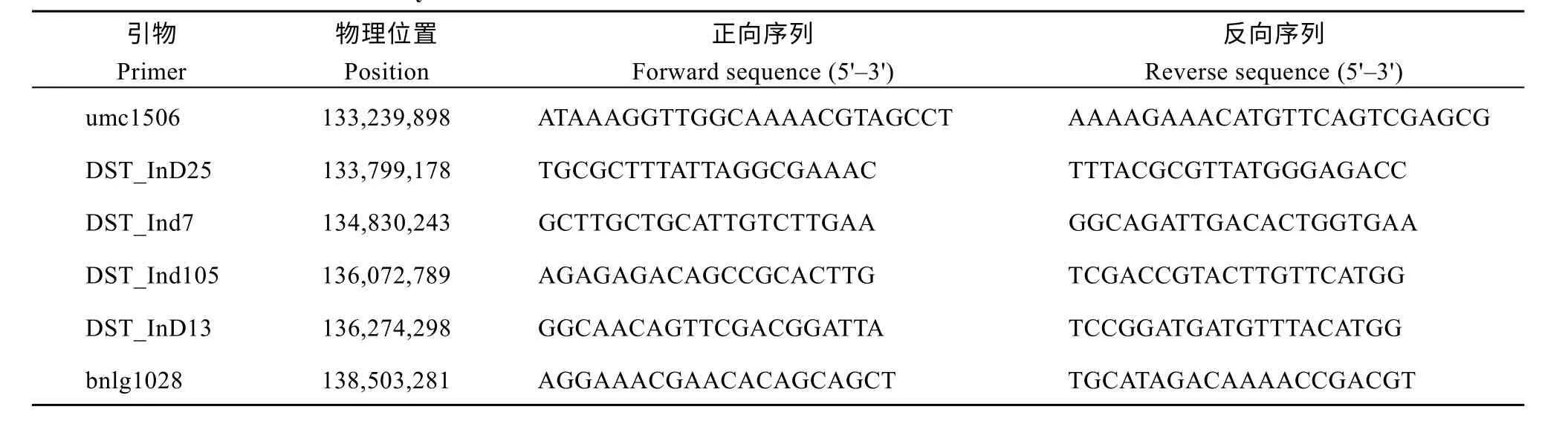
表1 本研究所用的引物Table 1 Primers used in this study
1.4 QTL 确证和精细定位
为了验证qMES20-10的真实性, 对 170 个BC3F3:4家系进行了表型和基因型鉴定, 表型2 次重复, 基因型用umc1506 和bnlg1028 两对标记检测,并用方差分析检测不同基因型家系之间的表型是否有差异。利用覆盖全基因组的芯片进一步检测了这些 BC3F3:4家 系 的 基 因 型, 用 QTL 定 位软 件IciMapping v4.0[25]对这些家系进行了全基因组的QTL 检测, 先用bin 模块去除冗余标记和偏分离标记, 再采用完备区间作图法(ICIM)进行QTL 检测。表型值为两次测量结果的平均值。实验群体设置为F2, LOD 阈值设置为2.5。方差分析采用R 语言中的anova()函数。
精细定位QTL 采用两种策略。一是AB-QTL[26]分析法, 从BC4F2:3群体中直接鉴定交换单株的表型,通过t测验比较交换单株与受体亲本之间的表型是否有差异, 从而判断QTL 的位置; 二是后代测验法[27],从BC4F2:3群体选取交换单株的穗子作为分离群体,单株鉴定基因型和表型, 比较分别带有3681-4 和X178 纯合基因型材料的表型是否有差异, 从而确定目标QTL 是否位于杂合区段。t测验采用SAS9.2 中的Proc TTEST。
1.5 RNA-Seq 和差异表达分析
从BC3F3:4家系中挑选目标QTL 区段分别为X178 纯合(NILX178)和3681-4 纯合(NIL3681-4)基因型的材料各3 份作为近等基因系, 再加上2 个亲本材料X178 和3681-4 (各3 次重复), 共8 份材料进行RNA-Seq 分析。每份材料按照上述表型鉴定的方法播种, 取幼苗中胚轴部分, 用 TRIol 法提取总RNA[28]。RNA-Seq 由北京贝瑞和康生物科技有限公司完成。
差异表达分析先用TopHat2[29]将测序得到的原始 Reads 锚定到 B73 参考基因组上, 再使用Cufflinks 软件的Cuffdiff[30]组件, 通过Mapped Reads 在基因上的位置信息, 对转录本和基因的表达水平进行定量。采用FPKM (fragments per kilobase of transcript per million fragments mapped)[31]作为衡量转录本或基因表达水平的指标。用DESeq2[32]进行样品组间的差异表达分析, 获得两个样品之间的差异表达基因集。在差异表达基因检测过程中, 将Fold Change≥2 且FDR < 0.01 作为筛选标准。其中,差异倍数(fold change)表示两样品(组)间表达量的比值。错误发现率(false discovery rate, FDR)是通过对差异显著性P值(P-value)校正得到的。基因富集分析使用在线分析软件 AgriGo (http://bioinfo.cau.edu.cn/agriGO/)[33]。
2 结果与分析
2.1 用BC3F3:4 家系确证qMES20-10
在170 个BC3F3:4家系中,qMES20-10的区间内X178 纯合、杂合、3681-4 纯合3 种基因型的家系分别有34、65、35 个, 符合1∶2∶1 的分离比(χ2测验:P= 0.935)。剩余的36 个家系在umc1506 与bnlg1028两个标记之间发生交换, 重组率为21.2%。3 种基因型材料的中胚轴长度平均值分别为7.62、8.52、9.87 cm。方差分析表明三者之间有极显著差异(图2-A)。
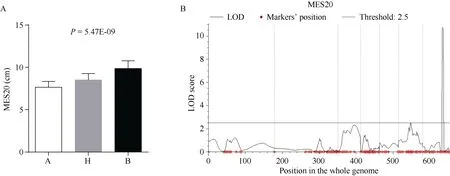
图2 耐深播主效QTL qMES20-10 确证Fig. 2 Validation of the major QTL qMES20-10 for deep-seeding tolerance
为了排除背景对后续定位的干扰, 进一步用玉米6 K 芯片鉴定了上述170 份BC3F3:4家系的基因型。从6K 芯片的5179 个SNP 标记中, 共筛选出1485个多态性SNP 位点, 覆盖了玉米的全基因组。用2次表型的平均值进行全基因组的QTL 检测, 发现只有10 号染色体上有一个主效QTL (图2-B), 位于SNP 标记PZE-110074955 和chr10.S_132532921 之间。2 个标记在B73 参考基因组(V2)的物理位置与该QTL 的初定位区间基本一致。在BC3F3:4家系中,该QTL 的LOD 值为11.36, 可解释的表型变异为38.66%。该位点的加性效应为0.82 cm, 显性效应为0.03 cm, 为典型的加性基因。通过以上两个实验,进一步确证了qMES20-10的真实性。
2.2 qMES20-10 的精细定位
利用组配的200 份BC4F2:3家系对qMES20-10进行精细定位, 通过初定位区间内的4 个分子标记DST_InD7、DST_InD105、DST_InD13、DST_InD25将这些BC4F2:3家系分成9 种重组类型, 分别对不同重组类型的家系进行表型鉴定, 检验每种重组类型的表型与受体亲本X178 是否有显著差异。如图3所示, 在DST_InD7 和DST_InD105 两个标记区段内含有3681-4 染色体片段的重组类型(R1-R4), 中胚轴长度显著高于受体亲本, 而含有X178 片段的重组类型(R5-R9)与受体亲本无显著差异。由此可将目标QTL 定位于DST_InD7 和DST_InD105 两个标记之间。这2 个标记在B73 参考基因组(V2)中的物理位置为10 号染色体133.3~136.0 Mb。
在上述BC4F2:3家系中, 结合分子标记辅助选择,选取在不同位置发生交换的交换单株, 利用其穗子作为分离群体进行后代测验。如图3 所示, 发现当DST_InD7 和DST_InD105两个标记之间的染色体片段为杂合状态时(R1, R5-R7), 3681-4 等位基因型植株的中胚轴长度显著大于X178 基因型的植株, 而当染色体其他位置的片段为杂合状态时(R2-R4),二者无显著差异。由此同样将目标 QTL 定位于DST_InD7 和DST_InD105 之间, 进一步验证了上述精细定位的结果(图4)。
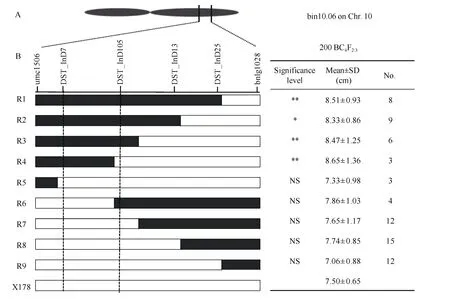
图3 耐深播主效位点qMES20-10 的精细定位Fig. 3 Fine mapping of the major QTL qMES20-10 for deep-seeding tolerance
2.3 近等基因系中差异表达基因分析
为了从转录组水平上解释两亲本之间以及近等基因系材料之间中胚轴长度的差异, 进而挖掘候选基因, 对近等基因系和亲本材料进行了RNA-Seq 测序。各样品的Reads 与参考基因组的比对效率从75.28%到80.45%不等, 大部分的Reads 都能比对到参考基因组上的唯一位置, 这部分Reads 所占比例从66.71%到76.59%不等。在两亲本X178 和3681-4之间共检测到8800 个差异表达基因, 接近玉米基因总量的1/4, 且这些基因在10 条染色体上大体分布均匀, 说明两个亲本在基因表达水平上的差异很大。而在BC3F3:4家系构建的近等基因系中只检测到115 个差异表达基因, 这些基因绝大部分分布在10号染色体, 表明经过连续的回交和自交后, 两个近等基因系之间基因表达的差异大大减少, 可能只在少数的染色体片段上有所差别。两组RNA-Seq 数据共同检测到的差异基因共73 个(图5)。
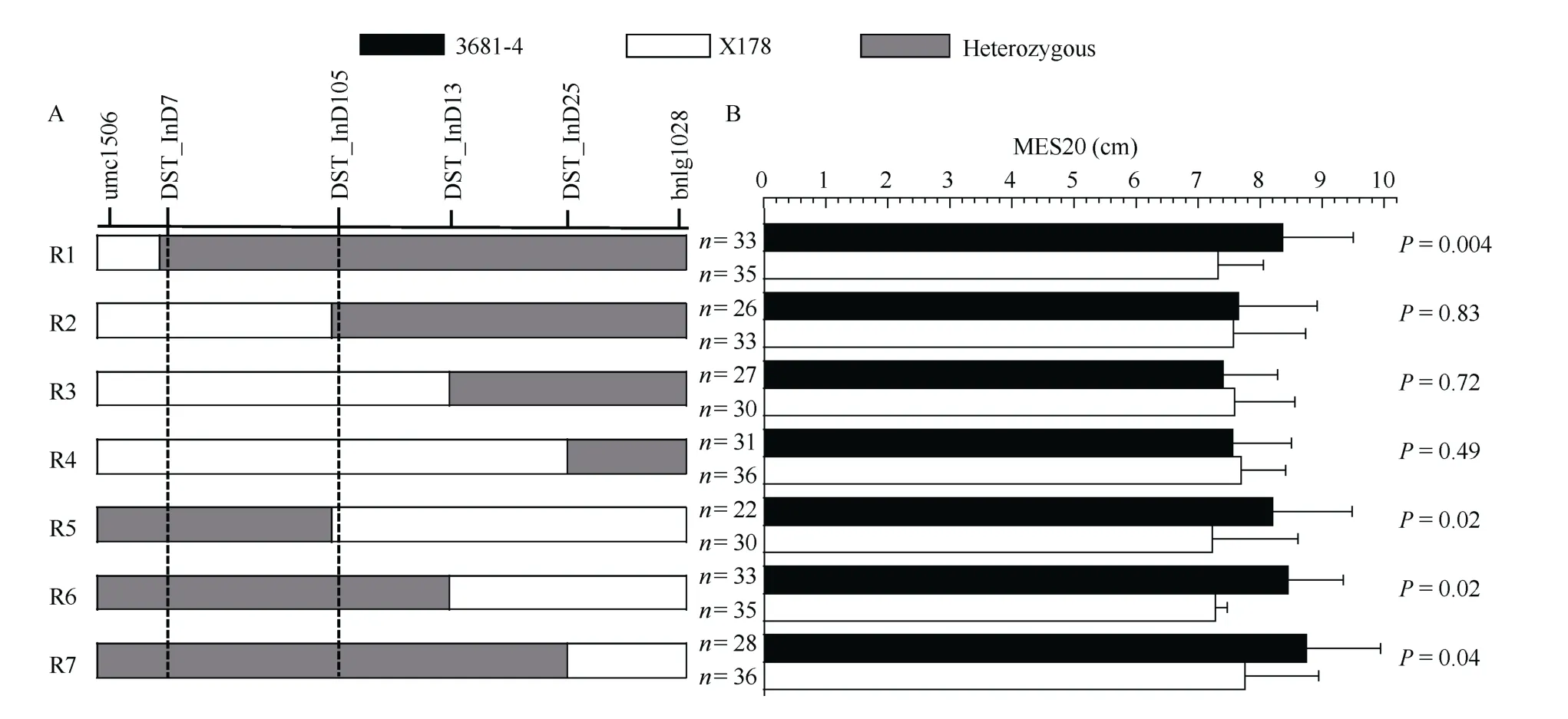
图4 后代测验验证定位区间Fig. 4 Validation of the mapping region using progeny test
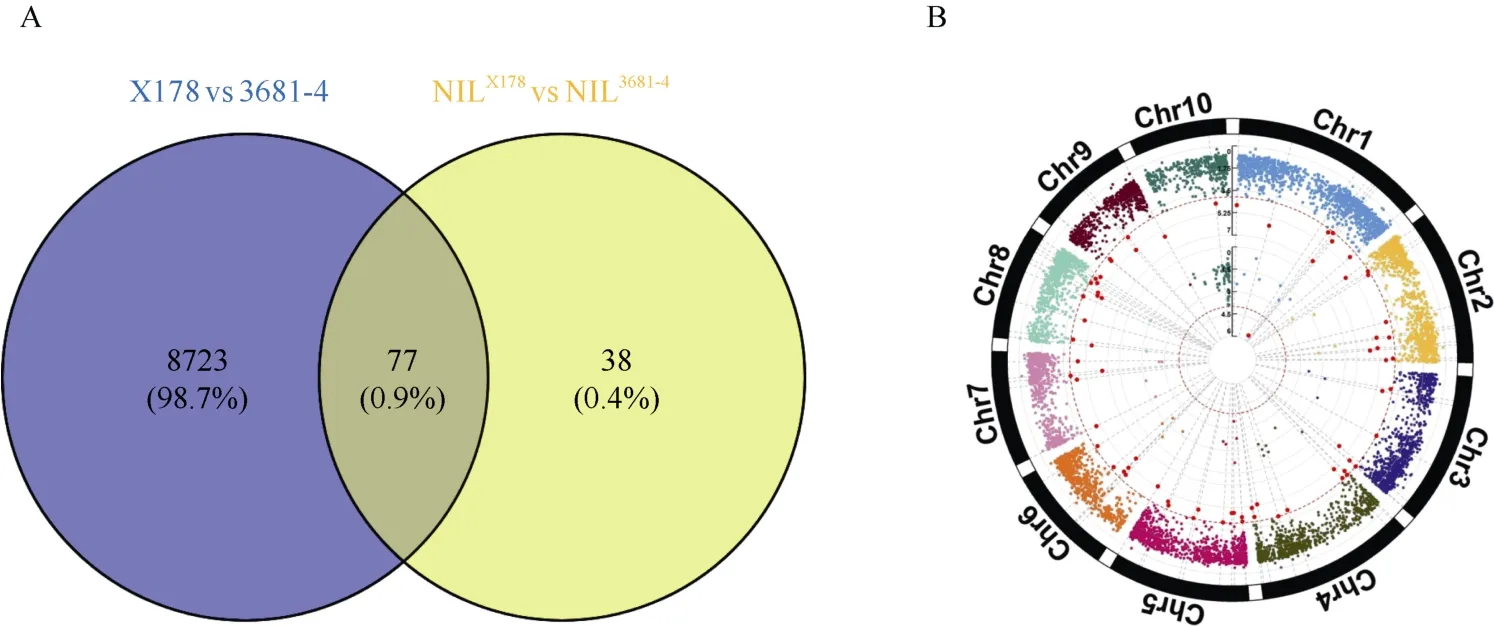
图5 差异基因统计和分布图Fig. 5 Statistics and distribution of differentially expressed genes
用AgriGo 对近等基因系中的差异表达基因作基因富集分析, 发现这些基因主要参与了对化学刺激的应激反应、氧化还原反应和对氧化胁迫的应激反应等(图6-A)。这表明深播条件对于玉米来说是一种逆境, 诱导了逆境相关基因的表达变化。根据KEGG 网站的注释, 这些差异表达基因中有两个与植物激素信号转导途径有关, 分别参与了细胞分裂素和脱落酸的信号转导; 一个基因参与了类胡萝卜素的合成, 在叶黄素循环中负责低光条件下的催化作用; 还有一个基因参与了卟啉和叶绿素的代谢途径, 负责将镁离子螯合到卟啉环上(图6-B)。表明候选基因可能是通过这些途径调控中胚轴伸长, 进而导致近等基因系的表型差异。

图6 近等基因系中差异基因GO 分析Fig. 6 GO analysis of differentially expressed genes in near-isogenic lines
3 讨论
目前玉米中已报道的耐深播相关的QTL 相对较少。Troyer[9]曾经定位到3 个深播条件下与中胚轴长度相关的QTL, 分别位于3 号、6 号、9 号染色体上,Liu 等[17]检测到32 个与深播发芽能力相关的QTL,在玉米10 条染色体上均有分布。尽管本实验中的耐深播QTLqMES20-10与他们报道的QTL 并没有重合, 但我们首先利用170 份BC3F3:4家系的基因型和表型, 对QTL 区段不同基因型家系的表型做了方差分析, 初步确证了qMES20-10的真实性(图2-A); 并利用覆盖玉米全基因组的芯片对该群体进行了全基因组的QTL 检测, 进一步确证了qMES20-10的真实性, 实验结果中仅 10 号染色体上存在一个主效QTL (LOD>2.5) (图2-B), 说明经过多代自交和回交后, 基本排除了其他位点的干扰, 所以本研究中构建的群体是对目标位点精细定位的理想材料。在精细定位的研究中, 利用BC4F2:3家系以两种策略都将qMES20-10定位到10 号染色体133.3~136.0 Mb 的区间(图3 和图4), 为该位点的进一步图位克隆打下基础。
对近等基因系进行差异表达分析, 进而挖掘候选基因, 是QTL 定位中寻找候选基因的常用方法[34]。同时差异表达分析还能预测候选基因通过怎样的代谢通路影响表型[35]。本研究的RNA-Seq 结果有助于本研究和其他后续研究确定与耐深播相关的候选基因。根据近等基因系中差异表达基因的GO 分析结果, 二者之间的差异表达基因主要影响了对化学刺激的应激反应、氧化还原反应和对氧化胁迫的应激反应等(图6-A)。深播条件下, 作物处于缺氧状态,细胞内可能会通过氧化还原反应产生较多的氧来维持平衡, 进而造成氧化胁迫, 诱导相关基因的表达。因此, 这些差异表达基因很好地解释了近等基因系中胚轴长度的差异。
赵光武等[16]研究表明, 深播条件下玉米中胚轴部位GA3和IAA 含量均显著提高, 且外源的GA3和IAA 均能促进中胚轴伸长, 这说明深播条件下对两种激素有响应的基因被诱导表达, 从而促进中胚轴伸长。而根据拟南芥中的报道, 细胞分裂素和脱落酸等植物激素也会影响下胚轴伸长[36-37], 说明植物激素在中胚轴伸长中有重要的调控作用。本研究根据KEGG 网站的注释信息, 差异表达基因影响了细胞分裂素和脱落酸的信号转导, 说明候选基因可能通过参与这些植物激素的调控影响中胚轴伸长。另外, 光信号通路也会影响拟南芥的下胚轴伸长[38],本研究中的差异表达基因还参与了叶绿素合成和类胡萝卜素合成, 表明候选基因也可能通过光信号通路调控中胚轴伸长(图6-B)。总之, 这些差异表达基因为进一步克隆qMES20-10位点和解释基因功能奠定了良好的基础。
4 结论
构建了X178 作为受体亲本的高代回交群体,将耐深播主效QTLqMES20-10进一步精细定位到10 号染色体上133.3~136.0 Mb 的区间, 并分析了近等基因系中基因差异表达情况。研究结果为该QTL的克隆奠定了基础。
