基于黄麻转录组序列SNP 位点的CAPS 标记开发与验证
2020-07-02陶爱芬游梓翊徐建堂林荔辉张立武祁建民方平平
陶爱芬 游梓翊 徐建堂 林荔辉 张立武 祁建民,* 方平平,*
1 福建农林大学金山学院, 福建福州 350002; 2 福建农林大学教育部作物遗传育种与综合利用重点实验室 / 福建省分子设计育种实验室, 福建福州 350002
黄麻属于椴树科(Tiliaceae)黄麻属(CorchorusL.)一年生草本植物, 其韧皮纤维具有易降解、透气性强、吸水性好等优点。近年来, 黄麻纤维被广泛应用于建材和可降解地膜等产业[1]。但与苎麻、亚麻等相比, 黄麻韧皮纤维细胞壁木质化程度较高、纤维素含量较低, 导致纤维粗、硬, 品质不佳, 使其只适合加工绳索和包装用麻袋等, 限制了黄麻在纺织业等工业领域的应用[2]。目前, 国内外研究人员已克隆了包括4CL 和COMT 在内的多个木质素生物合成途径中的关键酶基因[3]。而 MYB (V-myb avian myeloblastosis viral oncogene homolog)转录因子是植物转录因子家族中最大的成员之一, 它参与并对植物生长和发育的各个方面产生重大影响, 在植物次生代谢调节、激素和环境因子反应、细胞分化、器官形态发生和细胞周期调节中均有重要作用[4]。若能开发出与黄麻木质素合成和MYB 转录因子相关的标记, 则将为黄麻种质资源鉴定、育种亲本选配和遗传学研究等提供重要方法。
生物的遗传因子受到多个因素的影响, 并在基因组水平上产生一定的波动。而分子标记技术是直接反映DNA 分子水平多态性的技术, 与其他标记相比, 具有无表型效应、分布广、多态性高等优点, 并且不受环境的影响[5-6]。近年来取得长足发展的转录组测序技术, 具有通量高、分辨率高、不受限制等优点[7-8]。利用该技术可以获得大量潜在的 SNP(Single Nucleotide Polymorphism)和 SSR (Simple Sequence Repeat)标记, 并用于各种分子生物学研究[9-10]。与其他标记方法相比, SNP 标记遗传稳定性高、位点分布广泛且富有代表性, 因此已成为当前研究最多的分子标记之一[11-12]。虽然SNP 的检测方法众多, 但大部分方法均有其不足, 例如操作繁琐或者成本过高, 从而限制了其在植物遗传育种领域的应用[13-14]。其中较经济并且适合常规分子生物学实验的方法, 是把SNP 多态转化成酶切扩增多态性(Cleaved Amplified Polymorphic Sequences, CAPS)标记或衍生CAPS (Derived Cleaved Amplified Polymorphic Sequences, dCAPS)标记[15]。王彩芬等[16]和司文洁等[17]研究表明, CAPS 标记技术具有多态性高、共显性、所需DNA 量少、操作简便、结果稳定可靠等优点。CAPS 分子标记技术建立以来, 已经在水稻、小麦、玉米、大豆、番茄、马铃薯等作物中得到广泛应用[18-21]。
目前, 分子标记技术已广泛应用于黄麻遗传多样性分析、起源与演化、遗传连锁图谱构建等研究上[22-26]。关于黄麻分子标记的开发已取得一些进展,例如SSR 标记[27-29]、SNP 标记[30-31]都已被开发利用,但是关于黄麻CAPS 标记的开发, 未见有相关文献报道, 而与黄麻木质素合成基因和MYB 转录因子相关的CAPS 标记的开发与验证, 更是未见报道。鉴于CAPS 分子标记的优点及其在黄麻上开发应用的滞后性, 开展该研究是十分必要的。
1 材料与方法
1.1 材料
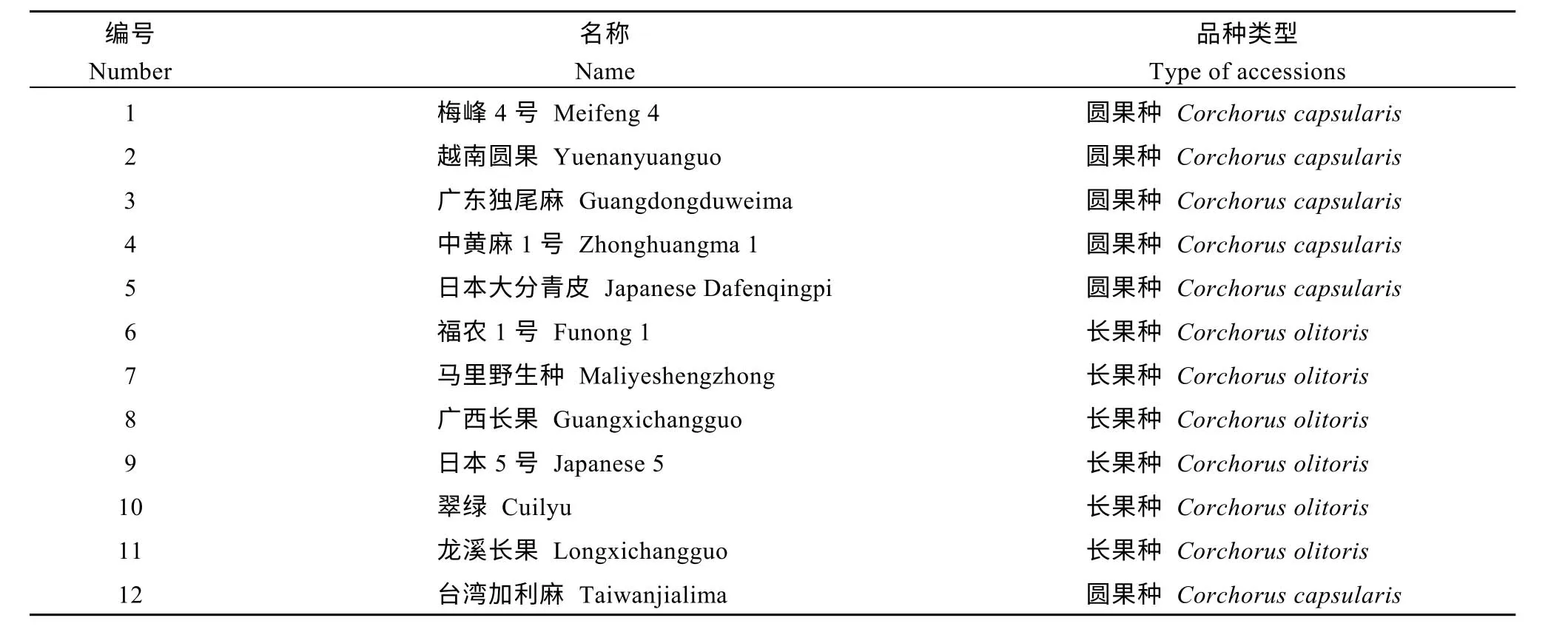
表1 用于CAPS 引物多态性验证的黄麻品种名称和类型Table 1 Name and type of jute accessions used in the validation of CAPS markers
1.1.1 植物材料 用于转录组测序的黄麻材料为黄麻179 和爱店野生种。2017年4月种植于福建农林大学田间科技园, 采用盆栽栽培, 每盆10 株, 3 次重复, 出苗后30 d, 真叶长到7~8 片时, 取茎和叶混合进行转录组测序。用于CAPS 多态性引物筛选验证的12 份黄麻种质资源的名称和类型见表1。所有材料均为盆栽, 于2017年8月份播种, 每盆10 株,置自然条件下生长, 长至5~6 片真叶时, 取幼嫩叶片混合后提取DNA。
1.1.2 试剂及仪器 DNA 提取试剂盒、限制性内切酶等购自北京百泰克生物技术有限公司, 其他常用试剂为实验室分析纯常规试剂; PCR 仪为德国Eppendorf 公司的MC-gradient; 电泳仪购自六一仪器厂(北京), 型号为DYY-6。
1.2 方法
1.2.1 黄麻转录组测序和SNP 位点鉴别 基于Illumina HiSeq 4000 测序技术平台, 构建黄麻转录组文库并进行建库测序, 获得黄麻转录组数据。在转录组测序的基础上, 采用GATK 针对RNA-Seq的SNP 识别流程, 进行SNP 多态性位点识别[32], 识别标准是, (1) 35 bp 范围内连续出现的单碱基错配不超过3 个; (2)经过序列深度标准化的SNP 质量值大于2.0。
1.2.2 与4CL、COMT 和MYB 转录因子相关的黄麻SNP 引物设计 在黄麻转录组序列中, 寻找与木质素合成酶基因4CL 和COMT 以及MYB 转录因子相关的unigene, 并采用Oligo 8 设计SNP 引物, 主要参数为: (1)引物长度在18~25 bp 之间; (2) PCR 预期扩增产物大小在100~500 bp 之间; (3)退火温度(Tm)在53~58℃之间, 上、下游引物的Tm值相差不大于4℃; (4)GC 含量在40%~60%之间。
1.2.3 CAPS 标记开发及内切酶的选择 采用dCAPS Finder 2.0 进行CAPS 引物开发和内切酶的选择, 主要步骤为, (1)在黄麻unigene 序列中寻找与CAPS 标记的PCR 扩增体系中SNP 引物相对应的unigene 片段; (2)选取其SNP 位点前后各30 bp 长度的碱基序列; (3)将SNP 位点原序列和突变序列分别输入 dCAPS Finder 2.0 的对话框中, 寻找可以对位点进行切割的限制酶, 其对应的标记即为基于SNP的CAPS 标记。
1.2.4 黄麻DNA 的提取 采用北京百泰克生物公司的BioTeke 试剂盒, 按说明书提取DNA。
1.2.5 CAPS 标记的PCR 扩增体系构建 采用束永俊等[14]的CAPS 标记的PCR 扩增程序, 反应体系为20 μL, 包含DNA 2 μL、上下游引物各1 μL、PRR MIX 10 μL、无菌水6 μL。PCR 程序为95℃预变性3 min; 95℃变性30 s, 53℃退火30 s, 74℃延伸45 s,共9 个循环; 95℃变性30 s, 53℃退火45 s, 72℃延伸45 s, 共14 个循环; 最后72℃延伸10 min, 4℃保存。
1.2.6 限制性核酸内切酶的酶切反应 按照内切酶的使用说明书构建限制性酶切反应体系, 反应体系可分为省时酶和非省时酶2 种。
1.2.7 琼脂糖凝胶电泳 用1.2%的琼脂糖凝胶检测扩增和酶切产物, 电泳电压为100 V, 电流为100 mA, 电泳时间为40 min, 电泳完成后用培清600 型凝胶成像仪观察条带并拍照记录。
1.2.8 数据统计分析 按照扩增和酶切条带的有无计数, 当某一条带出现时赋值为“1”, 不存在时赋值为“0”, 从而把图形资料转换成数据资料。参照Nei 等[33]的方法求得品种之间的遗传距离, 然后用DPS 软件中的类平均聚类法(UPGMA)对12 份黄麻种质进行聚类分析。
2 结果与分析
2.1 黄麻转录组中SNP 位点的数量与分布
黄麻转录组经组装后共获得72,674 条unigene序列, 检测到的SNP 位点总数为67,567 个, 序列总长度为29,705,997 bp。在黄麻unigene 序列中, 平均每440 bp 会有一个SNP 位点。SNP 位点分布密度如表2 和图1 所示。由表2 可知, 含0~1 个SNP 位点的unigene 序列最多, 有66,525 条, 占总数的91.53%;含2 个以上SNP 位点的unigene 序列数目急剧下降,其中含1~2 个SNP 位点的unigene 序列有2092 条,比例为2.88%; 含2~3 个SNP 位点的unigene 序列有1277 条, 占1.76%; 含3 个以上SNP 位点的unigene序列总数亦较少, 有2780 条, 比例为3.81%。表明绝大多数黄麻unigene 上含有单个SNP 位点, 含有2个及以上SNP 位点的unigene 数量不多, 总计约8%左右, 其中含5~8 个SNP 位点的unigene 数量尤其少, 比例非常低。
2.2 与4CL、COMT 及MYB 转录因子相关的CAPS 标记开发
在对与黄麻木质素合成基因4CL、COMT 及MYB 转录因子相关的unigene 分析的基础上, 共获得39 对与上述基因及转录因子相关的SNP 引物(表3)。其中和4CL 相关的SNP 引物最少, 仅有6 对, 和COMT 相关的有15 对, 数量居中, 而与MYB 转录因子相关的最多, 有 18 对。在此基础上, 采用dCAPS Finder 2.0 软件开发基于SNP 位点的CAPS标记, 共获得26 对有酶切位点的CAPS 标记(表4),开发比率为66.7%, 这26 对CAPS 标记分别具有能被SspI、ClaI 和MseI 等17 种内切酶识别的位点。
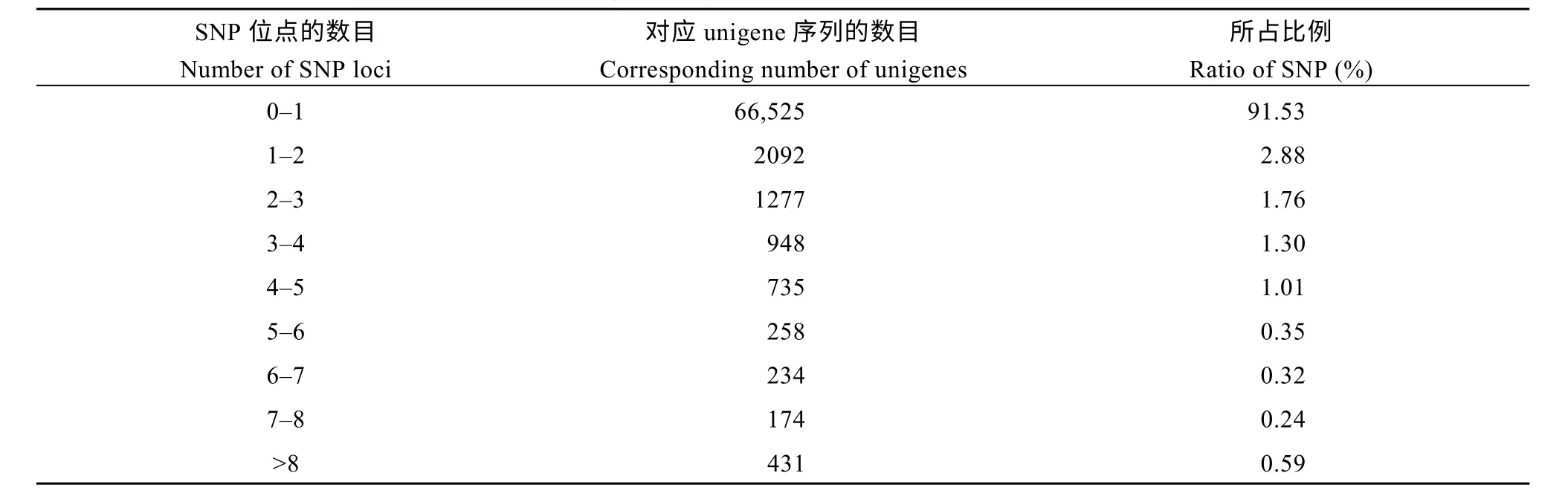
表2 黄麻unigene 中SNP 位点的分布密度表Table 2 Distribution density of SNP loci in the unigenes of jute
图1 黄麻unigene 中SNP 位点分布的趋势图Fig. 1 Trend of SNP loci distribution in the unigenes of jute
由表4 可知, 在26 对CAPS 引物中, 仅有3 对与黄麻木质素合成基因4CL 相关, 比例为11.5%,其对应的unigene 序列仅有一条, 即unigene_17597,能识别位点的内切酶种类也仅有一种, 为SspI; 有9 对CAPS 引物与COMT 相关, 占34.6%, 数量居中,它们分别与 unigene_04800、unigene_09002 和unigene_08713 等3 条unigene 序列相对应, 能识别变异位点的内切酶也分别有3 种, 即ClaI、BsiYI和AvaIII; 而与MYB 转录因子相关的CAPS 标记最多, 有14 对, 占总数的53.8%, 其对应的unigene 序列有 14 条, 各不相同, 除 MYBCAPS8 和MYBCAPS13 对应的内切酶均为SecI 之外, 其他内切酶种类也均有所不同。表明在3 种CAPS 标记中,与MYB 转录因子相关的CAPS 标记具有多样化的、可以被多种内切酶识别的SNP 变异位点。

表3 39 对SNP 引物的编号、名称及序列Table 3 Name and sequence of 39 pairs of SNP primers
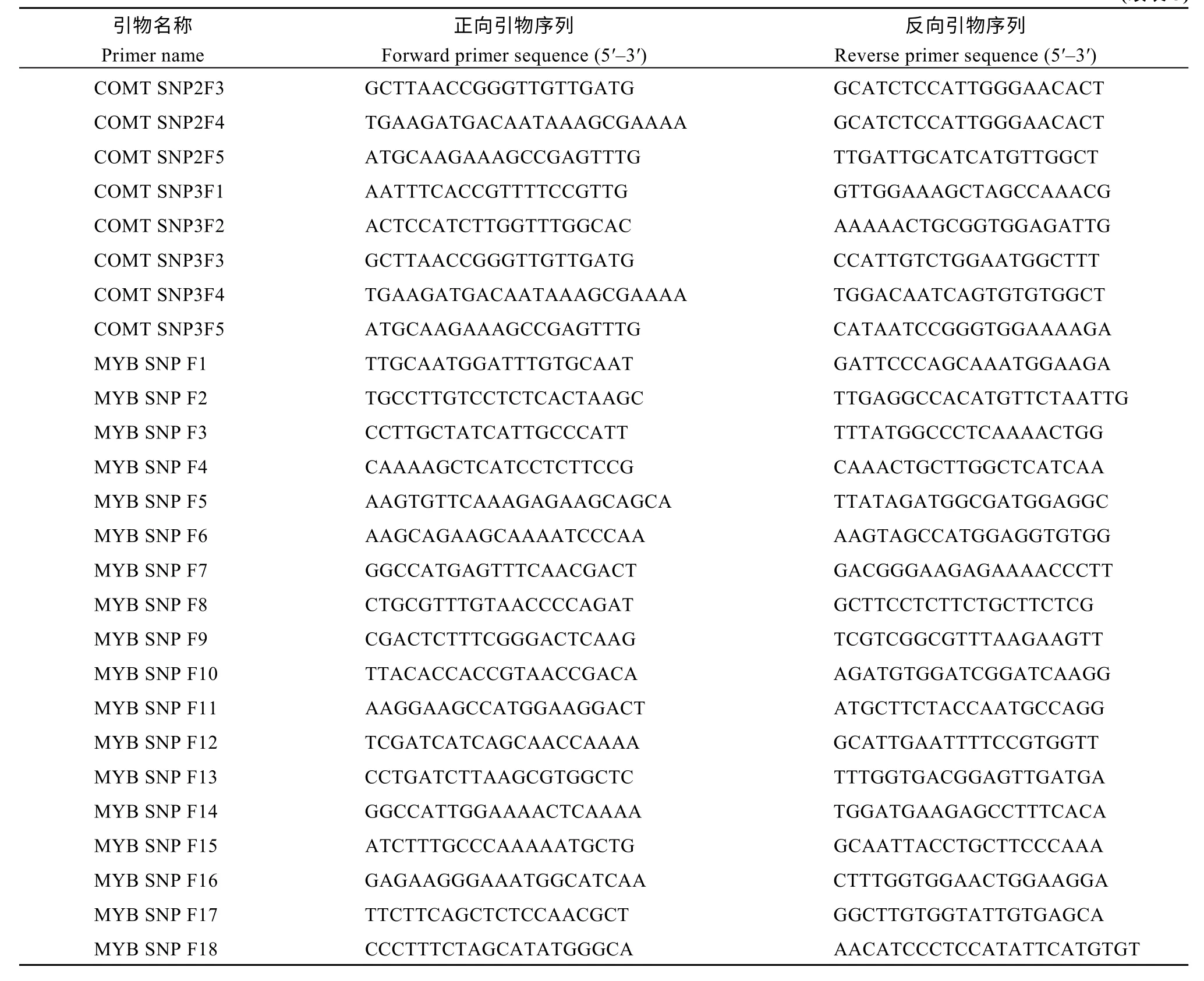
(续表3)
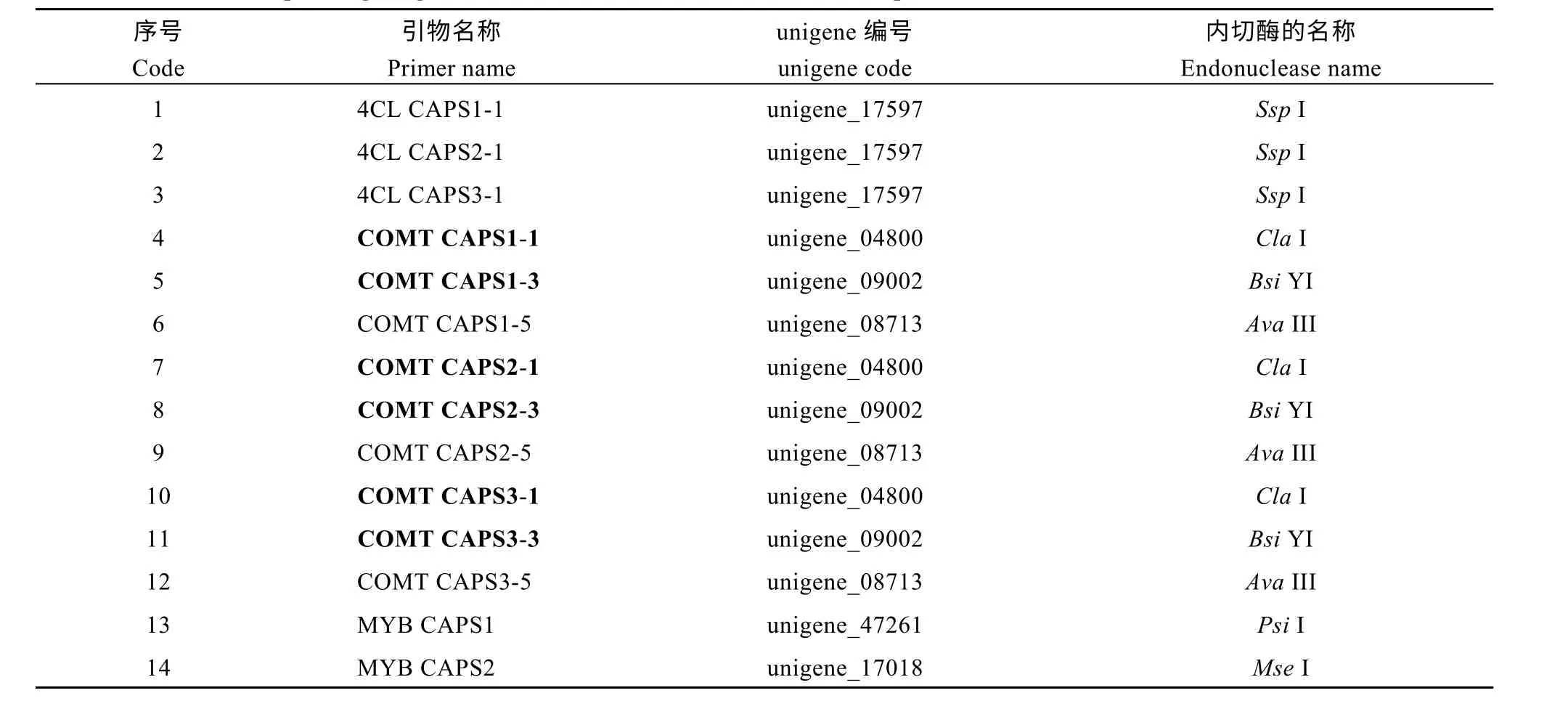
表4 CAPS 引物名称、对应的unigene 编号及内切酶名称Table 4 Name, corresponding unigene code, and endonuclease name of CAPS primers
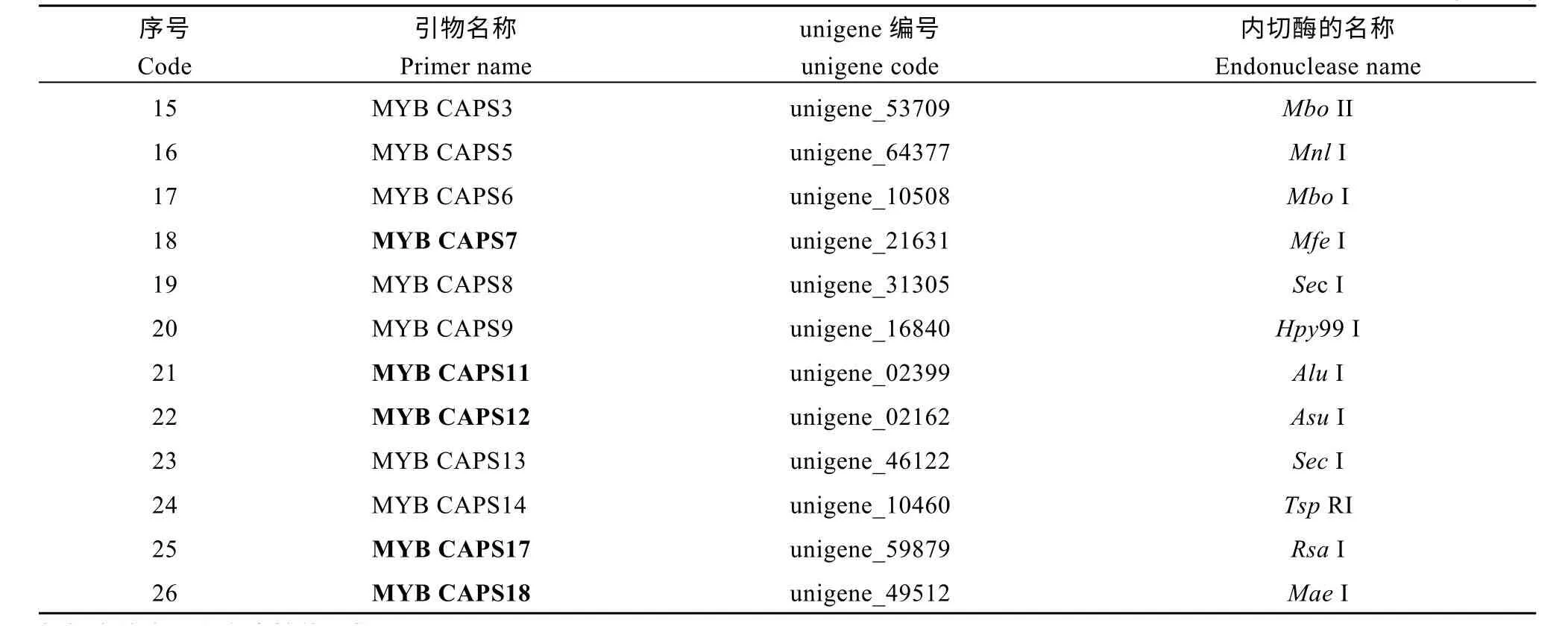
(续表4)
2.3 多态性CAPS 引物的筛选
对上述26 对CAPS 引物进行PCR 扩增和酶切处理表明, 有11 对CAPS 引物的PCR 产物可以被酶切开来, 得到多态性条带(表4 中黑体字标注, 扩增结果见图2), 占总数的42.31%, 而另外15 对引物的PCR 产物无法被酶切开, 虽然有清晰的条带但没有多态性。在多态性CAPS 引物中, 与木质素合成酶基因COMT 相关的引物有6 对(54.5%), 与MYB 转录因子相关的引物有5 对(45.5%), 二者数量相当, 而未获得和4CL 相关的多态性CAPS 标记引物。
2.4 多态性CAPS 标记的验证
以12 个黄麻品种为材料, 对所筛选的11 对CAPS 标记的有效性进行验证。聚类分析表明(图3),在遗传相似系数为0.5 处画线时, 12 个黄麻品种被区分为2 个不同的类群, 第I 个类群包括7 个品种, 除“日本5 号”外, 其他6 个品种均为圆果种黄麻; 第II个类群包括5 个黄麻品种, 均为长果种黄麻。由此可见, 所用的黄麻材料大致有按2 个不同栽培类型,即长果种和圆果种黄麻区分的趋势。来自日本的圆果种黄麻“日本大分青皮”和长果种黄麻“日本5 号”被聚在一起, 推测这2 个品种虽然属于不同的栽培类型, 但其木质素合成相关的基因以及MYB 转录因子均可能有很高的相似性, 因而聚在同一个小类群中。同时进一步细分发现, 在第I 个类群中, “台湾加利麻”和“龙溪长果”又各自成一类, 没有和其他品种的黄麻聚在一起, 推测这2 个品种的木质素合成相关的基因以及MYB 转录因子较为特殊, 与其他品种有所区别。综上所述, 所筛选出来的11 对CAPS多态性标记, 可以将12 份不同类型的黄麻品种较好地区别开来, 验证了所开发的CAPS 标记的有效性和可行性。
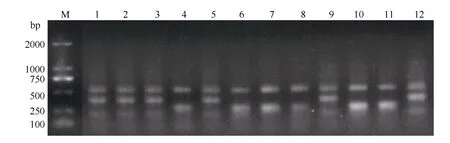
图2 COMT CAPS1-3 引物扩增和酶切结果Fig. 2 Amplified and enzyme digestion result of CAPS primer COMT CAPS1-3
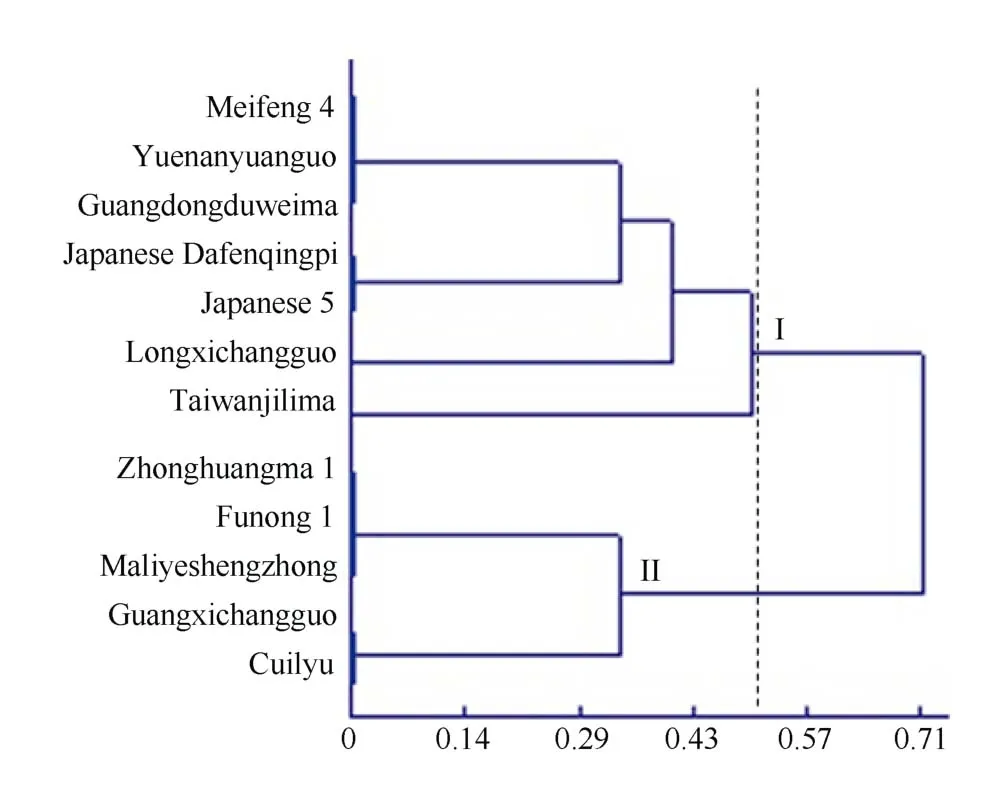
图3 12 份黄麻品种基于CAPS 标记的聚类图Fig. 3 Dendrogram of 12 jute accessions based on CAPS markers
3 讨论
3.1 黄麻转录组中SNP 位点的分布
SNP 是由单碱基突变引起的DNA 序列多态性,具有数量丰富、分布广泛、覆盖度高等特点[34]。黄麻转录组序列中共检测到67,567 个SNP 位点, 平均每440 bp 出现一个SNP 位点, 与其他作物相比较,出现的频率较低。前人研究表明, 水稻基因组中约268 bp 存在1 个SNP[35], 玉米基因组中每60~120 bp就存在1 个SNP[36], 大豆基因组中每185~266 bp 有1 个SNP[37], 而茶树基因组中平均每172 bp 有1 个SNP[38]。SNP 出现的频率, 第一, 与物种本身基因组的差异有关, 不同物种不同。第二, 存在于非编码序列中的SNP 会提高SNP 出现的频率[39], 而黄麻中存在于非编码序列的SNP 位点较少, 导致其出现的频率降低。第三, 试验样本大小及测序长度也会影响SNP 的频率[40], 今后应进一步增加黄麻转录组的测序长度, 以提高SNP 的频率。
3.2 所开发CAPS 标记的多态性
本试验的CAPS 标记是在前期黄麻转录组序列中SNP 位点的基础上开发的。SNP 标记因为在基因组中分布广泛、遗传稳定等特点, 在植物遗传育种研究方面有巨大的优势, 但是由于检测手段的限制,阻碍了SNP 标记的广泛应用。而CAPS 方法无需昂贵的设备、操作简单, 可以实现中通量的 SNP 检测,因而可以大大促进SNP 标记在植物遗传育种中的应用[38]。本研究以39 对SNP 引物为基础, 获得了26对CAPS 标记, 开发成功的比率为66.7%, 与柑橘(65%)和水稻(67.8%)的成功率相似[17,41], 但低于茶树和大豆SNP 快速转化为CAPS 方法的比例(81.8%和 86.21%), 高于大豆传统方法转化的比例(52.51%)[14]。造成这种差异的原因, 首先与酶切位点选择和分析的标准有关, 严格的酶切位点选择和分析标准, 可以提高转化的成功率[38]; 其次, 由于某些SNP 并未引起限制性内切酶识别位点的改变, 不能直接使用内切酶进行检测; 再次, 若在同一扩增产物中存在多个与SNP 所在酶切识别位点相同的酶切位点, 直接使用内切酶检测难以分辨[19]。另外, 多态性CAPS 引物所占的比率为42.3%, 也比较低。原因可能是本研究开发的CAPS 引物是和4CL、COMT等木质素合成酶基因及MYB 转录因子相关的, 针对性和目的性较强, 而所用的供试材料在这些位点仅有部分多态性, 从而导致多态性引物的比率降低。也有可能是物种本身的特异性引起的, 潜宗伟等研究亦表明, 分子标记引物的多态性比率因物种不同而有所差异[42]。
3.3 所开发CAPS 标记的有效性
所开发的CAPS 标记引物可以将供试的12 份黄麻种质较好地区别开来, 供试材料有大致按栽培类型, 即长果种黄麻和圆果种黄麻聚类的趋势。同时,来自日本的2 个品种聚在一起, 体现了按种质地理来源聚类的特点。另外, 基于CAPS 标记的聚类分析也能鉴别出个别特异的种质类型, 如台湾加利麻。以上结果均表明, 所开发的CAPS 标记在黄麻遗传多样性分析上是有效和可行的, 且可用于黄麻种质资源鉴定等研究, 为黄麻杂交育种亲本选配提供理论依据。综上所述, 本试验所开发的黄麻CAPS标记是黄麻遗传育种研究中的一项理想的分子标记方法, 可为黄麻分子标记辅助育种和性状改良提供有效方法。
4 结论
对黄麻转录组序列中的SNP 位点进行了分析,发现平均每440 bp 出现一个SNP 位点, 其分布频率低于其他作物, 推测与物种之间的基因组差异性有关, 而黄麻中存在于非编码序列的SNP 位点较少也是影响因素之一。同时, 设计了与黄麻木质素合成基因4CL、COMT 及转录因子MYB 相关的39 对SNP引物, 并以此为基础开发了26 对CAPS 标记, 开发成功率为66.7%, 其中11 对CAPS 标记具有多态性,多态性比例为43.2%。开发的CAPS 标记能较好地将12 份不同类型的黄麻种质按栽培类型区分开来,同时亦能鉴别出特异种质, 表明所开发的CAPS 是适用于黄麻种质资源鉴定的较理想的分子标记方法,可为黄麻杂交育种亲本选配提供理论依据, 同时也为黄麻分子标记辅助选择育种提供了有效方法。
