高光谱快速预测冷鲜鸡胸肉中乳酸菌
2020-06-17何鸿举蒋圣启王玉玲马汉军陈复生朱明明赵圣明周浩宇
何鸿举,蒋圣启,王 魏,王玉玲,马汉军,2,陈复生,朱明明,赵圣明,周浩宇
(1.河南科技学院食品学院,河南新乡 453003;2.河南科技学院博士后研发基地,河南新乡 453003;3.河南工业大学粮油食品学院,河南郑州 450001;4.河南科技学院生命科技学院,河南新乡 453003)
作为禽肉重要组成的鸡肉,因其脂肪和胆固醇含量低、蛋白质含量高且价格便宜等优点成为国内外消费者青睐的对象[1-3]。微生物指标直接反映了冷鲜鸡肉品质好坏和腐败程度。冷鲜鸡肉在运输、保藏和加工过程中,都会因为微生物的生长和繁殖导致品质下降[4]。乳酸菌(Lactic acid bacteria,LAB)是一群可以发酵糖类,并产生大量乳酸的革兰氏阳性杆菌或球菌,广泛存在于人、畜、禽的肠道以及许多食品、物料中[5-6]。冷鲜肉在冷藏过程中的腐败变质主要是由于嗜冷性微生物的大量增殖代谢造成的,乳酸菌的生长繁殖加速了冷鲜肉的腐败变质[7-8]。孙彦雨[9]研究发现,乳酸菌是真空冷鲜鸡胸肉的优势腐败菌之一,会分解鸡肉中的营养物质产生有机酸,使鸡肉pH下降,感官品质下降。梁慧等[10]发现乳酸菌是冷鲜鸡肉初始菌相中占比最大的菌群,达到35%。赖宏刚等[11]对冷鲜鸡肉中腐败微生物进行分离鉴定,结果表明乳酸菌是冷鲜鸡肉中主要腐败菌群之一。
传统检测乳酸菌的方法有平板法、PCR扩增产物法和凝胶电泳检测法等[12-14]。虽然这些方法结果准确可靠,但存在费时费力,消耗大量药品,以及对原有样品造成破坏性等缺点。这些传统方法已经无法满足现场快速分析检测和工业化实时在线检查的要求。此外,这些传统技术也不适用于测量大量样品的场合。因此,需要开发快速检测乳酸菌的相关方法和技术来满足生产者和消费者对乳酸菌快速无损检测的需求。
高光谱技术(Hyperspectral imaging,HSI)将光谱和成像结合起来,可同时获得样品的光谱信息和图像信息[15],该技术被广泛应用于肉类无损检测的各个领域。Barbin等[16]通过高光谱技术对鸡胸肉的持水力、色泽L*和pH进行建模预测,其预测模型的相关系数0.84、0.95和0.9;Yang等[17]用400~2500 nm的HSI对鸡肉进行等级分类,其模型相关系数达到了0.85。王莉等[18]针对滩羊肉的菌落总数和挥发性盐基氮进行400~1000 nm HSI的建模分析,得到预测模型的相关系数分别为0.87和0.88;王慧等[19]对鸡肉嫩度的高光谱数据分析建模,最终获得最优预测模型R为0.94,效果较好。目前,已报到的肉类乳酸菌无损检测研究较少,大多数见于近红外光谱技术领域。光谱技术只能够提供被测样品的光谱信息,其不足之处是不能反应样本的空间分布信息,且结合高光谱技术对鸡胸肉的乳酸菌含量进行无算检测的研究鲜有报道。
本试验基于高光谱成像技术的优势,通过分析波长900~1700 nm高光谱数据,构建快速稳定预测模型,旨在实现对冷鲜鸡肉中乳酸菌的无接触快速检测。为建立一种快速无损检测鸡肉中乳酸菌方法和鸡肉品质在线监测设备的开发提供数据支撑和理论依据。
1 材料与方法
1.1 材料与仪器
冷鲜鸡胸肉 河南众品股份有限公司;CM361乳酸菌培养基、CM0509蛋白胨 英国Oxoid公司;氯化钠 分析纯,天津市科密欧化学试剂有限公司。
HSI-eNIR-XC130型推扫式高光谱成像设备 台湾Isuzu Optics公司;3900-ER光源 美国Illumination Technologies公司;ImSpector V10E光谱仪 芬兰Spectral Imaging公司;CCD探测仪 爱尔兰Andor公司;OLE2镜头 德国 Schneider公司;移动平台 中国Isuzu Optics公司;SW-CJ-1D双人单面净化工作台 苏州净化设备有限公司;HV-85全自动高压灭菌锅 日本Hirayama公司;BF240微生物培养箱 德国Binder公司;Scientz-04拍打式均质机 宁波新芝生物科技股份有限公司。
1.2 实验方法
1.2.1 冷鲜鸡胸肉预处理 将新鲜的鸡胸肉置于带冰袋的无菌冷藏箱内,运至微生物实验室。分割样品前将案板和刀具放入超净工作台中开紫外光灯灭菌30 min。把整块鸡胸肉在超净工作台中分割成3.0 cm(长度)× 3.0 cm(宽度)× 1.0 cm(厚度)的小块,共获得119块样品;用一次性塑料盒将样品分装编号,置于0~4 ℃的冰箱内,每24 h取出若干样本,进行高光谱数据的采集和肉样中乳酸菌的测定。最长储存时间为7 d。
1.2.2 高光谱数据采集 将高光谱成像设备打开预热30 min使其光源稳定,减少光谱设备不稳定带来的误差。将待测样品从0~4 ℃冰箱中取出,待其温度恢复至室温。然后将样品置于设备载物台上扫描样品高光谱图像,基于本实验室前期和Wang等[20]研究成果,高光谱设备参数设置如表1所示。相机自身暗电流会使样品图像采集过程有附带很多噪声信息,需对原始图像进行黑白校正,减少部分噪声对高光谱数据的影响,具体校正方法和公式参考He等[21]研究。

表1 高光谱成像设备参数设置Table 1 Parameters setting of hyperspectral imaging system
1.2.3 乳酸菌测定 样品采集完高光谱图像之后,根据GB 4789.35-2016 《食品微生物学检验乳酸菌检验》的要求进行检测乳酸菌菌落总数[22]。
1.2.4 光谱数据提取及预处理 通过HSI Analyzer软件校正高光谱图像,提取图像中感兴趣区(Region of interest,ROI)内的每个像素点的光谱信息,计算所有像素点光谱信息的平均值。获得的原始光谱会附带外在的干扰和噪声信息,影响样品数据的真实性,因此需要对原始光谱进行预处理来消除或减弱外界光线和噪声等因素对真实数据的影响[23]。数据采用多元散射校正(Multiplicative Scatter Correction,MSC)、基线校正法(Baseline Correction,BC)、移动平均平滑(Moving Average Smoothing,MAS)、标准变量变换(Standard Normalized Variate,SNV)、归一化法(Normalize,NOR)、中值滤波平滑(Median Filter Smoothing,MFS)、卷积平滑(Savitzky Golay Smoothing,SG)和高斯滤波平滑(Gaussian Filter Smoothing,GFS)预处理原始光谱数据,对比每一种预处理效果,获取最佳预处理方法[24-26]。
1.2.5 预测模型构建、评价及优化
1.2.5.1 偏最小二乘模型构建及评价 偏最小二乘(Partial Least Squares,PLS)是一种高效率多元数据分析方法,融合了多元线性回归(Multiple Linear Regression,MLR)和主成分回归法(Principal Component Regression,PCR)的功能,具有适用范围广和预测能力强的优势[27-28]。本试验将900~1700 nm波长的反射率作为自变量,乳酸菌参考值作为因变量,通过PLS回归,构建预测鸡肉中乳酸菌含量的PLS模型。模型的性能通过相关系数(r)、校正误差(Root Mean Square Error of Calibration,RMSEC)、内部交叉验证均方根误差(Root Mean Square Error of Cross Validation,RMSECV)和外部预测均方根误差(Root Mean Square Error of Prediction,RMSEP)的数值大小来衡量。r越接近1,RMSE值越小,模型的精度越高,预测效果越好[29]。鲁棒性和剩余预测偏差(Residual Predictive Deviation,RPD)也可以评价模型的性能。鲁棒性是通过(RMSEC-RMSEP(表示,(RMSEC-RMSEP(的值越小,模型越稳定[30]。RPD是预测集所有样本参考值的标准偏差和预测标准偏差的比值,参考Malley等[31]的RPD反映效果表,RPD在1.75~2.25,反映模型是比较有用的;RPD在2.25~3,反映模型是比较成功的;RPD在3~4,反映模型是成功的;RPD大于4,反应模型是优秀的。以上评价参数指标按式(1)~(4)计算:
式(1)
式(2)
式(3)
式(4)

1.2.5.2 模型优化 本试验采用的900~1700 nm范围内共有486个波长。采用全部波长作为建模的输入变量,可以达到较好地的预测效果,但全波段数据量庞大且含有较多冗余信息,会降低模型运算效率,影响模型预测能力。虽然采用不同的预处理方法处理原始数据可以去除系统噪声的影响,但预处理不能消除冗余信息、并保留对乳酸菌含量模型贡献的有用信息。因此需要将与乳酸菌含量关系最大的最优波长从全波段中筛选出来进行建模,剔除无关信息,提取有用信息,减少数据计算量,提高建模效率和预测精度。
本试验采用回归系数法(Regression Coefficient,RC)[32]、逐步回归法(Stepwise)[33]和连续投影算法(Successive Projections Algorithm,SPA)[34]筛选最优波长。这三种方法被广泛应用于光谱模型变量选择上,可以在建模过程中筛选有用变量、剔除不相关变量达到变量降维的效果,从而减少原始光谱数据的奇异性的和不稳定性。通过比较三种化学计量学算法筛选出最优波长的数量、波长减少量以及优化后模型的性能,挑选出最好的筛选乳酸菌特征波长的方法。
1.3 数据处理
数据整理:每个样品的乳酸菌含量参考值对应每个样本的光谱信息,在Excel 2010软件中整理。
数据分集:将所有样本按乳酸菌含量参考值从小到大排列,随机取出样本总数的三分之二作为校正集,剩下三分之一作为预测集。
全波段模型构建:将样本的光谱信息作为自变量x,乳酸菌含量参考值作为因变量y。通过The Unscrambler 9.7建模软件建立全波段偏最小二乘预测模型。
最优波长筛选:SPA法是在MATLAB R2006a程序开发软件的工作区域中输入样本的光谱信息Xc,乳酸菌含量参考值Yc。然后输入SPA的程序相关指令,通过软件计算得到相应的最优波长。中Stepwise法是在MATLAB R2006a程序开发软件的工作区域中输入样本的光谱信息Xc,乳酸菌含量参考值Yc。然后输入Stepwise的程序相关指令,通过软件计算得到相应的最优波长。RC法在软件Unscrambler 9.7中通过挑选在全波段模型建立中回归系数最大的波长,即为最优波长。
模型优化:将样本的最优波长光谱信息作为自变量x,乳酸菌含量参考值作为因变量y。通过The Unscrambler 9.7建模软件建立优化的偏最小二乘预测模型。
2 结果与分析
2.1 乳酸菌测量结果
本试验共获得119个乳酸菌菌落总数参考值,按照1.3取其中40个作为预测集样品,其余的79个作为校正集样品,对应的光谱数据按同样的方式分组。统计结果如表2所示。
冷鲜鸡胸肉0~4 ℃储藏7 d的乳酸菌含量变化,如图1所示。该试验的初始乳酸菌为2.55lg CFU/g,随着储藏时间的延长,在1~5 d内乳酸菌快速生长,5~7 d生长趋于缓慢。鸡胸肉的品质在1~2 d内正常,无明显变质现象;在3~4 d内黏度开始增加,弹性下降,开始出现腐败气味,感官品质明显变差;在5~6 d内鸡胸肉的颜色变暗,出现乳黄色崩解液体;第7 d的乳酸菌数量达到7.50lg CFU/g,鸡肉已经完全发臭腐败[35]。可能由于刚开始冷鲜鸡胸肉营养丰富适合乳酸菌的生长,腐败微生物的总数较少,菌种之间的竞争作用较小,导致乳酸菌在前5 d快速生长。随着乳酸菌和其他腐败微生物的共同增殖,营养物质的慢慢耗尽,比例失调导致环境条件越来越不适宜乳酸菌的生长,衰亡的细胞数增多,导致在最后2 d生长速度变慢[36]。

图1 冷鲜鸡肉储藏期内乳酸菌含量变化Fig.1 Change of lactic acid bacteria contents infresh chicken breast during storage period
2.2 冷鲜鸡胸肉的光谱特征
图2整体上反映119个样品的光谱曲线高低位置不同,但趋势一致,说明肉样的化学成分含量不同。虽然光谱上找不到明显的乳酸菌吸收峰,但是乳酸菌的生长离不开鸡肉中的各种化学组分,可以通过化学计量学手段挖掘光谱信息,寻找到特征光谱信息与乳酸菌之间的定量关系。图2显示不同预处理之后鸡肉样品光谱和原始光谱在形状上有不同的变化,这表明每种预处理消除噪声的方法和消除效果不同。

图2 不同预处理下的鸡肉样品近红外光谱特征Fig.2 NIR characteristics of chicken samples under different pretreatment注:(a)原始光谱;(b)NOR预处理光谱;(c)MAS预处理光谱;(d)SNV预处理光谱;(e)SG预处理光谱;(f)MSC预处理光谱;(g)MFS预处理光谱;(h)BC预处理光谱;(i)GFS预处理光谱。
2.3 基于全波段光谱预测乳酸菌
本试验采用PLS回归算法挖掘鸡胸肉中乳酸菌参考值与全波段光谱信息之间的相关性,即建立F-PLS回归模型,结果如表3所示。

表3 F-PLS模型预测鸡胸肉中乳酸菌含量结果Table 3 Results of predicting lactic acid bacteria by F-PLS model in chicken breast
由表3得知,利用原始光谱和预处理光谱构建的9种F-PLS回归模型预测冷鲜鸡胸肉中乳酸菌含量效果良好。rC都达到了0.963以上,rP达到了0.913以上,模型精度都较高;ΔE均在0.200以下,模型稳定性较好;RPD在2.3~3.2之内,模型建立都比较成功。但每种预处理光谱所建立的F-PLS精度、稳定性和预测能力有各有不同。通过8种预处理光谱所建F-PLS模型的各种参数与原始光谱所建F-PLS模型进行对比,SG预处理光谱的RPD最大,为3.121,参考Malley等[31]的RPD反映效果表,说明该模型是成功的。且rC为0.981,rCV为0.936,rP为0.950,模型的相关性较好,预测能力较强;ΔE为0.152,维持在较低水平,模型的鲁棒性较好,性能稳定。SG预处理光谱所建F-PLS模型为最佳模型。
2.4 最优波长筛选结果
以SG预处理光谱和SG预处理光谱构建的F-PLS模型为基础,采用RC、Stepwise和SPA算法从486个波长中筛选最优波长优化F-PLS回归模型。结果如表4所示:三种筛选方法选出的最优波长数都在20~30之间,波长减少量在94%~96%之间。用Stepwise法筛选出来的最优波长数量最少为20个,与SPA筛选出的21个数量接近。相比较486个全波长数,RC法、Stepwise法和SPA法筛选出的最优波长数量分别减少了94.86%、95.88%和95.68%。SPA法筛选的波长具体位置如图3所示。在SPA法筛选最优波长位置图3中,筛选对象是900~1700 nm的486个波长,红色方块标识的位置即为最优波长所在位置,共21个。

表4 三种方法筛选最优波长结果比较Table 4 Comparison of optimal wavelengths selected by three methods
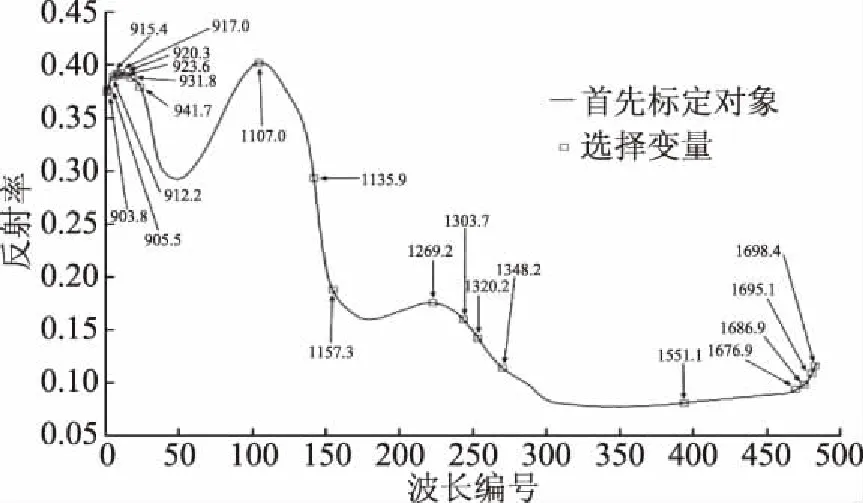
图3 SPA法筛选的最优波长位置图Fig.3 Optimal wavelength location selected by SPA
2.5 基于最优波长预测乳酸菌
利用三种方法筛选出的最优波长,分别建立优化模型,结果如表5所示。

表5 优化PLS模型预测鸡胸肉中乳酸菌含量结果Table 5 Results of predicting lactic acid bacteria by optimized PLS model in chicken breast
由表5可得,与全波段F-PLS回归模型相比较,优化后的PLS模型相关系数整体上都有所降低,误差也都有增加。其中用Stepwise法筛选出的20最优波长,虽然波长数量最少,但所构建的SW-PLS模型效果不理想。基于RC和SPA构建的RC-PLS模型和SPA-PLS模型相比,后者使用的波长数最少,RPD最大,为2.787,rP最大,为0.949,ΔE最小,为0.135lg CFU/g。说明基于SPA法筛选出的21个最优波长建立的SPA-PLS模型精度最高,预测稳定性最好。
3 结论
对近红外高光谱数据对冷鲜鸡胸肉的乳酸菌含量进行快速无接触检测,采用MSC等8种不同方法预处理原始光谱后,分别构建全波段F-PLS模型预测鸡肉乳酸菌,其中基于SG预处理光谱所构建的F-PLS模型效果最好。使用RC、Stepwise和SPA筛选最优波长,其中通过SPA筛选的21个最优波长建立的SPA-PLS模型预测效果最好,rP达到0.949,RMSEP为0.439lg CFU/g。结果表明,采用合适的化学计量学算法挖掘近红外高光谱数据构建模型快速无接触检测冷鲜鸡肉中乳酸菌是可行的。
