基于高通量测序技术分析东北豆酱的微生物多样性
2020-06-17马岩石裴芳艺
马岩石,姜 明,李 慧,裴芳艺
(齐齐哈尔医学院科研处,齐齐哈尔 161000)
我国制酱工艺历史悠久,历经多个朝代的发展,如今已经普及到了韩国、日本、印度尼西亚等东南亚国家和地区。豆酱是以大豆为主材料,通过发酵制备而成的一种具有独特风味的食品以及调味品。东北作为我国大豆的主要产区,其大豆年产量占全国总产量的50%左右,豆酱的制作也是非常普遍的[1]。
目前东北豆酱主要采用大豆发酵的方式进行制作,制作过程中各种微生物作用赋予了豆酱独特的风味,因此明确东北豆酱成品中的微生物组成是十分必要的[2]。目前国内对食品中微生物的研究在发酵过程中的风味物质检测和重要微生物的鉴定方面已经比较普遍[3-6],Yu等[7]对传统农家酱中细菌的生物胺含量进行了研究,发现组胺和酪胺与发酵时间呈显著正相关。齐欣[8]通过测定朝鲜族大酱中异黄酮提取物(IEOKSP)对几种癌细胞增殖的影响等试验得出,朝鲜族大酱中主要抗癌成分随着贮藏时间的延长,含量逐渐增多。
高通量测序如今越来越多的应用在农业、工业、食品、医学等微生物多样性的检测中[9-12],谷静思等[13]通过高通量测序的方法以及传统分离培养方法对臭豆腐中的细菌的微生物群进行分析,最终得到了臭豆腐中微生物群落的分布信息。Ercolini[14]介绍了高通量测序技术用于研究食品微生物群的当前情景和未来前景,并描述了选择最佳工作条件以满足食品研究特定需求的决策过程。本研究采用高通量测序技术[15],以东北市场常见的5种豆酱成品为研究材料,选取比较适宜序列检测的细菌16S rDNA V4区及真菌ITS1~2区[16-18]基因序列检测其微生物群落结构,能够更加全面、准确的了解其菌群结构,为进一步的豆酱的生产、保藏以及优势菌种的利用提供一定的参考。
1 材料与方法
1.1 材料与仪器
5种豆酱FJ01、FJ02、CD01、CD02、CD03 均为发酵时间45 d的市售产品,分别来自东北地区5个不同的生产地点,FJ01产于辽宁营口,工业生产,FJ02产于黑龙江绥化,工业生产,CD01产于吉林通化,手工制作,CD02产于吉林白城,工业生产,CD03产于哈尔滨,手工制作;DNA Marker5000、DNA Marker2000 上海生工生物工程有限公司;琼脂糖 西班牙 Biowest Agarose;普通琼脂糖凝胶DNA回收试剂盒DP209 天根生化科技(北京)有限公司;gold view II型核酸染色剂 Solarbiao;提取液等配制试剂 上海润捷化学试剂有限公司。
ZS-F6-20恒温水浴锅 山东桑泽仪器仪表有限公司;MixPlus涡旋振荡器 合肥艾本森科学仪器有限公司;5415D高速离心机 德国eppendorf;超微量分光光度计 Thermo Nano Drop2000;DYCP-32A琼脂糖水平电泳仪 北京六一仪器厂;T100 PCR仪、1708195 凝胶成像系统 美国Bio-rad。
1.2 实验方法
1.2.1 豆酱总DNA提取 根据Zhu等[19]提取DNA的方法,略有修改。取待测的5个豆酱样品各0.5 g装于2 mL EP管中,离心去上清液,在沉淀内加入0.75 mL提取缓冲液(100 mmol/L Tris-HCl,pH9.0,40 mmol/L EDTA),0.15 mL 10% SDS和0.45 mL苄基氯,50 ℃水浴35 min,每5 min取出上下摇动若干次,以保证提取物与试剂充分接触。水浴结束后,加入0.45 mL 3 mol/L NaOAc在冰上处理15 min。最后,10000 r/min离心10 min后,使用异丙醇沉淀DNA,梯度酒精洗涤晾干后用ddH2O溶解。分别用1%的琼脂糖电泳和超微量分光光度计检测DNA片段大小及浓度,并将其稀释至适宜的浓度[20],-20 ℃保存待用。
1.2.2 PCR扩增 以提取的豆酱总DNA为模板,细菌16S rDNA基因的V3~V4区通用扩增引物341F和806R[21],真菌ITS区通用扩增引物ITS3F和ITS4R[22-23](表1)为引物,结合高秀芝等[24]和Siddiqui等[25]的PCR反应体系和程序进行PCR反应。

表1 PCR扩增引物信息表Table 1 Information of PCR amplification primer
PCR反应体系(50 μL):10×PCR Buffer 5 μL,2 mmol/L dNTP mix 4 μL,45 pmol前后引物各5 unit/μL,Taq DNA聚合酶0.2 μL,DNA模板10 ng/μL 1 μL,ddH2O加至50 μL。反应程序:95 ℃预变性10 min;95 ℃变性30 s,48 ℃退火30 s,72 ℃延伸1 min,共35个循环;72 ℃终延伸7 min。
1.2.3 电泳检测及测序 通过1%琼脂糖凝胶电泳检测扩增产物,并对目的片段进行胶回收,将样品交由北京百迈客生物科技有限公司进行测序。
1.3 数据处理
原始测序序列使用Trimmomatic(v 0.32)软件对各样品序列做质控处理,利用软件Usearch(V 7.0)去除非靶区域序列、嵌合体及短片段序列;利用FLASH软件将测序得到的reads进行组装。在97%相似度条件下,按照序列间的距离对序列进行聚类,得到用于物种分类的 OTU(Operational Taxonomic Unit)序列。利用MOUTHUR软件进行OTU分布统计分析以及聚类比对的分类学分析。计算ACE/Chao/Shannon/Simpson/Coverage。利用RDP classifer(v 2.2)软件将OTU序列与数据库进行比对,获得物种注释,Excel 2007用以数据处理及分析统计作图。
2 结果
2.1 豆酱总DNA提取结果
通过琼脂糖凝胶电泳检测可知,5个样品在大于5000 bp处均获得目的条带,且条带清晰,说明豆酱总DNA提取成功,经超微量分光光度计检测可知基因组DNA的浓度及纯度OD260/OD280均在1.75~2.10的范围内,证明DNA纯度较好,没有污染,可用于后续的PCR扩增。
2.2 测序序列长度与分布
通过高通量测序结果可知,本研究五份豆酱样本中细菌(表2)共测得序列134115条,其中样本细菌长度分布在460~469 bp之间,通过过滤筛除低质量序列[26],得到有效序列共131462条,长度分布在427~430 bp之间;五份豆酱样本真菌(表3)共得到序列124301条,长度在332~350 bp范围内,得到有效序列120336条,长度309~312 bp范围内。

表2 细菌序列信息Table 2 Bacterial sequence information
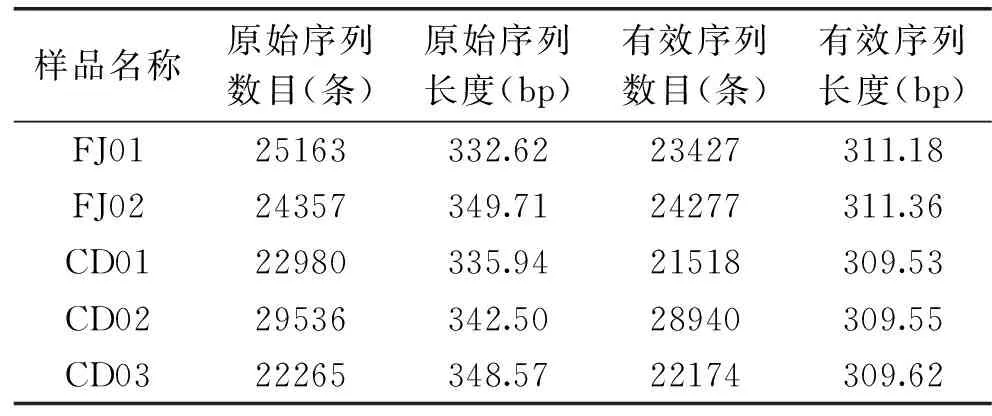
表3 真菌序列信息Table 3 Fungus sequence information
2.3 样品的OTU统计及分析
通过对序列结果随机抽样,统计序列数变化与OTU变化,从一个样本测序得到的所有序列中随机抽取序列(reads),统计每次抽取出来的序列有多少OUT(代表了有多少物种),最后以抽取的序列数目为横坐标,相应的OUT数目为纵坐标,绘制曲线图1,随着序列数增大,几个样品的曲线逐渐接近平行,即更大的序列不会导致OUT数目的显著性增长,证明以目前的数据进行分析是比较可靠的。

图1 样品稀释性曲线Fig.1 Dilution curve of sample注:A:样品中细菌OTU;B:样品中真菌OUT。
采用OUT统计工具bioinformatics.psb.ugent.be-Venn,将几个样本的测序结果按照操作提示录入工具中,然后分别对测序结果筛选出来的细菌真菌有效序列进行聚类,如图2,样品细菌在OTU聚类后共得到1732个OTU,其中有38个OTU是个样品共有的,样品FJ01特有333个OUT,FJ02特有193个OTU,CD01有117个,CD02有132个,CD03有190个。样品真菌得到1040个OTU,共有OTU为18个。样品FIJ01特有342个OTU,FJ02特有54个OTU,CD01有13个,CD02有41个,CD03有62个。

图2 样品OTU分布韦恩图Fig.2 Venn diagram of the sample OTUs distribution注:A:样品中细菌OTU;B:样品中真菌OUT。
2.4 群落多样性分析
由表4可知,本研究5个样品共得到细菌OUT 1732个,其中FJ01细菌样品最多,达到535个。CD02细菌最少,有267个。真菌OUT 1040个,其中FJ01样品最多,有515个。CD03真菌最少,仅111个。细菌总体数量和种类高于真菌,说明在豆酱成品当中,细菌占据微生物群落的主导地位。所有试验样品的OUT覆盖率(Coverage)均大于99%,即表明本次试验建立的文库能够较准确地反映出待测样品的微生物多样性情况。Chao、ACE指数用于计算菌群丰度(community richness),二者指数越大,说明群落丰富度越高的理论[27-28]。本试验细菌的Chao和ACE指数均大于真菌,说明试验样品中的细菌群落丰富度大于真菌群落;Shannon、Simpson用于计算菌群多样性(community diversity)指数,Shannon的值越大,说明群落多样性越高,Simpson指数值越大,则说明其群落多样性越低[29],因此结合表4中Simpson和Shannon指数,五种豆酱细菌多样性水平应该是CD01最高,CD02次之,CD03高于FJ01,最低的是FJ02;真菌多样性水平上,CD01远高于其他几个品种,这应该源于其生产方式,CD01属于手工自制酱,对灭菌做得不如工业生产。

表4 豆酱微生物群落多样性Table 4 Microbial community diversity in soy sause
2.5 群落组成
2.5.1 样品微生物在门上的分布 通过对五个样品的测序结果分析可知,样品中共有17个细菌门被检出,其种类丰富度较高,所占总序列数差异大,厚壁菌门(Firmicutes)和变形杆菌门(Proteobacteria)是主要的菌门。由图3A可得,厚壁菌门(Firmicutes)在样品FJ01、CD02、CD03中属于优势菌门,分别占总数的85%、79%、92%。变形杆菌门(Proteobacteria)是样品FJ02和CD01中的优势菌门,各占总数的79%和78%。除优势菌门以外还发现有以放线菌门(Actinobacteria)和拟杆菌门(Bacteroidetes)为主的其他菌门,但各自在样品中的总占比均较低。
样品在真菌中检测出丰富的菌门有子囊菌门(Ascomycota),比较丰富的有担子菌门(Basidiomycota)、接合菌门(Zygomycota),以及其他菌门。从图3B中可以看出,子囊菌门(Ascomycota)几乎是所有样品的优势菌门,分别占总数的99%、98%、70%、98%、90%。样品CD01中担子菌门所占比例也达到了近20%。

图3 样品在门上的分布情况Fig.3 Distribution of samples at the phylum level注:A:样品中细菌群落分布;B:样品中真菌群落分布。
2.5.2 样品微生物在属上的分布 在属的水平上,样品共检出39个细菌属。如图4A所示,样品FJ01和样品CD03中的优势菌属是葡萄球菌属(Staphylococcus),占各自总数的69%和64%。肠杆菌属(Enterobacter)在FJ01和CD03中也分别占到总数的19%和11%;在样品CD01中,肠杆菌属(Enterobacter)占到了总数的53%,乳酸杆菌属(Lactobacillus)占总数的20%,明串珠菌也达到了16%;FJ02中的优势菌群为芽孢杆菌属(Bacillus),其丰度达到了45%,色盐杆菌属(Chromohalobacter)32%,葡萄球菌属为7%;CD02中乳酸杆菌与肠杆菌属分别占到了总数的22%和20%,肠球菌占到了30%;魏斯氏菌属(Weissella)在除CD03以外中均有占到3%以上。肠杆菌、乳酸杆菌、色盐杆菌为5个样品共有细菌,各个样品间微生物群落丰度差距很大,这可能与几种材料产自不同地区有关。此外各样品中相对丰度大于1%的还有双歧杆菌(Bifidobacterium)、雷尔氏菌属(Ralstonia)、伯克霍尔德氏菌属(Burkholderia)。
样品中共检测出43个真菌属,如图4B所示各样品间的优势菌的种类和数量同样差异显著,曲霉属、接合霉属、赤霉属、毛霉属为5个样品共有的真菌属,但丰度上差异明显。FJ01中的优势真菌是接合酵母属,序列丰度为70%,FJ02、CD01、CD02、CD03 中该菌属的序列丰度分别为10%、18%、30%、11%,可见接合酵母属在几个材料的真菌中数量都相对比较丰富。在 FJ02中相对丰度最高的真菌属为曲霉属。FJ02、CD02、CD03中均含有德巴利氏酵母属(Debaryomyces)。CD02中菌属种类多,分布比较均匀都在10%~30%左右,因此其优势并不能判定其优势均属。CD03中优势菌属十分明显,属于赤霉菌属,占到了60%。

图4 样品在属上的分布情况Fig.4 Distribution of samples on genus level注:E:样品中细菌群落分布;F:样品中真菌群落分布。
3 讨论
本研究采用Illumina MiSeq高通量测序技术对东北不同产地的5份豆酱进行高通量测序,检测到豆酱食品中含有数量庞大的真菌和细菌微生物群体,包括葡萄球菌、乳酸杆菌、德巴利氏酵母属、芽孢杆菌等大量的发酵食品优势菌属。焦玉[30]通过对火腿肠的风味物质以及微生物检测中就发现,葡萄球菌是一类可以通过水解脂肪从而提高发酵香肠中游离脂肪酸的含量,促进脂肪分解的优势菌属;样品FJ01和CD03的葡萄球菌占比均超过60%。袁钰等[31]通过高通量测序对北京豆汁儿的微生物多样性和功能研究,也发现了乳酸杆菌在对豆汁儿中的氨基酸代谢具有重要作用,益于肠道菌群健康发酵;在糟鱼发酵过程中,芽孢杆菌、乳酸杆菌具有产蛋白酶和脂肪酶的特性,酵母菌具有产脂肪酶的特性,能够更好的促进发酵品质以及风味被李改燕[32]证实。
研究中对豆酱微生物群落结构分析后,发现几种样品的细菌真菌多样性在门和属的水平上均呈现出较大的差异,说明在豆酱生产中其微生物多样性可能受到地域的影响,同时也很好的解释了造成不同地区生产的豆酱风味差异的一部分原因是豆酱内部微生物的差异;不同加工方式对于豆酱中微生物多样性的影响也存在较大影响,例如实验中手工制作的豆酱CD01,或许因为在灭菌过程中处理得并不彻底而导致真菌多样性相对几种工业酱有显著的差距,其中不乏有对人体有害的菌类;此外实验结果对了解豆酱中微生物群落组成和微生物多样性能够提供一定的数据基础。在今后地方特色的豆酱生产、保藏以及优势菌种选育中,可以参考本研究中的各个样品的试验结果,来针对不同产地以及发酵天数的豆酱中优势菌属进行生产处理,以达到最佳的效果。
