基于加权基因共表达网络分析识别心肌肥厚中的枢纽基因
2020-06-06付一乐彭富华郑宁泽蔡少仪中山大学中山医学院药理学教研室广州510080共同第一作者通讯作者mailwangqin6mailsysueducn
付一乐,陶 亮,彭富华,郑宁泽,林 青,蔡少仪,王 琴(中山大学中山医学院药理学教研室,广州 510080;共同第一作者;通讯作者,E-mail:wangqin6@mail.sysu.edu.cn)
心肌肥厚(cardiac hypertrophy,CH)是心肌细胞对多种刺激因素所产生的适应性反应[1]。这是一个复杂的病理过程,其病理变化包括心肌细胞肥大、心肌间质细胞增殖以及心脏细胞外基质改建等多方面的改变,即心肌重构[2]。心肌肥厚是许多心脏疾病发展的一个重要阶段,但是心肌肥厚发生的分子机制非常复杂,人们对心肌肥厚发生的确切分子和细胞信号转导机制仍知之甚少[3]。
近年来,随着分子生物学和细胞生物学等技术的应用,有关心肌肥厚发生发展中机制方面的研究取得了一定进展,主要集中在JAK-STAT信号通路,Ca2+介导的信号转导通路、有丝分裂原活化蛋白激酶信号转导通路等[4,5]。但是,仅仅从局部关注单个或某几个基因已经不能满足这种具有高度复杂性的调控研究。立足于调控网络整体,心肌肥厚中某些基因异常表达且与其他多个基因关系密切,它们的表达可能在心肌肥厚的发生发展过程中扮演重要的角色,这类处于调控网络枢纽位置的基因称作枢纽基因(hub genes)[6,7]。近年来,随着基因芯片、RNA-Seq等高通量测序技术的兴起,产生了大规模组学数据,进一步数据分析与挖掘将有利于系统性地研究多个基因表达关联性。因此,根据基因芯片数据来研究心肌肥厚发生与发展过程中的枢纽基因具有明显意义[8]。
在生物信息学领域中,网络分析的应用日渐成熟。其中,作为系统生物学分析方法中极具潜力的一种新方法,加权基因共表达网络分析(weighted gene co-expression network analysis,WGCNA)以其在描述解析分子作用机制和网络关系等方面具有的独特优势迅速脱颖而出[9]。自开发以来,WGCNA已成功应用于多种生物环境,并取得了一系列的研究成果。
本研究采用WGCNA进行研究,从大量基因中筛选与心肌肥厚密切相关的枢纽基因。结合GO富集分析,进行功能注释,然后对基因模块进行KEGG信号通路分析,研究枢纽基因与心肌肥厚发生与发展间的关联性,这些工作能够为某些信号通路的调控机制提供参考,有利于构建评估心肌肥厚早期诊断预测平台,降低人群心肌肥厚发病率及死亡率。有理由相信,随着基因筛选方法的不断发展,WGCNA有望揭示心肌肥厚发生与发展的分子机制,并在识别潜在的治疗靶点等领域发挥越来越大的作用。
1 材料与方法
1.1 GEO全基因组表达数据
从NCBI的GEO下载心肌肥厚的全基因组表达数据GSE36961。该数据集发表于2012年9月,包括106个肥厚型心肌病患者的心脏组织和39个正常对照组的心脏组织,提取mRNA后,用Illumina HumanHT-12 V3.0芯片检测全基因组基因表达。
1.2 共表达网络的构建和模块的识别
利用R-3.6.2软件运行“WGCNA”包[9]。为降低运算量,计算每个基因的倍数差异值(取对数变换),并推断差异发生的概率P,当倍数差异值大于预设倍数,且筛选P<0.05时即判定为差异基因(即该基因表达无差异的概率小于0.05)。将筛选出的差异基因用于共表达网络的构建。采用样本聚类树方法,根据聚类图剔除离群样本来保证构建稳定的共表达网络。构建无尺度网络使基因共表达网络符合无尺度现象,以无尺度网络指数(R2)=0.9作为满足无尺度条件的标准,同时根据平均连接度确定软阈值(β)。利用拓扑重叠矩阵(TOM)、相异常度矩阵计算基因与基因间的关联程度;对基因构建层次聚类树图形,采用动态剪枝法计算基因模块颜色。计算基因模块的特征值(ME),引入临床信息,对ME进行分层聚类并绘制树状图,设置高度值0.25为分割线,合并相似程度较高的基因模块,再用剪切后的模块绘制新的聚类树和模块图。
1.3 基因富集分析
基因本体(GO)及京都基因与基因组百科全书(KEGG)通路富集广泛应用于高通量基因数据功能分析。DAVID数据库为研究人员提供了一套全面的功能注释工具,以了解大量基因背后的生物学意义[10]。本研究采用DAVID数据库对关键网络模块进行GO富集注释分析,其中包含生物过程(biological process,BP)、细胞成分(cellular component,CC)、分子功能(molecular function,MF)以及KEGG通路富集分析。
1.4 蛋白-蛋白互作网络构建及枢纽基因识别
STRING数据库是用于预测蛋白质-蛋白质相互作用(protein-protein interaction,PPI)的生物学数据库[11],可用于检索经实验验证或基于文献预测的蛋白/基因相互作用。为了进一步探讨关键网络模块的生物学功能,本研究应用STRING数据库构建靶点PPI网络。在数据库中将蛋白种类设置为“Homo sapiens”(人类)进行操作,最低相互作用阀值设为高置信度“high confidence≥0.4”,其余参数均保持默认。进一步将获得的PPI数据导入Cytoscape(Version 3.7.2)进行可视化分析,并应用Network Analyzer分析模块进行网络拓扑结构计算,依据节点度(degree)值识别网络枢纽基因,其值越大,表明与该节点相连边越多,在维持网络整体结构上可能占据重要地位。
1.5 心肌肥厚模型大鼠的建立
选用由北京维通利华实验动物技术有限公司提供的健康状况良好,清洁级、16周龄、雄性WKY大鼠3只为对照组(WKY组),自发性高血压大鼠3只为心肌肥厚模型组(SHR组),合格证号:SYXK(粤)2015-0107,饲养于中山大学SPF级实验动物中心,饲养温度20-25 ℃,湿度40%-60%,光照节律:06∶00-18∶00,保持通风良好。
1.6 左心室质量指数和全心质量指数的测定及心脏组织的HE染色
在取材之前测量大鼠体质量(BW),动物麻醉后,迅速取出大鼠心脏,滤纸吸干,沿房室分隔处分离心房及右心室,保留室间隔及左心室,称左心室质量(LVW)和全心质量(HW),计算左心室质量指数(LVWI)=左心室质量(LVW)/体质量(BW)和全心质量指数(HWI)=全心质量(HW)/体质量(BW)[12]。取2/3心脏组织10%福尔马林固定,石蜡包埋,沿左室长轴常规切片,行苏木素-伊红染色(HE染色),在光镜下观察心脏的病理改变[13]。
1.7 RT-qPCR检测枢纽基因表达
按照TRIzol试剂盒说明书从SHR模型组大鼠和WKY对照组大鼠左心室区域提取总RNA,用RT-qPCR检测APOB、CCND1、SOX9、MYC、APP mRNA水平表达。采用SYBR Premix Ex TaqⅡ试剂盒,应用实时定量PCR扩增仪(ABI StepOne Plus)检测每个扩增循环后SYBR Green荧光信号水平。PCR扩增条件:94 ℃ 30 s,58 ℃ 15 s,72 ℃ 10 s,共计40个循环。收集荧光信号,建立熔解曲线,运用熔解曲线检测扩增产物纯度。采用相对定量2-ΔΔCt法对获得的Ct值进行分析。用Student’st检验分析枢纽基因在心肌肥厚与正常对照之间的差异[13]。由上海生工生物工程公司合成引物,序列见表1。

表1 引物序列Table 1 Primer sequences for PCR
1.8 统计学方法
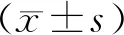
2 结果
2.1 GEO全基因组表达数据基本信息
GSE36961全基因组表达数据包括106个模型组与39个对照组,这些数据均已经过背景校正和标准化处理。为了减轻噪音和减轻计算机的运行负担,通过LIMMA包[14]滤过差异并不显著的基因,最后保留6 321个差异基因(|logFC|>1,FDR<0.05)作为本文的重点研究对象。
2.2 样本数据初筛
采用层次聚类法得到样本聚类图,发现145个样本表达谱数据中存在4个离群的样本(见图1),离群样本经过剪枝去除(剪枝高度为50),最后剩余106个心肌肥厚样本和35个正常组样本进入后续的网络分析。

采用层次聚类法得到样本聚类图,发现在聚类高度50(红线标记)时聚类树形图发生明显偏移,剪枝去除4个离群样本,最后剩余141个样本
2.3 阈值选择与网络拓扑结构分析
选择合适的领接矩阵权重参数βg以尽量满足无尺度分布。参数βg从1-20取值,对某节点连接度的对数logi与该节点出现的概率的对数logp(i)建立线性模型,参数βg即系数R的平方,按照候选的参数β,返回被检测的网络参数(见图2)。我们选取了首次接近0.90时的βg值来构建基因网络,最终我们选取β=7,这样既保证了网络接近于无尺度网络同时也是使曲线趋于平滑的最小阈值,并且它使得网络的平均连接程度不会太小,这有利于网络包含足够的信息。
图2 软阈值参数网络拓扑结构的分析Figure 2 Analysis of network topology for soft thresholding parameters
2.4 TOM聚类鉴定基因模块
选取β=7来计算网络拓扑重叠TOM,利用层次聚类法得到基因聚类树。根据动态分层剪切树算法来挖掘基因模块。设置每个基因模块中基因数目最小为30,设定基因的聚类高度上限为0.995。最终得到11个网络模块,模块中的基因个数从34到1 160不等(见图3)。另外,Grey模块表示未分配到任何一个模块的基因集,其中含有122个基因。
每个分支代表一个基因,下面的每种颜色代表一个网络模块
2.5 模块关联度分析与鉴定关键模块
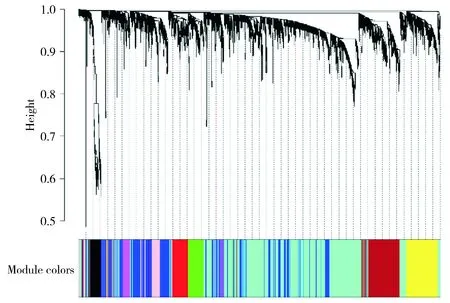
根据拓扑重叠程度,选择全部的基因制作热图(见图4)。通过WGCNA,我们识别了心肌肥厚中相关度高的基因模块,即共表达模块代表的基因在功能上具有紧密的关系。而且,通过层次聚类法对11个网络模块进行网络聚类,结果表明11个网络模块被分成了两组(见图5A)。进一步分析发现,在11个网络模块中,Blue模块和Turquoise模块与心肌肥厚组具有高相关性(见图5B),因此,我们选择Blue模块和Turquoise模块作为心肌肥厚疾病的关键模块作进一步的功能分析。
不同的颜色代表不同的模块,图中间的黄色密度表示不同模块之间的关联性

图B中每一列代表一个组别,每一行对应一个模块特征基因,颜色表示相关性大小
2.6 关键模块的功能分析与信号通路分析
经过GO富集分析,我们得到了Blue模块和Turquoise模块的详细功能(见图6A和图6B),发现Blue模块中的基因主要参与肌细胞发育、表皮细胞增殖、氨基多糖结合、胶原结合、TGF-βg受体结合等分子功能和生物学过程及信号通路,而Turquoise模块中的基因主要参与免疫系统调节、刺激反应、炎症反应、趋化因子和细胞因子介导的炎症反应信号通路等分子功能和生物学过程,这可能与心肌肥厚的发生有一定的联系。
另外,我们还对Blue模块和Turquoise模块进行了KEGG信号通路富集分析(见图6C和图6D),发现Blue模块富集于10条KEGG信号通路,包括TGF-β信号通路、Wnt信号通路、凋亡信号通路等,而Turquoise模块富集于64条KEGG信号通路,包括AMPK信号通路、PPAR信号通路、肾素-血管紧张素系统、IL-17信号通路、凋亡通路、缝隙连接、TNF信号通路和吞噬体等。

BP:生物过程;CC:细胞组成;MF:分子功能
2.7 枢纽基因识别与共表达网络可视化
通过比较模块内连通性和基因重要性可以帮助找到一些与心肌肥厚相关的枢纽基因。在模块中与其他基因关联性越多的基因,往往在模块中的作用越重要,Blue模块和Turquoise模块中在右上角的基因既与其他基因相关性高又对心肌肥厚重要(见图7A和7B)。因此我们用Cytoscape软件分别对Blue模块和Turquoise模块构建蛋白-蛋白相互作用网络,特别关注模块内连通性最大的30个基因。结果发现,Blue模块和Turquoise模块均存在多个枢纽基因,如APOB、SOX9、CCND1等(见图7C和7D)。
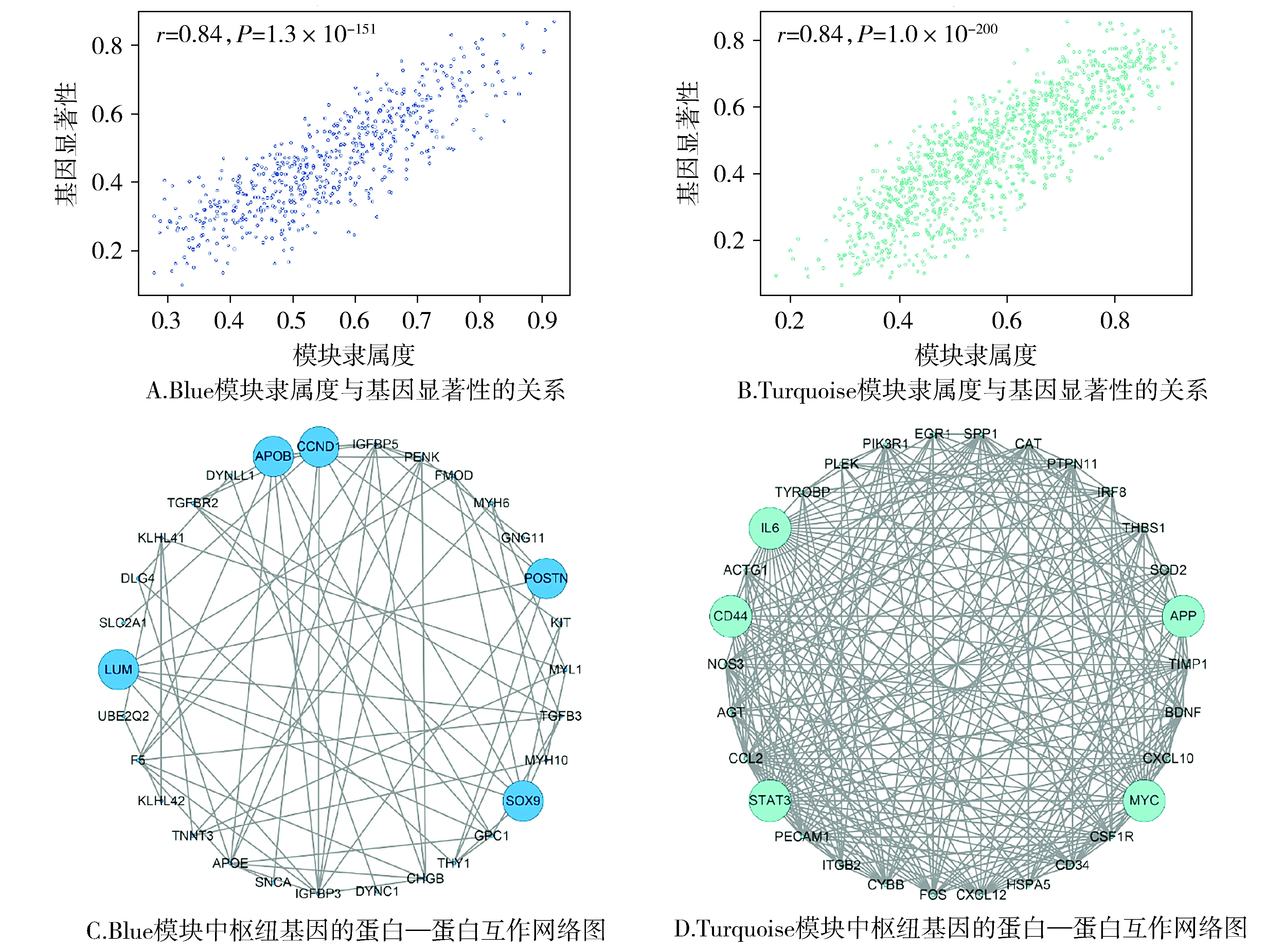
对Blue模块和Turquoise模块内连通性最大的30个基因构建PPI网络,应用Network Analyzer计算节点度(degree)
2.8 枢纽基因的验证
将3只WKY大鼠作为正常组,3只SHR大鼠作为模型组,通过比较左心室质量指数(LVWI)和全心质量指数(HWI)来观察SHR模型组的心肌肥厚情况。结果发现,模型组的LVWI和HWI分别为2.23和3.56,与正常组比较差异均具有统计学意义(P<0.05,见图8A),提示SHR大鼠的左心室已出现心肌肥厚。另外,HE染色光镜下可见:正常组心肌细胞结构清晰、排列整齐,细胞间隙均匀,未见心肌排列纤维增生;与正常组比较,模型组心肌细胞增大、排列紊乱,细胞间隙增宽,可见大量的胶原纤维增生,炎性细胞浸润在组织周围,提示SHR大鼠心肌细胞已出现病理损伤。RT-qPCR实验结果显示(见图8)。与WKY大鼠相比,SHR大鼠心肌组织中APOB、SOX9、CCND1、MYC和APP mRNA水平分别升高1.57倍、1.81倍、2.64倍、1.99倍和2.61倍,差异均具有统计学意义(P<0.05)。
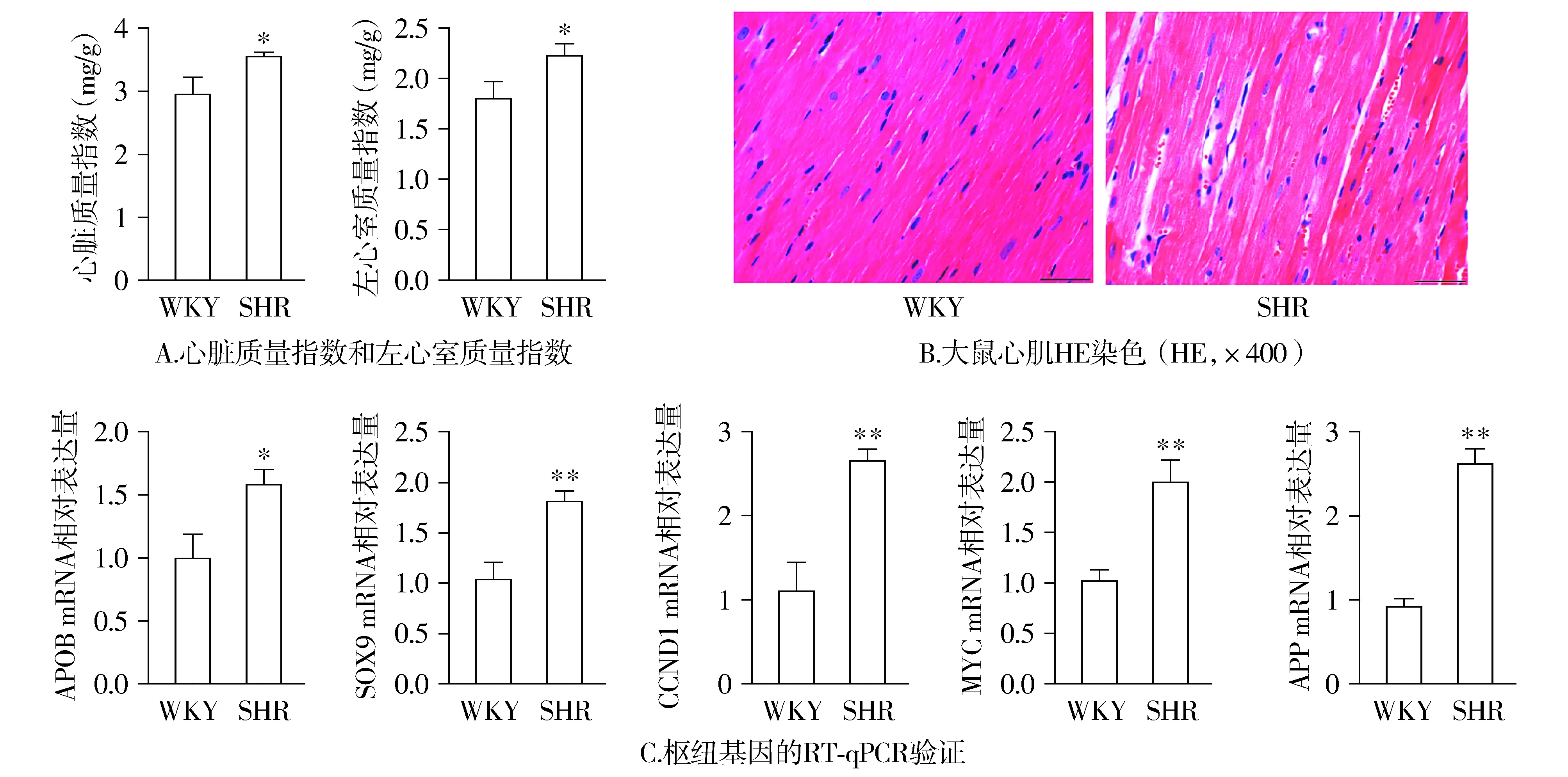
与WKY组相比,*P<0.05,**P<0.01
3 讨论
目前学术界普遍认为,心肌肥厚发生与发展的情况异常复杂[15],不仅与压力负荷、容量负荷、机械负荷相关,还与神经体液因子密切相关[16]。心脏为适应各种刺激而产生的心肌细胞体积增大和质量增加虽然有一定的代偿意义,但过度刺激将造成心肌缺血、心脏收缩力下降和输出功能障碍等,最终可致扩张性心肌病、心力衰竭和猝死[16]。在此过程中,心肌细胞内发生一系列信号转导方面的变化。近年来,人们发现Ca2+介导的信号转导通路、有丝分裂活化蛋白激酶信号转导通路、Janus激酶/信号转导分子和转录激活子信号转导通路在心肌肥厚的发病过程中发挥重要作用,但大部分的调控关系仍处于长期探索中[4]。
本研究首先从NCBI的GEO数据库下载心肌肥厚的表达数据集GSE36961(含145个样本),利用WGCNA模拟基因簇筛选出了与心肌肥厚相关的基因模块并对模块内基因进行GO功能注释和KEGG通路富集分析,这些基因主要富集在免疫系统调节、刺激反应、炎症反应、趋化因子和细胞因子介导的炎症反应信号通路等分子功能和生物学过程。需要指出的是,已经有研究表明NF-κB信号通路可能在心肌肥厚的发生发展中起重要作用[17]。NF-κB信号通路发挥作用主要是作为介导炎性反应的细胞内信号转导通路。炎性细胞因子,如:肿瘤坏死因子α(TNF-α)和IL-1等可能是介导NF-κB与心肌肥厚有关的介质。通过采用NF-κB活性抑制剂阻断NF-κB的激活,可以显著抑制心肌细胞肥大的进展[17]。另外需要值得注意的是,部分模块同时富集到凋亡通路、缝隙连接和吞噬体等信号通路,提示这些通路模块中的基因与心肌肥厚的发生发展也可能有着内在关联,这为心肌肥厚的发生发展提供了新的研究思路。
我们还进一步通过Cytoscape软件分别对Blue模块和Turquoise模块构建蛋白-蛋白相互作用网络,将枢纽基因可视化,发现了与心肌肥厚显著相关的基因模块内的枢纽基因,如APOB、CCND1、SOX9、MYC和APP等。通过进一步在自发性高血压大鼠心肌肥厚中验证枢纽基因,发现APOB、CCND1、SOX9、MYC和APP mRNA水平均显著上调。APOB基因编码产物是乳糜颗粒和低密度脂蛋白的主要载脂蛋白,并且是低密度脂蛋白受体的配体。以往研究发现,APOB异常与动脉粥样硬化性心血管疾病包括冠心病、脑卒中和周围动脉疾病密切相关。但是近年来有研究报道,降低APOB水平的阿托伐他汀药物在治疗心肌肥厚中也能够发挥改善作用[18],再结合我们当前的发现,APOB基因的异常可能在心肌肥厚的发生发展中有重要作用,这有待进一步的研究工作来验证。
综上所述,加权基因共表达网络分析方法是一个高效的系统生物学方法,应用本方法我们发现了心肌肥厚的枢纽基因APOB、CCND1、SOX9、MYC和APP。我们当前的工作有助于为心肌肥厚发生的调控机制的研究以及治疗诊断、靶点选择提供更多的参考信息。
