染色体核型分析和基因芯片检测在产前超声胎儿异常中的应用▲
2020-06-01李军红
李军红
(广西柳州市人民医院优生遗传门诊,柳州市 545001,电子邮箱:ljhong0725@sohu.com)
中国是出生缺陷高发国家之一,出生缺陷发病率为5.6%[1],除了明显的严重畸形外,还包括一些微小畸形,这些超声异常与胎儿染色体异常存在着一定的相关性,其中染色体畸变约占出生缺陷遗传病因的80%以上[2]。近年来超声医学不断发展,为临床诊断提供更多的人体组织生理和病理变化信息。超声检查兼有无创、便捷、价廉、实时、动态等优点,产前超声是筛查宫内胎儿畸形的首选影像学方法,具有较高的敏感度,有利于降低出生缺陷发生率,对干预出生人口质量具有重要的临床价值[3-5]。
长期以来,染色体核型分析技术是确诊染色体畸变的金标准,而在产前超声提示多发结构畸形胎儿中,染色体核型分析的阳性率更高[6]。但其检测周期长、分辨率低,无法检测5 Mb以下的拷贝数变异(copy number variation,CNV)。研究表明,将染色体微阵列分析(chromosomal microarray analysis,CMA)基因芯片技术运用于胎儿超声异常相关的CNV检测,可为超声提示一个或多个结构异常的病例诊断提供有效的信息[7]。本研究应用染色体核型分析和基因芯片检测等遗传学检测方法,对112例产前超声提示胎儿异常的羊水或绒毛组织样本进行检测,从而综合评估胎儿预后情况,减少出生缺陷。
1 资料与方法
1.1 临床资料 选取2018年1月至2019年7月我院产前超声提示胎儿异常(结构异常或软指标异常)的112例孕妇,纳入标准:(1)单胎妊娠;(2)均有完整的临床资料。排除标准:(1)非自然妊娠者;(2)未接受跟踪随访或失访者。孕妇年龄22~43岁,平均32.5岁;孕周11~25周,平均18周。所有孕妇经遗传咨询自愿签署知情同意书进行染色体核型分析及基因芯片检测。其中99例行羊膜腔穿刺术取羊水进行检测,13例引产后取胎盘绒毛组织进行检测。
1.2 标本取材 对妊娠17~25周的孕妇在超声引导下定位穿刺点,消毒腹部皮肤,无菌操作下采用一次性腰穿针经腹抽取羊水26 mL,其中16 mL用于染色体核型分析,10 mL用于基因芯片检测。对于妊娠11~17周的孕妇,将胎儿引产后取胎盘绒毛组织,用生理盐水冲洗干净后存放于生理盐水玻璃瓶中,用于染色体核型分析及基因芯片检测。
1.3 羊水细胞G显带染色体核型分析 采用ELITIST-5K型台式离心机(鑫奥仪器仪表有限公司)将羊水以1 500 r/min离心10 min后,弃去上清液,取2 mL接种于羊水培养基(广州白云山拜迪生物医药有限公司,批号:20190601)中,在5% CO2、37℃培养箱中培养1周,观察细胞生长情况,镜下见多个克隆时收集细胞备用。羊水细胞的收集、制片和显带:向培养瓶中加入20 μg/mL秋水仙碱50 μL,放入5% CO2、37℃培养箱中培养4 h,将贴壁的羊水细胞用0.25%乙二胺四乙酸胰酶消化5 min,脱落后倒入离心管,以1 500 r/min离心10 min后弃去上清液;加入0.075 mol/L氯化钾4 mL,以37℃水浴4 min,加入1.5 mL固定液(甲醇与冰醋酸按体积比3 ∶1混合),再次以1 500 r/min离心10 min,弃掉上清,在沉淀物中加入8 mL固定液(甲醇与冰醋酸按体积比3 ∶1混合),滴片,置于80℃烤箱中烘烤1.5 h,采用胰消化酶消化后以Giemsa染色显带。每份羊水标本于镜下观察30个细胞分裂像,采用Karyo 3.2分析系统进行染色体核型分析,当出现异常核型或嵌合型,则扩大分析至50个细胞分裂像。
1.4 绒毛组织染色体多重连接探针扩增分析
1.4.1 DNA提取:使用灭菌剪刀与镊子采集适量绒毛组织置入研磨管中,按照试剂盒说明书(MRC-Holland公司,批号:P290-B1)进行DNA提取。于紫外分光光度计中测量DNA浓度及纯度并稀释,要求浓度为(30±5)ng/μL,A260/280为1.6~2.0。
1.4.2 DNA裂解变性:取4 μL DNA样品小心加入做好标记的PCR管底部(为防止反应液挥发,使用进口PCR管),98℃加热20 min后马上取出,置于冰上3~5 min,注意动作要迅速以防止降温导致变性失败。
1.4.3 DNA杂交:在变性后的DNA样品中加入1.5 μL杂交液(0.75 μL多重链探针缓冲液+0.75 μL 探针混合液036/070,冰上配制,震荡混匀),放入PCR仪(罗氏公司)中杂交,反应条件为95℃ 5 min、60℃ 16~20 h;温度降至54℃。
1.4.4 连接反应:在杂交产物中加入16 μL连接酶混合液(ddH2O 12.5 μL+连接酶缓冲液A 1.5 μL+连接酶缓冲液B 1.5 μL+Ligase enzyme 0.5 μL,冰上配制,用移液器轻轻吹打混匀后),54℃温浴20 min,接着98℃加热5 min使连接酶失活,温度降至20℃。
1.4.5 PCR反应:加入5.05 μL PCR反应液(ddH2O 3.8 μL+SALSA PCR引物混合液1 μL+PCR聚合酶0.25 μL,用移液器轻轻吹打混匀),进行PCR 反应,反应条件为95℃ 30 s、60℃ 30 s、72℃ 1 min(共35循环)、72℃ 20 min,15℃维持。
1.4.6 上机检测:配制上样缓冲液,即80 μL甲酰胺+1 μL DNA Size Standard-600。96孔板中每孔加入9 μL上样缓冲液及1 μL PCR产物,吹打混匀。加样时小心加入96孔板中的底部(不可产生气泡),再加上1滴液状石蜡覆盖。参照Beckman GeXP遗传分析仪操作说明进行检测并采集数据,使用Coffalyser.Net软件分析结果。
1.5 羊水或绒毛组织CMA基因芯片分析 (1)取10~15 mL羊水或使用生理盐水收集的绒毛组织,1 000 r/min离心10 min,取沉淀物转入1.5 mL的EP管。加入20 μL蛋白酶K及4 μL RNase A反应30 min以溶解蛋白及去除RNA(组织样本需先加入200 μL ATL,56℃金属浴10 min对组织进行初步消化处理)。(2)样本加入200 μL AL缓冲液 ,并56℃金属浴10 min对样本进行消化,再加入200 μL无水乙醇沉淀DNA,转入离心柱含收集管组件中,8 000 r/min离心1.5 min,去除废液;往离心管中加入500 μL AW1缓冲液,8 000 r/min 离心1.5 min,去除废液;往离心管中加入500 μL AW2缓冲液,14 000 r/min离心 1.5 min,重复2次,第2次离心时长为3 min,以去除离子、蛋白等杂质;将收集管去除,离心柱转至1.5 mL EP管,滴加35 μL AE缓冲液至离心管中,放置5 min,8 000 r/min 离心1.5 min洗脱回收DNA,使用Thermo Scientific NanoDrop 2000紫外分光光度计测定DNA质量。(3)取400 ng样本及参比DNA转入八联管不同孔中,每孔加入2.5 μL随机引物,95℃ 3 min热片段化。配置含有荧光素的标记混合液,样本DNA的标记混合液包含5 μL 5×反应缓冲液、2.5 μL 10×dNTPs、1.5 μL Cy 5-dUTP荧光素、0.5 μL Exo(-)Klenow酶,参比DNA的标记混合液包含5 μL 5×反应缓冲液、2.5 μL 10×dNTPs、1.5 μL Cy 3-dUTP荧光素、0.5 μL Exo(-)Klenow酶。将含有荧光素的标记混合液加入每个样本及参比DNA中。放入37℃水浴孵育2 h,转至65℃水浴10 min灭活酶,后置于冰上或保持4℃温度内避光。使用480 μL 1×TE(pH值为8.0)对已标记样本和参比DNA进行两次纯化,之后对样本和参比DNA进行浓缩干燥,并使用定容1×TE(pH值为8.0)至8 μL,使用Thermo Scientific NanoDrop 2000紫外分光光度计测定DNA质量,然后将已纯化好标记这两种不同荧光素的样品和参比DNA按1 ∶1等浓度配比混合转入八联管中。(4)配置杂交混合液,包含2 μL Cot-1 DNA(1.0 mg/mL)、4.5 μL 10×aCGH Blocking Agent、22.5 μL 2×HI-RPM杂交缓冲液;将杂交混合液加入每个样本八联管中,放入98℃孵育3 min,再转入37℃ 孵育30杂交混合液,对非特异重复序列封闭进行预杂交。预杂交完成后将样本再转移进芯片孔中进行封片,放入65℃杂交炉杂交24 h。 (5)用玻璃洗缸提前备好两缸清洗液1和一缸清洗液2,清洗液2提前2 h预热至37℃。完成杂交的芯片下杂交炉后在清洗液1中拆开封片,将芯片架入在清洗液1中洗涤5 min,转架入清洗液2中洗涤1 min,缓慢提起。(6)清洗好的芯片装入芯片架,通过Agilent SureScan Dx微阵列芯片扫描仪进行激光扫描,Cy5激发后为红光,Cy3激发后为绿光,比较两种荧光激发后的光强,当红光更强时提示样本染色体区域可能存在拷贝数增加,绿光更强时提示样本染色体区域可能存在拷贝数缺失,使用Agilent CytoGenomics 2.7.22.0分析软件将信号数据荧光信号强度转换成Log Ratio值后呈现每个样本的结果。
本实验采用香港中文大学设计的Fetal DNA Chip Version 2.0,包含6万个探针。通过查询OMIM(https://www.omim.org/)、UCSC(http://genome.ucsc.edu/)、ISCA(https://www.iscaconsortium.org/)、DGV(http://dgv.tcag.ca/dgv/app/home)、DECIPHER(https://decipher.sanger.ac.uk/)等数据库,确定基因组DNA CNV的类型及临床意义,必要时需采集双亲的外周血,以确定其来源并进行临床分析。根据美国医学遗传学与基因组学会发布的《序列变异解读标准和指南》[8]对结果进行判读,检测结果分为致病性CNV、临床意义不明CNV及良性CNV。
2 结 果
在112例产前超声异常的胎儿中,检测出17例染色体核型异常,其中21-三体3例,18-三体1例,性染色体异常2例,平衡易位2例,染色体正常多态性变异8例,嵌合体1例;12例基因芯片异常,包括7例致病性CNV和5例临床意义不明CNV;有6例同时存在染色体核型异常和基因芯片异常;有6例染色体核型正常而基因芯片异常,包括4例临床意义不明CNV和2例致病性CNV;有11例染色体核型异常但基因芯片正常,包括染色体平衡易位2例,倒位4例,多态性变异5例。见表1。
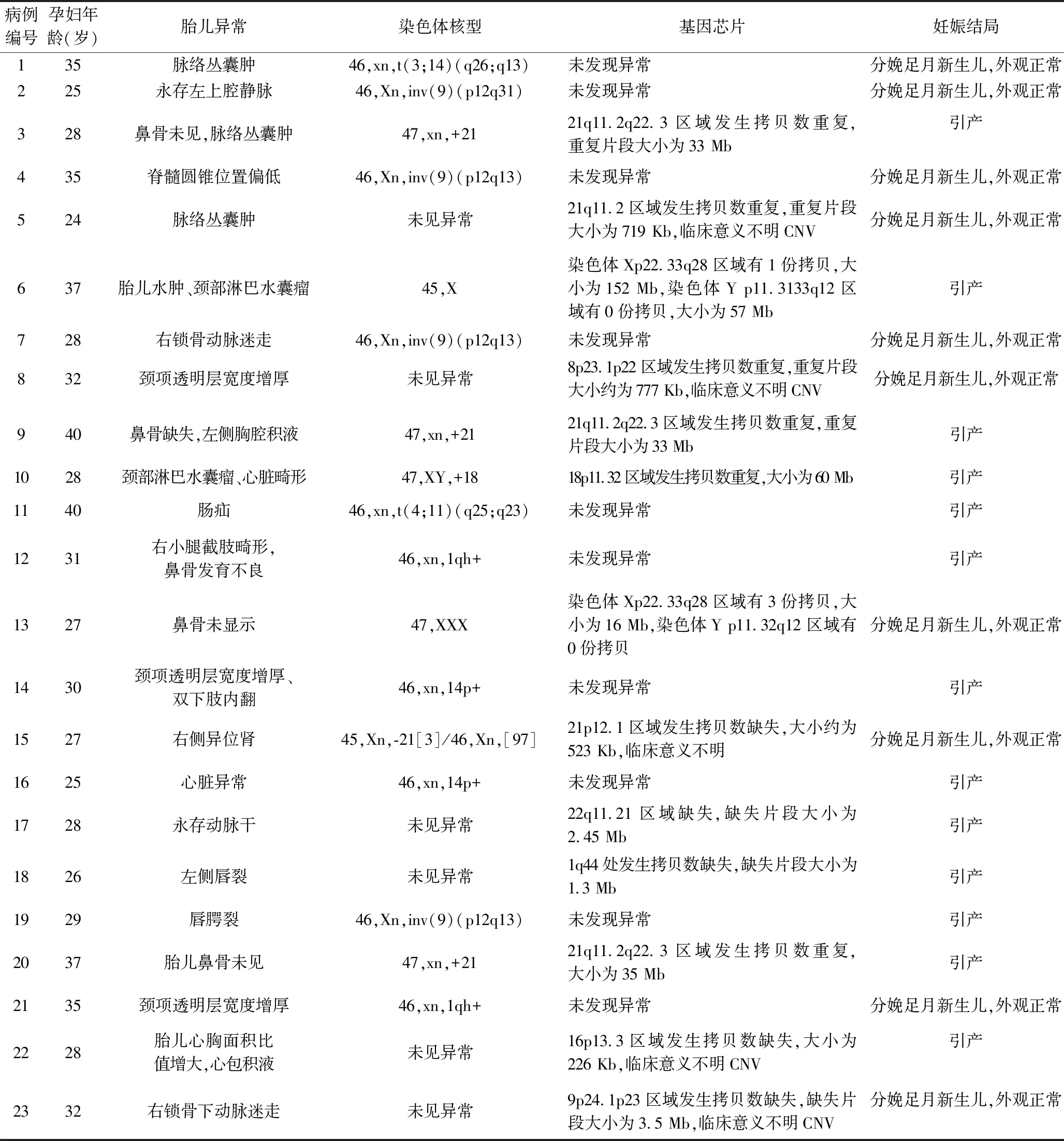
表1 23例染色体核型或基因芯片异常病例的检测结果及妊娠结局
3 讨 论
本研究中112例产前超声异常的胎儿中,共17例染色体核型异常,其中8例为异常染色体(包括21-三体3例,18-三体1例,性染色体异常2例,平衡易位2例),检出率为7.14%(8/112),高于一般人群染色体疾病的发病率(0.5%)[9],这提示产前超声检查异常的胎儿可能有较高的染色体异常风险。
长期以来,染色体核型分析技术是染色体畸变产前诊断的“金标准”,但其培养细胞及检测周期长,分辨率低,未能检测出5Mb以下的CNV。有学者发现,在染色体核型分析正常但超声结果异常的胎儿中,有6%~7%的胎儿存在明显致病性或可能致病性的CNV[10-12]。因此,传统的胎儿染色体检测方法已经不能满足临床对疾病诊断的需求,而结合基因芯片检测技术对染色体数目异常、大片段缺失、重复以及致病性基因组CNV等染色体畸变进行及时、准确的产前诊断,将有利于进一步减少活产儿的严重出生缺陷[13]。本研究中,共检出12例基因芯片异常,其中有6例基因芯片异常但染色体核型正常,占5.36%,包括4例临床意义不明CNV和2例致病性CNV。由此可见,进行CMA基因芯片技术检测有助于染色体CNV的检出。在本研究染色体核型正常而基因芯片异常的胎儿中,1例胎儿超声提示永存动脉干,检测染色体核型正常,但基因芯片发现染色体22q11.21区域缺失,缺失片段大小为2.45 Mb;我们将胎儿不良预后告知后,孕妇及家属最终选择终止妊娠,同时建议孕妇及其丈夫行基因芯片检测,确定胎儿异常病因来源以指导再次妊娠。因此,CMA基因芯片技术还有利于在产前临床咨询中正确评估胎儿预后,为孕妇是否继续妊娠提供更客观的理论依据[14]。
但是,由于染色体的平衡性变异,不涉及遗传物质的丢失或增加,故基因芯片无法检出,因此染色体核型分析仍是最佳的检测手段。本研究结果显示,有11例染色体核型分析异常但基因芯片正常,包括染色体平衡易位2例,倒位4例,多态性变异5例。虽然基因芯片是分析基因CNV的高分辨率检测工具,在检测速度、自动化程度、覆盖面和分辨率等方面均较传统的染色体核型检测有明显提高,但基因芯片仍有其局限性。因此,将CMA基因芯片技术联合传统的染色体核型检测,可弥补相互间的不足,达到最佳的检测效果,寻找出遗传学病因,从而有效减少出生缺陷儿[15],提高人口素质,减轻家庭和社会负担。
综上所述,产前超声异常的胎儿可能有较高的染色体异常风险,因此建议常规行染色体核型分析及基因芯片检测。同时使用两种检测方法可弥补相互间的不足,达到最佳检测效果,有利于发现遗传学病因,更好地评估胎儿预后,从而在遗传咨询中提供进一步的明确建议,减少出生缺陷患儿,这对优生优育工作、提高人口素质具有重要的现实意义。
