利用SRAP标记研究繁殖群体量对芝麻种质资源遗传完整性的影响
2020-04-16孙建颜廷献叶艳英梁俊超乐美旺饶月亮颜小文周红英
孙建 颜廷献 叶艳英 梁俊超 乐美旺 饶月亮 颜小文 周红英

摘 要:研究繁殖群体量对芝麻种质资源遗传完整性的影响,以确定适宜的繁殖群体量,为种质资源更新繁殖提供理论参考。利用SRAP分子标记技术对育成品种和地方种质2种不同类型的芝麻种质资源的不同群体量的遗传参数进行分析。结果显示:(1)育成品种:随机抽取了10、15、20、25、30、35和40株的7個群体量梯度,24对引物共扩增到DNA位点525个,平均每对引物扩增21.88个,多态性位点24个,占总位点数的4.57%。随着群体量的不断增加,扩增总位点数和遗传相似系数均表现为不断增加,多态性位点比率和遗传距离均呈现先升后降趋势。当群体量达到35~40株时,遗传相似系数达到一定高值(0.998 73和1.000 00),遗传距离降低到一定程度(0.000 64和0.000 00)。聚类结果显示群体量为35和40株的群体被紧密聚在一起,因此可认为育成品种的繁殖群体量达到35~40株时可以保持其遗传完整性;(2)地方种质:随机抽取10、15、20、25、30、35、40、45、50、55和60株的11个群体量梯度,24对引物共扩增到DNA位点552个,平均每对引物扩增23.00个,多态性位点44个,占总位点数的7.97%。随着群体量的不断增加,扩增总位点数波动上升,多态性位点比率和遗传距离呈先升后降然后再轻度变化的趋势。遗传相似系数则随群体量的增大表现为先下降后上升然后轻度下降后再次回升的趋势,并于群体量为40和60株时达到最大值0.99758。聚类结果显示群体量为50、55和60株的群体被紧密聚在一起,因此可认为地方种质的繁殖群体量达到50~55株时可保持其遗传完整性;(3)在芝麻种质资源繁育中,不同类型的种质资源由于其遗传背景的纯度存在差异,应选择不同的繁殖群体量以保持其遗传完整性。
关键词:芝麻;种质资源;繁殖群体量;遗传完整性;SRAP
中图分类号:S565.3 文献标识码:A
Effects of Regeneration Population on the Genetic Integrity of Sesame Germplasm Using SRAP Markers
SUN Jian, YAN Tingxian, YE Yanying, LIANG Junchao, LE Meiwang*, RAO Yueliang, YAN Xiaowen, ZHOU Hongying
Crops Research Institute, Jiangxi Academy of Agricultural Sciences / Jiangxi Research Station of Crop Gene Resource & Germplasm Enhancement, Ministry of Agriculture and Rural Affairs / Nanchang Branch of National Center of Oilcrops Improvement / Jiangxi Province Key Laboratory of Oilcrops Biology, Nanchang, Jiangxi 330200, China
Abstract: The effect of regeneration population on the genetic integrity of sesame germplasm was studied to determine the appropriate population size and provide theoretical references for germplasm renewal and reproduction.The genetic parameters of two different types of sesame germplasm including released cultivar and local germplasm were analyzed using SRAP markers. (1) Released cultivar: Seven gradients of population size including 10, 15, 20, 25, 30, 35 and 40 were selected randomly. 24 pairs of primers were co-amplified to 525 loci, with an average of 21.88 loci per primer pairs, including 24 polymorphic loci, accounting for 4.57% of the total locus. With the increase of population size, the total amplification loci and genetic similarity coefficient increased, and the ratio of polymorphic loci and genetic distance increased and then decreased. When the population size reached 3540 plants, the genetic similarity coefficient reached a certain high value (0.998 73 and 1.000 00), and the genetic distance decreased to a certain degree (0.000 64 and 0.000 00). The clustering results showed that the populations with 35 and 40 were closely clustered. So the genetic integrity could be maintained when the population size of released cultivar reached 3540. (2) Local germplasm: Eleven gradients of population size including 10, 15, 20, 25, 30, 35, 40, 45, 50, 55 and 60 were selected randomly. 24 pairs of primers were co-amplified to 552 DNA locus, with an average of 23.00 locus per primer pairs, and including 44 polymorphic loci, accounting for 7.97% of the total loci. With the increase of population size, the total amplification loci fluctuation increased, and the ratio of polymorphic loci and genetic distance first increased, then decreased and then slightly changed. With the increase of population size, the genetic similarity coefficient first decreased, then increased, then slightly decreased and then rose again, and reached the maximum value of 0.99758 when the population size was 40 and 60 plants. The clustering results showed that the populations of 50, 55 and 60 plants were closely clustered. So it could be considered that the genetic integrity of local germplasm could be maintained when the reproductive population reached 5055 plants. (3) In the regenerating of sesame germplasm, different germplasm types should have different population size to maintain the genetic integrity because of the different genetic purity.
Keywords: sesame (Sesamum indicum L.); germplasm; regeneration population; genetic integrity; SRAP
DOI: 10.3969/j.issn.1000-2561.2020.03.007
作物种质资源是农业科技创新、种业持续发展的物质基础,是保障国家食物供给安全、生态安全和能源安全的战略性资源[1]。我国十分重视作物种质资源收集保存和研究工作,自20世纪50年代以来先后收集保存340种作物50万份资源,资源总量居世界第2位[1]。随着种质资源种类和数量的增长,面临的种质更新繁殖和入库繁育等问题也越来越突出。不管是种质库中已有资源的繁殖更新,还是新收集种质的入库繁育,必须尽可能维持原有种质的遗传完整性。遗传完整性是指群体的基因型频率分布、等位基因频率分布等遗传结构得到完全的保持[2]。维持种质资源的遗传完整性就是在繁殖过程中保持亲代和子代之间最大的遗传相似性和最低的遗传变异,是种质资源安全保存和更新繁殖的核心问题[3]。遗传完整性一直是种质资源研究的重要内容,大量的研究從形态学[4-6]、生理生化[7-8]和分子的角度检测分析种质资源的遗传完整性。尤其是随着分子标记技术的发展,随机扩增多态性DNA(Random Amplified Polymorphism DNA, RAPD)[9-12]、限制性片段长度多态性(Restriction Fragment Length Poly morp hism,RFLP)[9-10]、扩增片段长度多态性(Amplified Fragment Length Polymorphism, AFLP)[13-15]、简单重复序列间扩增(Inter Simple Sequence Repeat, ISSR)[10, 12, 16]、相关序列扩增多态性(Sequence— Related Amplified Polymorphism, SRAP)[17-18]、简单重复序列(Simple Sequ ence Repeat, SSR) [19-20]、基于表达序列标签的简单重复序列(Expressed Se qu ence Tag-Simple Seq uence Repeat, EST-SSR)[21]和单核苷酸多态性(Single Nucleotide Polymorphism, SNP)[22]等越来越多的标记应用到种质遗传完整性的分子检测研究中。
种质资源的遗传完整性在繁殖过程中受种子老化、繁殖世代数、繁殖群体量、繁殖环境、选择压力、植株密度与授粉和收获方式等因素的影响[11, 23-24]。其中,繁殖群体量是指自然随机交配条件下的理想群体大小,当繁殖群体量足够大时才有可能保持其原有的遗传结构[25]。当然,在实际生产过程中,不可能将繁殖群体量无限扩大,适宜的群体量是繁殖时需要考虑的重要因素。适宜的繁殖群体量既要有利于群体遗传结构的保持,维持其遗传完整性,又要有利于降低繁殖工作量和成本,节省人力、物力[21, 25],因此,确定适宜的繁殖群体量就显得尤为重要。关于繁殖群体量对种质遗传完整性的研究已经在玉米[4]、棉花[5]、蚕豆[15]、燕麦[20, 25]、高粱[26]、烟草[27]等许多作物中开展。
芝麻(Sesamum indicum L.)是我国重要的优质油料作物,其天然异交率在5%~23%之间[28],在我国种植历史悠久,分布区域广泛,种质资源十分丰富。关于芝麻种质资源繁殖群体量的研究多以种子产量作为判定标准[29-30],而从遗传完整性角度进行研究的报道很少[21]。冯祥运等[29]对芝麻的3个育成品种和1个地方种质进行了繁殖更新群体大小的研究,根据农艺性状和种子量等确定有效繁殖群体的株数不少于25株,实际繁殖群体以40~50株为宜。马缘生等[30]利用8份芝麻种质进行了繁殖群体大小的研究,结合同工酶多样性验证分析,从产量的角度提出了芝麻繁殖群体不能少于25株,以40~50株为宜。张艳欣等[21]利用15对EST-SSR标记对中期库中的1份地方种质的群体量大小进行研究,认为群体量大小至少为30~35株可代表该种质遗传特性,田间种植应采用不小于60~70株进行繁殖。本研究以2种不同的种质类型,即育成品种(遗传纯度较高)和从农家收集的地方种质(遗传纯度一般)为材料,采用多态性较为丰富的SRAP标记,比较分析了不同类型种质资源繁殖群体量对其遗传完整性的影响,完善和丰富了芝麻种质资源繁育遗传完整性研究的相关内容,为芝麻种质资源更新繁殖提供了理论参考。
1 材料与方法
1.1 材料
本研究所用材料为2种种质资源类型,即育成品种和地方种质。育成品种为江西省农科院作物所育成的黑芝麻品种赣芝9号,种子由育种单位提供,为较大面积繁育的良种,品种纯度较好。地方种质采集于江西省进贤县梅庄镇一农户家,为农户家多年自留自种的地方品种,品种纯度一般。
1.2 方法
1.2.1 试验设计 将收集的种子于发芽皿中进行萌发,7 d后随机选择一定数量的幼苗子叶及上胚轴混合进行DNA提取以构成不同大小的群体。在预实验的基础上,育成品种随机选择10、15、20、25、30、35、40个单株幼苗,地方种质随机选择10、15、20、25、30、35、40、45、50、55、60个单株幼苗,均重复3次。
1.2.2 DNA提取与质量检测 DNA提取方法和质量检测参照孙建等[31]的方法,以种子发芽后的子叶和下胚轴为材料,采用CTAB法提取基因组总DNA,提取过程中以RNase A消化去除RNA,通过紫外分光核酸测定仪测定DNA质量和浓度,并在1%的琼脂糖凝胶上电泳检测其质量。质量合格后用ddH2O稀释到50 ng/μL放置20 ℃冰箱保存备用。
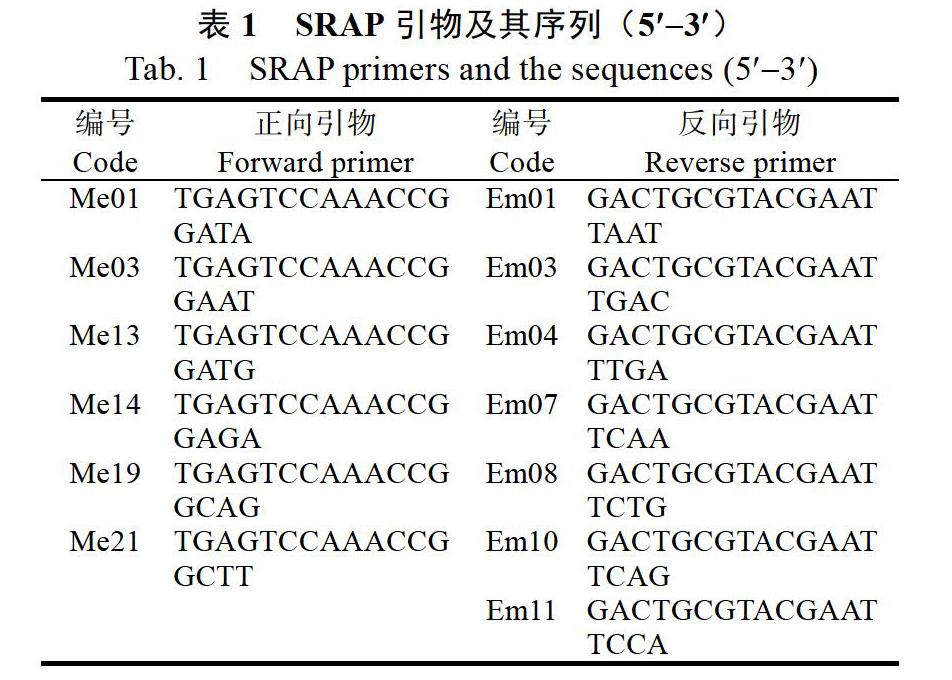
1.2.3 SRAP引物与PCR扩增 本研究使用SRAP正反引物共13条,引物序列信息详见表1。引物是根据Li等[32]设计引物的原则设计而成,由上海生工生物工程技术服务有限公司(Sangon)合成。参照孙建等[31]建立的SRAP的PCR体系(15 ?L):DNA 100 ng、正反引物各50 ng、1反应缓冲液、dNTPs 200 ?mol/L、MgCl2 2.0 mmol/L和Taq DNA聚合酶(购自MBI生物工程公司)1U。PCR程序参照Li等[32]得程序:94 ℃变性3 min;94 ℃ 1 min,35 ℃ 1 min,72 ℃ 1 min,共5个循环;94 ℃1 min,50 ℃ 1 min,72 ℃ 1 min,共35个循环;72 ℃延伸10 min,4 ℃保存。用6%的聚丙烯酰胺(PAGE)凝胶进行分离和银染显影。
1.3 数据处理
统计清晰的SRAP扩增位点数,在同一位点上有带记为“1”,没有带的记为“0”。按Nei等[33]的方法在NTSYS pc2.10e分析软件上计算遗传相似系数(GS)和遗传距离(GD),按照類平均法(UPGMA)进行聚类分析。群体间聚类先在POPGENE 32软件上计算群体间的遗传距离,然后利用NTSYS进行UPGMA聚类。不同群体量的平均遗传相似系数和平均遗传距离的差异显著性方差分析和部分图表制作在SPSS 18.0和Excel 2007软件上完成。
2 结果与分析
2.1 SRAP扩增多态性分析
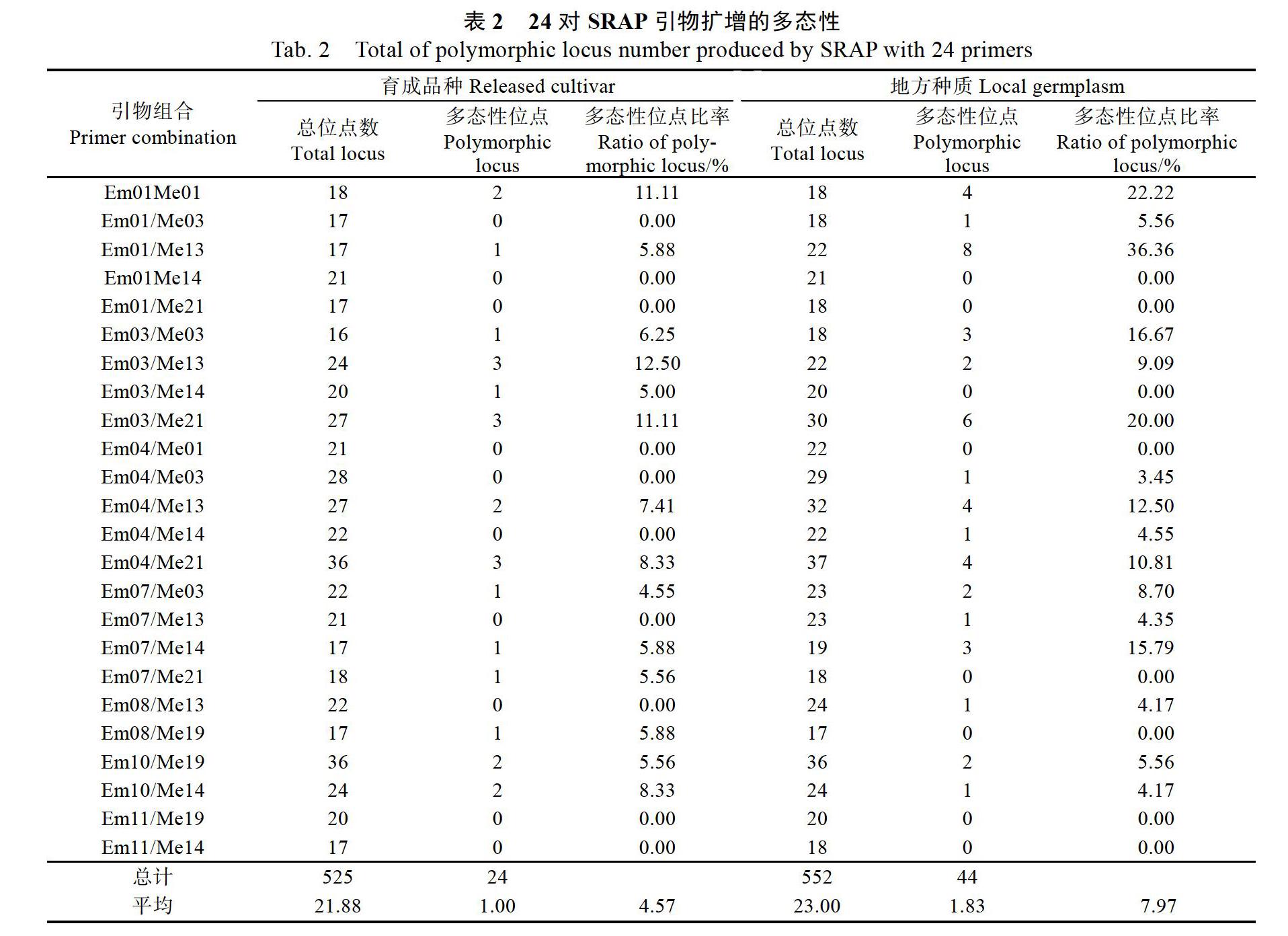
在育成品种中,24对引物共扩增到DNA位点525个(表2),每对引物有效扩增16~36个位点,平均21.88个。24对引物中14对引物扩增到了多态性位点,单对引物扩增的多态性位点最多为3个,平均每对引物扩增的多态性位点为1.00个,共计24个多态性位点。每对引物扩增的多态性位点比率为0~12.50%,平均为4.57%。图1为引物Em11/Me19扩增结果的截图。
由表2可知,在地方种质中,24对引物共扩增DNA位点数552个,每对引物扩增17~37个,平均23.00个。24对引物中16对引物扩增出了多态性,单对引物扩增出的多态性位点最多为8个,平均每对引物扩增出1.83个多态性位点,一共44个。每对引物扩增的多态性比率为0~36.36%,平均为7.97%。图2为引物Em10/Me19扩增结果的截图。
上面的数字表示群体植株数。
2.2 不同群体量对育成品种遗传完整性的影响
在育成品种中,扩增结果显示,平均扩增的DNA条带数和总位点数在群体量较小时有较小的波动变化,但总体上均呈现出随群体量的增加而不断增加的趋势,平均扩增的DNA条带数由506.00条(群体量为10株)增加到520.00条(群体量为40株),扩增的总位点数由511个增加到519个(表3)。多态性位点和多态性位点比率除在群体量为15株时表现为稍有增加之外,整体上表现为随着群体量的不断增加而减小的趋势,均在群体量为40株时降低到0,即在群体量为40株时,随机抽取的3次重复之间没有检测到多态性的存在。3次重复之间的平均相似系数也表现出随群体量的不断增大而不断升高的趋势,于群体量为40株时相似系数达到最高值1.00000,且群体量为35株和40株时平均相似系数没有显著差异。平均遗传距离总体上表现为随着群体量的不断增加而不断减小,于群体量为40株时平均遗传距离为0,且群体量35株和40株时的平均遗传距离没有显著差别。
图3中的扩增位点数(图3A)、多态性位点比率(图3B)、相似系数(图3C)和遗传距离(图3D)的变化折线图更形象地显示,随着群体量的不断增加扩增位点数和相似系数均不断增加,于群体量为35株和40株时达到一定高值;随着群体量的不断增加,多态性位点比率和遗传距离呈先升后降趋势,均在群体量15株时表现为轻度上升,于群体量为40株时降低至0。
可见,在供试的育成品种赣芝9号中,当群体量达到35~40株时,平均相似系数达到一定高值,平均遗传距离降低到一定程度,且相互之间无显著性差异,因此可认为繁殖群体量达到35~40株时可保持该品种的遗传完整性。
2.3 不同群体量对地方种质遗传完整性的影响
由表3可知,在地方种质中,平均扩增的DNA条带数和总位点数均表现为波动上升趋势,平均扩增的DNA条带数由群体量为10株时的516.67条上升至群体量为60株时的546.33条,总位点数由522个(群体量为10株)增加到548个(群体量为50和55株),群体量为60株时稍有减少,为547个。多态性位点数先由10个(群体量为10株和15株)增加到17个(群体量为25株),后降低到群体量为40株时的2个,然后回升至6个(群体量为50株和55株),最后在群体量为60时再次降低至2个。多态性位点比率也由1.91%(群体量为15株)和1.92%(群体量为10株)上升到群体量为25株时的3.21%,然后不断下降至群体量为40株时的0.37%,后又回升至群体量为50株和55株时的1.09%,最后再次下降至0.37%(群体量为60株)。平均相似系数先下降后上升,然后轻度下降后再次回升的趋势,于群体量为40株和60株时达到最大值0.99758,群体量为40~60株之间的相似系数差异不显著。平均遗传距离表现为先上升至群体量为25株时的0.01100,后下降至群体量为40株时的0.00123,随后轻微上升后又于群体量为60株时下降至最低点0.00122,群体量为40~60株时,不同群体量的平均遗传距离之间的差异不显著。
图3更为直观地显示出扩增位点数(图3A)、多态性位点比率(图3B)、相似系数(图3C)和遗传距离(图3D)随群体量大小而表现出的变化。总体上,扩增位点数波动增长至群体量为50~60株时达到一定高值。多态性位点比率则在群体量为40~60株时降低到一定程度,并在较低值水平
变化。相似系数表现为先降低后上升到一定高度后,然后轻度下降后再次上升,群体量在40~60株时的相似系数均表现为较高。遗传距离先升后降,同样在群体量为40~60株时下降至一定低值,并稍有变化。
可见,供试的地方种质每个单株间的遗传背景差异较大,随机抽取的不同样本量间的遗传多样性变化较大,但规律性总体上表现为随着群体量的增加,遗传完整性不断加强,结合各个指标间的变化趋势和相似系数、遗传距离的差异性显著性分析,群体量为40~60株之间的差异不显著,鉴于其变化的波动性,繁殖群体量在40~60株之间,具体株数还有待进一步分析。
2.4 聚类分析
对育成品种不同群体量的SRAP扩增结果进行聚类分析。不同群体量不同重复间的聚类结果显示(图4A),21个样品被聚成3大群,其中群体量为10株和15株的6个样品被聚在一起;群体量为20株、25株和30株(2个重复)的8个样品被聚在另一个群;而余下的群体量为35株和40株的3次重复以及群体量为30株的1次重复被较紧密地聚在一起,其中群体量为40株的3次重复、群体量为35株的2次重复分别被紧密聚在一起,可见群体量为35株和40株之间的关系最接近。群体间的聚类结果显示(图4B),7个取样群体被聚成2个大类,群体量为10、15、20和25株的群体被聚在一起,群体量为30、35和40株的3个群体被聚在另一起,其中35株的群体和40株的群体被更紧密地聚在一起,它们的相似度较为接近。
地方种质不同群体量不同重复间的聚类结果显示(图5A),33个样品首先被聚成两大群,群体量为10~30株的12个较小群体量的样品被聚在一起,其余的21个样品被聚在另一大群;在大群中,群体量为50株(2个重复)、55株(2个重复)和60株(3个重复)的7个样品聚类最为紧密,可见群体量为50~60株之间的相似关系最为接近。群体间的聚类结果显示(图5B),11个取样群体首先被聚成2个大类,群体量为10、15、20和25株的小群体量群体被聚在一起,余下的较大群体量群体被聚在一起。群体量为30~60株的7个取样群体又被聚为3类,分别是群体量为30株和35株聚为1类、40株和45株聚为1类、以及50、55和60株聚为1类,且群体量为50株和55株的群体最为紧密地聚在一起,它们的相似度较为接近。
3 讨论
SRAP标记操作简单、多态性好、可重复性高,是一种综合了多种DNA分子标记优点的标记技术,广泛应用于作物遗传多样性分析、遗传图谱构建及QTLs(基因)定位研究[34-36]。在芝麻研究中,SRAP标记同样得到了大量应用,车卓等[37-38]在100份黑芝麻种质资源和18份核心收集品的育成品种(系)的SRAP分析中,每对引物分别扩增出DNA条带14.0条和13.9条。孙建等[39]在67个中国白芝麻品种的遗传多样性研究中,21对SRAP引物共扩增出DNA条带561条,平均每对引物扩增带数为26.7条。张艳欣等[40]在对209份白芝麻种质资源进行遗传多样性分析中,13对SRAP引物共扩增出180条带,平均每对引物扩增出13.8条。孙建等[31]利用SRAP标记技术对20个中国主要黑芝麻品种DNA进行遗传基础分析,23对引物共扩增DNA带672条,平均每对引物扩增的带数为29.21条。本研究在育成品种中24对SRAP标记引物共扩增DNA位点
数525条,平均每对引物扩增位点21.88个,在地方种质中24对引物共扩增552个位点,平均每对引物扩增位点23.00个。该扩增结果显示,每对SRAP引物扩增的位点数介于前人在芝麻的研究结果之间[31, 37-40],与其他作物中的研究结果相近[41-42],可见本研究中的SRAP扩增结果有效。
分子标记是研究种质资源遗传完整性的一种科学高效的实用技术,具有检测位点多、结果真实可靠、不受环境影响等优点。利用分子标记技术分析繁殖群体量对种质遗传完整性的影响研究已经在很多作物得到广泛应用。计怀春等[5]应用68对SSR标记对3个棉花品种的不同群体量进行DNA扩增分析,获得236个位点,群体量为25、50、100和150株之间的遗传多样性指数和相似系数等指标均差异不显著,于是认为25株可作为棉花种质繁殖更新时的最低群体量。刘敏等[15]用AFLP标记比较分析9个蚕豆地方种质在繁殖群体量为20和50株时的遗传完整性指标,结果显示,50株的繁殖群体量更能保持其遗传完整性。杨苗苗等[20]利用SSR标记分析2个燕麦品种10~ 90株繁殖群体量对遗传完整性的影响,20对引物分别检测到43和32个等位基因,2个品种群体量分别在大于70株和50株时,遗传变化趋于平稳,认为不同品种的繁殖群体量存在差异。李俊等[25]利用20对SSR引物对燕麦育成品种燕科1号的繁殖群体量进行研究,认为繁殖群体量理论为50株,在实际生产中可加大到100株更能保持其遗传完整性。许玉凤等[26]在对高粱的繁殖群体量研究结果表明,25对SSR引物共检测到58个等位基因,群体量在40株以上时等位基因频率及多样性指数趋于一致,故建议高粱的繁殖群体量应在40株以上才能较好地保持其遗传完整性。潘应花等[27]以4份烟草种质和1份野生种为材料,利用SSR标记进行不同繁殖群体量的遗传完整性分析,结果表明普通烟草种质的繁育群体量应等于或大于10株、野生种应大于20株才能保持其遗传完整性。在芝麻繁殖群体量研究中,冯祥运等[29]和马缘生等[30]通过农艺性状和种子产量认为芝麻繁殖群体量应该大于25株,张艳欣等[21]利用15对EST-SSR引物对1份芝麻地方种质的研究认为至少30~35株可代表该种质的遗传特性。本研究中利用24对SRAP引物对2个不同类型的芝麻种质资源的不同繁殖群體量进行遗传多样性分析,结果显示,在育成品种中检测到DNA位点525个,群体量35株和40株时,其总位点数趋于稳定,多态性位点比率很低,遗传相似系数趋于1,遗传距离趋于0,且35株和40株的群体量之间的差异不显著,聚类分析也显示35株的群体和40株的群体被紧密聚在一起,故认为育成品种的繁殖群体量达到35~40株时,可保持其遗传完整性。在地方品种中共检测到DNA位点552个,群体量在50~60株时,总位点数变化较小,群体间的遗传相似系数和遗传距离差异不显著,遗传相似系数最高可达0.997 58,遗传距离低至0.001 22,且聚类结果将50~55株的群体紧密聚在一起,结合经济节约的原则,故认为地方种质的适宜繁殖群体量为50~55株。相较上述研究结果[5, 15, 20-21, 25-27],本研究检测的位点数多、设置的群体量梯度较密,且比较了不同类型种质的繁殖群体量,因此结果更为可靠有效。至于本研究中获得的适宜群体量要大于前人在芝麻上的研究[21, 29-30],这与选用的材料、标记类型和DNA位点数有关。
Rao N K, Bramel P J, Reddy K N, et al. Optimizing seed quality during germplasm regeneration in pearl millet[J]. Genetic Resources and Crop Evolution, 2002, 49(2): 153-157.
Chebotar S, Roder M S, Korzun V, et al. Molecular studies on genetic integrity of open pollinating species rye (Secale cereale L.) after long term genebank maintenance[J]. Theoretical and Applied Genetics, 2003, 107: 1469-1476.
李 俊, 杨苗苗, 卢 萍, 等. SSR分析燕科1号繁殖群体量对其遗传完整性的影响[J]. 中国草地学报, 2018, 40(5): 18-22.
许玉凤, 朱远英, 张志娥, 等. 高粱微卫星分析中遗传完整性样本量的确定[J]. 华北农学报, 2012, 27(3): 108-114.
潘应花, 刘艳华, 任 民, 等. 烟草种质不同群体量遗传完整性的SSR研究[J]. 植物遗传资源学报, 2013, 14(5): 979-984.
孙 建, 颜廷献, 高德学, 等. 芝麻繁育特性研究Ⅰ: 天然异交率的测定[J]. 中国油料作物学报, 2015, 37(4): 462-466.
冯祥运, 张秀荣, 刘越英. 基因库芝麻种子繁殖更新方法研究[J]. 中国油料, 1997, 19(1): 63-65.
马缘生, 范传珠, 王述民, 等. 五种作物基因库种子繁殖更新技术研究[J]. 植物遗传资源学科, 2002, 3(2): 1-7, 33.
孙 建, 乐美旺, 何才和, 等. 中国主要黑芝麻品种的遗传多样性分析[J]. 植物遗传资源学报, 2015, 16(2): 269-276.
Li G, Quiros C F. Sequence-related amplified polymorphism (SRAP), a new marker system based on a simple PCR reaction: its application to mapping and gene tagging in Brassica[J]. Theoretical and Applied Genetics, 2001, 103(2-3): 455-461.
Nei M, Li W H. Mathematical model for studying genetic variation in terms of restriction endonucleases[J]. Proceedings of the National Academy of Sciences of the United States of America, 1979, 76(10): 5269-5273.
刘 榮, 龚德勇, 刘清国, 等. 基于SRAP分子标记的13份贵州芒果种质资源遗传多样性分析[J]. 热带作物学报, 2019, 40(1): 87-91.
罗世杏, 唐玉娟, 黄国弟, 等. 利用SRAP分子标记分析杧果种质遗传多样性[J]. 热带作物学报, 2018, 39(12): 2369-2376.
王玉红, 金关荣, 陶爱芬, 等. 利用SRAP标记绘制中国芥菜基因源分子指纹图谱[J]. 热带作物学报, 2016, 37(9): 1719-1724.
车 卓, 张艳欣, 孙 建, 等. 应用SRAP标记分析黑芝麻核心种质遗传多样性[J]. 作物学报, 2009, 35(10): 1936-1941.
车 卓, 张艳欣, 孙 建, 等. 芝麻核心收集品中育成品种(系)的遗传多样性分析[J]. 植物遗传资源学报, 2009, 10(3): 373-377.
孙 建, 张秀荣, 张艳欣, 等. 中国芝麻主要品种遗传多样性特点及遗传基础演变[J]. 中国农业科学, 2009, 42(10): 3421-3431.
张艳欣, 张秀荣, 车 卓, 等. 应用SRAP标记分析白芝麻核心种质遗传多样性[J]. 中国油料作物学报, 2010, 32(1): 46-52.
文雁成, 王汉中, 沈金雄, 等. 用SRAP标记分析中国甘蓝型油菜品种的遗传多样性和遗传基础[J]. 中国农业科学, 2006, 39(2): 246-256.
李媛媛, 沈金雄, 王同华, 等. 利用SRAP、SSR和AFLP标记构建甘蓝型油菜遗传连锁图谱[J]. 中国农业科学, 2007, 40(6): 1118-1126.
