CRISPR系统相关蛋白的纯化与活性分析
2020-04-16田东芳崔香玲周睿张永欣马雪梅岑山
田东芳,崔香玲,周睿,张永欣,马雪梅,岑山
·论著·
CRISPR系统相关蛋白的纯化与活性分析
田东芳,崔香玲,周睿,张永欣,马雪梅,岑山
100124 北京工业大学生命科学与生物工程学院(田东芳、马雪梅);100050 北京,中国医学科学院北京协和医学院医药生物技术研究所免疫室(田东芳、崔香玲、周睿、张永欣、岑山)
对 CRISPR 相关蛋白 Cas3、Cas6 及 Cas9 进行表达纯化,并测定其活性。
在体外构建带有目的基因的质粒,导入大肠杆菌表达系统,通过大肠杆菌的过表达,裂解并提取蛋白,利用 His 标签,可将目的蛋白纯化出来;通过 3 种 Cas 蛋白对不同核酸的切割,分别设计 3 种核酸底物,再通过聚丙烯酰胺凝胶电泳,最终确定 Cas 蛋白的活性。
成功表达并纯化得到 Cas3、Cas6 及 Cas9 3 种蛋白,并根据其蛋白特性,与相应的核酸底物进行孵育,在电泳后通过凝胶成像,可观察到 3 种蛋白均具有活性。
表达纯化的 Cas3、Cas6 及 Cas9 具有生物学功能,为其功能机制研究奠定了基础。
CRISPR; Cas3; Cas6; Cas9
规律间隔成簇短回文重复序列及相关蛋白(clustered regularly interspaced short palindromic repeats and CRISPR associated proteins,CRISPR-Cas)是细菌和古生菌用来抵抗外源核酸的适应性免疫系统[1]。CRISPR 位点是由细菌自身的重复序列及用于识别外源核酸的间隔序列组成。CRISPR 免疫系统执行功能,需要经历 3 个阶段:适应、表达及干扰。在适应阶段,细菌将来源于噬菌体或者外源质粒的核酸序列作为间隔序列,整合到 CRISPR 位点的重复序列之间。这些间隔序列执行序列记忆功能,以保证在之后的相应噬菌体或质粒的入侵过程中进行针对性的防御[2]。在表达阶段,所有重复序列和间隔序列作为一个整体,被转录成 crRNA 前体,最终被相应 Cas 蛋白处理成成熟的 crRNA[3]。在干扰阶段,crRNA 作为向导,与入侵的核酸互补配对,同时由相应的 Cas 蛋白进行识别以及切割外源入侵核酸[4]。
根据基因组成、Cas 蛋白与基因结构之间的序列相似性,目前将 CRISPR 系统分为两类(1 类和 2 类),其中 1 类中包含 3 种型别(I 型、III 型和 IV 型),2 类中也包含 3 种型别(II 型、V 型和 VI 型)以及 19 个亚型[5]。两类 CRISPR-Cas系统在执行功能的效应蛋白上有根本的不同,1 类 CRISPR 系统是由 4 ~ 7 个 Cas 蛋白组成的多亚基效应复合物[6];2 类 CRISPR 系统是较为不常见的,几乎完全局限于细菌,由单一的大蛋白构成效应器[7]。I 型系统是最普遍的系统,包含 7 个亚型,都是依赖于多亚基效应复合物来靶向切割目标序列。其中,Cas3 蛋白是一种用于降解外源核酸的核酸内切酶[8-9],Cas6 是将长链的 crRNA 前体切割成成熟的 crRNA 的核酸内切酶[10-11]。在 II 型系统中,最普遍也是研究最多的就是 Cas9 蛋白,包含 HNH 和 RuvC 两个结构域,分别降解目标 DNA 的互补链和非互补链[12-14]。
CRISPR 系统作为一种基因编辑工具,凭借设计简单、耗时短、效率高的优点,其应用发展已经远远超过了锌指核酸酶和转录激活因子样效应物核酸酶技术,并在分子生物学领域得到广泛应用。目前基于 CRISPR 系统发展而来的应用工具,已经成功地作用于各种体外培养的细胞,通过对胚胎细胞的编辑而产生动物模型的技术也在不断成熟,以及在疾病治疗特别是遗传病治疗中也展现出其独有的优势,并且其作用范围在不断的扩展,作用方式还在不断的创新。CRISPR 系统虽然已在多个领域有所应用,但也只是利用了其一点价值,更多的价值正期待着我们去开发利用。
本研究中,选用了来源于 I-F 型的绿脓杆菌的 Cas3(Genebank:KYO77788.1)和 Cas6(Genebank:KYO77784.1)蛋白,以及来源于 II-A 型的化脓链球菌的 Cas9(Genebank:AII16589.1)蛋白。对其蛋白进行了重组表达以及纯化,并证明了所纯化蛋白的生物学活性,为其功能机制研究奠定了基础。
1 材料与方法
1.1 材料
基因3、6 和9 由生工生物工程(上海)股份有限公司合成;HisTrap HP 1 ml 预装柱购自美国 GE Healthcare 公司;肠激酶购自于美国 NEB 公司;分子伴侣质粒和 BCA 蛋白浓度测定试剂盒购自于日本 Takara 公司;T7 体外转录试剂盒购自于美国 Invitrogen 公司;质粒提取试剂盒购自于北京康为世纪生物科技有限公司。
1.2 方法
1.2.1 蛋白表达 将合成得到的3、6 以及9 基因分别进行 PCR 扩增,将目的基因克隆到 pET32a 表达载体上,提取质粒,并将带有目的基因的质粒转入大肠杆菌BL21(DE3)表达体系中。
1.2.1.1 Cas3 先在大肠杆菌 BL21(DE3)中,转进 pKJE7 分子伴侣质粒,再将带有分子伴侣的菌株制备成感受态细胞,转入带有目的基因的质粒。取带有两种质粒的单克隆菌落,过夜培养成母种,按照 1:100 转接比例,37 ℃孵育菌液浓度至600约为 0.5,并在转接时加入 2 mg/ml 的阿拉伯糖,用以诱导分子伴侣蛋白的表达。当菌液到达指定浓度时,加入 0.5 mmol/L IPTG,18 ℃过夜诱导表达。
1.2.1.2 Cas6 和 Cas9 取单克隆菌落过夜培养成母种,按照 1:100 转接比例,37 ℃孵育菌液浓度至600约为 0.5 时,加入 0.5 mmol/L IPTG,18 ℃过夜诱导表达。
1.2.1.3 纯化与透析 取 1 L 过夜培养的菌液离心,用 20 mmol/L HEPES,500 mmol/L NaCl 和1 mmol/L TCEP 重悬并超声,过镍柱分离纯化,在透析过程中加入肠激酶用于切除 Trx 促溶标签。
1.2.2 酶活验证
1.2.2.1 Cas3 配制10倍反应液:200 mmol/L Tris,3 mol/L NaCl,50 mmol/L MgCl2;将两条单链 DNA 退火成双链 DNA(5' GCTGTACGTCACT ATCGAAGCAATACAGGTAGACGCGGACATCAAGCCCGCCGTGAAGGTGCAGCTTCTCTACAGAGTGC 3';5' GCACTCTGTAGAGAAGCTGCACCAA GTGCCGCCGCTTGATGTCCGCGTCTACCTGTATTGCTTCGATAGTGACGTACAGC 3'),中间包含 10 bp 不互补区域;分别取 1 μl 反应液,1 μl Cas3 蛋白酶和 1 μl dsDNA,最终用 H2O 补足至 10 μl;37 ℃水浴,分别取30、60、90、120 和 150 min 时间节点;反应结束后用质粒提取试剂盒将 DNA 提取出来,上样于 20% 聚丙烯酰胺凝胶,120 V 电泳 1 h;电泳结束后用 SYBR Gold 染色 20 min,最终用凝胶成像系统检测 dsDNA 的切割情况。
1.2.2.2 Cas6 配制 10倍反应液:200 mmol/L HEPES,1 mol/L KCl;反应体系:1 μl(10 μmol/L)RNA(5' CUGCCGUAUAGGCAGC 3'),1 μl Cas6 蛋白酶和 1 μl 反应溶液,最终用 H2O 补足至10 μl;将上述溶液混合后,于 25 ℃反应 5 min,加入50 μl Trizol,10 μl 氯仿,剧烈振荡30 s,4 ℃离心后取上层水相加入 50 μl 异丙醇,室温静止10 min,并再次 4 ℃离心 10 min。弃上清加入50 μl 现配 75% 乙醇,离心后弃上清,充分晾干后,加入 10 μl 水,上样至尿素变性胶,先用 200 V 电泳 30 min,随后调整电压 400 V 电泳约 2.5 h。电泳结束后,用 SYBR Gold 染色 20 min,最后用凝胶成像系统成像。
1.2.2.3 Cas9 配制 10 倍反应液:200 mmol/L HEPES,pH 7.5;20 mmol/L MaCl2;1 mol/L KCl;底物 DNA(5' TCGGTGCGGGCCTCTTCGCTATTA CGCCAGCTGGCGAAAGGGGGATGTGCTGCAAGGCGATTAAGTTGGGTAACGCCAGGGTTTTCCCAGTCACGACGTTGTAAAACGACGGCCAGTGCCAAGCTTGCATGCCTGCAGGTCGACTCTAGAGGATCCCAATCCCAGCCAAGCGCACCTAATTTCCGAATTCGTAATCATGGTCATAGCTGTTTCCTGTGTGAAATTGTTATCCGCTCACAATTCCACACAACATACGAGCCGGAAGCATAAA 3')浓度为80 ng/μl;sgRNA(5' GGAAAUUAGGUGCGCUUGGCGUUUUAGAGCUAGAAAUAGCAAGUUAAAAUAAGGCUAGUCCGUUAUCAACUUGAAAAAGUGGCACCGAGUCGGUGCUU 3')的浓度600 ng/μl;加入 1 μl 反应液,1 μl Cas9 蛋白酶,1 μl dsDNA 和 1 μl sgRNA,用 H2O 补足至 10 μl,37 ℃孵育 15 min,反应结束后,用聚丙烯酰胺凝胶检测,120 V 电泳 40 min,然后用 SYBR Gold 染色 20 min后,用凝胶成像系统成像。
2 结果
2.1 蛋白的表达与纯化
Cas3 蛋白来源于绿脓杆菌,由 3231 bp 基因编码,包含 1076 个氨基酸残基,分子量约为121 kD。为了提高 Cas3 蛋白的溶解性,在表达体系中又转入了表达K-J-E 伴侣蛋白质的 pKJE7 分子伴侣质粒。首先加入阿拉伯糖诱导2.5 h 左右,诱导伴侣蛋白表达,之后再加入 IPTG 诱导目的蛋白,促进目的蛋白的溶解。过夜诱导约 18 h 后,收集菌体,进行蛋白纯化。pET32a 表达载体中带有肠激酶、凝血酶的酶切位点,因为 pET32a 带有约 18 kD 标签,标签的存在会影响酶活性,所以在透析时加入了肠激酶以切除标签(图 1A)。
Cas6 蛋白与 Cas3 蛋白一样也来源于绿脓杆菌,又称为 Csy4 蛋白,由 564 bp 基因编码,包含 187 个氨基酸残基,分子量约为 22 kD。同样在透析时加入肠激酶以切除标签,切除 Trx 标签后,由于融合蛋白的 C 端依然携带 His 标签,且融合蛋白与肠激酶位点之间存在载体序列所表达的蛋白,所以切割标签后的蛋白大小约为 25 kD(图 1B)。
Cas9 蛋白来源于化脓链球菌,是基因编辑中应用最广的蛋白。由 4107 bp 基因编码,包含 1368 个氨基酸残基,分子量约为 158 kD。目的蛋白能够在大肠杆菌中大量表达,且溶解性较好。在透析时加入肠激酶以切除标签(图 1C)。
2.2 浓度测定
在富集培养后,1 L菌液被浓缩至 30 ml 粗蛋白提取物,上至 HiTrap HP 柱,得到 3 种蛋白的浓度分别为 1.3、1.8 和 2.3 mg/ml(图 2)。通过 Quantity One 软件进行 SDS-PAGE灰度分析,其中目的条带所占信号值分别为 110.27、124.67 和 91.48,目的条带所在泳道所有信号值之和分别为 138.48、142.03 和 113.60,最终计算得到每种蛋白的纯度分别约为 80%、88% 和 81%。
2.3 Cas3 酶活验证
Cas3 能够在不改变自身构象的情况下,切割双链 DNA。有研究表明,crRNA 与 4 种相关蛋白组成了 Csy 复合物,它能够识别外源核酸,并将外源核酸进行解链,使之形成 R 环结构,Cas3 识别该 R 环结构并将其切割。根据文献[15]的方法,在 80 bp 底物中间,人为设计 10 bp 不互补区域,Cas3 能够识别该结构并切割(图 3A)。在37 ℃条件下,选取 30、60、90、120 和 150 min 5 个时间点,随着孵育时间的延长,切割产物逐渐增多(图 3B)。
2.4 Cas6 酶活验证
Cas6 是 RNA 内切酶,在表达阶段,它能够识别 CRISPR 位点中的重复序列,并在重复序列的下游将间隔序列切开,根据文献[10]的方法,我们选取了 16 nt 带有茎环结构的 RNA,并且下游包含 1 nt 的间隔序列碱基,当 Cas6 加入进去之后,能够立即切掉末端的间隔序列(图 4A)。在室温条件下,将底物与酶反应 5 min 后即可清晰地看到切割条带(图 4B)。
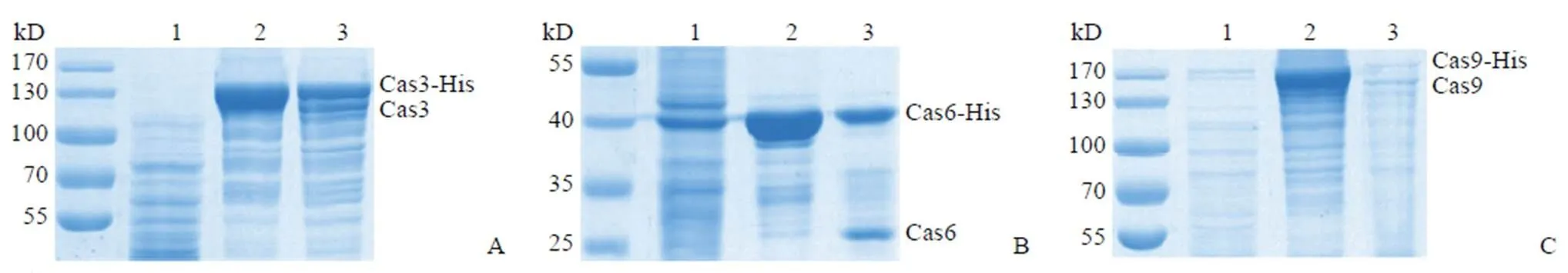
1:全菌;2:纯化;3:切割
Figure 1 Protein purification and cutting labels (A: Cas3; B: Cas6; C: Cas9)

图 2 蛋白浓度计算(A:Cas3;B:Cas6;C:Cas9)
Figure 2 Concentration of protein (A: Cas3; B: Cas6; C: Cas9)
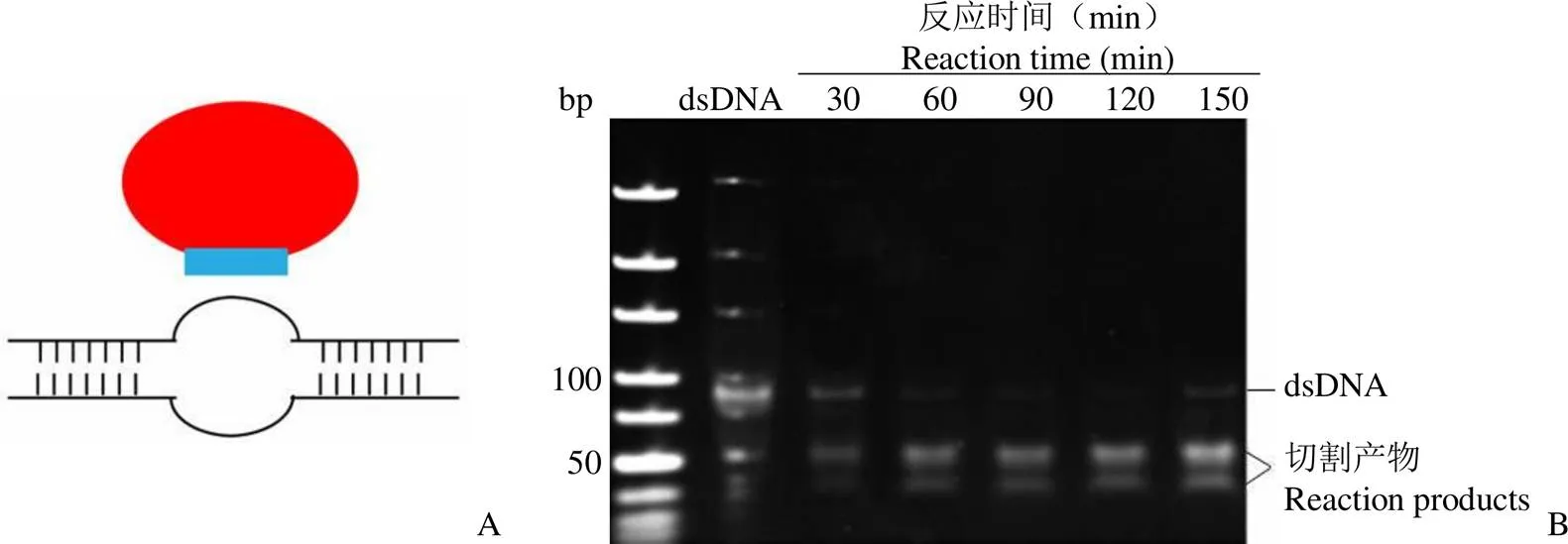
图 3 Cas3 的酶活验证(A:Cas3 切割核酸底物原理图;B:随着反应时间的延长,dsDNA 底物被切割,且切割产物有更多的积累)
Figure 3 Assay of Cas3 activity (A:Schematic diagram of Cas3 cutting nucleic acid substrate; B: With the extension of the reaction time, the dsDNA substrate is cleaved, and the cleaved products have more accumulation)

图 4 Cas6 的酶活验证(A:Cas6 切割核酸底物原理图;B:随着反应时间的延长,可明显观察到 RNA 底物被切割)
Figure 4 Assay of Cas6 activity (A: Schematic diagram of Cas6 cutting nucleic acid substrate; B: With increasing reaction time, cleavage of the RNA substrate is clearly observed)

图 5 Cas9 的酶活验证(A:Cas9 切割核酸底物原理图;B:没有 Cas9 加入时,是没有切割产物产生的)
Figure 5 Assay of Cas9 activity (A: Schematic diagram of Cas9 cutting nucleic acid substrate; B: When Cas9 is not added, there is no cleavage product)
2.5 Cas9 酶活验证
Cas9 蛋白包含两个结构域,在 sgRNA 的引导下,HNH 结构域切割与 sgRNA 互补的链,而RuvC 结构域切割非互补链(图 5A)。根据文献[16],通过体外转录,得到 sgRNA,PCR 扩增出Cas9 切割序列,在 37 ℃条件下切割 15 min,可观察到被切开的条带,从而证明了 Cas9 的活性(图 5B)。
3 讨论
自从基因编辑技术问世以来,CRISPR 系统一直是研究热点,利用 CRISPR 进行基因敲除,转录调节,基因治疗等,使 CRISPR 技术的应用越来越广泛,但目前我们对 CRISPR 系统的了解程度还是远远不够的,例如发挥作用所经历的 3 个阶段所涉及到的 Cas 蛋白,我们了解得还是不够充分。为了对 CRISPR 系统更好地了解,本研究选择了 I 型的 Cas3 和 Cas6,II 型的 Cas9 作为研究对象,通过各个蛋白对特定核酸底物的切割,证明了 3 种蛋白能够在体外进行切割。本文所应用的方法是通过大肠杆菌表达系统对目标蛋白进行大量表达,并通过 His 标签从总蛋白中将目标蛋白分离出来。再将目标蛋白与核酸底物切割后,通过聚丙烯酰胺凝胶电泳分析,根据核酸分子量大小,可明确看到核酸是否被切割,从而证明了目标蛋白的活性。综上所述,该工作也为后期的机制研究以及蛋白之间的相互作用奠定了基础。
[1] Barrangou R, Fremaux C, Deveau H, et al. CRISPR provides acquired resistance against viruses in prokaryotes. Science, 2007, 315(5819): 1709-1712.
[2] Sternberg SH, Richter H, Charpentier E, et al. Adaptation in CRISPR-Cas systems. Mol Cell, 2016, 61(6):797-808.
[3] Amitai G, Sorek R. CRISPR-Cas adaptation: insights into the mechanism of action. Nat Rev Microbiol, 2016, 14(2):67-76.
[4] van der Oost J, Jore MM, Westra ER, et al. CRISPR-based adaptive and heritable immunity in prokaryotes. Trends Biochem Sci, 2009, 34(8):401-407.
[5] Makarova KS, Wolf YI, Alkhnbashi OS, et al. An updated evolutionary classification of CRISPR-Cas systems. Nat Rev Microbiol, 2015, 13(11): 722-736.
[6] Mohanraju P, Makarova KS, Zetsche B, et al. Diverse evolutionary roots and mechanistic variations of the CRISPR-Cas systems. Science, 2016, 353(6299):aad5147.
[7] Shmakov S, Smargon A, Scott D, et al. Diversity and evolution of class 2 CRISPR-Cas systems. Nat Rev Microbiol, 2017, 15(3):169-182.
[8] Westra ER, Nilges B, van Erp PB, et al. Cascade-mediated binding and bending of negatively supercoiled DNA. RNA Biol, 2012, 9(9): 1134-1138.
[9] van Erp PBG, Patterson A, Kant R, et al. Conformational dynamics of DNA binding and Cas3 recruitment by the CRISPR RNA-guided cascade complex. ACS Chem Biol, 2018, 13(2):481-490.
[10] Haurwitz RE, Jinek M, Wiedenheft B, et al. Sequence- and structure- specific RNA processing by a CRISPR endonuclease. Science, 2010, 329(5997):1355-1358.
[11] Taylor HN, Warner EE, Armbrust MJ, et al. Structural basis of type IV CRISPR RNA biogenesis by a Cas6 endoribonuclease. RNA Biol, 2019, 16(10):1438-1447.
[12] Jinek M, Chylinski K, Fonfara I, et al. A programmable dual-RNA- guided DNA endonuclease in adaptive bacterial immunity. Science, 2012, 337(6096):816-821.
[13] Nishimasu H, Shi X, Ishiguro S, et al. Engineered CRISPR-Cas9 nuclease with expanded targeting space. Science, 2018, 361(6408): 1259-1262.
[14] Thavalingam A, Cheng Z, Garcia B, et al. Inhibition of CRISPR-Cas9 ribonucleoprotein complex assembly by anti-CRISPR AcrIIC2. Nat Commun, 2019, 10(1):2806.
[15] Rollins MF, Chowdhury S, Carter J, et al. Cas1 and the Csy complex are opposing regulators of Cas2/3 nuclease activity. Proc Natl Acad Sci U S A, 2017, 114(26):E5113-E5121.
[16] Dong, Guo M, Wang S, et al. Structural basis of CRISPR-SpyCas9 inhibition by an anti-CRISPR protein. Nature, 2017, 546(7658):436-439.
Purification and activity analysis of proteins related to CRISPR system
TIAN Dong-fang, CUI Xiang-ling, ZHOU Rui, ZHANG Yong-xin, MA Xue-mei, CEN Shan
College of Life Science and Bioengineering, Beijing University of Technology, Beijing 100124, China (TIAN Dong-fang, MA Xue-mei); Institute of Medicinal Biotechnology, Chinese Academy of Medical Sciences & Peking Union Medical College, Beijing 100050, China (TIAN Dong-fang, CUI Xiang-ling, ZHOU Rui, ZHANG Yong-xin, CEN Shan)
To expression and purify CRISPR-related proteins: Cas3, Cas6 and Cas9, and determine their activity to lay the foundation for the subsequent screening of their inhibitors.
The plasmid carrying the target gene was constructed, introduced into anexpression system, and the protein was extracted by over-expression and lysis of, and the target protein was purified by using the His tag; the cleavage of different nucleic acids by the three Cas proteins was performed. Three nucleic acid substrates were designed respectively, and then the activity of Cas protein was finally determined by polyacrylamide gel electrophoresis.
Three proteins, Cas3, Cas6 and Cas9, were successfully expressed and purified, and incubated with the corresponding nucleic acid substrate according to their protein properties. After electrophoresis, gel imaging showed that all three proteins were active.
Three active proteins, Cas3, Cas6 and Cas9, were obtained, which provided the basis for the subsequent screening of anti-CRISPR system.
CRISPR; Cas3; Cas6; Cas9
ZHANG Yong-xin, Email: yongxinzhang@imb.pumc.edu.cn; CEN Shan, Email: shancen@hotmail.com
中国医学科学院医学与健康科技创新工程(2017-I2M-1-012 YXZ)
张永欣,Email:yongxinzhang@imb.pumc.edu.cn;岑山,Email:shancen@imb.pumc.edu.cn
10.3969/j.issn.1673-713X.2020.02.011
2019-09-23
