谷子PEPC基因的鉴定及其对非生物逆境的响应特性
2020-04-16赵晋锋杜艳伟王高鸿李颜方赵根有王振华王玉文余爱丽
赵晋锋 杜艳伟 王高鸿 李颜方 赵根有 王振华 王玉文 余爱丽
谷子PEPC基因的鉴定及其对非生物逆境的响应特性
赵晋锋**杜艳伟**王高鸿 李颜方 赵根有 王振华 王玉文 余爱丽*
山西省农业科学院谷子研究所/ 特色杂粮种质资源发掘与育种山西省重点实验室, 山西长治 046011
磷酸烯醇式丙酮酸羧化酶(phosphoenolpyruvate carboxylase, PEPC)是C4植物光合作用关键酶, 并在植物多种代谢途径及逆境信号应答过程中起重要作用。本研究通过序列比对, 从谷子基因组中筛选出6个候选基因。SiPEPC蛋白特征参数相似度很高, 序列非常保守, 都含有PEPC基因特征功能域PEPcase Motif。SiPEPC成员主要被定位在细胞质、细胞核和线粒体。在SiPEPC成员启动子序列中含大量有光、激素、逆境以及其他生长调控相关的顺式应答元件。苗期逆境qRT-PCR表达谱分析表明, 5个基因()不同程度受ABA、PEG、高盐和低温诱导表达, 表明其参与了苗期对非生物逆境的响应。5个基因表达量在正常生长条件下随着谷子的生长呈增强趋势, 且在不同生育时期干旱胁迫下明显增加, 表明其参与了拔节、抽穗、灌浆期的干旱胁迫应答。拔节期弱光可诱导5个基因的表达, 而在拔节期中等强度光照以及抽穗期和灌浆期的中等光照和弱光照下表达量均急剧降低, 揭示光照强度严重影响基因的表达。
谷子; 磷酸烯醇式丙酮酸羧化酶; 非生物逆境; 表达分析

谷子[(L.) P. Beauv.]是C4禾本科重要粮食作物和饲草作物, 起源于我国, 在全球一些温带、亚热带和热带的干旱和半干旱地区广泛种植, 具有抗旱、耐瘠、适应性广等特点[22-24]。谷子在生长过程中会经常遇到干旱、盐渍、低温、高温、洪涝以及病虫害侵染等, 这些逆境严重影响了谷子的生长发育和产量品质[25]。2012年谷子基因组测序已完成并公布, 为开展谷子抗逆分子生物学研究提供了极为便利的条件[26-27]。迄今为止PEPC研究主要聚焦在C4型PEPC基因在提高C3植物光合作用和耐胁迫应答领域, 在C4作物玉米、高粱中研究较多, 但是在C4作物谷子中PEPC基因非生物逆境相关研究鲜有报道。本研究检索了谷子中的PEPC基因家族成员, 并对其氨基酸序列、蛋白特征、功能、亚细胞定位、启动子区域顺式应答元件等参数特征分析和预测, 随后检测了家族基因在幼苗期逆境胁迫下的动态表达模式及在拔节、抽穗、灌浆3个生育时期干旱胁迫和不同光照处理下的表达情况, 旨在为进一步分析PEPC基因在谷子逆境应答信号途径中的功能和机制以及为利用基因工程方法改善作物光合速率和提高产量提供数据支持。
1 材料与方法
1.1 试验材料
选用谷子品种‘豫谷1号’, 种子保存于山西省农业科学院谷子研究所生物技术课题。在组培室中培养谷子幼苗, 温度为(22±2)℃, 湿度为60%, 光照周期为16 h光照/8 h黑暗。
1.2 试验设计
待组培室幼苗长至三叶一心期时, 分别对其进行20% PEG-6000模拟干旱、盐(250 mmol L–1NaCl)、ABA (100 µmol L–1)和低温(4℃)胁迫处理, 苗期逆境表达谱分析取样时间点为处理后0、1、3、6、12和24 h, 整株取样[28]。旱棚种植‘豫谷1号’, 对照处理生育期内正常浇水; 干旱处理只浇3次关键水, 分别为拔节(6月20日)、抽穗(8月5日)和灌浆期(8月30日)浇透水, 其他时间采用自然控水方式控水。光照处理为当植株出苗后用黑色遮阳网分别遮挡1层(中等光照, 拔节期、抽穗期、灌浆期测量光照强度分别为221.8、411.6、164.6 µmol m-2s-1), 2层(弱光照, 拔节期、抽穗期、灌浆期测量光照强度分别为52.1、95.8、58.8 µmol m-2s-1)至成熟收获。光照强度用标智光强测定仪(GM1040)于晴天上午9:00—10:00测量10次数值, 取平均值。干旱处理试验与光照处理试验为同一对照。对照、中等光照处理和弱光照处理生育期内正常浇水, 其他农田管理措施相同。分别在拔节、抽穗、灌浆期时取旱棚干旱处理与光照强度处理材料和对照旗叶叶片, 立即在-80℃冰箱中速冻备用。
1.3 谷子PEPC基因的鉴定及基因结构、蛋白序列分析
在Phytozome数据库中以拟南芥和玉米的已知PEPC氨基酸序列为递交序列, 进行BlastP比对, 搜索具有完整阅读框的谷子同源序列, 获得谷子候选PEPC基因。基因参数和蛋白序列来源于Phytozome数据库。利用ExPASy网站在线工具预测蛋白分子量和等电点。利用PLACE在线软件对基因启动子区域顺式元件进行分析, 采用GSDS软件绘制基因结构图, 用在线工具CELLO v.2.5进行亚细胞定位预测, 预测置信度计算参考Yu等[29]描述方法。使用Blast工具在NCBI上查找不同物种氨基酸同源性序列, 用ClustalX1.83软件分析比对基因序列[30], 用Mega6.0软件邻接法构建不同物种N-J系统进化树[31]。
1.4 总RNA提取、cDNA合成及引物设计
按照说明书, 使用生工TRIzol试剂盒提取植物总RNA; 使用生工第1链cDNA合成试剂盒合成cDNA。所用引物由Primer Primer 5.0设计, 引物信息见表1。
1.5 Real-time PCR分析
样品cDNA均一化后作为实时定量PCR模板, 以谷子基因(Seita.7G294000)作为内参基因。反应体系为20 µL, 包含10 µL 2´荧光染料混合液、0.4 µL正向引物(10 µmol L–1)、0.4 µL反向引物(10 µmol L–1)、2 µL cDNA模板、7.2 µL无菌水。经预试验优化后PCR程序为95℃ 3 min; 95℃ 7 s, 57℃ 10 s, 72℃ 15 s, 45个循环。试验设计3次重复, 采用相对定量2–ΔΔCt方法计算基因在某种逆境处理下某个时间点相对于对照的转录水平变化[32]。SiPEPC成员在苗期不同逆境、不同生育期干旱和不同光照下Real-time PCR 表达分析结果利用百迈克云平台在线工具聚类热图绘制(Pretty Heatmaps)热图, 参数默认。
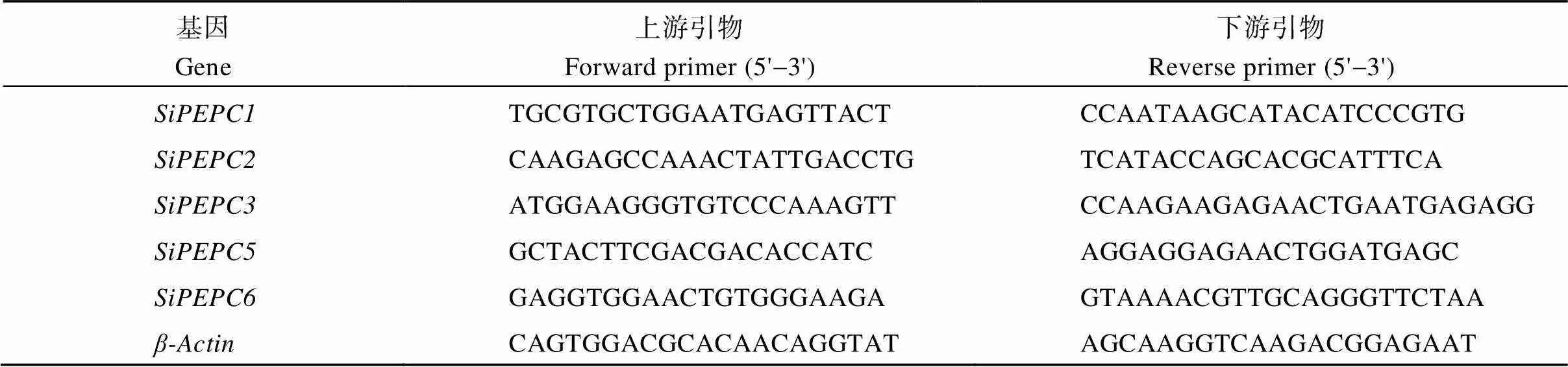
表1 本试验所用引物
2 结果与分析
2.1 谷子PEPC基因成员鉴定与参数分析
通过序列比对和分析, 从谷子中筛选出6个候选基因成员(表2)。根据基因定位在染色体1~9号上的先后位置顺序分别命名为。6个基因不均分布在1号、2号、4号、5号染色体上, 其中分别分布于1号、2号、4号染色体上, 而全部位于5号染色体上。基因有2种转录方式, 其他5个基因只有1种转录方式。功能域分析发现, 所有基因都含有PEPC基因特征功能域PEPcase, 功能域在基因中的详细位置见表2。候选SiPEPC蛋白参数分析表明, SiPEPC成员之间基因组长度变化明显, 在4676~8603 bp之间, 编码氨基酸数目在964~1032之间。成员间分子量范围在109.82~114.89 kD之间, 蛋白等电点范围在5.70~7.14之间, 不稳定指数范围在44.10~52.09之间, 脂肪系数范围在85.81~93.87之间, 平均疏水指数范围在-0.418~-0.277之间。GSDS在线软件分析SiPEPC成员基因结构发现,分别含9、9、8、6、9个内含子, 而含20个内含子(图1)。
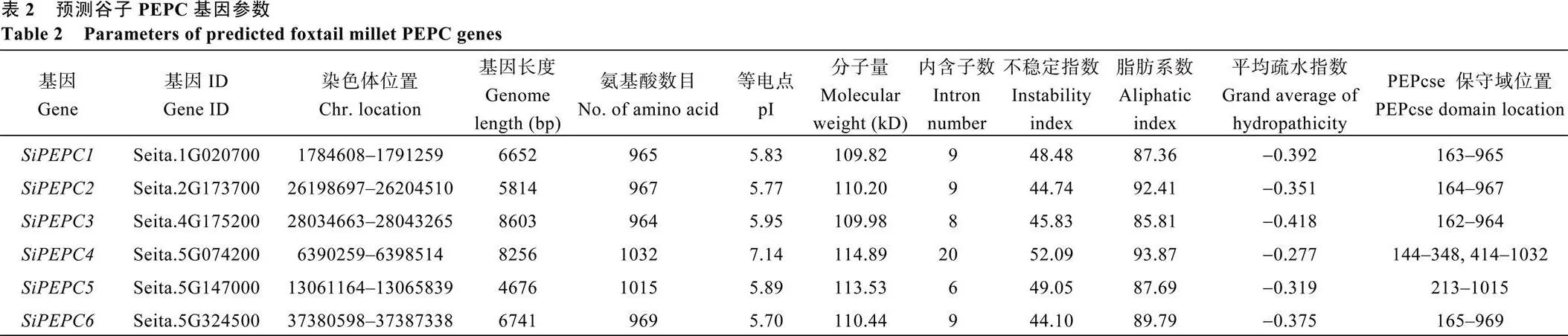
图1 SiPEPC成员的基因结构
Fig. 1 Gene structure of SiPEPC genes
2.2 SiPEPC蛋白亚细胞定位预测和启动子区域顺式元件分析
采用Psort在线工具对谷子SiPEPC家族成员的亚细胞定位预测表明, SiPEPC成员主要被定位在细胞质、细胞核和线粒体(表3)。利用PLACE在线软件对SiPEPC成员启动子顺式元件分析(表4), SiPEPC成员启动子区域主要包括大量的光应答元件、激素类应答元件(脱落酸、赤霉素、水杨酸、乙烯、茉莉酸甲酯、生长素)、逆境应答元件(逆境防御、低温、干旱、伤害)以及其他生长调控相关顺式元件, 包括缺氧特异性诱导、厌氧诱导必需、分生组织特异性表达、玉米蛋白代谢调控、种子特异性调控、昼夜节律元件等。
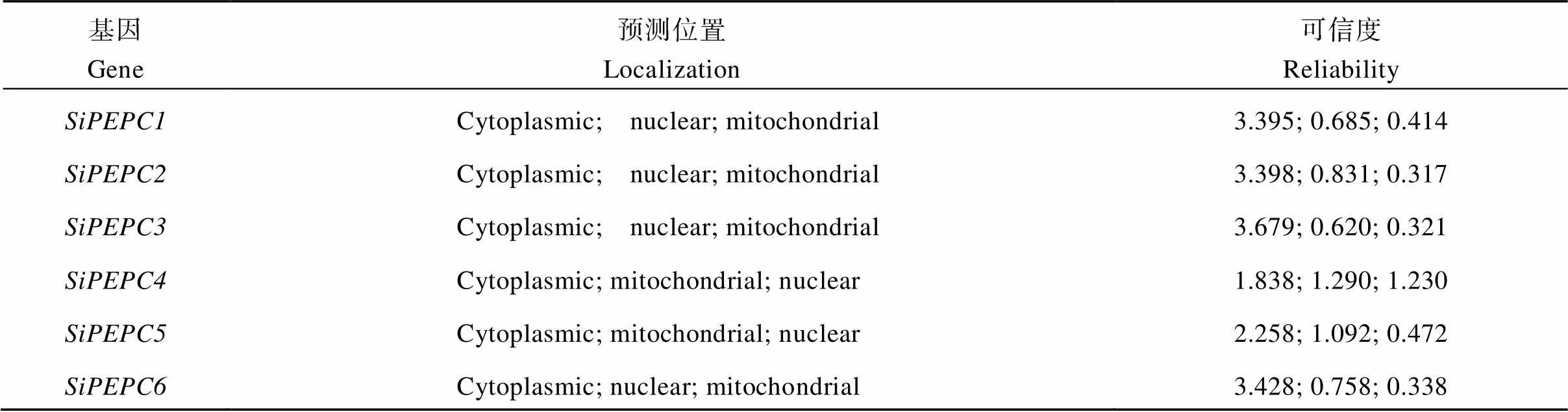
表3 SiPEPC蛋白亚细胞定位预测

表4 SiPEPC基因启动子区域顺式元件预测
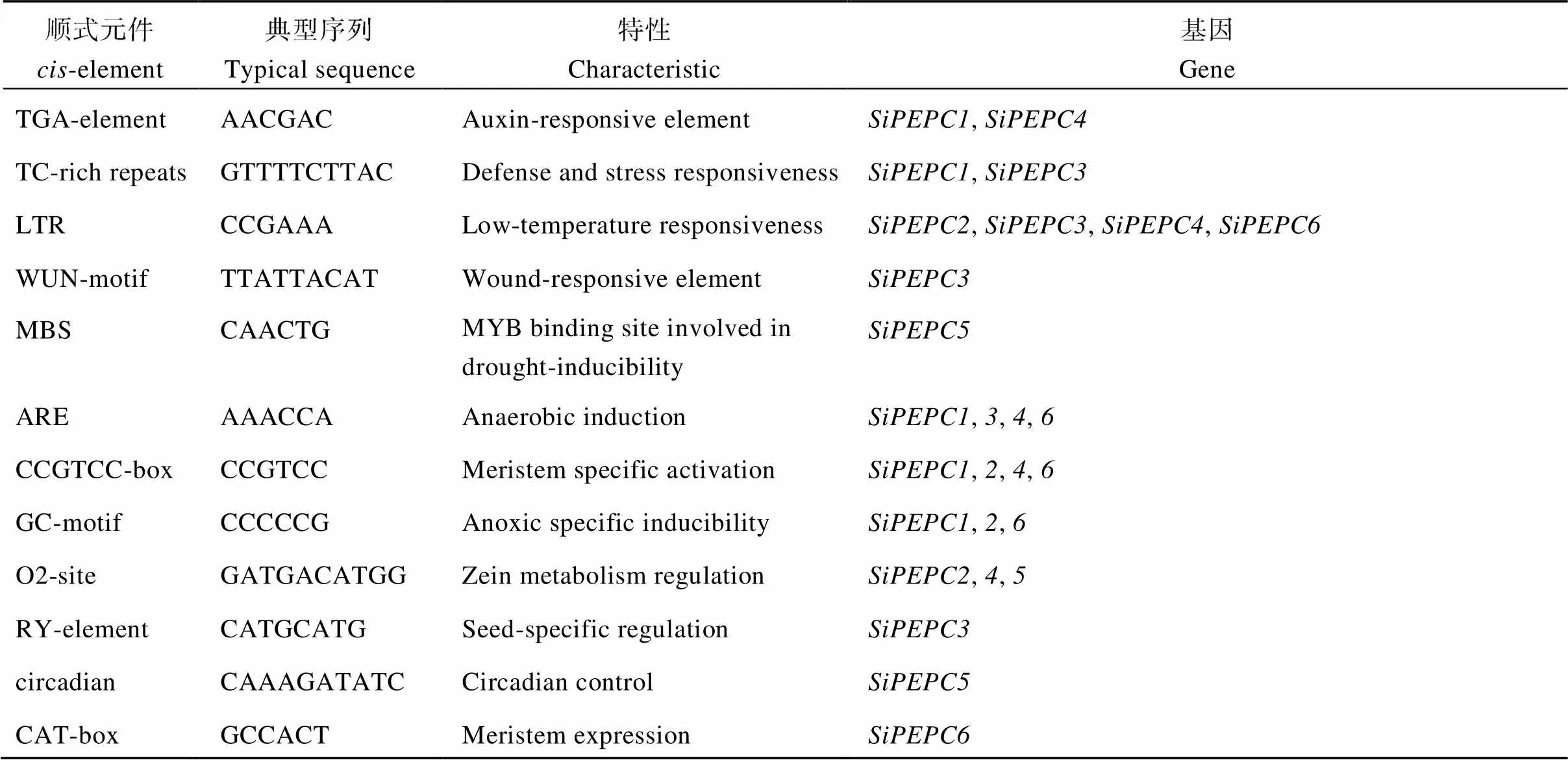
(续表4)
2.3 PEPC序列比对及进化分析
谷子PEPC成员氨基酸序列比对发现成员之间序列非常保守, 相似性较高, 其序列一致性为67.71%。所有成员都含有PEPC蛋白特征功能域PEPcase, 其中SiPEPC1、SiPEPC2、SiPEPC3、SiPEPC5、SiPEPC6序列相似性较高, 整体序列一致性为74.03%, 而SiPEPC4在特征功能域内由于有几个片段的插入和缺失, 因此与其他几个SiPEPC序列一致性较低, 在32.92%~ 34.06%之间。为进一步了解谷子SiPEPC进化关系, 推测其生物功能, 在NCBI数据库中检索了不同物种已知PEPC基因。序列比对发现所有物种PEPC整体一致性非常高, 为72.88%, 说明PEPC基因在进化过程中非常保守。利用 MEGA6.0 软件构建了谷子和不同物种PEPC蛋白成员的Neighbor-Joining进化树(图3)。
从图3可以看出, 谷子的基因与高粱、玉米基因聚合在一起, 揭示谷子与高粱、玉米亲缘关系较近(同属禾本科C4作物), 另一方面也暗示它们在某些功能上可能具有相似性。玉米、高粱、谷子的PEPC成员与拟南芥、马铃薯、花生、蓖麻、烟草PEPC成员相互嵌合在一起, 表明PEPC基因在单、双子叶植物分化以前就已经存在。另外发现一些同源基因对, 例如和、和、和、和和和, 这些相同或不同物种的PEPC基因聚在一起表明, 它们可能由共同祖先进化而来, 而且具有类似的生物功能。
2.4 SiPEPC家族基因苗期非生物逆境胁迫下表达分析
由图4可知, 谷子三叶一心期幼苗在4种胁迫处理下, 除由于表达量低未检测到信号外, 其他5个基因在不同时间点表达水平变化趋势不完全相同。在ABA胁迫处理下所有时间点表达量均下调, 其他基因表达量在4种胁迫处理过程中至少1个时间点有所上调。在ABA-12 h、Cold-24 h、PEG-12 h、NaCl-12 h时其相对表达量达最高, 分别为对照的8.20、9.70、4.85、9.93倍;在ABA-12 h、Cold-24 h、PEG-6 h、NaCl-6 h时其相对表达量达最高, 分别为对照的4.81、8.77、5.69、21.38倍;在ABA-6 h、Cold-24 h、PEG-24 h、NaCl-6 h时其相对表达量达最高, 分别为对照的13.91、1.34、3.55、3.22倍;在ABA-6 h、Cold-24 h、PEG-24 h、NaCl-12 h时其相对表达量达最高, 分别为对照的16.00、31.70、4.31、9.24倍; 而在ABA-12 h、Cold-24 h、PEG-12 h、NaCl-12 h时其相对表达量达最高, 分别为对照的0.93、5.19、3.53、15.68倍。试验表明5个基因都参与了苗期谷子对逆境胁迫的响应。
2.5 不同生育期干旱胁迫及不同光照强度下表达分析
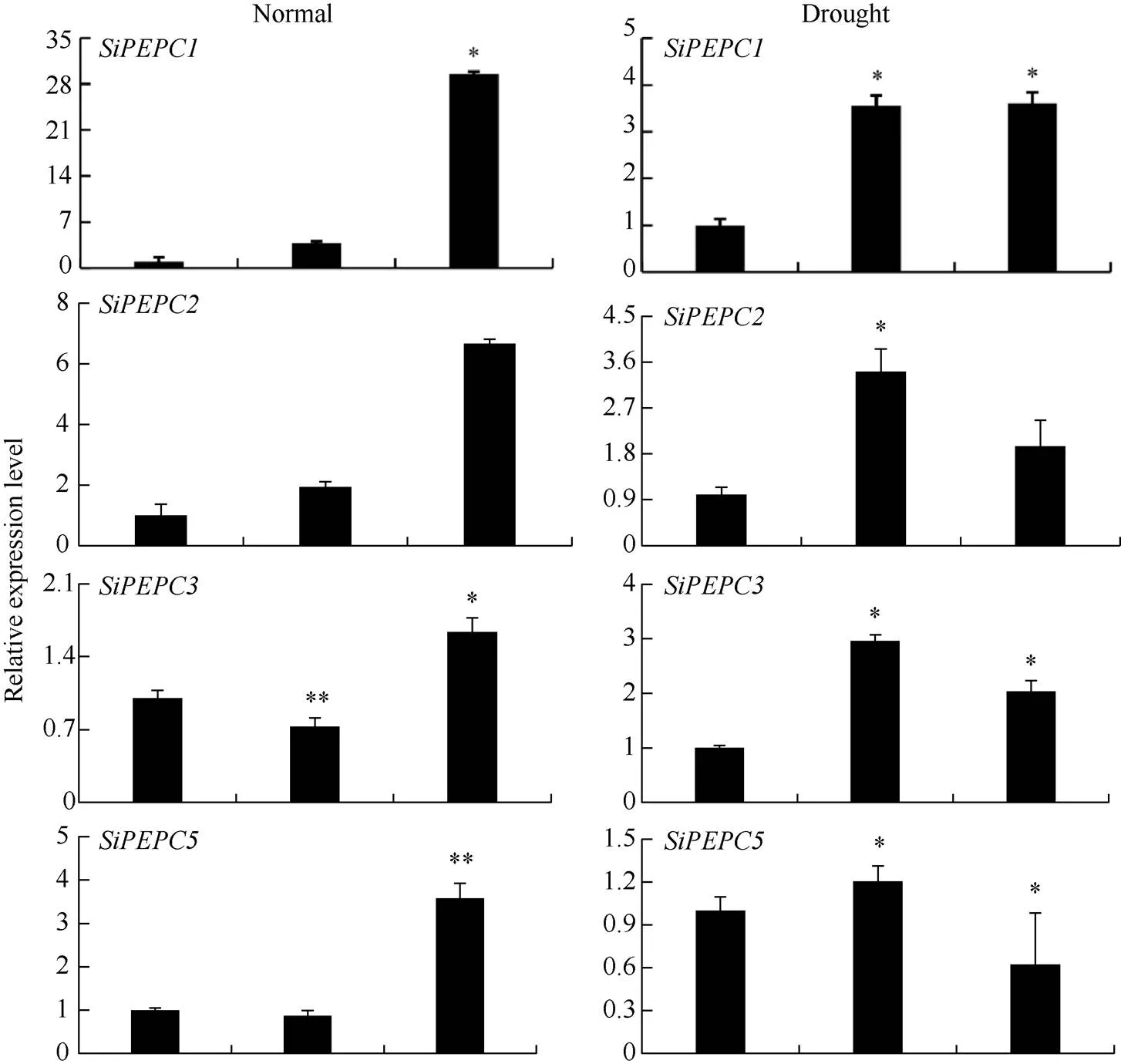
为进一步了解PEPC基因在谷子不同生育期干旱胁迫下的表达情况, 本研究检测了它们在3个关键生育时期(拔节期、抽穗期和灌浆期)干旱胁迫及不同光照强度下的表达情况。由图5可知,在正常抽穗期、灌浆期表达量与拔节期相比均有明显提升, 而在抽穗期表达量下降, 在灌浆期表达量较拔节期有明显提升。干旱胁迫下,在抽穗期、灌浆期表达量与拔节期相比均也有明显提升,在灌浆期、抽穗期表达量与拔节期相比呈现先升后降趋势, 而呈现先降后升趋势。表明大部分SiPEPC基因在正常生长条件下其表达随谷子生长发育进程呈现增强趋势, 在不同生育期干旱胁迫下其表达量趋势都有明显增加, 揭示这5个基因在关键生育时期都参与了对干旱胁迫的响应。

图2 PEPC家族蛋白序列的氨基酸序列多重比对
AtPEPC1、AtPEPC2、AtPEPC3: 拟南芥AtPEPC1、AtPEPC2、AtPEPC3; SbPEPC1、SbPEPC2、SbPEPC3: 高粱SbPEPC1、SbPEPC2、SbPEPC3; GmPEPC: 大豆GmPEPC; ZmPEPC1、ZmPEPC2: 玉米ZmPEPC1、ZmPEPC2; StPEPC: 马铃薯StPEPC; AhPEPC: 花生AhPEPC; RcPEPC: 蓖麻RcPEPC; NtPEPC: 烟草 NtPEPC。对应GenBank蛋白序列号分别为Q9MAH0.1、Q5GM68.2、Q84VW9.2、P29195.1、P29194.1、P15804.2、P51061.1、P04711.2、P51059.1、P29196.2、ABY87944.1、ABR29878.1、P27154.1。相同氨基酸残基用黑色表示, 相似氨基酸残基用灰色表示(相似性≥ 60%)。
AtPEPC1, AtPEPC2, AtPEPC3:AtPEPC1, AtPEPC2, AtPEPC3; SbPEPC1, SbPEPC2, SbPEPC3:SbPEPC1, SbPEPC2, SbPEPC3; GmPEPC:GmPEPC; ZmPEPC1, ZmPEPC2:ZmPEPC1, 2; StPEPC:StPEPC; AhPEPC:AhPEPC; RcPEPC:RcPEPC; NtPEPC:NtPEPC. The corresponding GenBank protein numbers are Q9MAH0.1, Q5GM68.2, Q84VW9.2, P29195.1, P29194.1, P15804.2, P51061.1, P04711.2, P51059.1, P29196.2, ABY87944.1, ABR29878.1, and P27154.1, respectively. The amino acids with an entire homology are shown by a black background, and those shared non-identical conserved identity by a graybackground (≥ 60% similarity).
由图6可知, 所有基因热图颜色从J-1期(拔节期对照)偏黑红色向在J-2期(拔节期干旱)的鲜红色转变, 即所有基因在J-2期表达量与J-1期相比均有所提高, 也就是说拔节期干旱诱导了所有基因的表达。但在J-3期(拔节期中等光照)条件下所有基因表达量全部下降, 而在J-4期(拔节期弱光照)所有基因表达量又全部上升。试验结果说明拔节期光照强度影响基因表达, 弱光条件能诱导基因在拔节期的表达。与拔节期相似, 在H-2期(抽穗期干旱)所有基因表达量较对照H-1期(抽穗期对照)均有明显提高, 但在H-3 (抽穗期中等光照)和H-4 (抽穗期弱光照)条件下所有基因表达量都是下降的。说明抽穗期干旱诱导了所有基因的表达, 抽穗期中等、弱光照抑制基因的表达。在灌浆期的各个处理下(F-1、F-2、F-3、F-4), 除了在F-2期(灌浆期)干旱表达量提升外, 其他所有基因在各处理下表达量均呈下降趋势。说明灌浆期大部分基因在干旱、中等和弱光照处理下表达量下降, 揭示基因在灌浆期可能对干旱、中等光照以及弱光照响应较弱。

图3 不同物种PEPC蛋白的进化关系
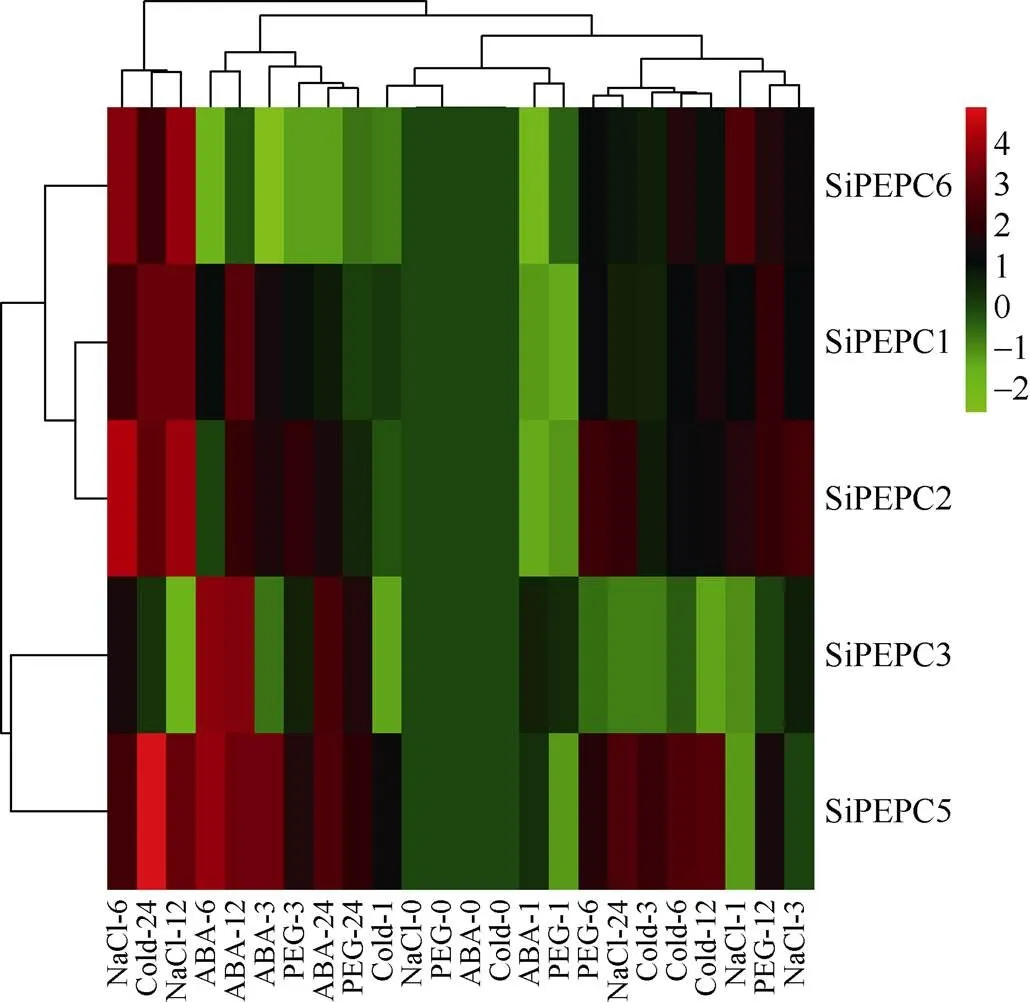
图4 SiPEPC基因苗期逆境表达谱
图中浅绿、深绿、黑色、浅红、深红5种颜色代表基因表达水平。绿色表示基因表达弱, 红色表示基因表达强。
Light green, dark green, black, light red and dark red are used to represent gene expression levels. Green indicates weak gene expression; red indicates strong gene expression.
(图5)
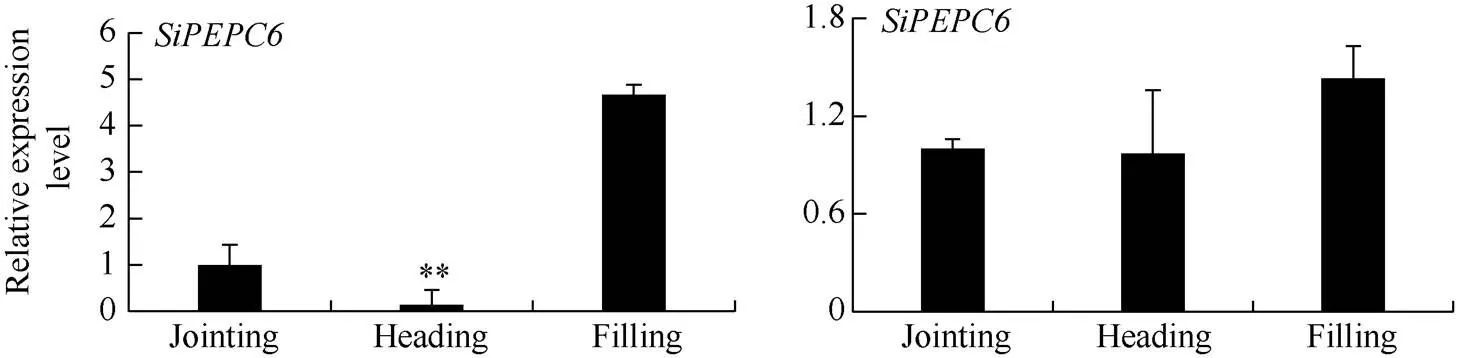
图5 正常和干旱条件下SiPEPC基因在不同发育阶段的相对表达量
*表示在0.05水平上显著; **表示在0.01水平上显著。
* Significantly different at<0.05; ** Significantly different at<0.01.

图6 光照和干旱条件下SiPEPC的相对表达量
图中浅绿、深绿、黑色、浅红、深红五色代表基因表达水平。绿色表示基因表达弱, 红色表示基因表达强。J-1: 拔节期对照; J-2: 拔节期干旱; J-3: 拔节期中等光照; J-4: 拔节期弱光照; H-1: 抽穗期对照; H-2: 抽穗期干旱; H-3: 抽穗期中等光照; H-4: 抽穗期弱光照; F-1: 灌浆期对照; F-2: 灌浆期干旱; F-3: 灌浆期中等光照; F-4: 灌浆期弱光照。
Light green, dark green, black, light red, and dark red are used to represent gene expression levels. Green indicates weak gene expression; red indicates strong gene expression. J-1: control at jointing stage; J-2: drought at jointing stage; J-3: medium light intensity at jointing stage; J-4: weak light intensity at jointing stage; H-1: control at heading stage; H-2: drought at heading stage; H-3: medium light intensity at heading stage; H-4: weak light intensity at heading stage; F-1: drought at filling stage; F-2: drought at filling stage; F-3: medium light intensity at filling stage; F-4: weak light intensity at filling stage.
3 讨论
本研究在谷子基因组中检索得到6个基因, 序列比对显示, 谷子PEPC基因序列相似性很高, 揭示它们在进化上非常保守。系统进化树(图3)表明, 在单、双子叶植物分离以前PEPC基因就已经存在。进化树中谷子成员多与高粱、玉米成员紧密嵌合在一起, 而与拟南芥、马铃薯、花生、蓖麻、烟草成员相对较远, 揭示了谷子与高粱、玉米亲缘关系相对较近, 而与而与拟南芥、马铃薯、花生、蓖麻、烟草亲缘关系相对较远。值得一提的是,基因由于在进化过程中存在一些片段的插入和缺失, 而且在基因组成上含有20个内含子, 远比其他基因内含子数目多, 因此推测在功能上可能与其他基因有所不同。随后的表达分析也证明了这一点, 在所有表达谱分析中都检测不到基因的表达, 说明与其他基因在功能上有明显不同, 可能参与了谷子生长与发育中的其他信号途径。另外在进化树中发现一些同源基因对, 揭示这些同源基因对可能由共同祖先进化而来, 另一方面也暗示它们在某些信号通路中可能具有相似的功能。
随后本研究对谷子6个基因生物信息学特征进行了系统的预测和分析, 期望能够发现对于功能研究有帮助信息。预测结果显示,成员很多性状和参数非常接近类似, 比如亚细胞定位分析成员都主要被定位在细胞质、细胞核和线粒体, 这些位置都是植物光合作用的关键位置, 揭示了基因可能在植物光合作用信号途径中起比较重要作用。这些结论不仅验证了它们同属PEPC基因家族, 而且预示不同基因可能共同参与或调控某些信号途径。顺式元件分析表明, 在启动子区域含有ABA、低温、干旱和防御应激反应等顺式元件。一般来讲如果基因启动子区域存在某种顺式元件则暗示该基因很可能参与相应的信号途径[33]。研究表明在非生物逆境, 比如干旱、盐、低温、高温以及伤害等胁迫下会导致植物细胞内的ABA水平升高, 而且众所周知, ABA是在非生物逆境应答中起重要作用的激素[34-36]。因此我们推测谷子家族基因很可能参与植物对非生物逆境胁迫的响应。
Real-time PCR检测结果表明, 5个谷子基因在4种处理下其表达量有显著变化, 除了几乎所有基因表达量在4种胁迫处理过程中至少1个时间点均有所上调。试验结果表明, 谷子家族基因广泛参与了植物苗期非生物逆境胁迫应答。本研究结果同已有关于PEPC在逆境应答方面研究结果是相一致的[19-21]。为了进一步了解在不同关键生育期干旱胁迫下的表达情况, 本研究检测了成员在关键生育期(拔节期、抽穗期和灌浆期)干旱胁迫及不同光照强度(中等光照强度Ⅰ和弱光照强度Ⅱ)下的表达情况。如图5所示, 试验结果表明基因成员不同处理下表达情况各有特点。例如在正常条件下在抽穗期、灌浆期表达量较拔节期均有明显提升, 而在抽穗期表达下降, 在灌浆期表达明显提升。在干旱胁迫下抽穗期、灌浆期表达较拔节期也有明显提升,在灌浆期、抽穗期表达量呈现先升后降趋势, 而呈现先降后升趋势。结果揭示基因在正常生长条件下随着谷子生长发育进程其表达趋势增强, 在不同生育期干旱胁迫下其表达量趋势都明显增加, 表明5个基因在不同生育期都参与了对干旱胁迫下的响应。不同生育期干旱、不同光照处理下表达分析(图6)表明, 所有基因在J-2期(拔节期干旱)表达量提高, 但在J-3期(拔节期中等光照)全部下降, 而在J-4期(拔节期弱光照)又全部上升。在H-2期(抽穗期干旱)所有基因表达量均有明显提高, 但在H-3 (抽穗期中等光照) H-4 (抽穗期弱光照)条件下都是下降的。在灌浆期的各个处理下(F-1、F-2、F-3、F-4), 只有在F-2期(灌浆期)干旱表达量提升, 其他所有基因在各处理下表达量均呈下降趋势。试验结果说明拔节期干旱和弱光条件能诱导基因的表达, 抽穗期干旱诱导了所有基因的表达, 灌浆期大部分基因在干旱、中等和弱光照处理下表达量下降,基因参与了拔节期和抽穗期的干旱胁迫响应, 不同光照强度影响基因在不同生育期的表达, 弱光可诱导基因在拔节期的表达。光强对PEPC有调节作用, 穗期玉米遮阴后其叶片的PEPC活性显著降低, 且随光照强度的减弱而加剧, 穗期遮阴结束后PEPC活性能恢复到对照水平[37]。本研究结果只表明弱光可诱导基因在拔节期的表达, 未出现随光照强度的减弱而诱导加剧情况, 而在谷子抽穗期和灌浆期大部分基因没有检测到弱光能诱导PEPC表达的现象。这可能是谷子与玉米物种差别或是PEPC基因在不同生育期功能差别造成, 需要我们下一步详尽的试验验证, 但是这些结果进一步验证了顺式元件分析结果, 证明了基因在植物逆境应答中起一定作用。顺式元件分析还显示在基因启动子区域存在茉莉酸甲酯、水杨酸、赤霉素顺式原件。研究表明病原体感染通常导致细胞中茉莉酸甲酯、水杨酸、乙烯等激素水平的增加[38], 因此推断基因可能在生物应激反应中起一些作用。此外还发现了缺氧特异性诱导(GC-motif)、分生组织表达(CAT-box)、厌氧诱导必需(ARE)、玉米醇溶蛋白代谢调控(O2-site)、种子特异性调控(RY-element)、昼夜节律(circadian)等核心元件, 这些顺式元件的存在暗示PEPC可能参与相应的生理生化过程。需要注意的是在启动子区域还发现了大量的光应答元件, 众所周知谷子是光温敏感性作物, 而且PEPC是光合作用中的关键酶, 预示可能参与调控谷子的光温应答调控并在其中起重要作用。
本文报道的谷子基因也为进一步阐明在谷子逆境应答中的功能、机制提供了试验依据。尽管这些这些结论与推测需进一步严谨详尽的试验证明, 但是仍然为我们理解C4植物的关键酶PEPC在非生物逆境下的表达特征及光强对植物PEPC基因表达特性影响提供有益线索, 对深入了解C4植物光合特点对提高C3植物的光合效率和对C3植物的改造将具有一定意义。
4 结论
从谷子全基因组中鉴定出6个PEPC基因成员, 集中分布在谷子的1号、2号、4号、5号染色体上。所有蛋白序列高度保守, 都具有PEPC基因特征保守功能域PEPcase。谷子PEPC家族基因成员主要存在于细胞质、细胞核和线粒体。家族基因广泛参与了谷子苗期非生物逆境胁迫应答。成员参与了谷子在拔节、抽穗、灌浆期的干旱应答, 而光照强度会严重影响基因在不同生育期的表达。
[1] Hibberd J M, Quick W P. Characteristics of C4 photosynthesis in stems and petioles of C3flowering plants.(London), 2002, 415: 451–454.
[2] Lara M V, Chuong S D X, Akhani H, Andreo S C, Edwards G E. Species Having C4single-cell-type photosynthesis in the chenopodiaceae family evolved a photosynthetic phosphoenolpyruvate carboxylase like that of kranz-type C4species., 2006, 142: 673–684.
[3] Merkelbach S, Gehlen J, Denecke M, Hirschet H J, Kreuzaler F. Cloning, sequence analysis and expression of a cDNA encoding active phosphoenolpyruvate carboxylase of the C3plant., 1993, 23: 881–888.
[4] Hart Y, Mayo A E, Milo R, Alon U. Robust control of PEP formation rate in the carbon fixation pathway of C4plants by a bi-functional enzyme., 2011, 5: 171.
[5] 魏绍巍, 黎茵. 植物磷酸烯醇式丙酮酸羧化酶的功能及其在基因工程中的应用. 生物工程学报, 2011, 27: 1702–1710. Wei S W, Li Y. Functions of plant phosphoenolpyruvate carboxylase and its applications for genetic engineering., 2011, 27: 1702–1710 (in Chinese with English abstract).
[6] Lebouteiller B, Gousset-Dupont A, Pierre J N, Bleton J, Tchapla A, Maucourt M, Moing A, Rolin D, Vidal J. Physiological impacts of modulating phosphoenolpyruvate carboxylase levels in leaves and seeds of., 2007, 172: 265–272.
[7] Muramatsu M, Suzuki R, Yamazaki T, Miyao M. Comparison of plant-type phosphoenolpyruvate carboxylases from rice: identification of two plant-specific regulatory regions of the allosteric enzyme., 2015, 56: 468–480.
[8] Sanchez R, Cejudo F J. Identification and expression analysis of a gene encoding a bacterial-type phosphoenolpyruvate carboxylase fromand rice., 2003, 132: 949– 957.
[9] Dong L Y, Masuda T, Kawamura T, Hata S, Izui K. Cloning, expression, and characterization of a root-form phosphoenolpyruvate carboxylase from: comparison with the C4-form enzyme., 1998, 39: 865–873.
[10] Besnard G, Pincon G, Dhonta A, D'Hont A, Hoarau J Y, Cadet F, Offmann B. Characterisation of the phosphoenolpyruvate carboxylase gene family in sugarcane (spp)., 2003, 107: 470–478.
[11] Sullivan S, Jenkins G I, Nimmo H G. Roots, cycles and leaves: Expression of the phosphoenolpyruvate carboxylase kinase gene family in soybean., 2004, 135: 2078–2087.
[12] 蔡小宁, 陈茜, 杨平, 任源浩. 磷酸烯醇式丙酮酸羧化酶的生物信息学分析. 安徽农业科学, 2008, 36: 914–916. Cai X N, Chen X, Yang P, Ren Y H. Bioinformatics analysis of phosphoenolpyruvate carboxylase., 2008, 36: 914–916 (in Chinese with English abstract).
[13] 焦进安. 植物磷酸烯醇式丙酮酸羧化酶的多生理功能. 植物生理学通讯, 1987, (1): 40–43. Jiao J A. Multiple function of phosphoenolpyruvate carboxylase in plants., 1987, (1): 40–43 (in Chinese).
[14] Bandyopadhyay A, Datta K, Zhang J, Yang W, Raychaudhur S, Miyao M. Enhanced photosynthesis rate in genetically engineeredrice expressinggene cloned from maize.(Oxford), 2007, 172: 1204–1209.
[15] 丁在松, 周宝元, 孙雪芳, 赵明. 干旱胁迫下 PEPC 过表达增强水稻的耐强光能力. 作物学报, 2012, 38: 285–292. Ding Z S, Zhou B Y, Sun X F, Zhao M. High light tolerance is enhanced by overexpressed PEPC in rice under drought stress., 2012, 38: 285–292 (in Chinese with English abstract).
[16] 焦德茂, 李霞, 黄雪清, 迟伟, 匡廷云, 古森本. 转基因水稻的光合CO2同化和叶绿素荧光特性. 科学通报, 2001, 46: 414–418. Jiao D M, Li X, Huang X Q, Chi W, Kuang T Y, Gu S B. Photosynthetic CO2assimilation and chlorophyll fluorescence characteristics of transgenic PEPC rice., 2001, 46: 414–418 (in Chinese).
[17] 方立锋, 丁在松, 赵明. 转基因水稻苗期抗旱特性研究. 作物学报, 2008, 34: 1220–1226. Fang L F, Ding Z S, Zhao M. Characteristics of drought tolerance inoverexpressed rice seedlings., 2008, 34: 1220–1226 (in Chinese with English abstract).
[18] Jeanneau M, Gerentes D, Foueillassar X, Zivy M, Vidal J, Toppan A, Perez P. Improvement of drought tolerance in maize: towards the functional validation of thegene and increase of water use efficiency by over-expressing C4-PEPC., 2002, 84: 1127–1135.
[19] Gonzalez M C, Sanchez R, Cejudo F J. Abiotic stresses affecting water balance induce phosphoenolpyruvate carboxylase expression in roots of wheat seedlings., 2003, 216: 985–992.
[20] Sanchez R, Flores A, Cejudo F J. Arabidopsis phosphoenolpyruvate carboxylase genes encode immunologically unrelated polypeptides and are differentially expressed in response to drought and salt stress., 2006, 223: 901–909.
[21] Garciá-Maurino S, Monreal J, Alvarez R, Vidal J, Echevarría C. Characterization of salt stress-enhanced phosphoenolpyruvate carboxylase kinase activity in leaves of sorghum vulgare: independence of osmotic stress, involvement of iontoxicity and significance of dark phosphorylation., 2003, 216: 648–655.
[22] 智慧, 牛振刚, 贾冠清, 柴杨, 李伟, 王永芳, 李海权, 陆平, 白素兰, 刁现民. 谷子干草饲用品质性状变异及相关性分析. 作物学报, 2012, 38: 800–807. Zhi H, Niu Z G, Jia G Q, Chai Y, Li W, Wang Y F, Li H Q, Lu P, Bai S L, Diao X M. Variation and correlation analysis of hay forage quality traits of foxtail millet [(L.) Beauv.]., 2012, 38: 800–807 (in Chinese with English abstract).
[23] Devos K M, Wang Z M, Beales J, Sasaki T, Gale M D. Comparative genetic maps of foxtail millet () and rice ()., 1998, 96: 63–68.
[24] Jayaraman A, Puranik S, Rai N K, Vidapu S, Sahu P P, Lata C, Prasad M. cDNA-AFLP analysis reveals differential gene expression in response to salt stress in foxtail millet (L.)., 2008, 40: 241–251.
[25] 赵晋锋, 余爱丽, 田岗, 杜艳伟, 郭二虎, 刁现民. 谷子CBL基因鉴定及其在干旱, 高盐胁迫下的表达分析. 作物学报, 2013, 39: 360–367. Zhao J F, Yu A L, Tian G, Du Y W, Guo E H, Diao X M. Identification of CBL genes from foxtail millet ([L.] Beauv.) and its expression under drought and salt stresses., 2013, 39: 360–367 (in Chinese with English abstract).
[26] Zhang G Y, Liu X, Quan Z W, Cheng S F, Xu X, Pan S K, Xie M, Zeng P, Yue Z, Wang W L, Tao Y, Bian C, Han C L, Xia Q J, Peng X H, Cao R, Yang X H, Zhan D L, Hu J C, Zhang Y X, Li H N, Li H, Li N, Wang J Y, Wang C C, Wang R Y, Guo T, Cai Y J, Liu C Z, Xiang H T, Shi Q X, Huang P, Chen Q C, Li Y R, Wang J, Zhao Z H, Wang J. Genome sequence of foxtail millet () provides insights into grass evolution and biofuel potential., 2012, 30: 549–554.
[27] Bennetzen J L, Schmutz J, Wang H, Percifield R, Hawkins J, Pontaroli A C, Estep M, Feng L, Vaughn J N, Grimwood J, Jen-kins J, Barry K, Lindquist E, Hellsten U, Deshpande S, Wang X W, Wu X M, Mitros T, Triplett J, Yang X H, Ye C Y, Mauro-herrera M, Wang L, Li P H, Sharma M, Sharma R, Ronald P C, Panaud O, Kellogg E A, Brutnell T P, Doust A N, Tuskan G A, Rokhsar D, Devos K M. Reference genome sequence of the model plant setaria., 2012, 30: 555–561.
[28] Shinozaki K, Yamaguchi-Shinozaki K. A novel-acting element in angene is involved in responsiveness to drought, low-temperature, or high-salt stress., 1994, 6: 251–264.
[29] Yu C S, Chen Y C, Lu C H, Hwang J K. Prediction of protein subcellular localization., 2006, 64: 643–651.
[30] Larkin M A, Blackshields G, Brown N P, Chenna R, Mcgettigan P A, McWilliam H, Valentin F, Wallace I M, Wilm A, Lopez R. Clustal W and Clustal X version 2.0., 2007, 23: 2947–2948.
[31] Tamura K, Stecher G, Peterson D, Filipski A, Kumar S. MEGA6: molecular evolutionary genetics analysis version 6.0., 2013, 30: 2725–2729.
[32] Livak K J, Schmittgen T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2–ΔΔCt., 2001, 25: 402–408.
[33] Narusaka Y, Nakashima K, Shinwari Z K, Sakuma Y, Furihata T, Abe H, Narusaka M, Shinozaki K, Yamaguchi-shinozaki K. Interaction between two-acting elements, ABRE and DRE, in ABA-dependent expression ofgene in response to dehydration and high-salinity stresses., 2003, 621: 137–148.
[34] Nakashima K, Yamaguchi-shinozaki K. ABA signaling in stress-response and seed development., 2013, 32: 959–970.
[35] Verma V, Ravindran P, Kumar P P. Plant hormone-mediated regulation of stress responses., 2016, 16: 86.
[36] Zhang J, Jia W, Yang J, Ismail A M. Role of ABA in integrating plant responses to drought and salt stresses., 2006, 97: 111–119.
[37] 张吉旺, 董树亭, 王空军, 胡昌浩, 刘鹏. 大田遮荫对夏玉米光合特性的影响. 作物学报, 2007, 33: 216–222. Zhang J W, Dong S T, Wang K J, Hu C H, Liu P. Effects of shading in field on photosynthetic characteristics in summer corn., 2007, 33: 216–222 (in Chinese with English abstract).
[38] Bari R, Jones J D. Role of plant hormones in plant defence responses., 2009, 69: 473–488.
Identification of PEPC genes from foxtail millet and its response to abiotic stress
ZHAO Jin-Feng**, DU Yan-Wei**, WANG Gao-Hong, LI Yan-Fang, ZHAO Gen-You, WANG Zhen-Hua, WANG Yu-Wen, and YU Ai-Li*
Millet Research Institute, Shanxi Academy of Agricultural Sciences / Shanxi Key Laboratory of Genetic Resources and Breeding in Minor Crops, Changzhi 046011, Shanxi, China
Phosphoenolpyruvate carboxylase (PEPC) is a key enzyme in photosynthesis of C4plants and plays an important role in a variety of metabolic and stress pathways. In this study, we identified six candidate PEPC genes from foxtail millet genome via sequence alignment. The characteristic parameters of allprotein were very similar and the sequences were very conservative. Allgenes contained the PEPcase motif, which is the characteristic domain ofgene.were localized in cytoplasm, nucleus and mitochondrion. Promoter analysis identified a variety of light, hormonal, stress, and other growth-related cis-elements in the promoter sequences ofmembers. The qRT-PCR expression profiles showed that the fivegenes (,,,,) were induced by ABA, PEG, high salt and low temperature at seedling stage, indicating that fivegenes ate involved in abiotic signaling pathway at the seedling stage. The expression of fivegenes increased with the growth of foxtail millet under normal growth conditions, and increased significantly under drought stress at different growth stages, indicating that fiveare involved in drought stress response at jointing, heading and filling stages. The weak light at jointing stage could induce the expression of fivegenes, while the expression level decreased sharply under moderate light intensity at jointing stage, moderate and weak light intensity at heading and filling stages, showing that light intensity seriously affects the expression ofgenes.
foxtail millet; phosphoenolpyruvate carboxylase; abiotic stress; expression analysis
10.3724/SP.J.1006.2020.94107
本研究由山西省农业科学院项目(YGJPY2009, YGG17021, YCX2019T05), 国家现代农业产业技术体系建设专项(CARS-06-13.5-A23)和国家农业环境数据中心观测检测任务(ZX03S0410)项目资助。
This study was supported by the Project Plan of Shanxi Academy of Agricultural Sciences (YGJPY2009, YGG17021, YCX2019T05), the China Agricultural Research System (CARS-06-13.5-A23), and the National Agricultural Environmental Data Center Observation and Detection Mission (ZX03S0410).
余爱丽, E-mail: yuailimail@163.com, Tel: 0355-2204195
同等贡献(Contributed equally to this work)
赵晋锋, E-mail: zhaojfmail@126.com, Tel: 0355-2204195
2019-07-24;
2020-01-15;
2020-02-14.
URL: http://kns.cnki.net/kcms/detail/11.1809.S.20200214.1339.002.html
