岩原鲤遗传多样性和种群历史动态研究
2020-04-06张晓宇张富铁姚富城王剑伟
张晓宇 张富铁 姚富城 王剑伟
(1. 中国科学院水生生物研究所中国科学院水生生物多样性与保护重点实验室, 武汉 430072;2. 中国科学院大学, 北京 100049)
岩原鲤(Procypris rabaudi), 隶属于鲤形目(Cypriniformes)鲤科(Cyprinidae)原鲤属(Procypris),是长江上游特有鱼类和重要经济鱼类。分布于金沙江、岷江、赤水河、嘉陵江和长江上游等干支流[1]。岩原鲤是生活在水流较缓区域的杂食性底层鱼类, 喜欢栖息于江河岩石缝间[2]。目前岩原鲤种群资源衰退严重, 面临着种群小型化趋势, 长江上游水电梯级开发、过度捕捞及其他人类活动的影响已成为其面临的主要威胁[3]。在《中国濒危动物红皮书》中岩原鲤被列为我国易危鱼类[4]。为了保护及合理开发岩原鲤资源, 开展群体遗传多样性研究十分必要。
近年来, 国内学者对岩原鲤开展了分子系统发育、遗传多样性和种群结构等方面研究。如: 宋君[5]于2002—2005年分别在合江、通江、苍溪、北碚、木洞和习水6个水体采集岩原鲤50尾, 分析其mtDNA控制区核苷酸序列后, 得出单倍型多样性指数Hd为0.736, 核苷酸多样性指数Pi为0.0036; 王爽等[6]用磁珠富集法构建得到岩原鲤GATA四碱基重复的10个微卫星位点, 并对25个岩原鲤个体进行检测, 结果表明观测杂合度和期望杂合度分别在0.460—0.788和0.375—0.739; 朱成科等[7]对2008年以前采集的三峡水库木洞、涪陵和万州3个群体45尾岩原鲤样本进行线粒体控制区序列分析, 结果说明三个群体属于同一种群, 整体上单倍型多样性指数Hd为0.857, 核苷酸多样性指数Pi为0.00472。何勇凤等[8]利用线粒体DNA细胞色素b基因标记对2008年以前在塘河和木洞采集的共57个个体进行分析, 两个群体遗传多样性水平相当, 两群体平均单倍型多样性指数Hd为0.8828, 核苷酸多样性指数Pi为0.0019。程飞等[9]基于10个微卫星位点对江津地区野生群体(n=50)、宜宾珍稀动物研究所养殖群体(n=51)和四川内江特种水产养殖场的养殖群体(n=45)进行分析, 三个种群的平均观测杂合度和期望杂合度分别为0.44、0.47、0.41和0.8、0.78、0.75, 认为人工养殖群体的增殖放流会降低野生群体的遗传多样性水平。
目前, 从分子水平上研究动物种间或种内的遗传变异和系统演化关系已成为必然趋势。线粒体DNA(mtDNA)结构简单, 进化速率快, 多用于鱼类系统发育和群体遗传研究[10], 其中细胞色素b基因(Cytb)为重要的蛋白质编码基因。Cytb基因具有种间差异; 其序列易于获得和检测, 遗传信息表达清晰; 在进化的过程中, Cytb基因序列变异速率适中, 在系统进化和分类研究、群体遗传结构研究等方面有很强的适用性[11,12]。除了何勇凤等[8]2008年的文章外, 以岩原鲤的Cytb基因序列分析为基础的遗传多样性研究尚无最新报道。甚至近10年来, 没人利用重新采集的岩原鲤样本进行遗传多样性方面的研究工作。而这十年中, 因金沙江水电梯级开发等因素, 长江上游水文形势急剧变化, 岩原鲤的遗传多样性水平可能也随之发生变化。那么岩原鲤种群的遗传多样性如何变化?现在应该如何加强长江上游特有鱼类保护工作?这些都是特有鱼类资源保护中急切需要了解的问题。本研究基于Cytb基因序列分析, 对我国长江上游岩原鲤赤水群体和万州群体2013—2017年间161尾样本进行了分析, 以期为该鱼的遗传多样性研究提供基础资料,并为其物种资源的保护提供详实可靠的科学依据。
1 材料与方法
1.1 材料
2013—2017年收集赤水和万州2个采样点的岩原鲤样本161尾。本实验所用样本均为随机采样。野外样本采集时以定置刺网捕捞为主, 同时对其形态进行初步观察鉴定。将样本保存于95%酒精中带回实验室, 样本储存于4℃的冰箱中, 并标注学名,编号, 日期, 保存于中国科学院水生生物研究所淡水鱼类博物馆。另外, 在GenBank中下载乌原鲤(P.mera, 登录号为YP_009110302.1)的Cytb序列作为系统树构建的外类群。
1.2 基因组DNA提取
基因组DNA的提取采用高盐抽提法[13], 提取过程中应注意尽量减少DNA的降解, 避免非核酸类成分污染, 保证核酸样品的完整性。DNA充分溶解后采用1%琼脂糖(德国Biofroxx生产)凝胶电泳检测DNA浓度和质量, 储存于-20℃冰箱。
1.3 PCR扩增及测序
扩增岩原鲤Cytb基因的引物序列为L14724和H15915[14]。引物由上海生物工程有限公司合成。其中上游引物为L14724(5′-GACTTGAAAAACCAC CGTTG-3′, 下游引物为H15915(5′-CTCCGATC TCCGGATTACAAGAC-3′)。
PCR反应体系(Transgene公司生产)总体积为20 μL, 其中10×buffer 2.0 μL, dNTP (2.5 mmol/L)1.5 μL,Taq聚合酶0.3 μL, 正反向引物(10 μmol /L)各1 μL, DNA 1 μL, 其余体积用双蒸水补足。PCR反应程序为: 94℃预变性4min; 94℃变性45s, 54℃退火45s, 72℃延伸1min, 30个循环; 72℃终延伸10min。
PCR产物用1%琼脂糖凝胶电泳检测, 对目的条带清晰的PCR产物直接送上海生物工程有限公司进行纯化及序列测定, 为保证所测序列的可靠性,所有样品均采用双向测序。
1.4 数据处理及分析
用CLUSTALX 1.81软件[15]对mt DNA Cytb基因序列进行同源序列比对分析, Seaview[16]软件辅以手工校正。
用Dnasp5.0软件[17]进行单倍型数目、多态位点数目、单倍型多样性、核苷酸多样性指数和平均核苷酸差异数等主要遗传多样性统计值的计算;利用Tajima’sD[18]和Fu’sFs[19]中性检验分析两个岩原鲤种群历史上是否发生扩张。
用MEGA6.0[20,21]软件计算核苷酸中的碱基含量, 计算转换与颠换比值(transitions/transvertions,Si/Sv)及基于Kimura 2-参数模型校正的种群间遗传距离和平均种群内遗传距离。利用MEGA6.0软件,以乌原鲤为外类群, 采用最大似然法(Maximum Likelihood, ML) 构建单倍型分子系统树, 并对各分支进行2000次重复检验。
利用遗传分化分析软件Arlequin 3.11[22]计算出遗传分化参数(Fst), 并利用分子变异分析方法进行AMOVA分析, 分析群体内及群体间变异程度; 为验证中性检验的结果, 采用曲线拟合的方式, 即核苷酸配对的可期望值和观察值的曲线是否拟合, 通过两个参数评估扩张的可靠性: SSD(Sum of Square Deviations)和r(Harpending’s raggedness index)[23];同时, 对于经历种群扩张的群体, 进一步估算种群扩张时间, 利用公式τ=2ut[24]计算,τ为Tau(种群扩张参数),u为每个世代每条序列的变异速率,t为每个世代种群的扩张时间; 其中u=2μk,μ为每个碱基的变异速率, 参照文献[25], 采用1% per Myr (1×108),k表示序列长度(bp)。扩张时间T=t×世代时间,T值由Arlequin软件计算得到。
2 结果
2.1 Cyt b序列分析
本研究获得161条岩原鲤个体线粒体Cytb基因序列, 比对校正后序列长度为1012 bp。序列中无碱基的短缺或插入。161条序列检测到18个变异位点,其中简约信息位点11个, 单一突变位点7个, 总体变异较小。161条序列的平均碱基组成如下: T=27.9%,C=28.2%, A=30.2%, G=13.7%。所有Cytb基因序列的转换和颠换均未达饱和, 转换数明显大于颠换数,平均转换/颠换比(Ti/Tv)比值为2.80。(A+T)含量(58.1%)大于(G+C)含量(41.9%), G的含量最低, 存在较强的碱基组成偏倚性, 符合线粒体蛋白质编码基因的特点, 与其他鱼类线粒体DNA的研究结果相
似[26]。
2.2 遗传多样性
整个群体的单倍型多样性(Hd)和核苷酸多样性(Pi)分别为0.590和0.00132, 表现出较高的单倍型多样性和较低的核苷酸多样性(表 1)。在各地理群体中, 万州群体120个样本中单倍型个数为12, 单倍型多样性指数为0.628, 核苷酸多样性指数为0.00141;赤水群体41个样本中单倍型个数为7, 单倍型多样性指数为0.460, 核苷酸多样性指数为0.00101。万州群体表现出较高的单倍型多样性和较低的核苷酸多样性, 而赤水群体遗传多样性水平明显低于万州群体。如表 2、表 3所示, 2013年赤水群体遗传多样性水平最低, 2015年万州群体遗传多样性水平最高; 随着年份的增加, 万州群体的单倍型多样性和核苷酸多样性均呈现逐年降低的趋势, 但是赤水群体的单倍型多样性和核苷酸多样性呈现逐年增加的趋势。
2.3 分子系统进化
以Network软件中接法构建得到岩原鲤各单倍型网络图(图 1)。所有161个样本的Cytb基因序列共识别出15个单倍型。其中, 赤水(CS)种群41条序列共检测到7个单倍型, 万州(WZ) 种群120条序列共检测到12个单倍型。种群间共享单倍型共4个,其中单倍型H-2在群体间都有分布且数量也大, 可能是原始单倍型, 其他单倍型直接或间接由单倍型H-2通过碱基突变发展而来。
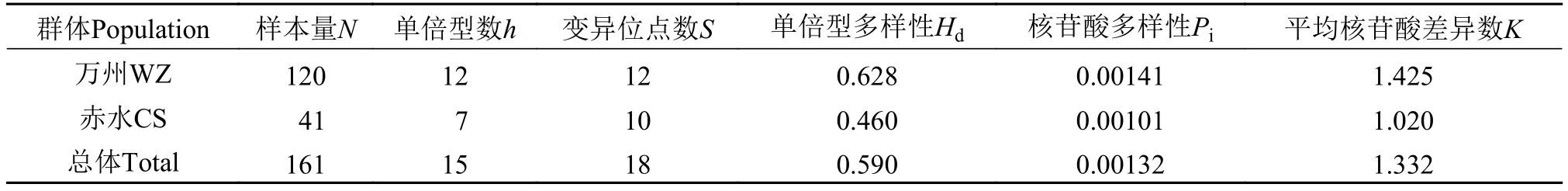
表 1 基于Cyt b基因的岩原鲤群体遗传多样性分析Tab. 1 Genetic diversity of P. rabaudipopulations based on Cyt b gene

表 2 基于Cyt b基因的万州不同年份岩原鲤群体遗传多样性分析Tab. 2 Genetic diversity of P. rabaudi in different years in Wanzhou based on Cyt b gene

表 3 基于Cyt b基因的赤水不同年份岩原鲤群体遗传多样性分析Tab. 3 Genetic diversity of P. rabaudiin different years in Chishui based on Cyt b gene
以乌原鲤为外类群, 利用邻接法(Neighbor Joining, NJ)和最大似然法(Maximum Likelihood,ML)构建单倍型系统发育树。结果显示两种方法做出的系统发育树拓扑结构相似, 以ML树为例(图 2)。在15个单倍型中较为明显的两支: Hap3、Hap6、Hap8和Hap4聚为一支; Hap1和Hap14聚为一支。其他单倍型相互混杂, 各自聚为单系。其中单倍型Hap1、Hap2、Hap4、Hap5为共享单倍型, Hap3、Hap6和Hap7为赤水群体所特有, 其余Hap8、Hap9和Hap10等8个单倍型为万州所特有。单倍型系统发育树和网络图结果一致, 均未呈现明显的地理分布格局。

图 1 岩原鲤各单倍型间的进化网络图(灰色代表万州群体, 黑色代表赤水群体)Fig. 1 Statistical parsimony network based on Cyt b haplotype frequencies of P. rabaudi (Gray means Wanzhou population and black indicates Chishui population)

图 2 基于Cyt b基因序列构建的岩原鲤单倍型ML树Fig. 2 Phylogenetic tree of haplotypes by ML analysis based on Cyt b gene sequences
2.4 群体遗传结构
以Mega软件的Kimura 2-parameter遗传距离模型计算得到2个地理种群内和种群间的遗传距离。群体内的遗传距离显示, 2个种群之间的遗传距离为0.001; 各种群内平均遗传距离均为0.001。
使用Arlequin 3.11计算岩原鲤各地理群体间遗传分化系数(Fst), 分析结果表明: 2个地理群体间没有出现遗传分化现象(Fst=0.01749,Fst<0.05,P>0.05)。DnaSP软件分析单倍型数据所得两个地理群体间的遗传分化系数 (Gst) 为 0.01002(P>0.05),基因流(Nm)为24.71, 表明两个地理群体间无分化,基因交流很频繁。
采用Arlequin 3.11软件对万州群体和赤水群体进行分子变异分析(AMOVA)。分析结果显示: 岩原鲤群体间的遗传变异占1.75%, 群体内的变异占98.25%(表 4)。由此可见, 分子之间的遗传差异主要发生在群体内的个体之间。
2.5 种群扩张
使用Dnasp软件对2个群体所有的161个岩原鲤个体进行Tajima’sD检验和Fu’sFS检验结果见表 5。结果表明: Tajima’sD检验中赤水和万州群体均为负值, 但均未达到显著性水平(P>0.05)。在Fu’sFS检验中, 万州群体和赤水群体的FS值为负, 赤水群体未达到显著性水平 (P>0.05), 而万州群体达到显著性水平(0.01<P<0.05)。以上结果表明, 万州群体可能经历了种群扩张事件。
使用Arlequin 3.11软件基于碱基差异数绘制预期值和实际观测值的拟合曲线(图 3)。结果显示万州群体实际观测结果与期望的假定模型吻合度较高。同时万州群体的SSD值未达到显著性水平; 粗糙指数r值较小且不显著。以上结果均说明万州群体出现种群扩张现象。
采用1% per Myr的突变速率, 本研究推算出万州群体的大约扩张时间为0.15百万年前。

表 4 基于Cyt b基因对岩原鲤不同地理群体的AMOVA分析Tab. 4 AMOVA analysis for P. rabaudi between two populations based on Cyt b gene

表 5 基于Cyt b基因的岩原鲤群体中性检验Tab. 5 Statistical tests for neutrality of P. rabaudi based on Cyt bgene
3 讨论
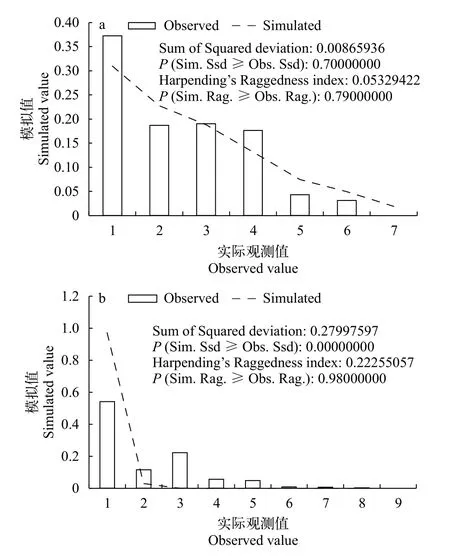
图 3 基于Cyt b基因的万州群体(a)、赤水群体(b)碱基错配分布图Fig. 3 Mismatch distribution of P. rabaudi in Wanzhou (a) and Chishui (b) based on Cyt b gene
3.1 群体遗传多样性
遗传多样性是种群进化的基础, 能够影响种群自身的长期生存能力[27]。通常高的遗传多样性会使物种拥有更强的抵御不良环境的能力和进化的潜力, 遗传多样性对物种的演化和保存有重要意义[28]。本研究中2015—2017年万州群体的单倍型多样性和核苷酸多样性逐年降低, 而2013—2017年赤水群体的单倍型多样性和核苷酸多样性逐渐升高。程飞等[9]指出人工繁殖岩原鲤群体的放流会造成野生群体遗传多样性的下降, 万州群体遗传多样性水平总体呈现下降趋势, 这可能是由于万州地区近年来频繁的岩原鲤人工增殖放流造成的。2013—2017年间重庆万州曾多次开展增殖放流活动。其中万州段长江流域水域2013年放流较大规格的岩原鲤等8种鱼类, 共计502.83万尾(万州区增殖放流项目);2015—2017年放流不同规格的岩原鲤等7个种(类),共计1784万尾(万州区鱼类增殖放流项目)(数据来源于三峡后续工作规划鱼类增殖放流项目实施效果评价报告, 国务院三峡办移民管理咨询中心和水利部中国科学院水工程生态研究所于2017年12月编; 未公开资料)。我们通常在每年5月份和10月份进行万州渔获物调查和分子样收集工作。其中发现是有小部分岩原鲤个体与放流岩原鲤个体大小相近。因此作出“遗传多样性降低可能是增殖放流引起的”的推断。赤水种群遗传多样性总体呈上升趋势, 可能是赤水群体原有群体遗传多样性水平较低, 但是随着地理群体间频繁的基因交流, 赤水群体遗传多样性水平慢慢升高。从线粒体DNA分析的结果来看, 与长江上游其他特有物种相比, 如厚颌鲂[29](单倍型多样性和核苷酸多样性分别为0.673和0.00446)、半䱗[30](单倍型多样性和核苷酸多样性分别为0.895和0.00487), 岩原鲤的单倍型多样性和核苷酸多样性值均相对较小, 群体的遗传多样性水平还较低。
国内关于岩原鲤线粒体基因遗传多样性的研究较少, 特别是近十年来没有相关研究工作。本研究中样本采集时间为2013—2017年, 覆盖了中间大部分空缺时间。结合2008年之前的基于线粒体序列的遗传多样性研究资料: 宋君[5]根据mtDNA控制区核苷酸序列得出岩原鲤群体的单倍型多样性指数Hd为0.736, 核苷酸多样性指数Pi为0.0036; 朱成科等[7]根据mtDNA控制区核苷酸序列得出岩原鲤群体的单倍型多样性指数Hd为0.857, 核苷酸多样性指数Pi为0.00472; 何勇凤等[8]根据mtDNACytb序列得出岩原鲤群体的单倍型多样性指数Hd为0.8828, 核苷酸多样性指数Pi为0.0019。本研究中2个地理群体整体的单倍型多样性和核苷酸多样性分别为0.590和0.00132, 可以发现长江上游岩原鲤群体的遗传多样性整体上呈下降趋势。其原因可能是: (1)岩原鲤的自然繁殖率低, 自然分布区域有限, 本身在自然界中的资源就偏少。(2)过度捕捞和非法捕捞, 如使用电动捕鱼机和网眼密集的渔网,严重破坏了自然资源。段辛斌等[31]指出, 与水库蓄水前相比, 现在渔船数量增加, 捕捞强度增加, 捕捞效率显著提高。三峡库区渔业资源呈现持续下降趋势, 过度捕捞是造成这一现象的主要原因。过度捕捞在一定程度上导致了水库鱼类资源的减少, 其中包括岩原鲤种群[32]。(3)水体污染和非法采砂严重影响岩原鲤的生存环境, 这种环境的持续变化不利于岩原鲤种群的生存, 直接造成资源量减少、遗传多样性下降。
之前宋君[5]曾十分全面地对2003—2005年合江(采自长江支流;Hd为0.667,Pi为0.0026), 通江(渠江;0.750, 0.0029), 苍溪(嘉陵江; 0.758, 0.0032), 木洞(长江干流; 0.933, 0.0051), 习水(长江支流; 1.000,0.0056), 北碚(嘉陵江; 0.250, 0.0014)六个群体共50尾野生岩原鲤个体进行线粒体控制区序列分析。而在本文章中涉及的地理群体分别为2013—2017年赤水群体(0.460, 0.00101)和万州群体(0.628,0.00141)。一般来讲, 鱼类线粒体基因组中控制区的进化速率最快[33]。2003—2005年, 除了北碚群体其他五个群体的单倍型多样性较高, 且有两个群体(木洞、习水群体)也具有较高的核苷酸多样性。但是在2013—2017年, 赤水群体的单倍型多样性和核苷酸多样性都是较低的, 同时万州群体的单倍型多样性和核苷酸多样性水平(Cytb水平)同之前年份的数据(D-Loop水平)来看也是相对较少的。后面我们可以进一步利用线粒体控制区序列来比较两个时间段的遗传多样性差异。
3.2 群体遗传结构
遗传距离的大小指示着遗传变异水平的高低,也代表着样本亲缘关系的远近。本研究中岩原鲤2个地理群体间遗传距离为0.001, 远远小于0.05, 根据Shaklee[34]的结果, 说明岩原鲤群体间的遗传分化极低; 两个地理群体的群体内平均距离也很小, 也说明无论是群体间的序列差异还是群体内部的差异都是很少的。
群体分化系数(Fst)是一个反映种群遗传分化水平的重要参数, 常用来表示两个群体间的基因流和遗传漂变的程度。固定系数(Fst)值一般和群体的遗传分化程度呈正相关。本研究Fst值为0.01749且不显著, 按照Wright[35]分类标准,Fst值小于0.05, 说明2个岩原鲤群体间不存在遗传分化。
分子方差分析结果表明大部分的遗传变异来自群体内, 说明岩原鲤群体间差异不显著, 群体间的差异远小于群体内的差异。本研究基因流值(Nm=24.71)较大, 按照Allwndorf[36]结果: 说明2个群体间基因交流很频繁, 也进一步说明岩原鲤群体间未出现分化。
ML系统树进化分析和单倍型网络图表明2个岩原鲤群体的单倍型相互混杂, 群体之间并没有明显的地理分布格局, 其中万州群体中单倍型形成单系较多, 这个结果可能是由于万州群体的样本数量较多。
综合看来, 赤水和万州群体在总体上并未出现遗传分化, 我们的采样点位于重庆市万州区长江干流部分和贵州省赤水市赤水河支流部分, 两个地点之间并未建造大坝或存在其他阻隔, 因此两者之间存在较广泛的基因交流, 应作为一个整体进行资源保护。
3.3 种群历史动态
本研究中2个地理群体的整体单倍型多样性和核苷酸多样性分别为0.590和0.00132, 说明整个岩原鲤地理群体具有高的单倍型多样性(Hd>0.5)和低的核苷酸多样性 (Pi<0.5%), 根据Grant等[37]结果, 我们猜测岩原鲤种群曾受到环境急剧变化的影响发生瓶颈效应, 使得一个原本有效种群数量较小的种群在经历快速扩张后, 种群能够快速增长并获得突变积累, 但核苷酸变异所需要的时间往往大于单倍型突变积累所需要的时间, 因此形成了高Hd和低Pi模式。
Tajima’sD与Fu’sFs中性检验是较常用的两种检验显著偏离中性突变的方法。负的Tajima’sD值和差异显著性被认为群体在历史上有扩张的迹象,Fu’sFs中性检验是建立在溯祖理论基础上的中性检验, 对于经历过扩张的种群, Fu’sFs也为显著的负值。万州群体表现为与中性进化偏离的显著性差异, 说明万州历史上曾发生过种群扩张。而赤水群体符合中性假设, 说明历史上赤水岩原鲤群体相对稳定, 未受到外部环境的强烈干扰。
3.4 岩原鲤种群的保护
本研究基于Cytb基因序列研究岩原鲤不同地理群体的遗传多样性, 探讨了2个地理群体间近期的遗传多样性变化趋势, 旨在揭示长江上游岩原鲤资源现状, 为其今后的保护与开发提供基础数据,为长江上游鱼类的生物多样性保护提供详实可靠的科学资料。2个岩原鲤群体处于遗传多样性水平不高、遗传分化尚未形成的阶段, 其中赤水市赤水河岩原鲤群体单倍型多样性和核苷酸多样性不高,低于万州群体。据调查, 在赤水河和长江干流中,岩原鲤渔获量逐渐减少, 处于易危状态。
岩原鲤作为长江上游特有鱼类和重要经济鱼类, 近年来种群资源急剧下降。因此, 从现在开始应该进一步加强长江上游岩原鲤种质资源的调查收集和遗传背景的研究, 及时了解岩原鲤种群的遗传多样性和分化程度。同时, 还需要规范长江上游渔业管理体系, 禁止过度捕捞, 特别是对一些资源现状比较严峻的特有鱼类进行保护。最后, 对优质种质资源进行人工繁殖和增殖放流, 以促进基因交流保证较高的遗传多样性和种群活力。等到岩原鲤的遗传多样性水平恢复到正常水平、种质资源复壮后, 我们可以对其合理利用, 适当地投放到市场发挥其作为经济鱼类的重要价值。当前赤水和万州2个地理群体既要作为一个整体单元去保护,同时也要考虑各地的生境特征和水文变化来制定合理的保护措施。
致谢:
衷心感谢中国科学院水生生物研究所鱼类生态学与资源保护学科组辛苦采样的工作人员, 感谢俞丹博士对文章提出宝贵的修改意见!
