基于高光谱成像的大米中蛋白质含量的可视化研究
2020-03-31王朝辉赵层赵倩王艳辉赖翰卿王晓东王靖会
王朝辉,赵层,赵倩,王艳辉,赖翰卿,王晓东,王靖会
(1.吉林农业大学食品科学与工程学院,吉林长春130118;2.广东地球土壤研究院,广东广州510385;3.吉林省长春市净月开发区福祉街道办事处,吉林长春130122;4.吉林农业大学信息技术学院,吉林长春130118)
中国稻米的产量占世界稻米产量的30%以上[1],而吉林省“梅河大米”是中国粳米地理标志产品,其产地位于世界黄金粮食生产带(北纬45°)。在实际生活中梅河大米的种类繁多,通常采用凯氏定氮法和分光光度法等化学类方法测定不同品种中大米的蛋白质含量,但是这些传统的化学方法不但对样品本身具有破坏性,而且步骤繁琐,检测周期过长。红外光谱技术作为一种快速无损的检测手段,已经广泛的应用于大米主要成分(蛋白质[2]、脂肪[3]、淀粉[4]、水分[5])的检测,但是其只能根据光谱信息得到组分的含量,无法实现更加直观的表达即含量的可视化。
高光谱是包括图像信息和光谱信息[6]的三维立方体数据,得到的高光谱图像既包含了大米的内部信息(内部物理结构、化学成分的信息)也包含了大米的外部信息(粒型、缺陷等),能弥补近红外不能快速识别某一物质的空间分布情况[7-8]即图像的缺失。目前,高光谱成像技术在农产品方面的主要应用有瘀伤识别分类[9]、籽粒的识别[10]、品种的检测[11]、含水量预测[12]等,而高光谱成像技术在大米中的主要应用有垩白检测[13]、水分含量检测[14]、掺假大米的检测[15]等。虽然高光谱检测技术在组分的检测方面已有诸多应用,但关于高光谱成像技术应用于大米中蛋白质含量分布可视化的研究还未见相关报道。
本文选取了吉林省梅河市4 个产区的3 个品种(稻花香、秋田小町、吉粳60)的大米为研究对象,利用高光谱成像技术对采集的大米进行检测,求取大米感兴趣区域的平均光谱,为了降低光谱的信噪比和得到相对稳健的模型,对光谱进行了卷积平滑(savitzkygolay,S-G)、均值中心化(mean centering)、多元散射校正 (multiplicative scatter correction,MSC)3 种算法的预处理,建立了大米蛋白质含量的偏最小二乘回归(partial least-squares regression,PLSR)、主成分回归(principal component regression,PCR)、误差反向传播神经网络(back propagation neural networks,BP-NN)3 种预测模型。采用SPA 选取特征波长,建立特征波长模型,并将大米高光谱图像转变为蛋白质含量分布图,实现不同品种产地大米蛋白质含量的可视化。
1 材料与方法
1.1 材料与仪器
以吉林省梅河市种植的稻米品种(稻花香、秋田小町、吉粳60)为研究对象,通过空间分层随机布点的原则采集了包括稻花香、秋田小町、吉粳60 3 个品种在内的120 个稻米样品。试验前先将采集的稻米进行脱壳、去糙,之后剔除霉变、未成熟和粒型不完整的籽粒,选择大小均匀、饱满的籽粒进行高光谱扫描和进行大米蛋白质含量的化学测定。采集的120 个大米样品中86 个用于校正集,34 个用于检验集。
ImSpector V10E-QE 型高光谱图像系统:芬兰Spectral Imaging 有限责任公司;9589(EKE-ER)全光谱卤素灯:荷兰Philips 公司;PSA200-11-XZolix 电控位移台:北京Zolix 公司;C8484-05G 相机:日本Hamam atsu Photonics;GZ02DS20 可升降样品台:北京广正仪器有限公司;JLGJ4.5 型检验砻谷机、JNMJ3 型检验碾米机:台州市粮仪厂;JXFM110 锤式旋风磨:上海嘉定粮油仪器有限公司;K1100 全自动凯氏定氮仪、SH220 石墨消解仪:济南海能仪器股份有限公司;PHS-3C 型酸度计:上海仪电科学仪器股份有限公司。
1.2 高光谱图像的采集与校正
测量的光谱范围是408.360 0 nm~1 007.220 0nm,包含477 个波段,系统采集大米样品前通过对比多次预实验结果来调整曝光时间、物距等各项参数。采集图像时物距为13.5 cm,曝光时间为15 ms,位移台移动速度为1.62 mm/s。设定好仪器参数后将放有大米样品的黑色底板平铺在载物台上(黑色底板的反射率接近于0,大米样本按照网格单粒随机的方式摆放至载物台黑板上),进行大米高光谱数据的采集。高光谱成像系统的结构示意图如图1 所示。

图1 高光谱成像系统示意图Fig.1 Schematic diagram of hyper-spectral imaging system
在高光谱图像采集过程中由于会受到外界(自然光光源分布不均)和系统(暗电流噪声)等因素的干扰[16],导致得到的高光谱图像包含大量的无效信息,为了减少由于上述因素导致的噪声增强现象,因此在采集大米高光谱图像之前,必须先进行原始大米图像的黑白校正。校正公式如下
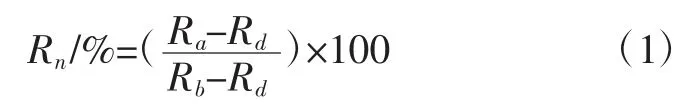
式中:Ra为原始大米的图像;Rd为黑板图像;Rb为白板图像;Rn校正后大米图像。
1.3 蛋白质含量的测定
将采集完光谱数据的大米样品进行磨粉过筛,采用GB5009.5-2016《食品安全国家标准食品中蛋白质的测定》测量大米中的蛋白质含量,之后把称量好的大米样品和催化剂放入准备好的消化管中(大米样品重量 0.5g 左右,CuSO4和 K2SO4的重量分别是 0.2 g 和3 g),准备完成后在消化管中倒入10 mL 的浓硫酸进行消化,并做空白管试验,当消化管内溶液的颜色变为绿色透明时证明其消化完全,则可通过全自动凯氏定氮仪对其进行蛋白质含量的测定,每个样品测量3 次取其平均值作为该大米样品蛋白质含量的化学值。
1.4 数据处理
1.4.1 感兴趣区域与平均光谱的提取
在ENVI5.0 软件中对获得的高光谱图像进行感兴趣区域的选取,选取3×15 网格内所有单粒米样中间部位11×11 像素的正方形区域作为1 个感兴趣区域(region of interest,ROI),ROI 全部像素点的平均光谱作为建立模型的光谱数据。
1.4.2 光谱预处理
为了获得相对稳定和准确的模型,从原始光谱中摒弃噪音等过多无用信息的干扰,从而放大差异,提高分辨率。采用卷积平滑(savitzky-golay,S-G)、均值中心化(mean centering)、多元散射校正(multiplicative scatter correction,MSC)3 种光谱预处理方法。通过3种预处理算法的对比,以期找到适合本次试验的光谱预处理方法。
1.4.3 模型的建立
采用偏最小二乘回归(partial least-squares regression,PLSR)、主成分回归(principal component regression,PCR)、误差反向传播神经网络(back propagation neural networks,BP-NN)3 种常用的算法建立蛋白质的预测模型,采用不同建模算法,最终呈现的建模效果也各不相同。本文以大米为试验对象,以校正集和检验集样品的相关系数(Rc2、Rp2)和均方根误差(RMSEC、RMSEP)为评价模型性能的指标,通过对比最终得到稳健性好的大米蛋白质预测模型。
1.4.4 偏最小二乘回归模型
偏最小二乘回归 (partial least square regression,PLSR)是多因变量对多自变量的回归模型,是常用的一种光谱建模分析方法[17]。PLSR 通过对多自变量和多因变量进行压缩分解,分解时得到两者最强对应关系的主成分,加强模型的预测能力。
1.4.5 主成分回归模型
主成分回归(principal component regression,PCR)是一种多元线性回归分析方法,利用主成分分析将样品光谱矩阵进行分解,把得到的变量组合成相互无关的新变量[18],用较少的变量代表原有的变量,能有效解决预测变量高度相关和共线的问题。
1.4.6 误差反向传播神经网络模型
误差反向传播神经网络(back propagation neural networks,BP-NN)是一种按误差逆传播算法训练的多层前馈网络[19],是目前应用最广泛的神经网络模型之一。本文选用有导师学习算法来调整神经元间的联接权,使得网络输出更符合实际。
1.4.7 连续投影算法(successive projection algorithm,SPA)特征波长的选取
连续投影算法(successive projection algorithm,SPA)是一种使矢量空间共线性最小化的前向变量选择算法[20],采用SPA 对原始高光谱数据进行特征波长的选取,解决了高光谱数据自身维度高,变量多等问题,由于后期模型的建立使用消除了大量冗余信息的高光谱数据,因此可以根据挑选的特征波长(保留光谱信息中最优的变量组)建立简化高效的模型。
1.4.8 大米中蛋白质含量的可视化
通过对比得到最适合本研究的大米蛋白质含量的预测模型,将像素点的光谱数据带入预测模型,可得到各像素点位置的蛋白质含量。通过各像素点位置蛋白质含量的高低,使用MATLAB 对灰度图像进行各像素点颜色的设定。光谱预处理、预测模型的建立和大米蛋白质可视化的实现均在MATLAB2016a 软件中完成。
2 结果与分析
2.1 大米的原始光谱曲线
图2 是选取ROI 全部像素点的平均值得到的反射率光谱(408.360 0 nm~1 007.220 0 nm),120 个大米全波长光谱的趋势大致相同,说明大米在全波长的情况下具有相同的反射特性,但在反射强度上存在差异。

图2 120 个大米样本光谱图Fig.2 Spectral map of 120 rice samples
2.2 蛋白质含量统计分析
对凯氏定氮法测量的大米蛋白质的化学值进行统计分析,得到校正集、检验集和全部样品的最小值、最大值、平均值和标准偏差,结果如表1 所示。

表1 校正集和检验集样品蛋白质含量Table 1 Calibration set and test set sample protein content
从表1 中可以看出校正集蛋白质含量范围大于检验集的蛋白质含量范围,说明校正集和检验集的划分合理。
对120 个大米样品的蛋白质含量进行统计分析得到如图3 所示的柱形图。

图3 不同品种和产地的蛋白含量柱状图Fig.3 Bar graph of protein content of different varieties and regions
如图所示3 个品种的大米在吉乐乡产区的蛋白质含量明显高于其他产区,不同品种大米的蛋白质含量在产区间的分布规律也不相同,稻花香从曙光到黑山头蛋白质含量表现为依次递增,而秋田小町和吉粳60从曙光、湾龙再到黑山头蛋白质含量表现为先增加后减少。对品种和产地进行双因素方差分析,得到关于品种因素、产地因素和品种产地的交互对应的P 值,因为所得三者的P 值均远远小于0.01,因此品种和产地对大米蛋白质含量均有显著影响。
2.3 光谱预处理
通过对比不同预处理算法建立的PLSR 模型的相关系数和均方根误差,得到最优的光谱预处理算法。表2 是光谱预处理的结果,通过对比可以发现MC 预处理后的光谱所建立模型最优,其校正集和检验集的相关系数为0.927 5 和0.907 9,SG 和MSC 预处理后所得模型检验集相关系数下降为0.807 0 和0.897 1。

表2 基于不同预处理方法的PLSR 模型结果Table 2 Results of PLSR models based on different preprocessing methods
2.4 全波段模型的建立
本文以477 个波段的光谱数据为自变量,以测量的蛋白质含量为因变量,建立PLSR、PCR、BP 神经网络预测模型,基于全波段不同建模算法的结果见表3。
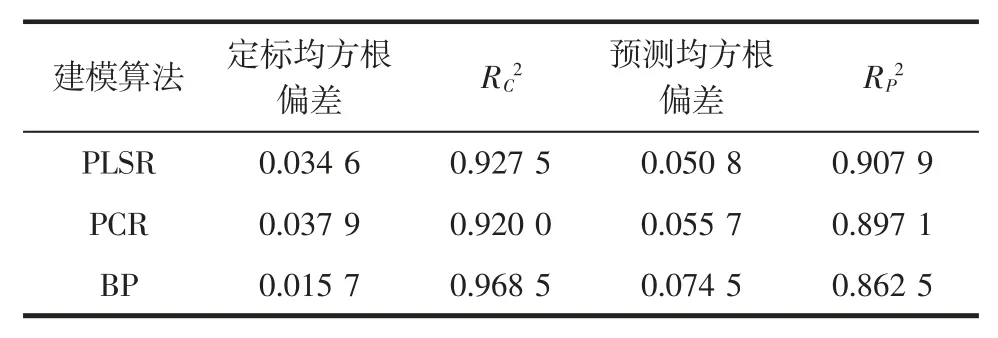
表3 基于全波段不同建模算法的结果Table 3 Results based on different modeling algorithms for the full band
如表3 所示,建立PLSR 模型时确定的最优潜在变量个数是13,其校正集的相关系数和均方根误差RMSEC 分别为0.927 5 和0.034 6,其检验集的相关系数和均方根误差RMSEP 分别为0.907 9 和0.050 8;建立PCR 模型时确定的最优主成分个数是17,其校正集的相关系数和均方根误差RMSEC 分别为0.920 0和0.037 9,其检验集的相关系数和均方根误差RMSEP 分别为0.897 1 和0.055 7;建立BP 神经网络模型时,神经网络隐含层神经元数20,其校正集的相关系数和均方根误差RMSEC 分别为0.968 5 和0.015 7,其检验集的相关系数和均方根误差RMSEP 分别为0.862 5 和0.074 5。通过对3 种建模算法的结果对比得到最优的建模算法为PLSR。
蛋白质PLSR 模型的实测值和预测值的散点图见图4。

图4 蛋白质PLSR 模型的实测值和预测值的散点图Fig.4 Scatter plot of measured and predicted values of protein PLSR model
2.5 SPA特征波长的选取
特征波长的计算过程在MATLAB 中实现,设定最小和最大的选定波长数为5 到50,最终得到的结果如图5 所示。

图5 SPA 筛选特征波长Fig.5 SPA screening characteristic wavelength
通过SPA 筛选出27 个特征波长,分别为:416.690 0、429.840 0、426.250 0、459.910 0、469.590 0、519.590 0、861.890 0、881.170 0、892.750 0、910.760 0、927.480 0、939.060 0、959.650 0、966.080 0、971.230 0、975.080 0、978.940 0、982.800 0、990.510 0、993.090 0、995.660 0、1 000.800 0、1 003.370 0、948.070 0、747.900 0、487.810 0、1 005.940 0 nm。
PLSR 通过计算分别得到合适的X 和Y 的潜变量P 和Q,通过迭代法调整得分矩阵T 和U,同时保持残差矩阵E 和F 的值接近0,求得T 和U 的内在联系,间接得到X 和Y 的映射关系[21]。

基于SPA 筛选的27 个特征波长的反射值作为自变量X,全自动凯氏定氮仪测定的大米蛋白质含量为因变量Y 建立PLSR 模型,得到27 个特征波长的反射值和蛋白质含量的内在关系,并用此关系在已知波谱反射值的情况下对蛋白质含量值进行预测。图6 所示SPA-PLSR 模型的校正集和检验集的相关系数分别为0.923 2 和 0.903 9。
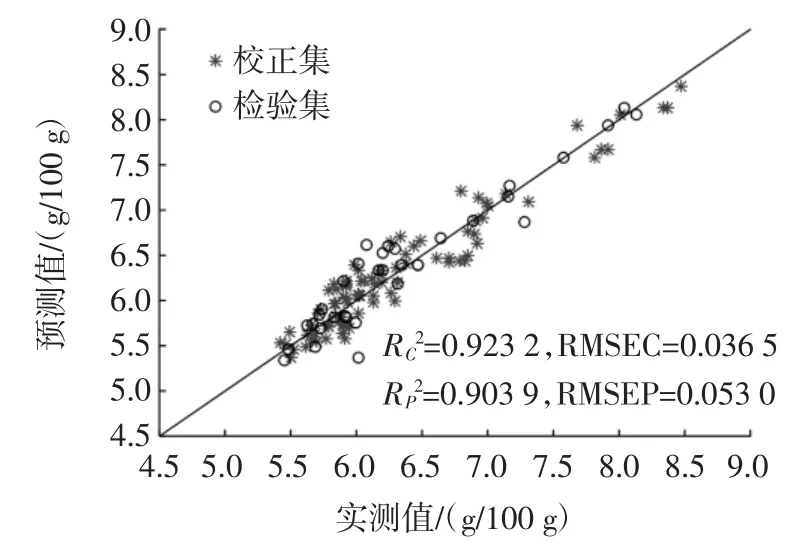
图6 蛋白质SPA-PLSR 模型的实测值和预测值的散点图Fig.6 Scatter plot of measured and predicted values of the protein SPA-PLSR model
2.6 大米中蛋白质含量分布的可视化
选取稻花香、秋田小町和吉粳60 的大米高光谱图像进行成像分析,通过ENVI5.0 从大米样本的高光谱图像中提取27 个特征波长下的高光谱图像,然后提取特征图像中每个像素点的光谱反射值,将提取的所有像素点的光谱反射值代入2.5 已建立的PLSR 模型,最终预测到每个像素点的蛋白质含量。将高光谱灰度图像转入MATLAB2016a 中,高光谱灰度图像中所有像素点的灰度值用得到的对应像素点的蛋白质含量值代替,生成如图7 所示的大米蛋白质含量分布图。


图7 不同品种产地蛋白质含量分布可视化图Fig.7 Visualization of protein content distribution in different varieties of production areas
如图7 所示3 个品种在吉乐乡产区的蛋白质含量值最高,在曙光、湾龙和黑山头产区蛋白质含量无明显差异,因为吉乐乡采集的大米地处山区与其他3 个产区相比距离最远,地貌差异最大,因此形成的大米蛋白质含量差异明显,表明产地对大米蛋白质的含量是有影响的,与图3 所示的规律基本相同。
从全部大米蛋白质含量分布图可以看出米粒内部大米蛋白质的分布是不均匀的,但是总分布趋势基本相同,胚内多于胚乳,大米蛋白质含量的高低主要表现为胚乳内蛋白质含量分布情况,这与Little and Dawson(1960)公布的大米蛋白质染色图得到的结论基本相同。
3 结论
采用高光谱成像技术对大米中蛋白质含量分布的可视化进行了可行性研究,通过MC 光谱预处理方法和SPA 特征波段的选取,得到了简化高效的PLSR蛋白质含量预测模型,基于该定量模型的基础上对不同品种和不同产地大米中蛋白质含量的分布进行可视化研究。由于同一品种间大米形状相近,难以通过普通RGB 图像区分,通过对蛋白质含量分布成像可以为大米产地的识别提供思路,而对比不同品种间大米的蛋白质含量分布图,可以为后期选育大米品种提供依据。
