玉米HD-ZIP I亚家族基因鉴定及表达分析
2020-03-23梁思维姜昊梁翟立红万小荣李小琴
梁思维 姜昊梁 翟立红 万小荣 李小琴 蒋 锋,* 孙 伟,*
玉米HD-ZIP I亚家族基因鉴定及表达分析
梁思维1姜昊梁1翟立红2万小荣1李小琴1蒋 锋1,*孙 伟1,*
1仲恺农业工程学院农业与生物学院, 广东广州 510225;2湖北文理学院医学院, 湖北襄阳 441053
转录因子是植物响应逆境胁迫的重要调节因子, 在其整个生长发育过程中发挥着重要的作用。HD-ZIP家族蛋白是植物中特有的一大类转录因子, 包含4个亚家族(HD-ZIP I~IV), 其中HD-ZIP I亚家族成员主要参与干旱、渗透压等极端环境和ABA及乙烯等激素处理的响应过程。本文采用隐马可夫模型(HMM)在玉米参考基因组中鉴定到17个HD-ZIP I亚家族成员, 这些基因不均匀分布于玉米6条染色体上, 与水稻的亲缘关系要近于拟南芥。玉米HD- ZIP I 亚家族基因在玉米7种组织中表现出多种表达模式, 具有明显的组织表达特异性。另外, HD-ZIP I亚家族基因对高盐、淹水及冷害等不同的逆境胁迫处理呈现出不同的响应模式及响应程度差异。5种不同激素处理后, 玉米HD-ZIP I亚家族基因也表现出复杂的响应模式。这些结果为进一步解析玉米HD-ZIP I亚家族基因的生物学功能和作用机理提供了一定的参考价值。
HD-ZIP I 亚家族; 逆境胁迫; 表达分析; 玉米
植物生长发育是一个复杂且多样的生物学过程,这一发育过程常受到多种生物及非生物等胁迫因素的影响。在应对这些胁迫中, 植物启动了细胞、分子以及生理生化水平等多种调节机制[1-2]。植物最早感知和传递的非生物逆境胁迫信号是通过信号分子途径, 然后影响一系列相关基因的表达水平[3]。其中, 转录因子在植物应激反应中发挥着重要的调节作用。目前已有研究表明, 胁迫响应的转录因子的诱导表达, 如、、、和[4-7], 会提高植株对逆境胁迫的适应能力, 越来越多的证据表明, HD-ZIP家族蛋白参与植物对逆境胁迫的响应[8]。
HD-ZIP家族蛋白是植物中特有的一大类转录因子, 在拟南芥、水稻、小麦、大麦、马铃薯、向日葵、番茄和苜蓿中均有报道[9-17]。HD-ZIP蛋白包括两个重要的功能域, 即DNA结合的HD功能域(domain)和与之紧密相连、与蛋白二聚化作用相关的ZIP功能域[18]。拟南芥基因组中共存在48个HD-ZIP家族蛋白[9,19], 并根据DNA结合特异性、理化功能特性和其他的功能域将它们分成4个亚家族(HD-ZIP I ~ HD-ZIP IV)[18-19], 与植物响应相关的HD-ZIP蛋白大多属于第I亚类, 这一亚类家族成员的表达水平主要受干旱、渗透压等极端环境和ABA及乙烯等激素的影响[20]。研究表明, 在干旱和冷害胁迫条件下, HD-ZIP I家族基因AtHB6、AtHB7 、AtHB12、SlHB2和GhHB1通过气孔关闭、增加水分吸收和一系列应激相关基因的调控等途径来增强植物的耐受性, 减轻胁迫导致的不良反应[21-23], 而苜蓿MtHB1通过调节根的表面积来适应高盐胁迫[11]。水稻OsHOX22会影响ABA的生物合成, 并能通过ABA介导的信号传导途径来调控干旱和高盐胁迫响应过程[24]。向日葵基因受到干旱胁迫诱导表达, 进而促进水杨酸(JA)和乙烯(Eth)的产生, 从而提高植物对干旱胁迫的适应[14,25]。冷害胁迫下, 向日葵HaHB11和拟南芥AtHB13能够诱导产生稳定细胞膜结构和抑制冰生成的蛋白质, 从而增强拟南芥对低温胁迫的抵抗能力[26-27]。此外, HD-ZIP I 亚家族基因作为转录因子, 其作用的发挥依赖于上下游基因的调控。研究表明,在黑暗条件下上调表达, 在盐胁迫和低温冷害胁迫下表达量下降[19]。研究表明, 在短日照条件下, PIF1能够诱导的表达, 表明AtHB1在PIF1的下游发挥作用, 促进下胚轴的伸长[28]。AtHB1与基因启动子区结合, 导致铁供给条件下基因表达降低[29]。AtHB1通过与()启动子结合参与拟南芥抵抗高温、高盐和干旱胁迫响应过程[30]。另外, 水稻OsHOX4能够与的启动子区结合, 参与水稻赤霉素(GA)信号传导和干旱胁迫反应的负调节过程[31-32]。总之, HD-ZIP I亚家族蛋白在植物应对非生物逆境胁迫过程中发挥着重要的作用。
目前, 尽管模式植物中的HD-ZIP I家族部分成员已经有所报道, 参与非生物逆境胁迫响应和激素调节过程, 玉米和基因在拟南芥和水稻中过量表达会降低这些植物对干旱和盐胁迫的敏感性, 也增强玉米对干旱及盐胁迫的耐受性[33-34], 但是关于玉米HD-ZIP I亚家族蛋白的研究相对较少。本文将进一步展开对玉米HD-ZIP I亚家族成员的系统研究, 阐明该亚家族基因在逆境胁迫和激素处理下的表达变化规律, 为进一步解析玉米HD-ZIP I亚家族基因的生物学功能和作用机制提供一定的参考。
1 材料与方法
1.1 材料种植与处理
将玉米测序自交系B73种子在室温下水浸4 h后播于32盆沙钵中, 于温室培养至二叶一心期, 每盆留下长势一致的幼苗5~6株。玉米幼苗经100 μmol L–1脱落酸(ABA)、赤霉素(GA3)、萘乙酸(NAA)、激动素(KT)和乙烯(Eth)等激素处理以及渍害胁迫、高盐胁迫和冷害胁迫, 共8种处理, 每种处理2盆, 处理4 h, 2盆做空白对照。激素处理是用叶面喷施; 渍害胁迫是将水淹没至玉米幼苗的第1片叶片处; 高盐处理是用0.2 mol L–1的NaCl溶液; 冷害处理是在低温光照培养箱(10℃)中进行; 冷害后恢复处理是在冷害胁迫4 h后再将玉米幼苗恢复至室温环境培养。
1.2 玉米HD-ZIP I亚类基因鉴定与分析
根据TAIR (The Arabidopsis Information Resource, http://www.arabidopsis.org/)网站提供的拟南芥HD- ZIP I亚类基因的氨基酸序列, 采用隐马可夫模型(HMM)在MaizeGDB (http://www.maizegdb.org/)中检索。利用该家族亚类的结构特点, 通过BLASTp对蛋白功能结构域HD和ZIP进行检索, 筛选HD-ZIP I亚类蛋白。进一步根据MaizeGDB上查找的基因信息(B73 RefGen_V3), 将基因标注于玉米10条染色体上,并利用在线分析工具GSDS[35](http://gsds.cbi.pku. edu.cn/)和SMART[36](http://smart.emblheidelberg.de/)绘制基因蛋白结构图, 再根据分析得到的HD和ZIP结构域位置信息, 完善基因蛋白结构图, 对个别显示错误的结构图进行手工修正。
1.3 植物中HD-ZIP I亚类基因同源性分析
系统进化树分析所用HD-ZIP I 亚类蛋白质序列, 玉米的来自MaizeGDB (http://maizegdb.org/)、水稻的来自TIGR (http://www.tigr.org/)和拟南芥的来自TAIR (http://www.arabidopsis.org/)。采用Clustal X 2.0软件[37]进行HD-ZIP I亚类蛋白的多重序列比对和Neighbor- Joining进化树的构建, 并用MEGA software version 5.1[38]展示其结果。Bootstrap分析采用1000次重复。
1.4 玉米HD-ZIP I 表达分析
将胁迫和激素处理后的玉米幼苗分别在液氮中研磨至粉末状, 参照试剂盒说明书, 加入适量的Tripure Isolation Reagent (Promega, USA)进行总RNA的提取。用RNase-free DNase预处理所有样品提取的总RNA, 分别选取1 μg用M-MLV RTase cDNA Synthesis Kit (TaKaRa, Japan)进行反转录。在Bio- Rad CFX96 Real-time System进行Real-time PCR扩增。使用iTaq Universal SYBR Green Supermix (Bio- Rad, USA)试剂盒并按其提供的方法, 扩增体系为25 μL。反应程序为94℃ 3 min, 94℃ 10 s, 58℃ 30 s, 共40个循环。每个样品以肌动蛋白(,)为内参, 重复3次, 结果用2–DDCt进行相对定量分析[39]。选用PRIMER 5设计所有引物, 且横跨一个内含子(附表1)。
文中所用该亚类基因表达数据来自玉米表达数据库qTeller (http://qteller.com/qteller3/), 选取幼苗的地下部分(Seedling_roots)、地上部分(Seedling_shoots)、完全展开叶片(Leaves)、幼嫩的雌穗(Tassels)、幼嫩的雄穗(Ears)和授粉后5 d/10 d的籽粒(Seeds_5DAP、Seeds_10DAP)等7个组织的表达量数据, 运用R软件(R version 3.1.2) gplots包中heatmap.2制作heatmap图。
2 结果与分析
2.1 玉米HD-ZIP I亚家族基因的鉴定
根据HD-ZIP家族蛋白的HD和ZIP两个结构域的特点, 基于HMM模型在玉米基因组数据库中检索, 共筛选到55个符合该家族基因结构特点的多肽序列。其中, 17个多肽序列仅含有HD和ZIP两个结构域, 不含其他已知序列结构(或功能域), 属于HD-ZIP I亚家族(表1), 参考MaizeGDB Gene Data (https://www.maizegdb.org/gene_center/gene)命名所有基因。将17个基因序列与玉米基因组比对, 发现这些基因分布于玉米6条染色体上(图1), 即1号、2号、4号、5号、7号和9号染色体。其中, 有6个基因位于1号染色体上, 4个基因位于2号染色体上, 另外在4号、7号和9号染色体上分别存在2个基因, 在5号染色体仅有1个基因。这些基因在DNA序列长度上差异显著,基因最长, 为4811 bp,基因最短, 仅为1307 bp (表1)。有趣的是, 不同基因所含外显子数目基本相同, 除基因有4个外显子外, 所有基因均含有2~3个外显子(表1和图2)。这些基因的编码序列(ORF)长度差异不大, 在720~1134 bp之间; 翻译的氨基酸序列长度相近, 在239~377 aa之间(表1)。由此可见, 造成这些基因DNA序列长度差异的关键原因不是外显子的数目和长度, 而是内含子序列的长度。

表1 玉米HD-ZIP I亚家族基因信息
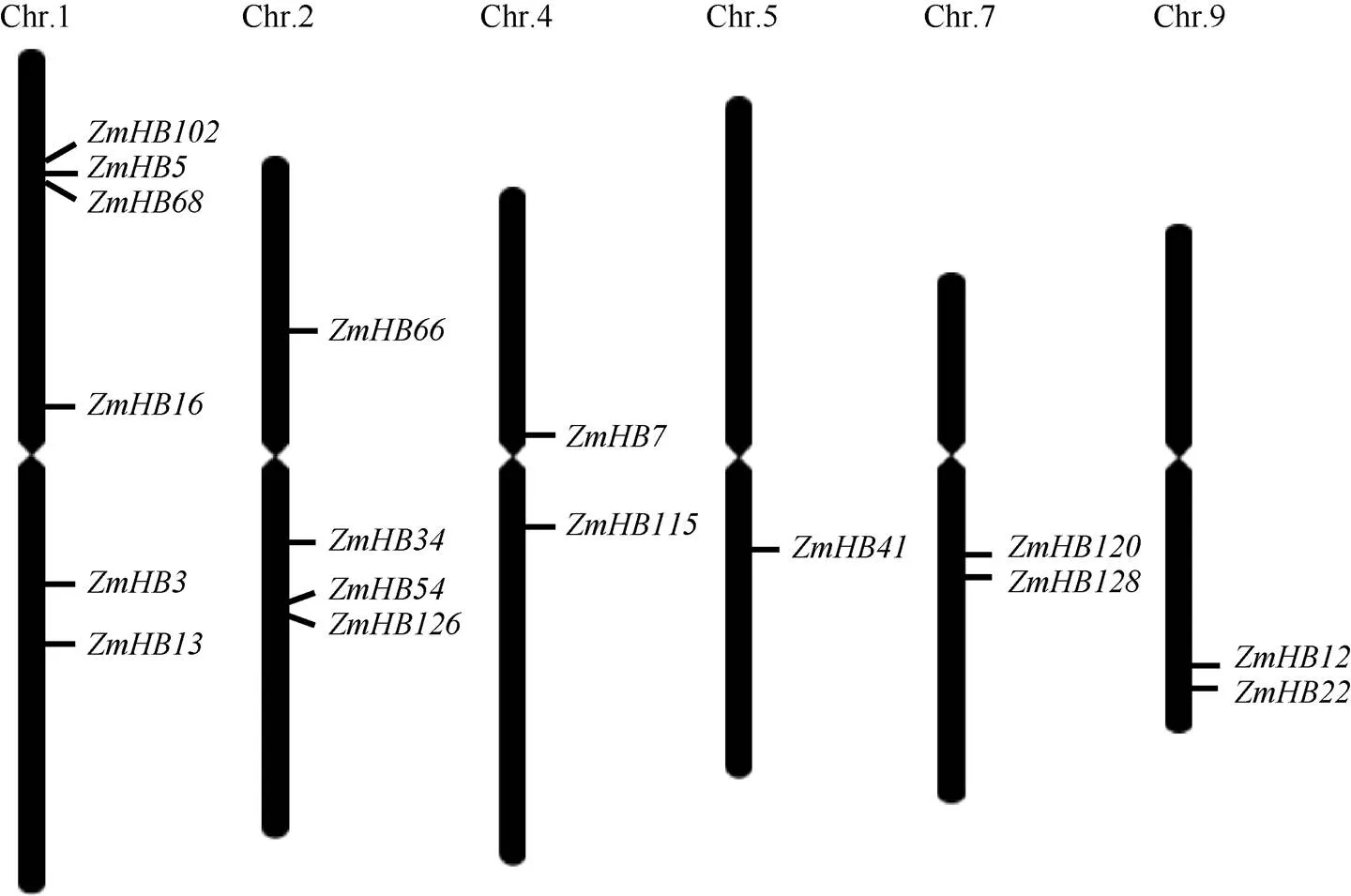
图1 玉米HD-ZIP I亚家族基因在染色体上的分布
2.2 玉米HD-ZIP I亚家族蛋白结构域和系统进化分析
进一步分析玉米HD-ZIP I亚家族基因结构和蛋白功能结构域, 发现17个蛋白中都含有同源异型结构域(HD domain), 属于Homodomian蛋白(图2)。在HD的羧基端, 也同样存在保守的锌指蛋白结构域(ZIP domain)(图2)。可见, 这些基因具有明显的HD-ZIP家族的结构特点。除此之外, 没有分析到其他具有明显结构的功能域, 这也是HD-ZIP I亚家族蛋白与其他3个亚家族蛋白在结构上的区别, 是鉴定HD-ZIP I亚家族基因的依据之一。
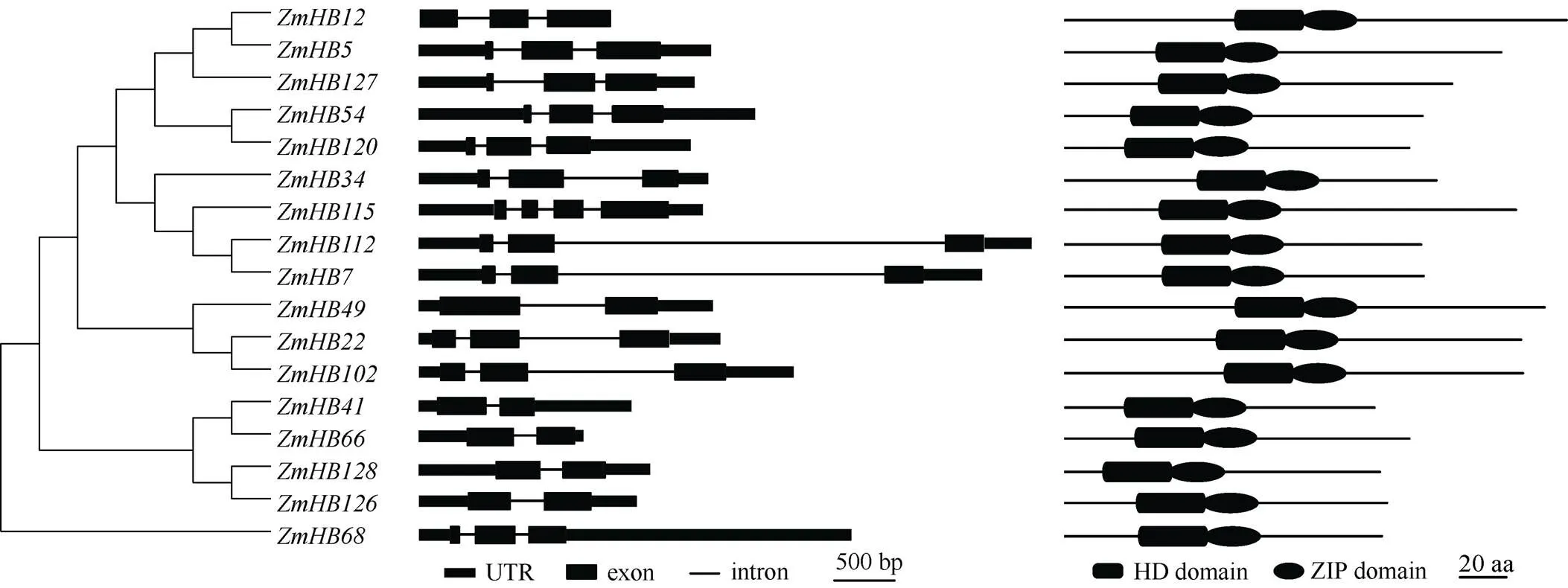
图2 玉米HD-ZIP I亚家族基因及蛋白结构域分析
左图中直线表示内含子, 黑色长方形(宽)表示外显子, 黑色长方形(窄)表示非编码调控区(untranslated region, UTR); 右图中黑色圆角矩形表示HD结构域, 黑色椭圆形表示ZIP结构域。
The line indicates intron, wide black rectangle indicates exon, and narrow black rectangle indicates untranslated region (UTR) in left figure; the black round corner rectangle indicates HD domain and black hexagon indicates ZIP domain in right figure.
根据HD-ZIP I亚家族蛋白的氨基酸序列及其保守性, 玉米、水稻和拟南芥中分别存在17、14和17个HD-ZIP I亚家族蛋白基因。同时, 利用CLC Sequence Viewer软件分析并构建玉米、水稻和拟南芥该亚家族的系统进化树(图3)。虽然该亚家族基因在单子叶(玉米和水稻)和双子叶(拟南芥)植物中的数目差异不大, 但三者在蛋白序列组成和排列上区别较大。从系统进化树可以看出, 这些基因在玉米和水稻中基本上是成组出现, 即1个水稻基因对应1个或2个玉米基因, 或是1个玉米基因对应1个或2个水稻基因, 比如与、与和等。这与拟南芥基因存在明显的区别, 在不同的分支中, 拟南芥的基因基本上是成簇出现, 比如、、和。这些结果说明, 玉米和水稻基因编码的蛋白在序列上的相似程度要高于拟南芥。这些基因在系统进化上的差异也证实了玉米和水稻的亲缘关系要近于拟南芥, 同时也可说明这些基因在单双子叶植物之间存在功能的分化。
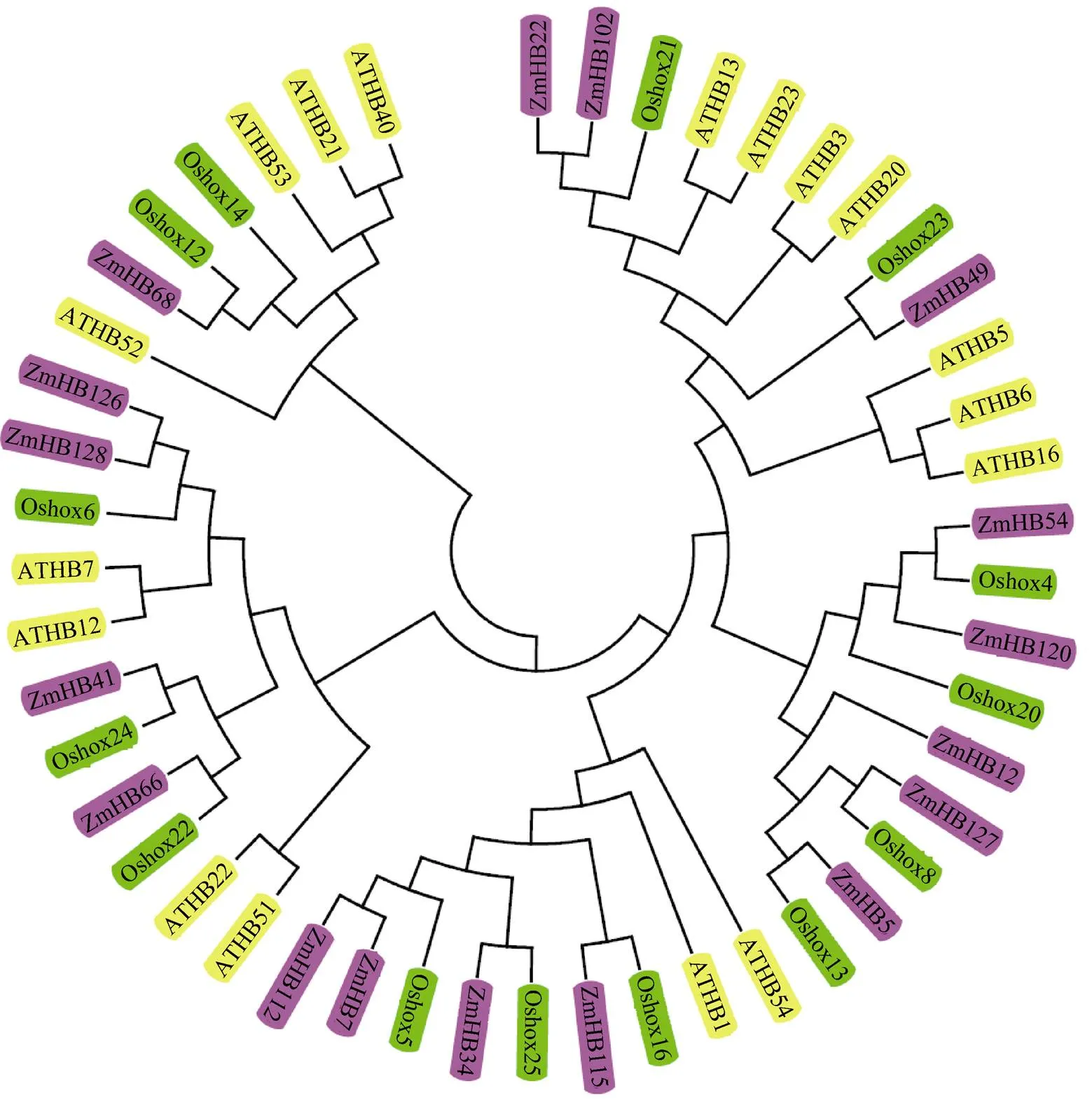
图3 玉米、水稻和拟南芥HD-ZIP I 亚家族蛋白的系统进化分析
所有HD-ZIP I亚家族基因的蛋白序列均来自于各自的基因组数据库: 玉米来自MaizeGDB (http://maizegdb.org/)、水稻来自TIGR (http://www.tigr.org/)和拟南芥来自TAIR (http://www.arabidopsis.org/)。利用ClustalX 2.0软件进行氨基酸序列的多重比对并用MEGA 5.1 软件构建Neighbor-Joining进化树(Bootstrap分析采用1000次重复)。黄色背景表示拟南芥基因, 绿色背景表示水稻基因, 紫色背景表示玉米基因。
The HD-ZIP I subfamily protein sequences of maize, rice, andwere obtained from the MaizeGDB (http://maizegdb.org/), TIGR (http://www.tigr.org/), and TAIR (http://www.arabidopsis.org/) databases, respectively. Multiple alignment of amino acid sequences was carried out by ClustalX 2.0 software and Neighbor-Joining evolutionary tree was constructed by MEGA 5.1 (1000 replications of bootstrap test).genes are highlighted in yellow background, rice genes are highlighted in green background, and maize genes are highlighted in purple background.
2.3 玉米HD-ZIP I基因表达分析
从玉米表达数据库qTeller中下载的HD-ZIP I亚家族基因转录组数据, 探究不同基因在幼苗、幼根、成熟叶、雌雄穗和授粉后5 d/10 d的种子等7个组织中的表达模式差异。其中,在7个组织中的FPKM值均为0, 故不予分析。其余16个基因按照log2(FPKM)值作图(图4)表明, 该亚家族各个基因在不同组织器官中的表达量存在显著差异。总体而言, 各基因在雌穗(Ear)和授粉后的种子(Seed_DAP)中的表达量要明显高于其他各时期的组织, 说明HD-ZIP I亚家族基因在玉米雌穗发育过程和授粉后的籽粒发育早期发挥着重要的生物学作用。在生长发育早期的幼苗(seedling)中, 除、、、外, 各基因的表达量都相对较低, 说明HD-ZIP I亚家族基因中可能只有部分基因参与玉米幼苗的生长发育过程。在生长发育后期, 雌穗中各基因的表达丰度比雄穗和成熟叶片中的高, 说明该亚家族基因对雌穗发育的重要性。进一步观察发现, 雌穗、中高丰度的基因在幼苗中表达水平相对较低(图4), 但是和基因刚好相反, 表达量是幼苗中高于雌穗中, 说明这些基因的表达具有一定的组织特异性。在玉米的各个组织器官中, 均有HD-ZIP I亚家族基因的表达, 说明这些基因可能参与多个组织器官发育的调控, 影响整个植株的生长。
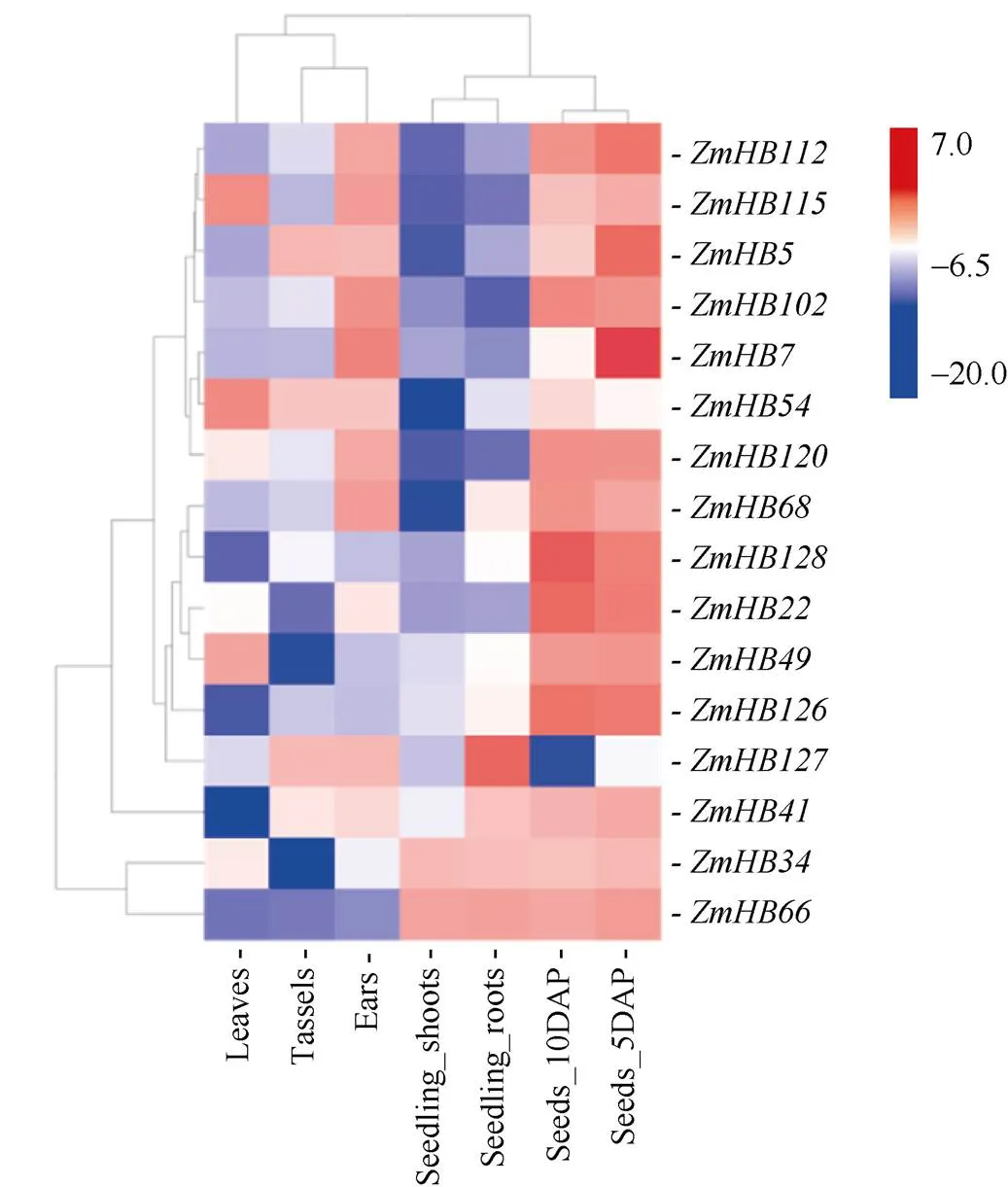
图4 玉米HD-ZIP I亚家族基因表达谱
Leaves: 完全展开叶; Tassels: 幼嫩的雄穗; Ears: 幼嫩的雌穗: Seedling_shoots: 幼苗地上部分; Seedling_roots: 幼苗地下部分; Seeds_5DAP: 授粉后5 d的籽粒; Seeds_10DAP: 授粉后10 d的籽粒。方框内颜色代表HD-ZIP I亚家族基因表达数据的ln的对数值(ln (RPKM))。
Leaves: fully expended leaves; Tassels: immature tassels; Ears: immature ears; Seedling_shoots: the shoots of maize seedlings; Seedling_roots: the roots of maize seedlings; Seeds_5DAP: the seeds at 5 days after pollination; Seeds_10DAP: the seeds at 10 days after pollination. Colors in square represent the logarithm of the HD-Zip I subfamily gene expression level (ln (RPKM)).
2.4 玉米HD-ZIP I响应胁迫和激素应答
对经过高盐、淹水及冷害等逆境胁迫处理前后的B73玉米幼苗进行HD-ZIP I 亚家族基因表达分析。结果显示, HD-ZIP I 亚家族中17个基因对不同的逆境胁迫呈现出不同的响应模式及响应程度差异(图5),、、、、、和七个基因受高盐胁迫诱导表达, 而、和三个基因的表达则被显著抑制。渍害胁迫时, 仅、和三个基因表达量明显升高,、、和四个基因表达受到抑制。
冷害胁迫4 h后, 大部分基因的表达都受到显著抑制, 仅基因诱导表达且达到对照组的表达量的2倍以上。对于冷害胁迫后恢复室温4 h时, 除、和三个基因外, 其他所有基因的表达量均呈现回升的趋势: 大部分基因表达都恢复到正常表达水平, 甚至表现出高水平的表达趋势。与冷害处理时的表达量相比, 冷害恢复后、和三个基因的表达量虽然有所上升, 但也没有达到对照(无处理)水平, 仅为对照组表达量的1/2。另外,、、、、和六个基因在冷害胁迫后基因的相对表达量受到一定程度的抑制, 但是在恢复至室温后这6个基因的表达量迅速升高, 甚至达到对照组的4~10倍。
不同激素处理后, HD-ZIP I亚家族17个基因表现出复杂的响应模式(图6)。17个基因中有9个在5种激素处理后表现出相同的变化趋势。与对照组相比,基因没有显著变化,、和三个基因表达量都明显升高, 而、、、和五个基因均呈现显著下调表达趋势。另外, 有6个基因显著受到一种或多种激素调控, 包括显著上调或显著下调。其中,在ABA处理后表达量上调了50%,在GA3处理后表达量增加了接近3倍,的表达量在除NAA外其他4种激素处理条件下均显著提高。同时,受NAA诱导下调表达,在ABA和NAA处理条件下的表达受到显著抑制,的丰度在ABA、Eth、GA3和NAA处理后都显著降低。除此之外,和两个基因对于不同激素处理后表现出不同的响应特点, 它们的表达均受ABA和KT诱导, 受NAA的抑制。
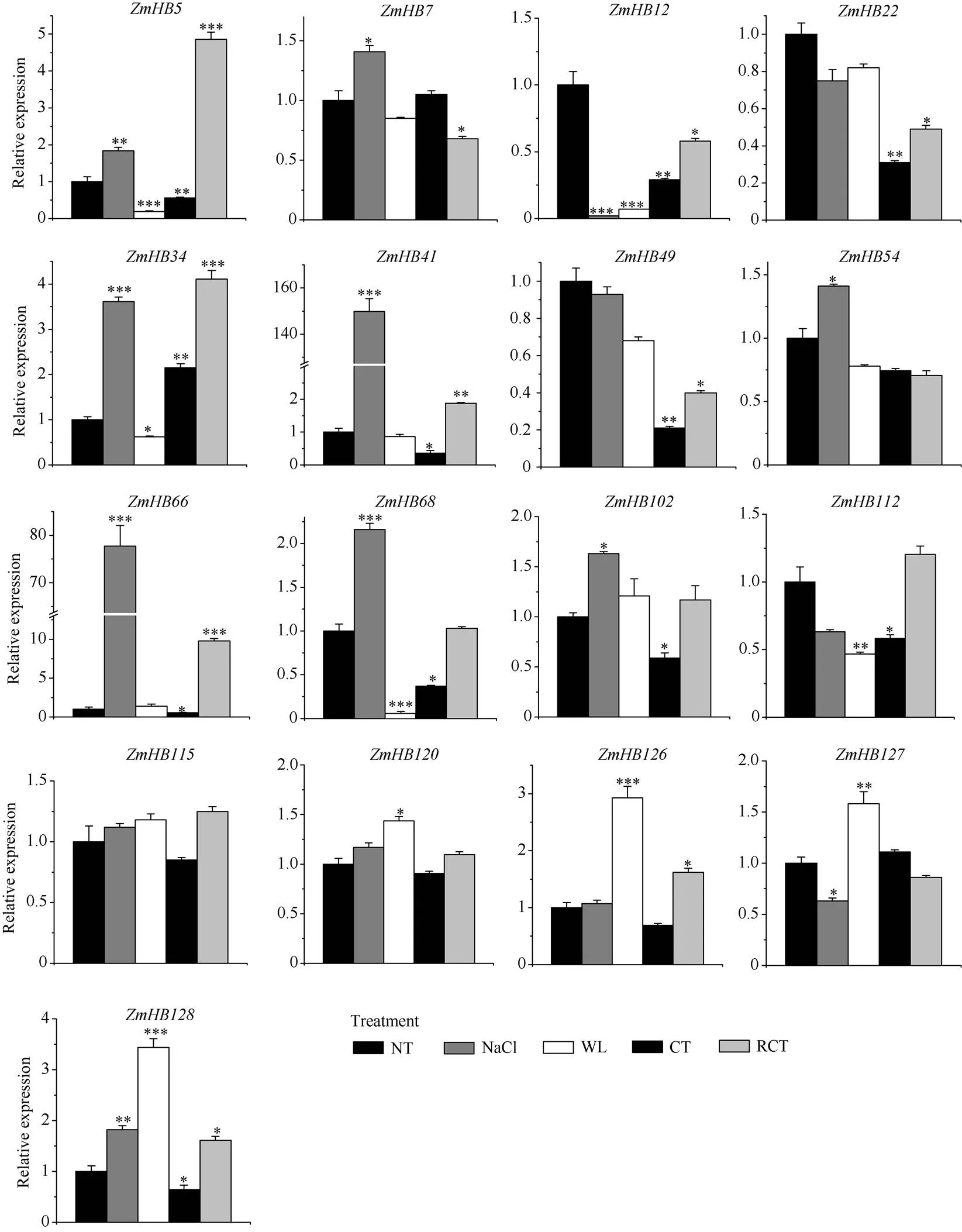
图5 逆境胁迫下玉米HD-ZIP I亚家族基因的表达模式
NT: 对照组; NaCl: 高盐处理; WL: 渍害胁迫; CT: 冷害胁迫; RCT: 冷害胁迫后恢复处理。纵坐标表示基因的相对表达量, 以对照组(NT)为1, 各基因的不同处理分别与对照组进行比较后获得的相对表达量, 差异显著性分析采用的是方差分析的方法, *< 0.05、**< 0.01、***< 0.001。
NT: non-treatment; NaCl: NaCl treatment; WL: waterlogging stress; CT: chilling stress; RCT: recuperative treatment after chilling stress. The ordinate indicates the relative expression of genes, using the expression of the non-treatment group (NT) as 1. The significance of difference was evaluated using analysis of variance. *< 0.05, **< 0.01, and ***< 0.001.
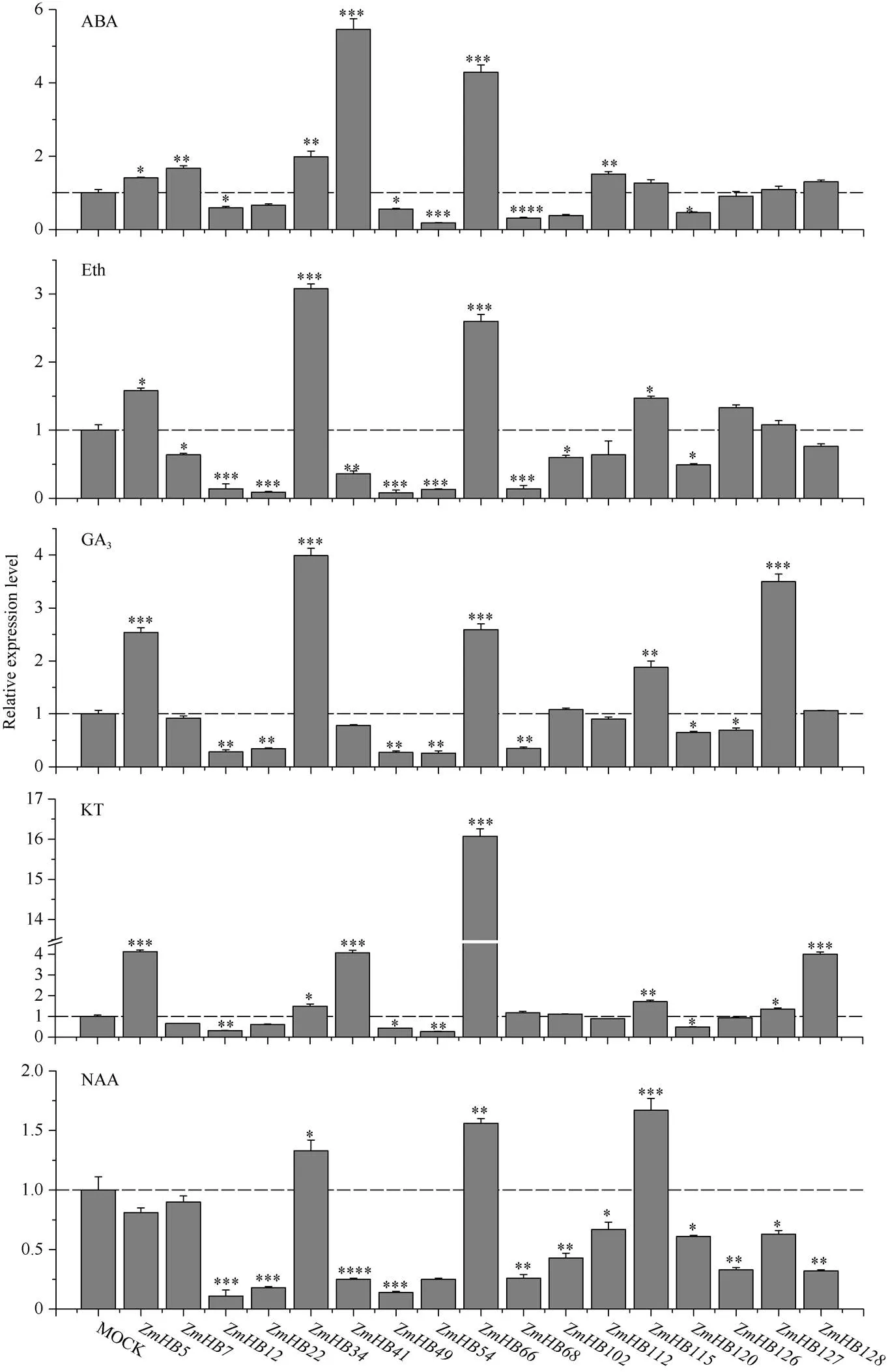
图6 玉米HD-ZIP I 亚家族基因对不同激素处理的响应
MOCK: 对照组; ABA: 脱落酸; Eth: 乙烯; GA3: 赤霉素; KT: 激动素; NAA: 萘乙酸。纵坐标表示基因的相对表达量, 以对照组(MOCK)为1, 差异显著性分析采用的是方差分析的方法, , *< 0.05、**< 0.01、***< 0.001。
MOCK: control; ABA: Abscisic Acid; Eth: Ethylene; GA3: Gibberellin; KT: Kinetin; NAA: 1-naphthylacetic acid. The ordinate represents the relative expression of genes, using the expression of the non-treatment group (MOCK) as 1. The significance of difference was evaluated using analysis of variance. *< 0.05, **< 0.01, and ***< 0.001.
3 讨论
同源异型-亮氨酸拉链(HD-ZIP) 蛋白是一个庞大而又保守的基因家族, 是植物所特有的一类转录因子。在植物中, HD-ZIP蛋白存在于各个组织和器官中, 它们在高等植物的生长、发育、形态建成以及生物和非生物胁迫等逆境应答中起着重要的调控作用[18]。本研究共鉴定到17个玉米HD-ZIP I亚家族基因, 与模式植物拟南芥(17)和水稻(14)相比[10,13], 三者在数目上差异不大。但是, 系统进化分析发现单双子叶植物在进化关系上还是有一定差异的, 即单子叶植物玉米和水稻中很多旁系同源基因基本是一一对应的, 而双子叶植物拟南芥在一定程度上是分开进化的(图3)。因此, 这个结果在一定程度上解释了单双子叶植物在进化进程中的差别。
另外, 无论是旁系同源基因还是直系同源基因的产生, 在一定程度上会造成基因功能的分工或是产生全新功能的基因。前人在水稻和拟南芥的HD- ZIP I亚家族基因的研究中很少谈及它们在植物生长发育中不同组织器官中的表达模式及功能[10,13]。本研究对玉米HD-ZIP I亚家族基因在7个组织器官中的表达数据进行分析发现, 这些基因表现出多种表达模式。整体来看, 大部分基因在雌穗中有较高的表达, 在授粉后的籽粒中呈现逐渐上升的表达趋势, 尤其是授粉后5 d的籽粒中, 比如、和(图4)。成熟叶片中高水平表达的、和三个基因以及幼苗根中高表达的基因都说明这些基因可能在玉米特定组织器官的建成和发育过程中发挥着重要的调控作用。在拟南芥中、和与()一起促进()的表达进而诱导脱落酸的富集, 最终抑制花蕾的发育[40]。水稻中的同源基因和也被证实参与水稻花序的发育过程[41]。研究发现玉米中仅存在1个同源基因, 表达数据显示其仅在雌穗和授粉后的种子中表达丰度较高, 但其是否具有与拟南芥和水稻相似的生物学功能还需要进一步验证。
大量研究表明, HD-ZIP I亚家族基因表达受环境胁迫诱导表达, 主要能够响应干旱、高盐、ABA和冷害等逆境胁迫[8,10,25,42]。对经过高盐、淹水及冷害等逆境胁迫处理前后的B73玉米幼苗进行HD-ZIP I 亚家族基因表达分析显示, HD-ZIP I 亚家族17个基因可以分成4种响应模式, 即基本不响应(、、、); 抑制表达模式(、、、); 诱导表达模式(、、、、、); 两种响应模式都存在(、、)。整体分析发现, 尽管如此, 在同一响应模式下的所有基因对不同的逆境胁迫的响应程度存在一定的差异, 基因表达量有一定程度的上下浮动, 但整体变化趋势相似。
在拟南芥中, 高浓度的NaCl (100 mmol L–1)处理会显著诱导和的表达, 其表达量上调12倍和25倍[42]。水稻、和是拟南芥和的同源基因, 干旱胁迫可以诱导这些基因上调表达, 但它们在水稻中的表达模式不完全相同, 其中突变后可减缓水稻叶片黄化现象[10]。向日葵中的同源基因受高盐、缺水和ABA诱导表达, 而且过表达的拟南芥表现出较强的耐干旱、耐高盐以及延缓衰老的表型[14,25]。玉米中和的同源基因有4个, 分别是、、和。本研究显示, 这4个基因受多种胁迫诱导表达, 其中和在NaCl处理后表达量上调几十倍,和能够响应淹水处理, 表达量增加2~3倍(图5)。在冷害胁迫时这4个基因的表达能够维持基本不变, 在冷害胁迫后恢复至室温时迅速增加。另外, 盐胁迫也诱导和的表达, 其表达量上调2~3倍, 同样拟南芥中的同源基因、、和也诱导上调表达。这些结果表明, 玉米与拟南芥和水稻的同源基因在响应逆境胁迫方面有相似的功能。
研究表明, 拟南芥中和也会受干旱和ABA处理诱导表达[42]。玉米中的同源基因、和也能够响应ABA诱导, 表达量呈现明显的上调趋势(图6)。与拟南芥不同的是, 拟南芥受干旱和ABA诱导表达, 而玉米中的同源基因、和在ABA处理后却抑制表达。这些结果说明不同物种的同源基因对于相同的逆境胁迫处理时的响应也是比较复杂的, 有相似的模式, 也有不同的模式。本研究进行的是玉米幼苗的处理, 结合基因组织表达分析发现, 与其他基因相比, 这些逆境胁迫响应上调表达的基因在幼苗的根中的相对表达量较高(图3)。说明这些基因不仅在玉米生长发育(尤其是幼苗时期)过程中发挥重要作用, 而且也是玉米响应逆境胁迫的关键基因。
4 结论
利用隐马可夫模型(HMM)在玉米基因组中鉴定到17个HD-ZIP I亚家族基因, 这些基因不均匀分布于玉米6条染色体上, 与水稻的亲缘关系要近于拟南芥。玉米HD-ZIP I亚家族基因在玉米7种组织中表现出多种表达模式, 具有明显的组织表达特异性。另外, 多种逆境胁迫和激素处理后, 玉米HD- ZIP I亚家族基因表现出复杂的响应模式。

附表1 本文中所用引物序列信息
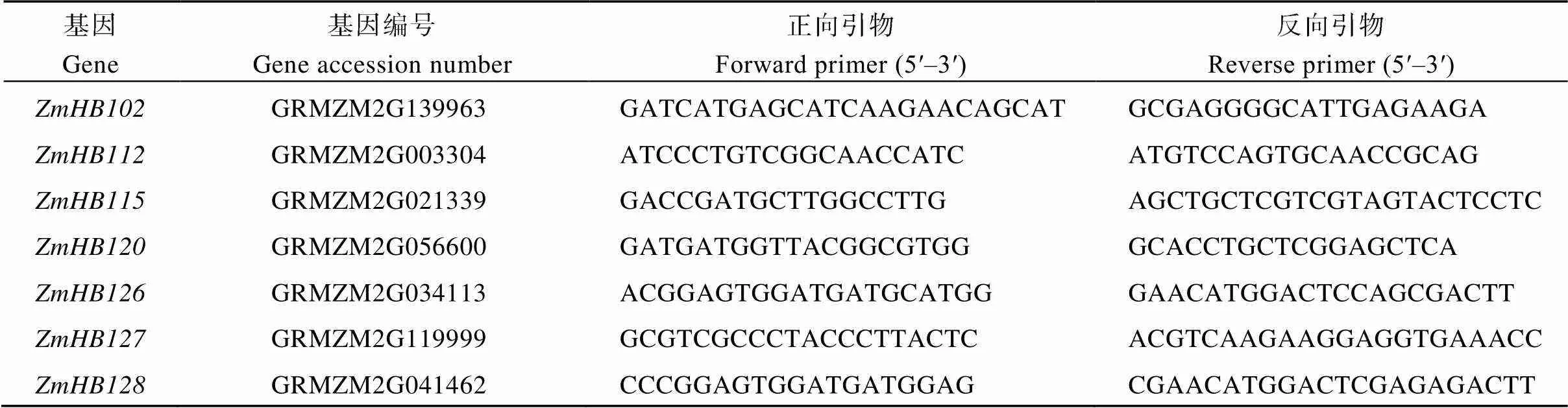
(续附表1)
[1] Verslues P E, Agarwal M, Katiyar-Agarwal S, Zhu J, Zhu J K. Methods and concepts in quantifying resistance to drought, salt and freezing, abiotic stresses that affect plant water status.,2006, 45: 523–539.
[2] Yamaguchi-Shinozaki K, Shinozaki K. Transcriptional regulatory networks in cellular responses and tolerance to dehydration and cold stresses.,2006, 57: 781–803.
[3] Zhu J K. Salt and drought stress signal transduction in plants.,2002, 53: 247–273.
[4] Kim S, Kang J Y, Cho D I, Park J H, Kim S Y. ABF2, an ABRE-binding bZIP factor, is an essential component of glucose signaling and its overexpression affects multiple stress tolerance.,2004, 40: 75–87.
[5] Dai X, Xu Y, Ma Q, Xu W, Wang T, Xue Y, Chong K. Overexpression of an R1R2R3 MYB gene,, increases tolerance to freezing, drought, and salt stress in transgenic.,2007, 143: 1739–1751.
[6] Gao T, Wu Y, Zhang Y, Liu L, Ning Y, Wang D, Tong H, Chen S, Chu C, Xie Q.overexpression greatly improves drought tolerance in transgenic rice.,2011, 76: 145–156.
[7] Hu T, Ye J, Tao P, Li H, Zhang J, Zhang Y, Ye Z. The tomato HD-Zip I transcription factor SlHZ24 modulates ascorbate accumulation through positive regulation of the D-mannose/L-galactose pathway.,2016, 85: 16–29.
[8] Gong S, Ding Y, Hu S, Ding L, Chen Z, Zhu C. The role of HD-Zip class I transcription factors in plant response to abiotic stresses.,2019, doi: 10.1111/ppl.12965.
[9] Mukherjee K, Brocchieri L, Burglin T R. A comprehensive classification and evolutionary analysis of plant homeobox genes.,2009, 26: 2775–2794.
[10] Agalou A, Purwantomo S, Overnas E, Johannesson H, Zhu X, Estiati A, de Kam R J, Engstrom P, Slamet-Loedin I H, Zhu Z, Wang M, Xiong L, Meijer A H, Ouwerkerk P B. A genome-wide survey of HD-Zip genes in rice and analysis of drought-responsive family members.,2008, 66: 87–103.
[11] Ariel F, Diet A, Verdenaud M, Gruber V, Frugier F, Chan R, Crespi M. Environmental regulation of lateral root emergence inrequires the HD-Zip I transcription factor HB1.,2010, 22: 2171–2183.
[12] Lin Z, Hong Y, Yin M, Li C, Zhang K, Grierson D. A tomato HD-Zip homeobox protein, LeHB-1, plays an important role in floral organogenesis and ripening.,2008, 55: 301–310.
[13] Johannesson H, Wang Y, Hanson J, Engstrom P. Thehomeobox geneis a potential regulator of abscisic acid responsiveness in developing seedlings.,2003, 51: 719–729.
[14] Manavella P A, Arce A L, Dezar C A, Bitton F, Renou J P, Crespi M, Chan R L. Cross-talk between ethylene and drought signalling pathways is mediated by the sunflower Hahb-4 transcription factor.,2006, 48: 125–137.
[15] Li W, Dong J, Cao M, Gao X, Wang D, Liu B, Chen Q. Genome-wide identification and characterization of HD-ZIP genes in potato.,2019, 697: 103–117.
[16] Li Y, Xiong H, Cuo D, Wu X, Duan R. Genome-wide characterization and expression profiling of the relation of the HD-Zip gene family to abiotic stress in barley (L.).,2019, 141: 250–258.
[17] Yue H, Shu D, Wang M, Xing G, Zhan H, Du X, Song W, Nie X. Genome-wide identification and expression analysis of the HD-Zip gene family in wheat (L.).(Basel), 2018, 9(2), doi: 10.3390/genes9020070.
[18] Ariel F D, Manavella P A, Dezar C A, Chan R L. The true story of the HD-Zip family.2007, 12: 419–426.
[19] Henriksson E, Olsson A S, Johannesson H, Johansson H, Hanson J, Engstrom P, Soderman E. Homeodomain leucine zipper class I genes in. Expression patterns and phylogenetic relationships.,2005, 139: 509–518.
[20] Romani F, Ribone P A, Capella M, Miguel V N, Chan R L. A matter of quantity: common features in the drought response of transgenic plants overexpressing HD-Zip I transcription factors., 2016, 251: 139–154.
[21] Perotti M F, Ribone P A, Chan R L. Plant transcription factors from the homeodomain-leucine zipper family: I. Role in development and stress responses.,2017, 69: 280-289.
[22] Hu J, Chen G, Yin W, Cui B, Yu X, Lu Y, Hu Z. Silencing of SlHB2 improves drought, salt stress tolerance, and induces stress-related gene expression in tomato.,2017, 36: 578–589.
[23] Ni Y, Wang X, Li D, Wu Y, Xu W, Li X. Novel cotton homeobox gene and its expression profiling in root development and in response to stresses and phytohormones.(Shanghai), 2008, 40: 78–84.
[24] Zhang S, Haider I, Kohlen W, Jiang L, Bouwmeester H, Meijer A H, Schluepmann H, Liu C M, Ouwerkerk P B. Function of the HD-Zip I genein ABA-mediated drought and salt tolerances in rice.Plant Mol Biol2012, 80: 571–585.
[25] Dezar C A, Gago G M, Gonzalez D H, Chan R L., a sunflower homeobox-leucine zipper gene, is a developmental regulator and confers drought tolerance toplants.,2005, 14: 429–440.
[26] Cabello J V, Giacomelli J I, Gomez M C, Chan R L. The sunflower transcription factor HaHB11 confers tolerance to water deficit and salinity to transgenicand alfalfa plants.,2017, 257: 35–46.
[27] Cabello J V, Arce A L, Chan R L. The homologous HD-Zip I transcription factors HaHB1 and AtHB13 confer cold tolerance via the induction of pathogenesis-related and glucanase proteins.,2012, 69: 141–153.
[28] Capella M, Ribone P A, Arce A L, Chan R L.HomeoBox 1 (AtHB1), a Homedomain-Leucine Zipper I (HD-Zip I) transcription factor, is regulated by PHYTOCHROME-INTERACTING FACTOR 1 to promote hypocotyl elongation.,2015, 207: 669–682.
[29] Parveen S, Pandey A, Jameel N, Chakraborty S, Chakraborty N. Transcriptional regulation of chickpea ferritin CaFer1 influences its role in iron homeostasis and stress response.,2018, 222: 9–16.
[30] Ebrahimian-Motlagh S, Ribone P A, Thirumalaikumar V P, Allu A D, Chan R L, Mueller-Roeber B, Balazadeh S. JUNGBRUNNEN1 confers drought tolerance downstream of the HD-Zip I transcription factor AtHB13.,2017, 8: 2118.
[31] Dai M, Hu Y, Ma Q, Zhao Y, Zhou D X. Functional analysis of rice HOMEOBOX4 () gene reveals a negative function in gibberellin responses.2008, 66: 289–301.
[32] Zhou W, Malabanan P B, Abrigo E.regulates GA signaling by interacting with DELLA-like genes and GA oxidase genes in rice.,2015, 201: 97–107.
[33] Zhao Y, Ma Q, Jin X, Peng X, Liu J, Deng L, Yan H, Sheng L, Jiang H, Cheng B. A novel maize homeodomain-leucine zipper (HD-Zip) I gene,, positively regulates drought and salt tolerance in both rice and.,2014, 55: 1142–1156.
[34] Wu J, Zhou W, Gong X, Cheng B. Expression of, a maize homeodomain-Leucine Zipper I gene, confers tolerance to drought stress in transgenic rice.,2016, 34: 845–853.
[35] Guo A Y, Zhu Q H, Chen X, Luo J C. GSDS: gene structure display server.(Beijing), 2007, 29: 1023–1026.
[36] Letunic I, Doerks T, Bork P. SMART: recent updates, new developments and status in 2015.2015, 43 (Database issue): D257–D260.
[37] Larkin M A, Blackshields G, Brown N P, Chenna R, McGettigan P A, McWilliam H, Valentin F, Wallace I M, Wilm A, Lopez R, Thompson J D, Gibson T J, Higgins D G. Clustal W and Clustal X version 2.0.,2007, 23: 2947–2948.
[38] Tamura K, Peterson D, Peterson N, Stecher G, Nei M, Kumar S. MEGA5: molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods.,2011, 28: 2731–2739.
[39] Livak K J, Schmittgen T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2(-Delta Delta C(T)) Method.,2001, 25: 402–408.
[40] Gonzalez-Grandio E, Pajoro A, Franco-Zorrilla J M, Tarancon C, Immink R G, Cubas P. Abscisic acid signaling is controlled by a BRANCHED1/HD-ZIP I cascade in Arabidopsis axillary buds.,2017, 114: E245–E254.
[41] Shao J, Haider I, Xiong L, Zhu X, Hussain R M F, Overnas E, Meijer A H, Zhang G, Wang M, Bouwmeester H J, Ouwerkerk P B F. Functional analysis of the HD-Zip transcription factor genesandin rice.,2018, 13: e0199248.
[42] Olsson A S, Engstrom P, Soderman E. The homeobox genes ATHB12 and ATHB7 encode potential regulators of growth in response to water deficit in.,2004, 55: 663–677.
Genome-wide identification and expression analysis of HD-ZIP I subfamily genes in maize
LIANG Si-Wei1, JIANG Hao-Liang1, ZHAI Li-Hong2, WAN Xiao-Rong1, LI Xiao-Qin1, JIANG Feng1,*, and SUN Wei1,*
1College of Agriculture and Biology, Zhongkai University of Agriculture and Engineering, Guangzhou 510225, Guangdong, China;2Medical College, Hubei University of Arts and Science, Xiangyang 441053, Hubei, China
Transcription factors (TFs) are indispensable regulators of plant response to abiotic stress and play an important role in the whole growth and development process. HD-ZIP proteins constitute a large family of transcription factors that are found only in plants and are divided into four subfamilies (HD-ZIP I–IV). HD-ZIP I subfamily genes mainly participate in response to extreme environments such as drought and osmotic stress and treatments of ABA and ethylene. Here, we identified 17 HD-ZIP I subfamily genes in the maize genome using the hidden Markov model (HMM), which distributed non-uniformly on six chromosomes of maize and were more closely related to rice than to. Furthermore, these HD-ZIP I subfamily genes exhibited multiple expression patterns in seven tissues, showing strong tissues-specific expression. Moreover, maize HD-ZIP I subfamily genes showed different response patterns and degrees to different stresses, such as high salinity, waterlogging and cold stress. In addition, maize HD-ZIP I subfamily genes also showed a complex response pattern under treatment of five different hormones. These results provide valuable reference information for dissecting function and molecular mechanism of HD-ZIP I subfamily genes in maize.
HD-ZIP I subfamily; abiotic stress; expression analysis; maize
2019-07-13;
2019-12-26;
2020-01-15.
10.3724/SP.J.1006.2020.93040
孙伟, E-mail: starking521@126.com, Tel: 020-89003026; 蒋锋, E-mail:breakthrough@139.com, Tel: 020-89003026
联系方式: E-mail:hellosiwei@163.com, Tel: 020-89003026
本研究由国家自然科学基金项目(31901565), 广东省重点领域研发计划项目(2018B020202013)和广东省科学技术厅项目(粤科产学研字[2016]176号)资助。
This study was supported by the National Natural Science Foundation of China (31901565), the National Key Research and Development Program of Guangdong Province (2018B020202013), and the Program of the Science and Technology Bureau of Guangdong Province ([2016]176)
URL: http://kns.cnki.net/kcms/detail/11.1809.S.20200115.1008.008.html
